Los polimorfismos de riesgo para el desarrollo de enfermedad de Alzheimer (se han estudiado en pacientes con demencia, pero aún no se han explorado en trastorno neurocognitivo leve (TNL) en nuestra población, ni se han considerado en relación con variables cognitivas, las cuales pueden ser biomarcadores predictivos de enfermedad.
ObjetivoEvaluar los desempeños cognitivos y los polimorfismos en los genes SORL1(rs11218304), PVRL2(rs6859), CR1(rs6656401), TOMM40(rs2075650), APOE(isoformas ɛ2, ɛ3, ɛ4), PICALM(rs3851179), GWAS_14q(rs11622883), BIN(rs744373), CLU (rs227959 y rs11136000) en pacientes con TNL y en sujetos sanos.
MetodologíaEstudio descriptivo, exploratorio y transversal, en una cohorte prospectiva de participantes seleccionados mediante muestreo no probabilístico, evaluados por neurología, neuropsicología y genética, y clasificados como cognitivamente sanos y pacientes con TNL, según criterios. La cognición se evaluó por medio de la batería Neuronorma y se analizó en relación con las variantes polimórficas por medio de medidas de tendencia, intervalos de confianza y estadísticos no paramétricos.
ResultadosSe identificaron diferencias en los desempeños en tareas de lenguaje y memoria en relación con las variantes de BIN1, CLU y CR1, junto con tendencias en las variantes de PICALM, GWArs, SORL y PVRL2, mientras que en APOE y TOMM40 no se encontraron tendencias.
DiscusiónLas tendencias en los desempeños cognitivos en relación con variantes polimórficas podrían indicar que, en ausencia de demencia, los mecanismos que regulan estos genes podrían tener un efecto sobre la cognición; sin embargo, esta aproximación tiene un carácter exploratorio y sus resultados permiten generar hipótesis que requieren ser exploradas en muestras de mayor tamaño.
Alzheimer disease risk polymorphisms have been studied in patients with dementia, but have not yet been explored in mild cognitive impairment (MCI) in our population; nor have they been addressed in relation to cognitive variables, which can be predictive biomarkers of disease.
ObjectiveTo evaluate cognitive performance and presence of polymorphisms of the genes SORL1(rs11218304), PVRL2(rs6859), CR1(rs6656401), TOMM40(rs2075650), APOE (isoforms ɛ2, ɛ3, ɛ4), PICALM(rs3851179), GWAS_14q(rs11622883), BIN1(rs744373), and CLU (rs227959 and rs11136000) in patients with MCI and healthy individuals.
MethodologyWe performed a cross-sectional, exploratory, descriptive study of a prospective cohort of participants selected by non-probabilistic sampling, evaluated with neurological, neuropsychological, and genetic testing, and classified as cognitively healthy individuals and patients with MCI. Cognition was evaluated with the Neuronorma battery and analysed in relation to the polymorphic variants by means of measures of central tendency, confidence intervals, and nonparametric statistics.
ResultsWe found differences in performance in language and memory tasks between carriers and non-carriers of BIN1, CLU, and CR1 variants and a trend toward poor cognitive performance for PICALM, GWAS_14q, SORL1, and PVRL2 variants; the APOE and TOMM40 variants were not associated with poor cognitive performance.
DiscussionDifferences in cognitive performance associated with these polymorphic variants may suggest that the mechanisms regulating these genes could have an effect on cognition in the absence of dementia; however, this study was exploratory and hypotheses based on these results must be explored in larger samples.
El trastorno neurocognitivo leve (TNL) tiene una presentación clínica heterogénea, se asocia a múltiples etiologías y puede evolucionar a un trastorno neurocognitivo mayor, por lo que se ha propuesto como un estado de transición entre el envejecimiento normal y la demencia1. Actualmente los estudios sobre los factores de riesgo genético para TNL son escasos, pero debido a que representa un alto riesgo para el desarrollo de trastornos neurocognitivos como la enfermedad de Alzheimer (EA)2, se considera que puede tener un importante componente genético3 y que los polimorfismos de riesgo para EA podrían ser explorados en TNL.
Los genes asociados a la EA se vinculan con diversos mecanismos fisiopatológicos que implican el metabolismo de lípidos, el estrés oxidativo, la respuesta inmune y mecanismos de transporte y degradación. En cuanto al transporte de lípidos, se vincula el gen APOE, que codifica la apolipoproteína E, una proteína plasmática constituyente de las lipoproteínas que mantienen la estructura de la membrana y regulan el metabolismo intracelular del colesterol4. Por otra parte, el gen TOMM40 codifica una proteína perteneciente a un complejo involucrado en la entrada de proteínas a la mitocondria. Se ha encontrado que TOMM40 colocaliza con APP dentro de la mitocondria en cerebros de pacientes EA y que existe alta acumulación de APP en portadores del genotipo APOEe3/4, lo cual apoya la hipótesis de la influencia de APOE sobre el transporte de APP en la mitocondria y de la interacción con TOMM40 para facilitar su acceso, por lo que se propone a este gen como candidato importante para el desarrollo de EA5.
Respecto a los mecanismos de transporte y degradación, se encuentra el gen SORL1 que codifica un receptor asociado al transporte de proteínas, el cual dirige fosfatasas, cinasas y receptores de señalización a su lugar dentro de la célula, garantizando la función adecuada de células y tejidos6. Otro gen involucrado es BIN1, que codifica una proteína adaptadora que participa en la endocitosis y la recuperación de las vesículas sinápticas, cuyas isoformas participan en procesos de apoptosis, inflamación y homeostasis del calcio7. Mientras que PICALM codifica una proteína esencial para el tráfico intracelular de neurotransmisores, proteínas y lípidos3,8.
En relación con mecanismos involucrados en la respuesta inmune, se ha asociado el gen CR1, que codifica para una glicoproteína que facilita la eliminación de antígeno, genera respuesta inflamatoria y puede mediar la limpieza de partículas de β amiloide9. También se encuentra el gen PVRL2, que codifica para una glicoproteína de membrana que sirve de entrada a algunas cepas virales y participa en los mecanismos de propagación de virus entre las células10. Por su parte, el gen CLU forma parte del grupo de proteínas chaperonas que facilitan el plegamiento de nuevas proteínas recién sintetizadas y está involucrada en procesos de apoptosis celular11.
Respecto a las variables clínicas, algunos de estos genes involucrados en la fisiopatología de la EA han sido estudiados en relación con la cognición en pacientes con EA, TNL y sujetos sanos12, pues se ha demostrado que ciertas variantes genéticas pueden tener un efecto sobre los desempeños cognitivos, haciendo de las variables cognitivas medidas útiles para detectar nuevos loci de riesgo que podrían considerarse como blancos terapéuticos y biomarcadores predictivos de enfermedad13. Por ejemplo, algunas variantes de APOE se han asociado a mejor desempeño cognitivo14, mientras que la variante épsilon 4 (ɛ4) se vincula con disminución en memoria episódica15. En el gen TOMM40 algunas variantes se asocian a mejores desempeños y otras variantes como la poli-T larga se relacionan con déficit en aprendizaje verbal y funciones ejecutivas16. Así mismo, el gen BIN-1 ha mostrado un efecto en la memoria de trabajo17.
En nuestra población se ha encontrado que el gen APOE explica solo una porción del riesgo de EA18, por lo que se han explorado otros polimorfismos genéticos (SNP) de riesgo, entre los que se ha encontrado asociación significativa con TOMM40(rs2075650). Destacan combinaciones alélicas de los genes TOMM40 y el alelo 4 de APOE, que pueden llegar a anticipar el inicio de la enfermedad hasta en 13 años19. Hasta el momento, no se han realizado estudios en nuestra población sobre variantes genéticas de riesgo para EA en pacientes con TNL, por lo cual el objetivo del presente trabajo es estudiar las variantes en los polimorfismos SORL1(rs11218304), PVRL2(rs6859), CR1(rs6656401), TOMM40(rs2075650), APOE(rs7412; rs440446), PICALM(rs3851179), GWAS_14q(rs11622883), BIN(rs744373), CLU (rs227959 y rs11136000) junto con los desempeños cognitivos en un grupo de participantes cognitivamente sanos y pacientes con TNL.
MetodologíaEstudio longitudinal de tipo observacional y descriptivo, con un alcance exploratorio debido al tamaño de la muestra, cuyos resultados solo se pueden interpretar en el contexto de la muestra estudiada y no se pueden extrapolar a la población general. Ha sido realizado por el Grupo de Neurociencias y aprobado por el Comité de Ética de la Facultad de Medicina de la universidad. Todos los participantes firmaron el consentimiento informado.
Selección de la muestraLos participantes fueron seleccionados mediante muestreo no probabilístico, entre diciembre de 2015 y noviembre de 2017. Se determinaron como criterios de inclusión: edad igual o mayor a 50 años; contar por lo menos con un año de escolaridad; no cumplir criterios para trastorno neurocognitivo mayor, ausencia de síndrome confusional, de antecedente de trauma craneoencefálico, de enfermedad cerebrovascular y de otras condiciones asociadas a alteración cognitiva y no presentar limitaciones sensoriales que impidan el desarrollo de las pruebas. Se excluyó a aquellos participantes que no cumplían con los criterios de inclusión. La verificación de los criterios de selección fue realizada por el equipo de profesionales de la salud, expertos en evaluación de trastorno neurocognitivo.
ProcedimientoLas variables clínicas y sociodemográficas fueron recolectadas durante la evaluación neurológica a partir de un cuestionario estructurado. El proceso de tamización se realizó por medio de los instrumentos Montreal Cognitive Assessment Test20, INECO Frontal Screening21, escala Yesavage22 y el Inventario Neuropsiquiátrico23. La funcionalidad fue medida con las escalas Lawton modificada24 y Bayer-Actividades de la vida diaria25. La valoración cognitiva se realizó con la batería neuropsicológica Neuronorma, normalizada para nuestra población26,27 y compuesta por pruebas cognitivas que miden 5dominios cognitivos: atención: TMT A y B, SDMT, test de stroop palabra y color, span verbal y visual directo; memoria: test de Grober & Buschke (FCSRT), Evocación de la figura de Rey; lenguaje: test de denominación de Boston, test de Token, fluidez verbal; habilidades construccionales: Figura de Rey; funciones ejecutivas: fluidez verbal fonológica, Torre de Londres (TOL), test de stroop interferencia, Wisconsin Card Sorting Test (WCST).
Los participantes fueron clasificados como cognitivamente sanos si obtenían desempeños esperados en las escalas de tamiz y su desempeño en las pruebas se encontraba, como máximo, una desviación estándar por debajo de la media, según lo esperado para la población por edad y escolaridad26,27. Los criterios utilizados para el diagnóstico de TNL fueron definidos a partir del DSM-528. Las variables genéticas consideradas en el estudio fueron: SNP en SORL1(rs11218304), PVRL2(rs2075650), CR1(rs6656401), TOMM40(rs2075650), APOE(rs7412 y rs440446), PICALM(rs3851179), GWAS_14q(rs11622883), BIN(rs744373), CLU (rs227959 y rs11136000). El ADN de los individuos fue obtenido de una muestra de sangre periférica usando el kit ReliaPrep Blood gDNA Miniprep System (Promega, California, EE. UU.). La metodología de genotipado se realizó por medio de la técnica de sondas Taqman (Applied Biosystems, Foster City, EE. UU.), que consiste en la discriminación de los 2alelos de un SNP específico. Cada reacción de este ensayo contiene 2primers no marcados (forward y reverse), una sonda 1 VIC® dye-MGB, que detecta la secuencia de un alelo y otra sonda 1 6FAM™ dye-MGB, que detecta el otro alelo. Los ensayos se realizaron en un termociclador BioRad CFX96 y las condiciones fueron: 50°C por 2 min, 95°C por 10 min, 50 ciclos de 95°C por 15 s y 60°C por un minuto. La discriminación alélica se analizó mediante el software CFX Manager (BioRad).
Análisis de los datosPara la caracterización clínica y sociodemográfica de la población se utilizaron medidas de frecuencia absoluta y relativa, así como medidas de dispersión de acuerdo con la naturaleza de los datos. Los resultados de la evaluación psicométrica fueron presentados en escalares de acuerdo con el cuantil en el que se encontraban los resultados de cada participante respecto a la distribución en la población general.
Se desarrolló un análisis psicométrico de los resultados por subgrupos a partir de la caracterización genética; los resultados se presentan como medidas de frecuencia relativa respecto del grupo. La unidad de análisis considerada para el estudio fueron las variantes polimórficas. Para todas las mediciones se reportó el respectivo intervalo de confianza (IC) al 95% aproximado mediante los cuantiles 2,5% y 97,5% calculados mediante técnicas de remuestreo bootstrap, seleccionando 1.000 muestras aleatorias con reemplazo del 75% de los datos observados29.
Los análisis de las variables demográficas, cognitivas y genéticas se realizaron mediante la prueba no paramétrica U de Mann-Whitney29. La relación entre las frecuencias alélicas y el diagnóstico se evaluó por medio de la prueba exacta de Fisher. Para analizar los desempeños cognitivos en relación con las variables genéticas, se utilizó el método de Kruskal-Wallis, con el fin de explorar la existencia de diferencias significativas en los desempeños cognitivos en función de las variantes polimórficas. Adicionalmente se realizó un análisis de frecuencias de los desempeños cognitivos clasificados por categorías de desempeño (alto, medio, límite y bajo) de acuerdo con sus variantes alélicas, considerando como unidad de análisis el sujeto con la dupla alélica. Se realizó inspección visual a partir de una tabla de frecuencias absolutas y relativas. Las frecuencias que mostraron tendencias sobre algunas pruebas específicas y consistencia por debajo del rendimiento de las pruebas fueron evaluadas comparando sus intervalos de confianza al 95%, para explorar si estas diferencias podrían ser relevantes y si en alguna medida se asociaban con los hallazgos obtenidos a través de los estadísticos no paramétricos.
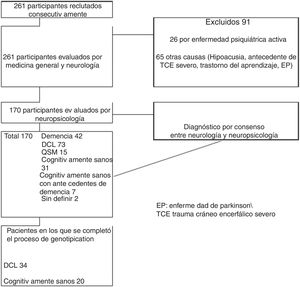
ResultadosDe los 261 participantes evaluados, 91 casos fueron excluidos por no cumplir los criterios de selección, 31 (11,8%) participantes se consideraron cognitivamente sanos y sin antecedentes familiares de demencia, de los cuales, se realizó genotipificación en 20 sujetos. Otros 73 (28%) participantes se diagnosticaron con TNL, entre los que se genotipificaron 34. Los detalles de la selección de la muestra se encuentran en la figura 1 (flujo de pacientes).
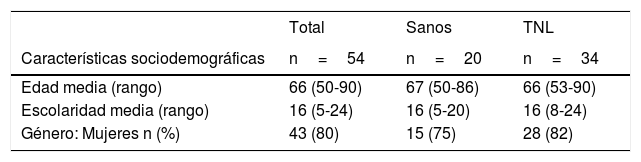
La comparación entre grupos (TNL: 34 y sanos: 20) mostró diferencias en la distribución por género, con mayor número de mujeres en ambos grupos (TNL 82,35%; IC: 69,5-95 y sanos 75%; IC: 56-94), la mediana de la edad en TNL fue de 66 años (rango 53-90) y de 67 años en el grupo de sanos (rango 50-86). En ambos grupos de participantes, el nivel de escolaridad mínimo fue de 5 años. El antecedente de riesgo vascular más frecuente en ambos grupos fue dislipidemia, mientras que el menos frecuente fue diabetes mellitus. Las características clínicas y sociodemográficas de la muestra se resumen en la tabla 1.
Características sociodemográficas y clínicas de la muestra
| Total | Sanos | TNL | |
|---|---|---|---|
| Características sociodemográficas | n=54 | n=20 | n=34 |
| Edad media (rango) | 66 (50-90) | 67 (50-86) | 66 (53-90) |
| Escolaridad media (rango) | 16 (5-24) | 16 (5-20) | 16 (8-24) |
| Género: Mujeres n (%) | 43 (80) | 15 (75) | 28 (82) |
| Total | Sanos | TNL | ||||
|---|---|---|---|---|---|---|
| Características clínicas | N=54 (%) | IC 95% | n=20(%) | IC 95% | n=34(%) | IC 95% |
| Hipertensión arterial | 17 (31,5) | (21,1-49,9) | 6 (30) | (13,1-46,9) | 11 (32) | (19,1-45,6) |
| Diabetes mellitus | 8 (15) | (6,8-22,8) | 2 (10) | (0-21,1) | 6 (17) | (6,9-29,4) |
| Dislipidemia | 28 (52) | (40,6-63,1) | 10 (50) | (31,6-68,4) | 18 (53) | (38,8-67,1) |
| Tabaquismo | 15 (27) | (17,7-37,8) | 4 (20) | (5,2-34,8) | 11 (32) | (19,1-45,6) |
| Sedentarismo | 11 (20) | (11,3-29,4) | 6 (30) | (13,1-46,9) | 5 (15) | (4,7-24,7) |
IC: intervalo de confianza.
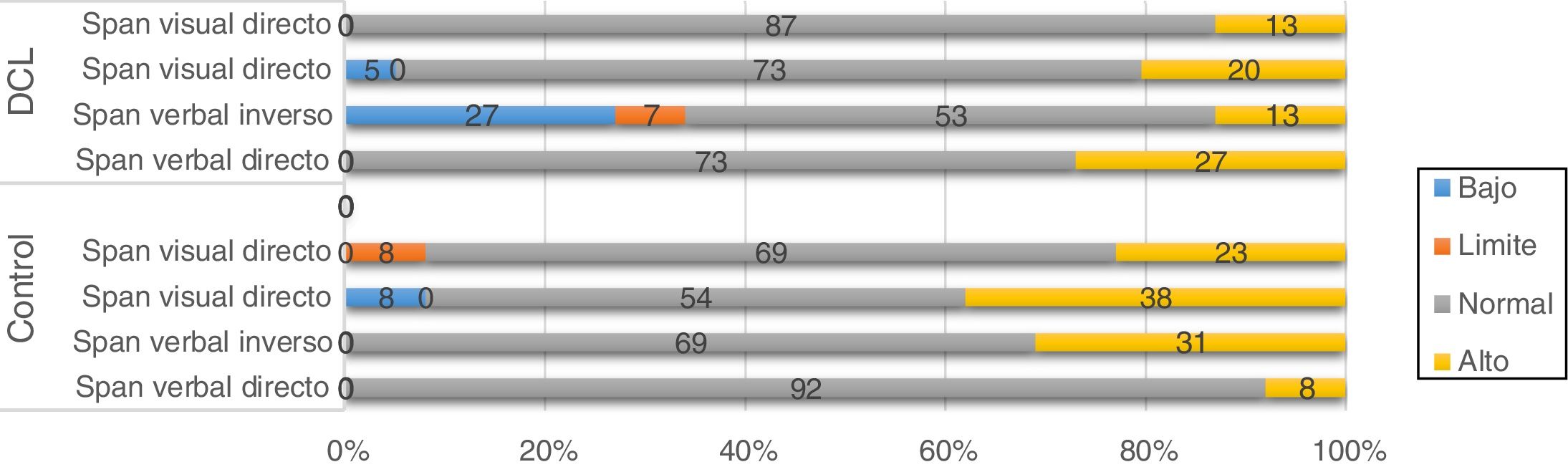
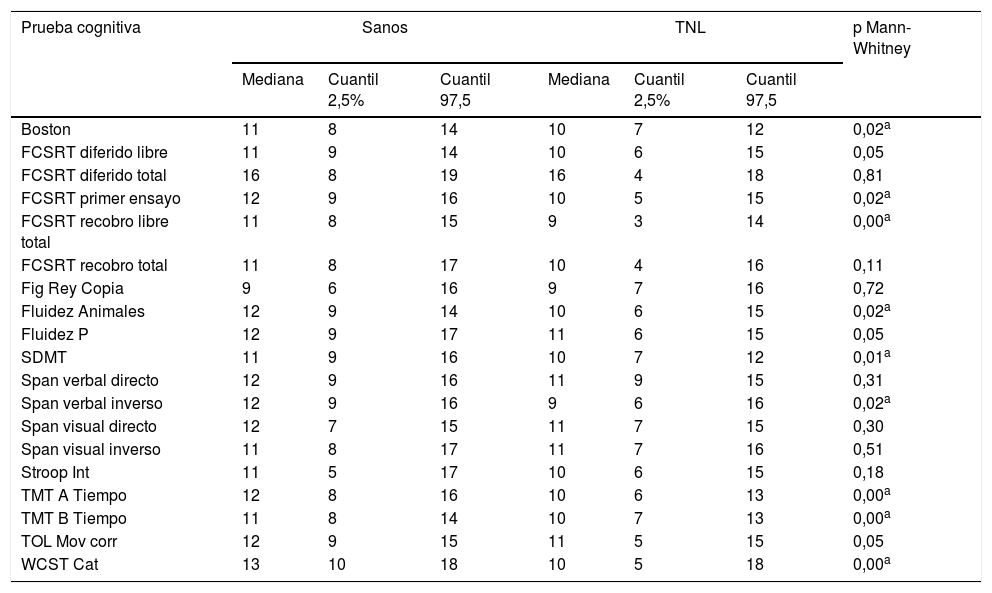
En tareas de tamiz cognitivo se identifican diferencias entre los grupos respecto a MOCA, con una mediana de 25 (IC: 20-28) en sujetos sanos, y de 23 (IC: 17-27) en pacientes con TNL; así como en el test de tamización frontal INECO, con una mediana de 27 (IC: 24-30) en sujetos sanos y una mediana de 26 (IC: 21-29) en pacientes, con un valor p: 0,02 que indica significación en las comparaciones. En cuanto a las pruebas de la batería Neuronorma, se encuentra una tendencia a puntuaciones escalares altas (tabla 2) y diferencias significativas entre los grupos en la mayoría de las tareas, excepto en la copia de la Figura compleja de Rey-Osterrieth, la interferencia del stroop, tareas de memoria a corto plazo (span verbal y visual directo), tareas de memoria de trabajo (span visual inverso), así como ensayos de acceso facilitado con clave en tareas de memoria verbal explícita.
Desempeños cognitivos evaluados por medio de la batería Neuronorma.Co en valores escalares
| Prueba cognitiva | Sanos | TNL | p Mann-Whitney | ||||
|---|---|---|---|---|---|---|---|
| Mediana | Cuantil 2,5% | Cuantil 97,5 | Mediana | Cuantil 2,5% | Cuantil 97,5 | ||
| Boston | 11 | 8 | 14 | 10 | 7 | 12 | 0,02a |
| FCSRT diferido libre | 11 | 9 | 14 | 10 | 6 | 15 | 0,05 |
| FCSRT diferido total | 16 | 8 | 19 | 16 | 4 | 18 | 0,81 |
| FCSRT primer ensayo | 12 | 9 | 16 | 10 | 5 | 15 | 0,02a |
| FCSRT recobro libre total | 11 | 8 | 15 | 9 | 3 | 14 | 0,00a |
| FCSRT recobro total | 11 | 8 | 17 | 10 | 4 | 16 | 0,11 |
| Fig Rey Copia | 9 | 6 | 16 | 9 | 7 | 16 | 0,72 |
| Fluidez Animales | 12 | 9 | 14 | 10 | 6 | 15 | 0,02a |
| Fluidez P | 12 | 9 | 17 | 11 | 6 | 15 | 0,05 |
| SDMT | 11 | 9 | 16 | 10 | 7 | 12 | 0,01a |
| Span verbal directo | 12 | 9 | 16 | 11 | 9 | 15 | 0,31 |
| Span verbal inverso | 12 | 9 | 16 | 9 | 6 | 16 | 0,02a |
| Span visual directo | 12 | 7 | 15 | 11 | 7 | 15 | 0,30 |
| Span visual inverso | 11 | 8 | 17 | 11 | 7 | 16 | 0,51 |
| Stroop Int | 11 | 5 | 17 | 10 | 6 | 15 | 0,18 |
| TMT A Tiempo | 12 | 8 | 16 | 10 | 6 | 13 | 0,00a |
| TMT B Tiempo | 11 | 8 | 14 | 10 | 7 | 13 | 0,00a |
| TOL Mov corr | 12 | 9 | 15 | 11 | 5 | 15 | 0,05 |
| WCST Cat | 13 | 10 | 18 | 10 | 5 | 18 | 0,00a |
Los valores registrados corresponden a los puntajes escalares obtenidos en cada prueba cognitiva con base en baremos ajustados a nuestra población.
Boston: test de denominación por confrontación visual de Boston; CST Cat: número de categorías correctas en el test de Wisconsin; FCSRT: Free and Cued Selective Reminding Test (Grober & Buschke-en la versión adaptada al español); Fig Rey: copia de la figura compleja de Rey-Osterrieth; SDMT: Symbol Digit Modalities Test; Stroop Int: interferencia en el test de stroop; TOL Mov corr: movimientos correctos en la Torre de Londres.
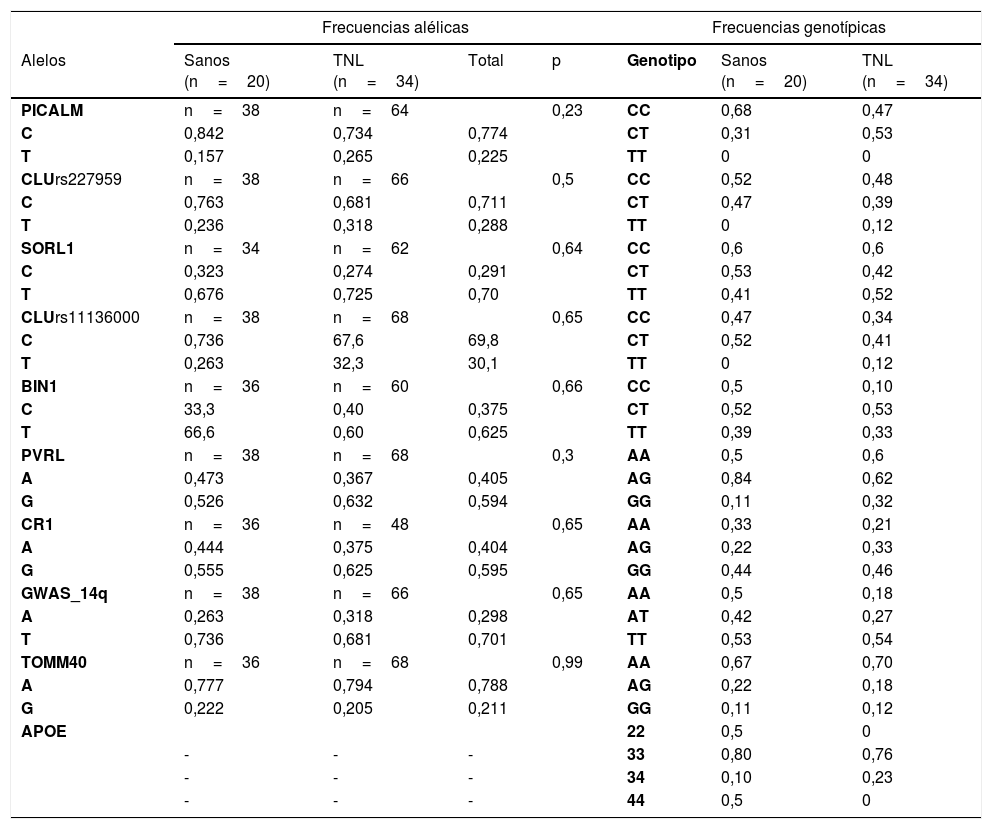
En relación con la caracterización genética de la muestra, todos los polimorfismos están en equilibrio de Hardy Weinberg, a excepción de PVRL2(rs6859) en el grupo de TNL. No se encontraron asociaciones significativas entre las variantes polimórficas de los genes estudiados y el diagnóstico. La frecuencias alélicas y genotípicas se encuentran en la tabla 3. Se compararon los desempeños cognitivos en función de la variantes polimórficas por medio de estadísticos no paramétricos.
Distribución de las frecuencias alélicas y genotípicas
| Frecuencias alélicas | Frecuencias genotípicas | ||||||
|---|---|---|---|---|---|---|---|
| Alelos | Sanos (n=20) | TNL (n=34) | Total | p | Genotipo | Sanos (n=20) | TNL (n=34) |
| PICALM | n=38 | n=64 | 0,23 | CC | 0,68 | 0,47 | |
| C | 0,842 | 0,734 | 0,774 | CT | 0,31 | 0,53 | |
| T | 0,157 | 0,265 | 0,225 | TT | 0 | 0 | |
| CLUrs227959 | n=38 | n=66 | 0,5 | CC | 0,52 | 0,48 | |
| C | 0,763 | 0,681 | 0,711 | CT | 0,47 | 0,39 | |
| T | 0,236 | 0,318 | 0,288 | TT | 0 | 0,12 | |
| SORL1 | n=34 | n=62 | 0,64 | CC | 0,6 | 0,6 | |
| C | 0,323 | 0,274 | 0,291 | CT | 0,53 | 0,42 | |
| T | 0,676 | 0,725 | 0,70 | TT | 0,41 | 0,52 | |
| CLUrs11136000 | n=38 | n=68 | 0,65 | CC | 0,47 | 0,34 | |
| C | 0,736 | 67,6 | 69,8 | CT | 0,52 | 0,41 | |
| T | 0,263 | 32,3 | 30,1 | TT | 0 | 0,12 | |
| BIN1 | n=36 | n=60 | 0,66 | CC | 0,5 | 0,10 | |
| C | 33,3 | 0,40 | 0,375 | CT | 0,52 | 0,53 | |
| T | 66,6 | 0,60 | 0,625 | TT | 0,39 | 0,33 | |
| PVRL | n=38 | n=68 | 0,3 | AA | 0,5 | 0,6 | |
| A | 0,473 | 0,367 | 0,405 | AG | 0,84 | 0,62 | |
| G | 0,526 | 0,632 | 0,594 | GG | 0,11 | 0,32 | |
| CR1 | n=36 | n=48 | 0,65 | AA | 0,33 | 0,21 | |
| A | 0,444 | 0,375 | 0,404 | AG | 0,22 | 0,33 | |
| G | 0,555 | 0,625 | 0,595 | GG | 0,44 | 0,46 | |
| GWAS_14q | n=38 | n=66 | 0,65 | AA | 0,5 | 0,18 | |
| A | 0,263 | 0,318 | 0,298 | AT | 0,42 | 0,27 | |
| T | 0,736 | 0,681 | 0,701 | TT | 0,53 | 0,54 | |
| TOMM40 | n=36 | n=68 | 0,99 | AA | 0,67 | 0,70 | |
| A | 0,777 | 0,794 | 0,788 | AG | 0,22 | 0,18 | |
| G | 0,222 | 0,205 | 0,211 | GG | 0,11 | 0,12 | |
| APOE | 22 | 0,5 | 0 | ||||
| - | - | - | 33 | 0,80 | 0,76 | ||
| - | - | - | 34 | 0,10 | 0,23 | ||
| - | - | - | 44 | 0,5 | 0 | ||
Los valores están registrados en porcentajes y corresponden a la distribución de las frecuencias alélicas y genotípicas de cada gen en función de cada grupo diagnóstico.
Los valores en la prueba exacta de Fisher p <0,05 indican que no existen diferencias significativas en la distribución de las frecuencias alélicas según el diagnóstico.
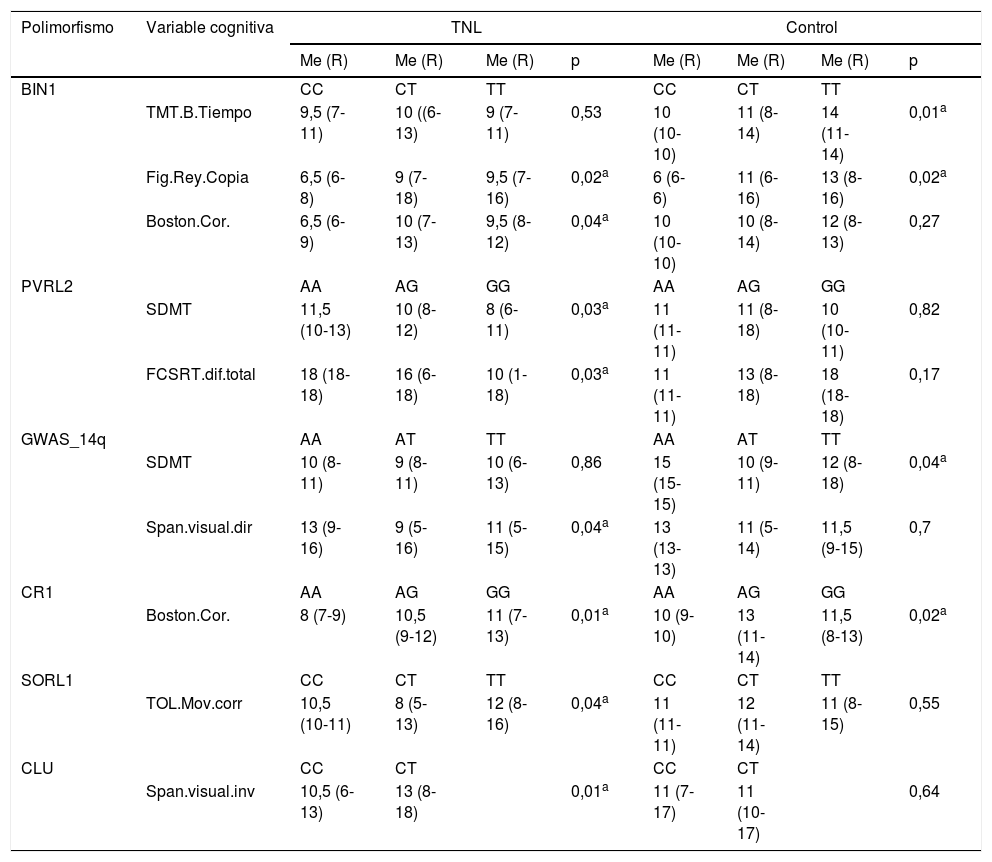
Los análisis no paramétricos (tabla 4) mostraron que en el grupo de TNL existen diferencias entre las variantes de los genes BIN1 (p: 0,04) y CR1 (p: 0,01) respecto a lenguaje (Boston), entre las variantes de PVRL2 (p: 0,03) en atención (SDMT) y memoria (FCSRT.diferido.total) y en GWAS_14q (p: 0,04) en relación con el span visual directo. Las funciones ejecutivas (TOL.mov.cor y span visual inverso) muestran desempeños diferenciales respecto a las variantes de SORL (0,04) y CLU (0,01), respectivamente. En los sujetos sanos se encontraron diferencias respecto a las variantes de GWAS_14q (p: 0,04) y BIN1 (p: 0,01) en tareas atencionales (SDMT, TMT B, respectivamente) y en las variantes de CR1 (p: 0,01) frente al test de Boston, prueba que también registró diferencias significativas en TNL.
Pruebas cognitivas con diferencias significativas en función de las variantes genotípicas
| Polimorfismo | Variable cognitiva | TNL | Control | ||||||
|---|---|---|---|---|---|---|---|---|---|
| Me (R) | Me (R) | Me (R) | p | Me (R) | Me (R) | Me (R) | p | ||
| BIN1 | CC | CT | TT | CC | CT | TT | |||
| TMT.B.Tiempo | 9,5 (7-11) | 10 ((6-13) | 9 (7-11) | 0,53 | 10 (10-10) | 11 (8-14) | 14 (11-14) | 0,01a | |
| Fig.Rey.Copia | 6,5 (6-8) | 9 (7-18) | 9,5 (7-16) | 0,02a | 6 (6-6) | 11 (6-16) | 13 (8-16) | 0,02a | |
| Boston.Cor. | 6,5 (6-9) | 10 (7-13) | 9,5 (8-12) | 0,04a | 10 (10-10) | 10 (8-14) | 12 (8-13) | 0,27 | |
| PVRL2 | AA | AG | GG | AA | AG | GG | |||
| SDMT | 11,5 (10-13) | 10 (8-12) | 8 (6-11) | 0,03a | 11 (11-11) | 11 (8-18) | 10 (10-11) | 0,82 | |
| FCSRT.dif.total | 18 (18-18) | 16 (6-18) | 10 (1-18) | 0,03a | 11 (11-11) | 13 (8-18) | 18 (18-18) | 0,17 | |
| GWAS_14q | AA | AT | TT | AA | AT | TT | |||
| SDMT | 10 (8-11) | 9 (8-11) | 10 (6-13) | 0,86 | 15 (15-15) | 10 (9-11) | 12 (8-18) | 0,04a | |
| Span.visual.dir | 13 (9-16) | 9 (5-16) | 11 (5-15) | 0,04a | 13 (13-13) | 11 (5-14) | 11,5 (9-15) | 0,7 | |
| CR1 | AA | AG | GG | AA | AG | GG | |||
| Boston.Cor. | 8 (7-9) | 10,5 (9-12) | 11 (7-13) | 0,01a | 10 (9-10) | 13 (11-14) | 11,5 (8-13) | 0,02a | |
| SORL1 | CC | CT | TT | CC | CT | TT | |||
| TOL.Mov.corr | 10,5 (10-11) | 8 (5-13) | 12 (8-16) | 0,04a | 11 (11-11) | 12 (11-14) | 11 (8-15) | 0,55 | |
| CLU | CC | CT | CC | CT | |||||
| Span.visual.inv | 10,5 (6-13) | 13 (8-18) | 0,01a | 11 (7-17) | 11 (10-17) | 0,64 | |||
Me: mediana; R: rango.
En esta tabla solo se resumen las variables que arrojaron valores significativos respecto a la interacción entre variantes genotípicas y rendimientos cognitivos. Los valores registrados corresponden a los puntajes escalares obtenidos en cada prueba cognitiva con base en baremos ajustados a nuestra población.
Además de los análisis estadísticos, se categorizaron los desempeños cognitivos a partir de las puntuaciones escalares (bajo: <6; límite: 7; normal: 8-13; alto:>14) y se hizo un análisis de frecuencias por grupos de desempeño discriminando por variantes polimórficas. A continuación se presentan los resultados que son consistentes entre los estadísticos no paramétricos y el análisis de frecuencias.
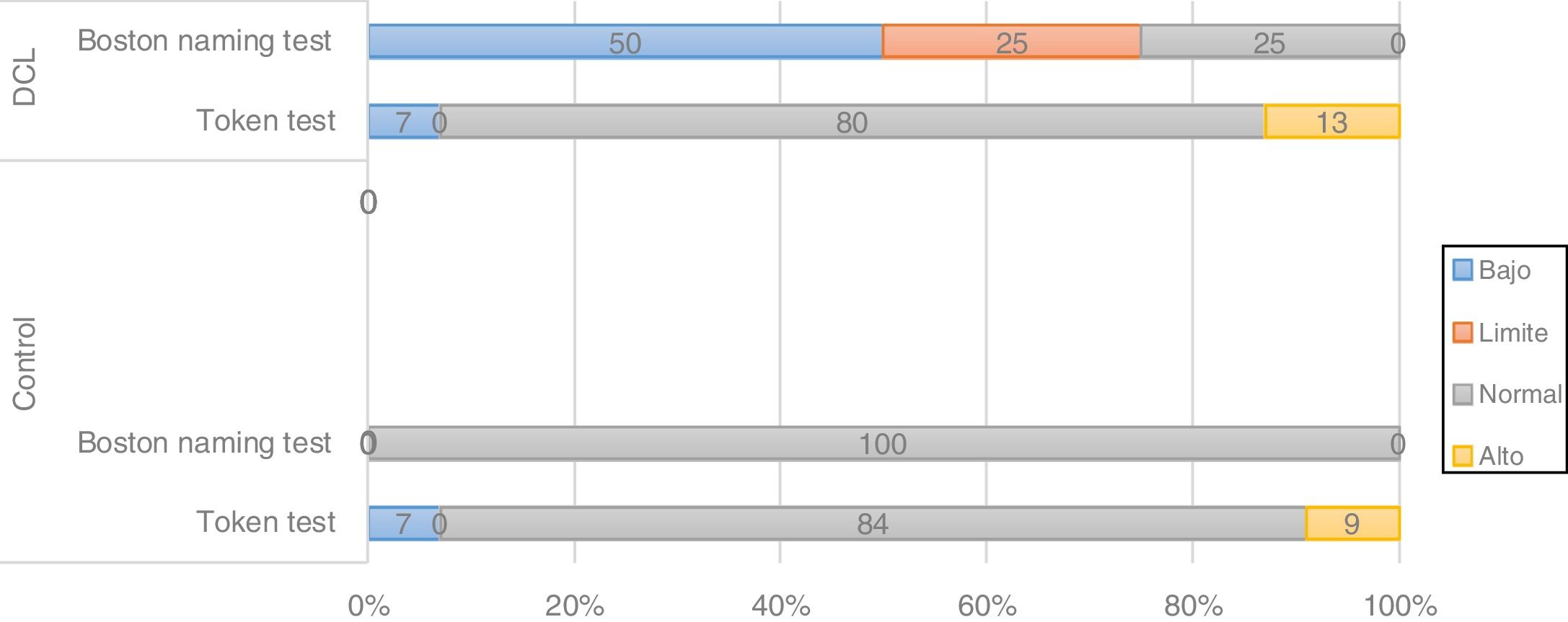
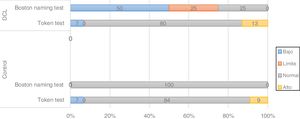
Para el gen BIN1 en la variante CC, el análisis de frecuencias mostró diferencias en la distribución de los desempeños cognitivos entre sujetos sanos y TNL, en donde el 50% de los pacientes con TNL portadores de la variante CC mostró rendimiento bajo y el 25% tuvo rendimiento límite en una tarea de lenguaje (Boston) (fig. 2), mientras que en portadores de las variantes CT y TT el rendimiento fue bueno en el 80 y en el 100% de los casos, respectivamente (datos no graficados). En sujetos sanos se encuentra que para todas las variantes de este gen el 100% de los casos se ubican en desempeños normales-altos, lo que podría indicar riesgo para la variante CC en TNL.
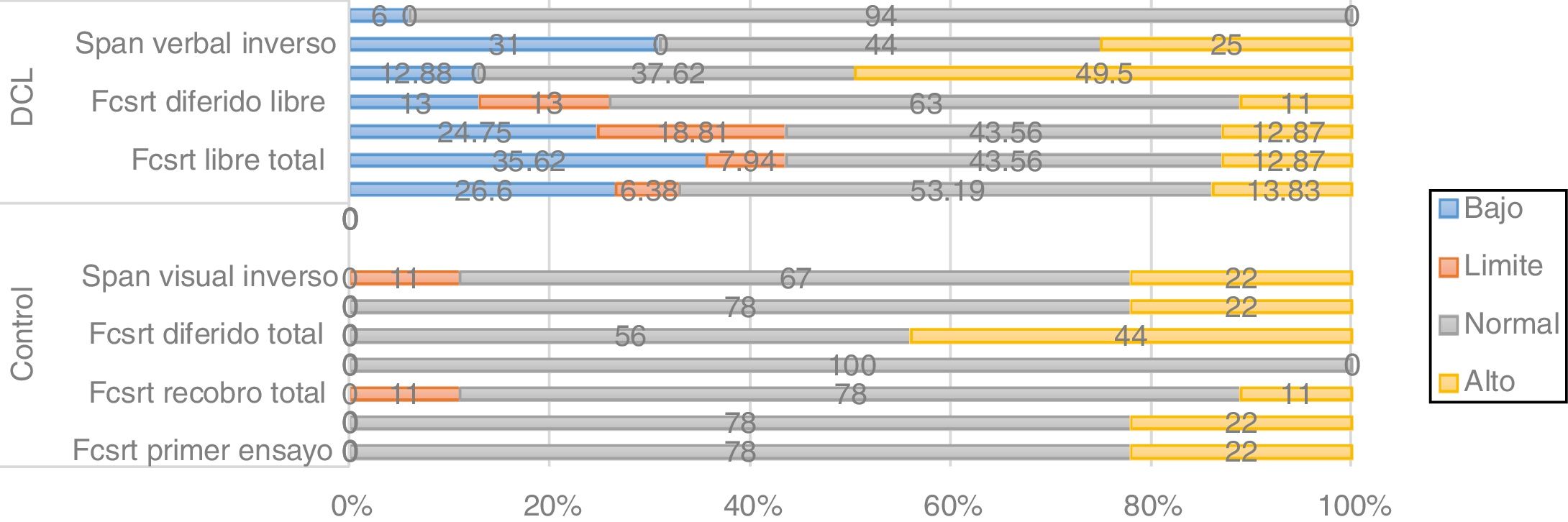
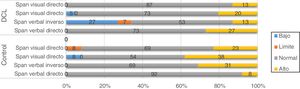
Los análisis de frecuencias también señalan tendencias en las variantes del gen CLU(rs11136000) en pacientes con TNL portadores de la variante CC, los cuales presentaron rendimiento bajo y límite en el 31% de los casos en pruebas de memoria (fig. 3). Otra tarea mnésica como el span visual inverso arrojó también diferencias en las comparaciones no paramétricas, por lo que se considera la hipótesis de que variantes de este gen se asocien a este proceso cognitivo.
En cuanto al gen CR1 en sujetos con TNL, los análisis de frecuencias muestran que portadores de la variante GG presentan desempeño bajo en cerca del 50% de los casos frente a 2pruebas que miden memoria (FCSRT .recobro.total con 46% entre límite y bajo y FCSRT.recobro.libre.total con 46%). En cuanto a los análisis no paramétricos, se encontraron diferencias en el desempeño en Boston, tanto en pacientes como en sujetos con TNL en función de las variantes de este gen, lo cual se asocia a los resultados obtenidos en el análisis de frecuencias, en tanto que el test de Boston implica la memoria semántica.
Para las variantes SORL, PVRL2, GWAS_14q, PICALM, aunque se encuentran tendencias relacionadas en los desempeños de algunas tareas en los análisis de frecuencias, tales como la tendencia a más bajos desempeños en la variante CC de PICALM en una tarea de memoria de trabajo (fig. 4), estas no son consistentes con los resultados obtenidos en los estadísticos no paramétricos. Además, implican amplios intervalos de confianza, por lo que se considera que podrían corresponder con la variabilidad en cognición presente en este tipo de pacientes. Por otra parte, para los polimorfismos de APOE y TOMM40 no se identifica ninguna tendencia (los resultados no se muestran aquí).
DiscusiónEn este estudio se exploran variantes genéticas y cognitivas en pacientes con TNL, considerando que dentro de este grupo existe un alto riesgo de desarrollar trastornos neurocognitivos mayores como la EA, y que el abordaje de los desempeños cognitivos en función de configuraciones genéticas puede fortalecer el carácter preventivo de la investigación13. Los resultados señalan algunas tendencias en los desempeños cognitivos en relación con determinadas variantes polimórficas, la cuales, aunque solo tienen un carácter exploratorio debido al tamaño recucido de la muestra, pueden permitir la generación de hipótesis acerca de la interacción entre estas variables genéticas y cognitivas que orienten estudios con tamaño de muestra mayor.
Para el caso de BIN1, el genotipo CC mostró tendencias en los desempeños en una tarea de lenguaje (Boston) que también se observaron en los estadísticos no paramétricos. Este gen es el segundo locus de riesgo para el desarrollo de Alzheimer de inicio tardío después de APOE30 y se ha asociado a peores desempeños cognitivos en sujetos jóvenes sanos17,30, lo que reafirma la hipótesis de su efecto sobre variables clínicas como la cognición.
En cuanto al polimorfismo CR1(rs6656401) se ha reportado asociación con declive cognitivo global y el genotipo GG, con déficit en memoria episódica tanto en pacientes como en personas sanas31, también ha sido asociado con los depósitos de placas neuríticas amiloides32 y fallas en memoria semántica, habilidad visoespacial y velocidad perceptual9. En nuestro estudio mostró tendencias con desempeños en el test de Boston, el cual implica procesos de memoria semántica y, dados los hallazgos del presente estudio, se puede considerar como una herramienta útil para abordar pacientes con TNL y su asociación con biomarcadores.
Con relación al gen PICALM(rs3851179) existen pocos reportes directamente relacionados con cognición, sin embargo, el alelo A es una variante protectora que participa en la eliminación de β amiloide (Aβ), mientras que los genotipos GG se asocian a bajo desempeño cognitivo y a cambios funcionales en la circunvolución frontal inferior derecha y el giro frontal superior bilateral, regiones relacionadas con la memoria episódica y las funciones ejecutivas3. De hecho, se ha encontrado que portadores de la variante GG presentan peores desempeños en tareas de fluidez verbal33. Y aunque en nuestra muestra la variante CC mostró tendencias con desempeños bajos en memoria de trabajo en los análisis de frecuencias, estas tendencias no fueron consistentes con los estudios no paramétricos.
En nuestro estudio, CLU(rs11136000) mostró diferencias significativas en tareas de memoria visual a corto plazo en función de sus variantes. En otros reportes este gen se ha relacionado con déficit en memoria episódica, el cual aumenta cuando se acompaña de ciertas isoformas de APOE31. Respecto a APOE, nuestros resultados no indican tendencias con las variantes cognitivas, lo cual se puede asociar a que en nuestra muestra el alelo de riesgo es poco frecuente, mientras que el genotipo 33 fue el más prevalente y, por tanto, puede reducir el impacto que suman algunas variantes de APOE sobre la cognición.
Un polimorfismo asociado de manera importante con la EA es GWAS_14q(rs11622883), debido a su posible participación sobre los mecanismos implicados en el aumento de β amiloide34; sin embargo, hasta el momento no se ha estudiado su influencia sobre la cognición. Nuestros análisis de frecuencias indican discretas tendencias en relación con tareas que implican velocidad de procesamiento y atención, pero estas corresponden a una exploración inicial que debe abrir paso a estudios con tamaño de muestra mayor. Así mismo, para la variante C de SORL1(rs11218304), aunque se encuentran tendencias a bajos desempeños en tareas de planeación, memoria y velocidad de procesamiento en portadores de la variante CC, estas no llegaron a ser significativas en los estadísticos no paramétricos. Este polimorfismo en particular no ha sido estudiado previamente en relación con cognición, pero su importancia en la fisiopatología de los trastornos neurodegenerativos lo hace un blanco de investigación que debe tener continuidad.
Respecto a PVRL2(rs6859), se encontraron tendencias hacia bajos desempeños en funcionamiento ejecutivo, atención y memoria. Este gen ha sido vinculado con procesos de longevidad35, bajo riesgo de desarrollo de EA y baja tasa de progresión36. Frente a la cognición, previamente ha sido relacionado como parte del efecto acumulativo que genera el clúster APOC3-APOE-TOMM40-PVRL2 localizado en el cromosoma 19q13.32, sobre la varianza de puntuaciones del MOCA test37. Sin embargo, su nivel de participación en esta interacción es incierto y requiere de más estudios.
Finalmente, aunque el gen TOMM40 es candidato importante para el desarrollo de EA, ha mostrado relación con la cognición38,39 y se ha asociado con la edad de inicio de la enfermedad40, en el presente estudio no mostró tendencias respecto a los procesos cognitivos en función de sus variantes. Sin embargo, dado que en nuestra población este gen ha mostrado relación con la edad de inicio de la enfermedad, se considera que deben continuar los estudios respecto a su asociación con variables clínicas, incluyendo la cognición.
Dentro de las limitaciones del estudio cabe mencionar que, aunque los criterios de exclusión de nuestra muestra se enfocaron a reducir la influencia de variables asociadas a declive cognitivo no neurodegenerativo, en los análisis de los datos no se controlaron los efectos de las variables de confusión, tales como dislipidemia, hipertensión arterial, diabetes mellitus, tabaquismo y sedentarismo, las cuales constituyen factores de riesgo para enfermedad neurodegenerativa41.
Aunque los resultados de este estudio indican tendencias sobre desempeños cognitivos diferenciales con relación a las combinaciones alélicas de algunos polimorfismos, los intervalos de confianza son amplios, lo cual se puede asociar al tamaño de la muestra y no permite considerar estas diferencias respecto a la población general. Por lo tanto, estos resultados se deben interpretar a manera de hipótesis, las cuales requieren estudios con un tamaño de muestra mayor que permita alcanzar el poder estadístico necesario para su comprobación, y que permita explorar en profundidad la influencia de las variables genéticas sobre el TNL en nuestra población.
Fuentes de financiaciónEl presente trabajo ha sido financiado por Colciencias en el marco de la Convocatoria para proyectos de ciencia, tecnología e innovación en salud 2015.
Conflicto de interesesNinguno.
A todos los pacientes, sus familias y los sujetos cognitivamente sanos que participaron en este estudio.