La esclerosis múltiple (EM) es una enfermedad desmielinizante que afecta a adultos jóvenes, grupo en que supone la segunda causa de discapacidad en nuestro medio. Su etiología precisa no está dilucidada, pero se acepta que se presenta en pacientes predispuestos genéticamente que se ven expuestos a determinados factores ambientales. El descubrimiento del papel regulador de la microbiota intestinal en diversas enfermedades autoinmunes ha abierto una nueva línea de investigación en este campo, lo que se discute en esta revisión.
DesarrolloRevisamos los estudios publicados acerca del papel de la microbiota en el desarrollo de la EM y su modelo animal, la encefalomielitis autoinmune experimental (EAE). En ratones, se ha demostrado que los microorganismos intestinales regulan la polarización de las células T helper de Th1-Th17 hasta Th2, la función de las células T reguladoras y la actividad de las células B, participando en la génesis de la EAE, así como en su prevención y tratamiento. Por el contrario, en humanos la evidencia es aún escasa, fundamentalmente en base a estudios de casos control que apuntan a la existencia de diferencias en determinadas comunidades bacterianas.
ConclusionesExiste múltiple evidencia del papel de la microbiota en la EAE. La extrapolación de los resultados a la EM está en las primeras fases de investigación, y hacen falta estudios que definan qué poblaciones bacterianas se asocian a la EM, su papel en la patogenia y las posibilidades terapéuticas que esto nos ofrezca.
Multiple sclerosis (MS) is a demyelinating disease that affects young adults; in that age group, it represents the second leading cause of disability in our setting. Its precise aetiology has not been elucidated, but it is widely accepted to occur in genetically predisposed patients who are exposed to certain environmental factors. The discovery of the regulatory role played by intestinal microbiota in various autoimmune diseases has opened a new line of research in this field, which is discussed in this review.
DevelopmentWe reviewed published studies on the role of the microbiota in the development of both MS and its animal model, experimental autoimmune encephalomyelitis (EAE). In mice, it has been shown that intestinal microorganisms regulate the polarisation of T helper cells from Th1-Th17 up to Th2, the function of regulatory T cells, and the activity of B cells; they participate in the pathogenesis of EAE and contribute to its prevention and treatment. In contrast, evidence in humans is still scarce and mainly based on case-control studies that point to the presence of differences in certain bacterial communities.
ConclusionsMultiple evidence points to the role of microbiota in EAE. Extrapolation of these results to MS is still in the early stages of research, and studies are needed to define which bacterial populations are associated with MS, the role they play in pathogenesis, and the therapeutic possibilities this knowledge offers us.
La esclerosis múltiple (EM) es una enfermedad desmielinizante que se caracteriza por la aparición de lesiones inflamatorias en diferentes topografías de la sustancia blanca del sistema nervioso central (SNC) que se resuelven dejando una cicatriz gliótica, con desmielinización, pérdida de oligodendrocitos y daño axonal1. Afecta a más de 2,5 millones de personas en el mundo y a alrededor de 46.000 en España, con una prevalencia aproximada de 100 afectados por cada 100.000 habitantes2. Su incidencia, al igual que ocurre con otras enfermedades de causa autoinmune, está aumentando en los últimos años3. Es una enfermedad que suele presentarse en adultos jóvenes4, donde representa la segunda causa de discapacidad en nuestro medio (tras los traumatismos)5, lo que, unido a una expectativa de vida prolongada (6años menos que la población general)6, hace que presente una larga evolución, con frecuencia de más de 30años7, con las implicaciones clínicas, sociales y económicas que comporta este hecho.
La etiología precisa de la EM no está aún dilucidada, aunque los datos epidemiológicos indican que tanto los factores genéticos como los ambientales son importantes8,9, iniciándose en pacientes predispuestos genéticamente que se ven expuestos a determinados factores ambientales10 que actúan fundamentalmente durante la infancia11. Entre estos factores están bien estudiados la infección por el virus de Epstein-Barr12-15, el tabaquismo16–18, la latitud8 y la vitaminaD19–21. Recientemente se están estudiando otros factores no tan documentados, como la ingesta de sodio22,23, el índice de masa corporal elevado en la adolescencia12, la leptina, la hormona producida por el tejido graso24, la vitaminaA25 y el alcohol26, o relacionados con la teoría de la higiene, defectos en el contacto con helmintos27 o con Helicobacter pylori28. Entre los nuevos factores ambientales en estudio se encuentran los microorganismos constituyentes de la flora bacteriana intestinal, objeto de esta revisión.
La microbiota intestinalNuestro tracto intestinal contiene más de 100 billones (1014) de microbios, la gran mayoría en el colon (1011-1012), superando el número de células humanas en un factor de 102–4. Esta compleja comunidad microbiana es conocida como la microbiota gastrointestinal, y está constituida por bacterias, arqueas, eucariotas, hongos y virus29,30. Así, se ha propuesto que el ser humano es un «meta organismo» con 10-100 veces más células bacterianas que humanas, que se integran desde el punto de vista metabólico e inmunológico31. La composición de la microbiota, además de por la localización, es influida por la edad, el sexo, la raza y otros factores como dieta, medicación (especialmente antibióticos), estrés, tabaquismo o infecciones gastrointestinales, así como propios de cada individuo30–32. Incluso dentro de cada persona se presentan grandes variaciones en su composición si se mide a diferentes tiempos33. Aunque a día de hoy resulta imposible definir el concepto de microbiota saludable, sí sabemos que la riqueza y la diversidad de la microbiota son indicadores de su salud34, y que su empobrecimiento se asocia a obesidad y marcadores metabólicos35. En cuanto a la composición cualitativa, están surgiendo numerosos estudios que tratan de relacionar a determinadas clases de microorganismos con diferentes estados fisiológicos. Se propone que los hay que mejoran el estado metabólico, la resistencia a infecciones, cáncer o autoinmunidad, la inflamación, la señalización endocrina y la funcionalidad cerebral (eje cerebro-intestino). Hoy en día se consideran bacterias asociadas a salud: Bacteroides, Bifidobacterium, Clostridium grupo XIVa y IVa (productoras de butirato), Eubacterium, Faecalibacterium, Lacobacillus y Roseburia32.
En resumen, podemos decir que la relación entre la microbiota y el ser humano está redefiniéndose de una relación comensal a mutualista, donde las bacterias aportan funciones biológicas no codificadas genéticamente en nuestro organismo que van desde la actividad metabólica hasta la homeostasis inmunológica (tabla 1)36,37, hablándose de la microbiota como un órgano virtual fundamental en la respuesta inmunológica. Hay en marcha numerosos estudios relacionados con el papel de la flora intestinal en la salud humana, en infecciones, enfermedades inflamatorias, neoplásicas, y lo que aquí nos ocupa, autoinmunes38.
Funciones biológicas de la microbiota intestinal en el ser humano
| Integridad de la barrera epitelial |
| Barrera a la colonización de patógenos |
| Desarrollo de la respuesta inmune |
| Fuente de energía |
| Biosíntesis de vitaminas |
| Transformación de sales biliares |
| Metabolismo de xenobióticos |
| Maduración y desarrollo del sistema nervioso central |
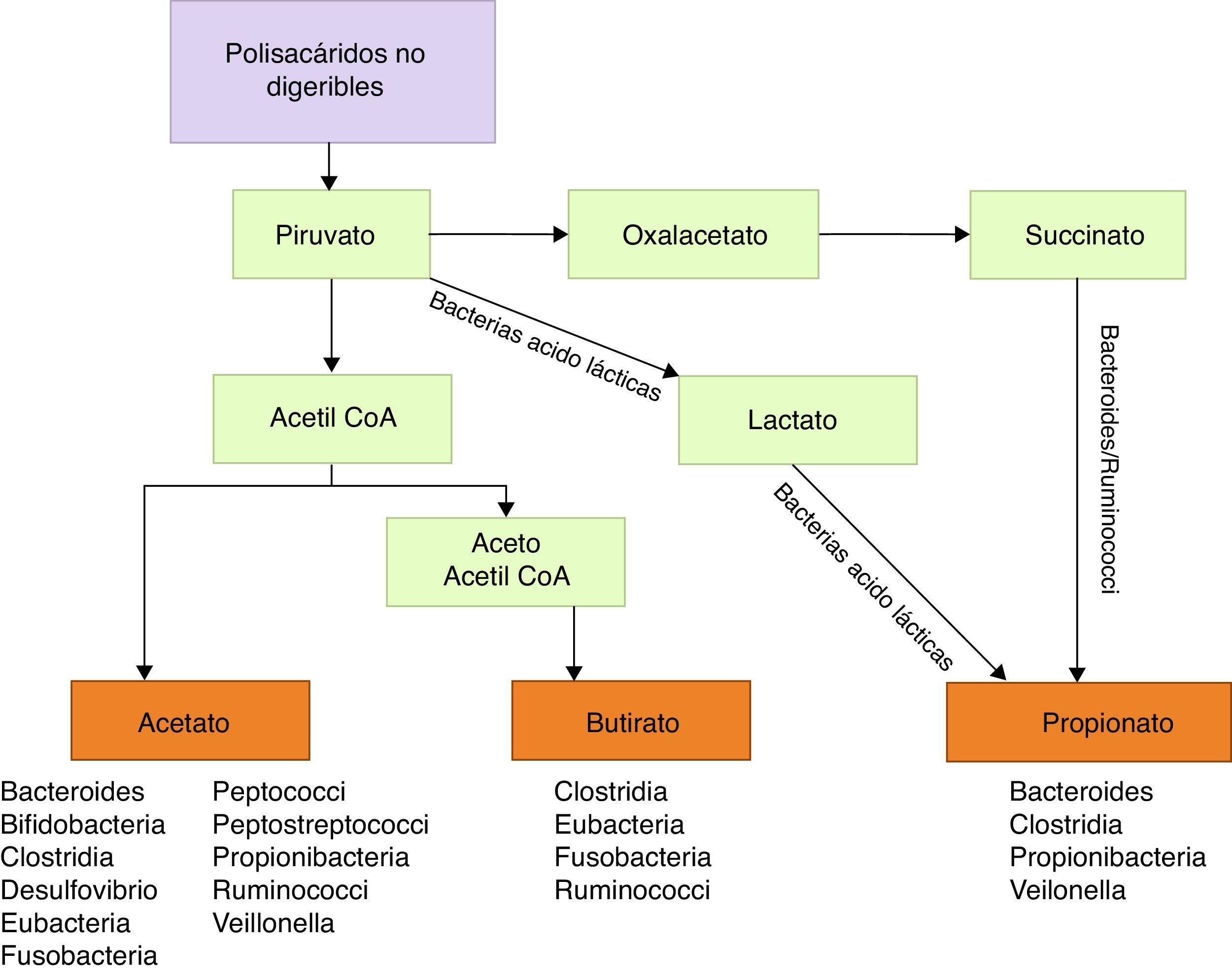
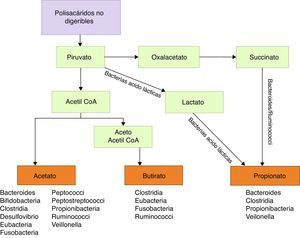
Entre las funciones metabólicas podemos citar la degradación de polisacáridos vegetales hasta ácidos grasos de cadena corta, como butirato, propionato y acetato, con propiedades antiinflamatorias y sustrato energético principal de colonocitos, por tanto implicados en la función barrera de la mucosa intestinal39 (fig. 1). Algunas bacterias de la flora, particularmente lactobacilli, se han implicado en el metabolismo del colesterol40,41 y en la producción de vitaminaK y vitaminas de grupoB41. También están implicadas en el metabolismo de xenobióticos, fármacos, antibióticos o productos bioactivos42, condicionando su farmacocinética y la producción de determinados tóxicos implicados en algunas neoplasias43,44.
En los últimos años ha surgido el concepto eje microbiota intestinal-cerebro, que hace referencia a la conexión, tanto aferente como eferente, entre el cerebro y el sistema gastrointestinal mediante señales neuroendocrinas, nutrientes y señales inmunológicas45. Se ha demostrado la influencia de la microbiota en la funcionalidad del SNC, manifiesta tanto en condiciones fisiológicas como durante la enfermedad46, implicándose en el desarrollo cerebral47, en el eje hipotálamo-hipófisis-adrenal48,49, en la expresión de receptores de serotonina 5-HT1 y en el recambio de neurotransmisores como la propia serotonina, la dopamina o la noradrenalina, o cambios en proteínas que regulan el desarrollo y la función de las sinapsis neuronales47. Con todo esto, se ha implicado a la microbiota en el desarrollo del control motor, de la ansiedad o de funciones cognitivas relacionadas con el desarrollo cerebral en las primeras etapas de la vida50,51.
La microbiota y la respuesta inmuneLa microbiota contribuye a diversas funciones inmunológicas. En la barrera intestinal previene la colonización y el crecimiento de microorganismos patógenos52,53, madura la barrera inmune, tanto estimulando la respuesta innata a través de los receptores toll-like receptors (TLR) y NOD-like receptors (NLR)54–56 como la adaptativa, con rol importante en la secreción de mucinas, péptidos antimicrobianos, defensinas e IgA53. En cuanto al desarrollo de la respuesta inmunológica sistémica, trabajando con ratones libres de gérmenes se ha observado que la microbiota interviene en la regulación y la maduración de las placas de Peyer, de nódulos linfáticos mesentéricos y de los centros germinales. También regula el número de células plasmáticas productoras de IgA, de células Tγδ intestinales y de linfocitos T CD4+ en la lámina propia o intraepiteliales, e interviene en la expresión génica de TLR y del complejo mayor de histocompatibilidadii53.
La microbiota también condiciona el desarrollo de las células T efectoras y la producción de citocinas, destacando la influencia sobre los linfocitos Th17 y Treg, implicados en la respuesta autoinmune y su regulación, y por tanto en enfermedades autoinmunes en general y en la EM en particular. Los ratones germ free (ratones criados para carecer de flora intestinal) presentan reducción en sus linfocitos Th1 y Th17, balanceando la respuesta inmune T hacia Th2, lo que se revierte al reconstituir en estos animales la flora intestinal normal57,58. Se ha propuesto que la microbiota está implicada en el paso de los linfocitos Th17 estacionarios a linfocitosT proautoinmunes, de modo que los microorganismos mutualistas inducen la producción de Th17 en estado estacionario, que en presencia de un microambiente proinflamatorio, promovido por determinadas citocinas como IL-12, IL-23, IL-1β o TGF-β3, pasarían a Th17 patogénico, productor de IFN-γ, contribuyendo a la progresión del ambiente inflamatorio intestinal59,60. Se ha demostrado que la implicación de una simple bacteria, la bacteria filamentosa segmentada (BFS), puede contribuir a esta actividad Th17 proautoinmune61.
En cuanto a los Treg, la microbiota es indispensable para su desarrollo y función62. Estos linfocitos regulan la inflamación que se genera frente a estímulos microbianos a través de la IL-1059. Numerosos agentes microbianos se han relacionado con la inducción de Treg63–65, destacando el papel de Bacteroides fragilis —y concretamente su polisacáridoA (PSA)— con el desarrollo de linfocitos T reguladores Foxp3+ productores de IL-10, y con la prevención y cura de colitis experimentales en modelos de animales, muestra de su papel clave en la regulación de la tolerancia inmunológica66,67.
Los ya mencionados ácidos grasos de cadena corta, especialmente el butirato, balancean al sistema inmunológico hacia un «estado antiinflamatorio» aumentando la producción de IL-10 e IL-468, reduciendo la adherencia vascular de los leucocitos mediados por VCAM-169, inhibiendo la función del IFN-γ, y por tanto su capacidad proinflamatoria70, regulando los linfocitos Treg71–73 y la función inflamatoria de los leucocitos74,75.
Microbiota y enfermedades desmielinizantesAdemás de la relación descrita de la microbiota en el desarrollo cerebral, están apareciendo evidencias que relacionan la flora intestinal con diversas enfermedades neurológicas, fundamentalmente dentro de espectro de las autoinmunes. Existe algún grado de evidencia del papel de la microbiota en trastornos del espectro autista76, el síndrome de Guillain-Barré77 y trastornos psiquiátricos como depresión, ansiedad o esquizofrenia78. En cuanto a las enfermedades desmielinizantes, hay una amplia línea de investigación en la relación de la microbiota intestinal con la neuromielitis óptica, y fundamentalmente con la encefalomielitis autoinmune experimental (EAE), modelo animal de la EM, y una línea incipiente que está tratando de extrapolar los resultados de la EAE a la EM.
Los pacientes con neuromielitis óptica acuaporina positiva presentan una respuesta sérica de anticuerpos contra antígenos gastrointestinales mayor que los controles sanos, especialmente en mielitis extensa, lo que parece relacionado con el control de la microbiota sobre la inflamación autoinmune79. También se ha demostrado la existencia periférica de células T específicas para acuaporina4, capaces de desarrollar respuestas Th17, que presentaban una reactividad cruzada con una secuencia homóloga en un epítopo presente en Clostridium perfringens, representante de la flora comensal, insinuando un mecanismo de mimetismo celular responsable de activar la mencionada respuesta Th1780.
Microbiota y encefalomielitis autoinmune experimentalLas primeras evidencias que comenzaron a relacionar a factores bacterianos intestinales con la tolerancia periférica y la prevención o tratamiento de la EAE se hicieron mediante «vacunación» con el factor antigénico de colonización i (CFA/I) fimbria de Escherichia coli enterotoxigénico, normalmente a través de inmunizaciones orales con cepas de Salmonella typhimurium atenuadas y modificadas para expresar el mencionado antígeno. Los ratones que recibían dicha profilaxis sufrían una enfermedad leve, de la que se recuperaban. Histopatológicamente presentaban menos infiltrados en la sustancia gris y blanca de la médula espinal, junto con pérdida de expresión de célulasT secretoras de IFN-γ y de un aumento de secreción de IL-4, IL-10, IL-1381 y TGF-β82. Se demostró que esta «vacuna» presentaba propiedades antiinflamatorias asociadas, implicando a los linfocitos TregFoxP3+ en dicho mecanismo de protección83,84.
El siguiente paso fue implicar a la microbiota directamente en el desarrollo de la EAE. Surgieron estudios en que se relacionaba su alteración mediante tratamiento con antibióticos de amplio espectro con una mejoría en la EAE. Así, administrando una mezcla de kanamicina, colistina y vancomicina a ratones antes de la inducción de la enfermedad se suprimía su desarrollo, acompañado de una menor producción de citocinas proinflamatorias (IL-6, IL-17, IFN-γ y TNF-α) y una reducción en las células Th17 mesentéricas85. Posteriormente, Ochoa Repáraz et al.86 repitieron la experiencia con ampicilina, vancomicina, neomicina y metronidazol por vía oral e intraperitoneal, más un grupo control no tratado. Mostraron que tan solo en el grupo tratado por vía oral se reducía la severidad de la EAE y se acumulaban linfocitos TregFoxP3+ en nódulos linfáticos periféricos, cervicales, además de en los mesentéricos, sugiriendo una regulación de células Treg mas allá del intestino. Implicaban en este mecanismo a las células dendríticas CD103+ y mostraban un aumento en los niveles de IL-13 y una disminución de IL-17. Además, cuando los animales eran recolonizados con bacterias comensales, el efecto protector se perdía. También demostraron que los ratones con EAE tratados con antibióticos modificaban sus célulasB, induciendo una subpoblación B CD5+ que le confería protección frente a la EAE, y que in vitro, estimuladas con lipopolisacárido, generaban IL-1087. La simple transferencia de estas célulasB CD5+ a ratones no tratados con antibióticos reducía notablemente la severidad de la EAE.
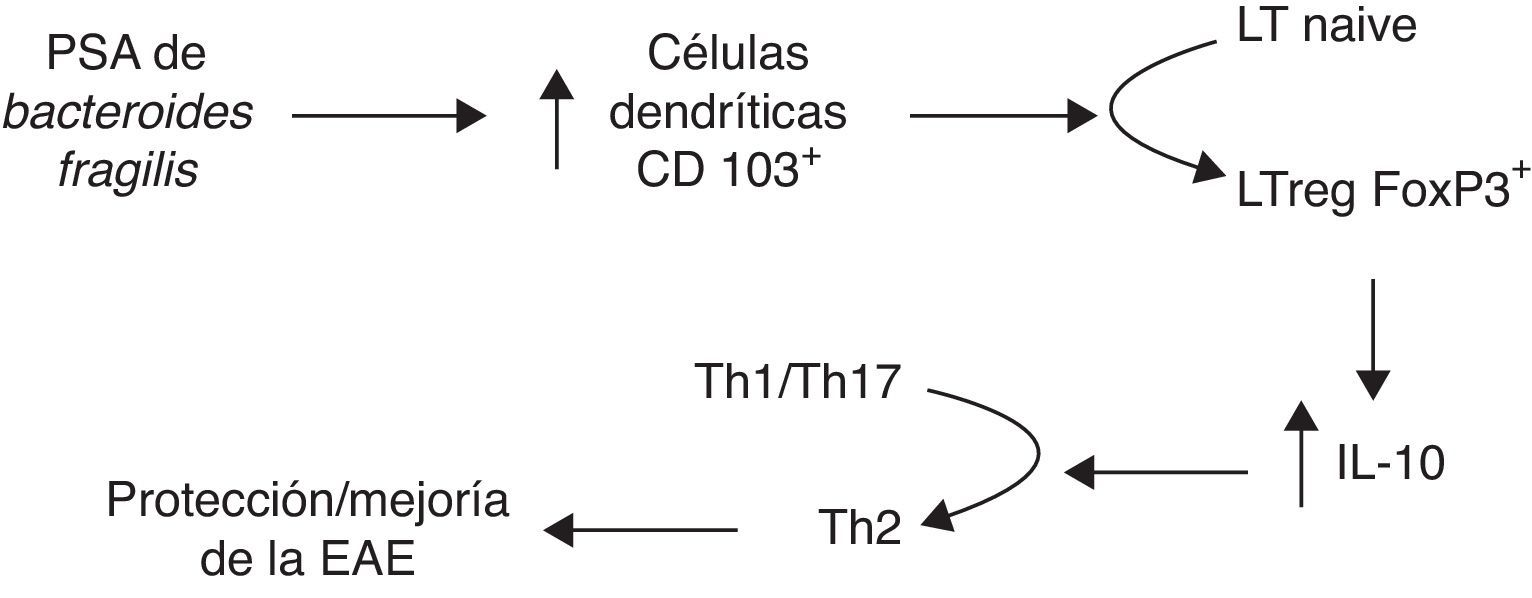
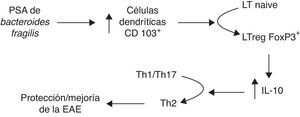
Otra línea abierta de investigación es a través del PSA de Bacteroides fragilis, conocido por modular el sistema inmunológico a través de la corrección en las desviaciones en la relación Th1/Th257, y su papel en la enfermedad inflamatoria intestinal66. Por esto, el grupo de Ochoa Repáraz88 testó este mismo antígeno PSA en la EAE. En primer lugar reconstituyeron la flora intestinal con B.fragilis salvajes, capaces de producir PSA, o con cepas deficientes en dicho polisacárido en ratones con la flora suprimida por antibióticos orales. Aquellos reconstituidos con las cepas productoras de PSA mantenían la resistencia a la EAE y tenían un mayor ratio de conversión en linfocitos TregFoxp3+ productores de IL-10. Posteriormente se testó la administración por vía oral de PSA purificado, demostrándose un efecto tanto profiláctico como terapéutico sobre la EAE que se perdía en ratones deficientes en IL-10. También observaron que los nódulos linfáticos cervicales presentaban un aumento en células dendríticas CD103+ y linfocitos TregFoxp3+, proponiendo el modelo mostrado en la figura 2 como mecanismo por el que el PSA mejora la enfermedad en ratones89. Recientemente, en 2014 y 2015, este grupo ha presentado 2 trabajos estudiando el mecanismo que media la protección del PSA frente a la EAE, observando que los toll-like receptor2 (TLR2) median la expansión de linfocitos T CD4 CD39+, y que este CD39 confiere un fenotipo inmunomodulador a linfocitosT en global y a los TregsFoxp3+ en particular90,91.
También se han estudiado microorganismos responsables de generar la enfermedad, como la BFS, capaz de inducir una respuesta pro autoinmune Th1761. Lee et al.92 trataron de inducir la EAE en ratones germ free y observaron que presentaban una forma de enfermedad atenuada, con niveles más bajos de IFN-γ y de IL-17ª tanto intestinal como medular, con una respuesta inflamatoria Th1/Th17 reducida en el SNC y niveles elevados de células TregFoxP3+. Demostraron que la simple colonización con BFS generaba de nuevo la EAE, acompañada de un aumento de IL-17 en el intestino, y de linfocitos Th17 en el SNC, el colon y el intestino delgado, demostrando que esta única bacteria intestinal podía generar una inflamación a nivel neurológico y, consecuentemente, la enfermedad.
También trabajando con ratones germ free, capaces de desarrollar la EAE de forma espontánea, Berer et al.93 demostraron que se requería la presencia del autoantígeno diana, el MOG, y de microbiota para desarrollar la enfermedad. Demostraron que los ratones sin gérmenes presentaban un descenso de linfocitos Th17 en la pared intestinal, la lámina propia y las placas de Peyer, y un descenso en la producción de IFN-γ e IL-17 en el bazo. Observaron que la producción de anticuerpos contra MOG estaba disminuida en ratones sin microbiota y que rápidamente aumentaba si se recolonizaban.
Otro grupo de bacterias comensales habituales relacionadas con la regulación de la respuesta inmune y estudiadas en la EAE son las acidolácticas. Dentro de este grupo se encuentra Pediococcus acidilactici, que administrada por vía oral induce una respuesta mediada por IL-10 que disminuye la severidad de la EAE, tanto de modo terapéutico como profiláctico, a través de la inhibición de IL-17 e IFN-γ, y una disminución de los infiltrados celulares en el SNC. En este caso, los linfocitos responsables relacionados, en lugar de ser TCD4+FoxP3+ (con un aumento leve), fueron los linfocitosT reguladores tipo1 (Treg1)94.
Más allá de las bacterias, se ha publicado en 2015 un trabajo con Candida kefyr, una levadura, y su impacto sobre la EAE y sobre la microbiota de los ratones95. La administración de la levadura producía una mejora clínica mediada por la inducción de células dendríticas CD103+ y células TregFoxP3+. Sabemos que, de hecho, estas células dendríticas estimulan a linfocitos Treg vía ácido retinoico y TGF-β96. El análisis de la microflora de los ratones tras la administración de C.kefyr mostró un aumento de Lactobacillales y Prevotella a costa de disminuir los Bacteroides, disminución que los autores consideraban responsable de la mejoría, mediada por el descenso en la producción de IL-695.
Por último, se ha investigado el potencial del uso de probióticos en la EAE. Se ha trabajado con Bifidobacterium animalis durante la lactancia de roedores que posteriormente se inducían para la EAE, observándose una reducción de la duración de los síntomas clínicos, curiosamente solo en los ratones macho97. Usando una combinación de 3 cepas de lactobacillus, demostraron que la combinación, pero no cada una por separado, revertía la EAE en ratones, asociado a un aumento de linfocitos TregFoxP3+ y de IL-4, IL-10 y TGF-β1 en los nódulos linfáticos mesentéricos y el bazo98. Usando otras mezclas de probióticos (lactobacillus, bifidobacterium y streptococcus) se han obtenido resultados similares, y también asociando en el mecanismo a la IL-10 y al desarrollo de Treg, lo que, como ya hemos comentado, genera una menor polarización de los linfocitos T helper hacia Th1/Th1799.
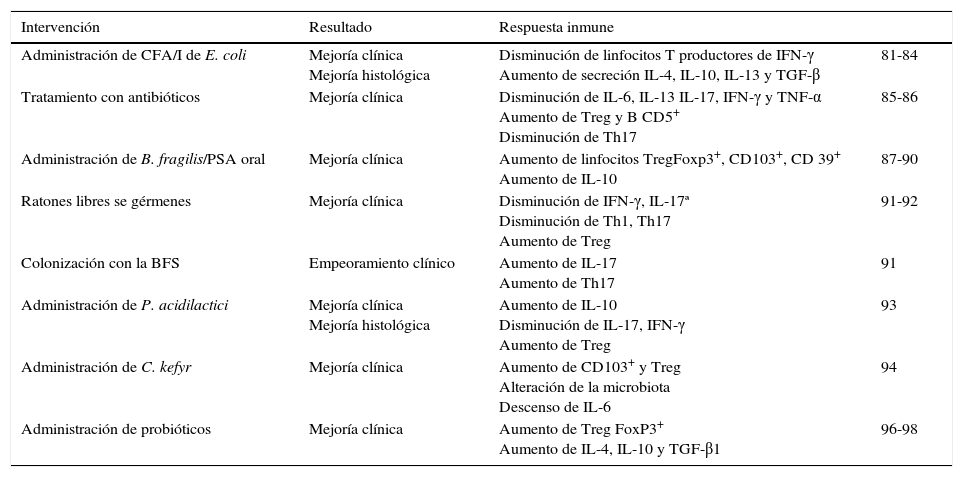
A modo de resumen, hemos visto diversos estudios, recogidos en la tabla 2, que en conjunto proveen una fuerte evidencia de la modulación de la respuesta inmunológica en la EAE por parte de la microbiota, que influye en la génesis, la prevención y el tratamiento de la enfermedad en modelos animales100. En cuanto a los mecanismos inmunológicos implicados en esta modulación, giran fundamentalmente en torno a 3 pilares: la polarización de las células T helper de Th1-Th17 hacia Th2, la función de las célulasT reguladoras y la actividad de las célulasB101.
Resumen de los estudios de microbiota en la encefalomielitis autoinmune experimental
| Intervención | Resultado | Respuesta inmune | |
|---|---|---|---|
| Administración de CFA/I de E. coli | Mejoría clínica Mejoría histológica | Disminución de linfocitos T productores de IFN-γ Aumento de secreción IL-4, IL-10, IL-13 y TGF-β | 81-84 |
| Tratamiento con antibióticos | Mejoría clínica | Disminución de IL-6, IL-13 IL-17, IFN-γ y TNF-α Aumento de Treg y B CD5+ Disminución de Th17 | 85-86 |
| Administración de B. fragilis/PSA oral | Mejoría clínica | Aumento de linfocitos TregFoxp3+, CD103+, CD 39+ Aumento de IL-10 | 87-90 |
| Ratones libres se gérmenes | Mejoría clínica | Disminución de IFN-γ, IL-17ª Disminución de Th1, Th17 Aumento de Treg | 91-92 |
| Colonización con la BFS | Empeoramiento clínico | Aumento de IL-17 Aumento de Th17 | 91 |
| Administración de P. acidilactici | Mejoría clínica Mejoría histológica | Aumento de IL-10 Disminución de IL-17, IFN-γ Aumento de Treg | 93 |
| Administración de C. kefyr | Mejoría clínica | Aumento de CD103+ y Treg Alteración de la microbiota Descenso de IL-6 | 94 |
| Administración de probióticos | Mejoría clínica | Aumento de Treg FoxP3+ Aumento de IL-4, IL-10 y TGF-β1 | 96-98 |
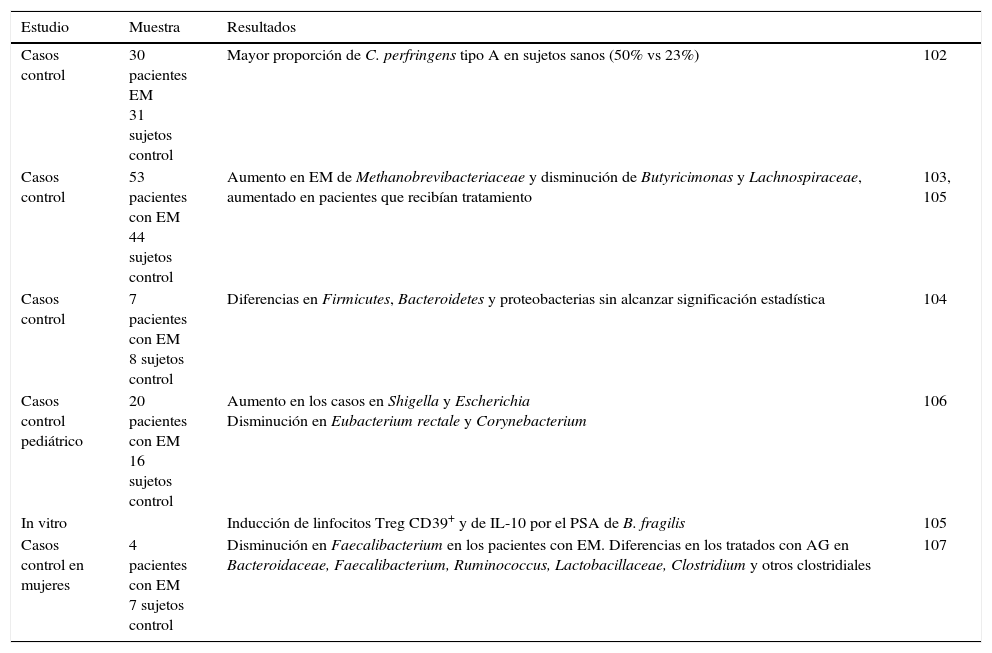
A diferencia de los modelos experimentales en ratones, los estudios de la microbiota en la EM, es decir en humanos, están en las primeras fases de investigación. La bibliografía procede fundamentalmente de comunicaciones a congresos102, evidencias indirectas y algún pequeño estudio publicado (tabla 3).
Resumen de los estudios de microbiota en esclerosis múltiple
| Estudio | Muestra | Resultados | |
|---|---|---|---|
| Casos control | 30 pacientes EM 31 sujetos control | Mayor proporción de C. perfringens tipo A en sujetos sanos (50% vs 23%) | 102 |
| Casos control | 53 pacientes con EM 44 sujetos control | Aumento en EM de Methanobrevibacteriaceae y disminución de Butyricimonas y Lachnospiraceae, aumentado en pacientes que recibían tratamiento | 103, 105 |
| Casos control | 7 pacientes con EM 8 sujetos control | Diferencias en Firmicutes, Bacteroidetes y proteobacterias sin alcanzar significación estadística | 104 |
| Casos control pediátrico | 20 pacientes con EM 16 sujetos control | Aumento en los casos en Shigella y Escherichia Disminución en Eubacterium rectale y Corynebacterium | 106 |
| In vitro | Inducción de linfocitos Treg CD39+ y de IL-10 por el PSA de B. fragilis | 105 | |
| Casos control en mujeres | 4 pacientes con EM 7 sujetos control | Disminución en Faecalibacterium en los pacientes con EM. Diferencias en los tratados con AG en Bacteroidaceae, Faecalibacterium, Ruminococcus, Lactobacillaceae, Clostridium y otros clostridiales | 107 |
En primer lugar podemos citar un ensayo, diseñado para relacionar la producción de toxina épsilon de Clostridium perfringens tipoB, no comensal en humanos, con la EM, donde se detectó una mayor proporción de C.perfringens tipoA, este sí parte de la microbiota comensal, en los controles sanos (50%) que en los pacientes con EM (23%)103.
En la reunión de la Academia Americana de Neurología de 2014 se presentaron 2 trabajos en forma de póster que hacían referencia a microbiota en la EM. El grupo de Jhangi el al.104 presentó un aumento en pacientes con EM respecto a controles sanos de Methanobrevibacteriaceae (Archaea) que se ha relacionado con procesos inflamatorios y una disminución de 2 organismos potencialmente antiinflamatorios, productores de butirato, Butyricimonas (perteneciente al phylum Bacteroidetes) y la familia Lachnospiraceae (del phylum Firmicutes), aumentado en pacientes que recibían tratamiento. En el segundo trabajo, encontraron diferencias en Firmicutes, Bacteroidetes y proteobacterias en pacientes con EM y controles, pero no alcanzaron significación estadística (trabajaron con 7 enfermos y 8 controles)105.
Durante la reunión ACTRIMS-ECTRIMS de 2014 en Boston se presentaron diversos posters reunidos bajo la denominación «microbiome», que englobaba 10 presentaciones entre las que había trabajos de investigación básica, con animales y 4 realizados en humanos106. En el primero, en población pediátrica, compararon 20 casos con 16 controles, encontrando un enriquecimiento significativo en los casos en Shigella y Escherichia, bacterias asociadas a infecciones gastrointestinales, y un empobrecimiento en Eubacterium rectale y Corynebacterium107. Un segundo trabajo, del grupo de Jhangi, trataba de determinar si existían diferencias en la microbioma intestinal en EM, y si esta se asociaba a algún fenotipo celular inmune. A lo aportado en el poster de la Academia Americana de Neurología, añadían que la presencia de Archaea se asociaba un fenotipo activado de células presentadoras de antígeno y a la expresión de IFN-γ.
El grupo de Ochoa Repáraz presentó un trabajo en que cultivaron células presentadoras de antígeno y linfocitosT en presencia de PSA, encontrando que el antígeno era capaz de inducir Treg CD39+HLA-DR-DR+FoxP3+ e IL-10, y que estos linfocitos Treg inducidos por PSA eran más capaces de suprimir el TNF-α que los controles. Por último, se presentó el grupo MS Microbiome Consortium (MSMC), que nace con el objetivo de trabajar de modo multicéntrico en la evaluación del efecto de la disbiosis en la EM.
Ya en 2015 se ha publicado un pequeño subestudio realizado con mujeres enroladas en un ensayo para demostrar la utilidad de la suplementación con vitaminaD3 en el tratamiento de la EM. Reclutaron 15 sujetos (8 sanos como control y 7 con EM), de los cuales 2 no llevaban tratamiento y 5 estaban tratados con acetato de glatiramero (AG) (3 de los cuales entregaron mal la muestra). Tomaron muestras de heces antes y tras la suplementación con vitaminaD. El estudio, muy limitado por el tamaño muestral, puso de manifiesto diferencias entre algunos géneros bacterianos entre los pacientes con EM y los sujetos control. Destaca la familia Faecalibacterium, considerada antiinflamatoria y relacionada con la enfermedad inflamatoria intestinal, que era menos abundante en los pacientes con EM y que aumentó su presencia en pacientes suplementados con vitaminaD que no estaban en tratamiento con AG. Comparando los pacientes tratados con AG frente a los no tratados, también se encontraron diferentes comunidades bacterianas intestinales en Bacteroidaceae, Faecalibacterium, Ruminococcus, Lactobacillaceae, Clostridium y otros clostridiales, lo cual puede estar relacionado con la modulación del sistema inmunológico por el fármaco108.
ConclusionesEn los últimos años están apareciendo numerosas publicaciones que implican a la microbiota intestinal en diversas funciones humanas, entre las que destaca la regulación inmunológica. Esto ha derivado en una intensa investigación del papel de estos microorganismos en diversos estados de salud y enfermedad, especialmente en las enfermedades autoinmunes.
En el campo de la investigación en EM existe a día de hoy una sólida evidencia científica del papel de la microbiota en la génesis, la prevención y el tratamiento de la EAE, modelo animal más aceptado de la enfermedad. El paso necesario de extrapolar estos resultados a la EM no ha hecho más que empezar, con la publicación de algunos estudios casos control.
Nos encontramos, por tanto, ante el inicio de un prometedor campo de investigación en la EM en que se habrán de identificar poblaciones bacterianas asociadas a la EM, definir su papel en la patogenia y las posibilidades terapéuticas que esto nos ofrezca.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.