Varias especies de Mycoplasma y Ureaplasma diversum pueden causar enfermedades en el ganado bovino lechero, asociadas o no a manifestaciones clínicas. En nuestro país, ha sido detectada la presencia de solo tres especies de este grupo hasta el momento: Mycoplasma bovis, Mycoplasma californicum y Mycoplasma canadense. El objetivo del presente trabajo fue identificar otras especies de la familia Mycoplasmataceae. Se estudiaron treinta y cinco aislamientos compatibles con Mycoplasma spp. obtenidos a partir de diferentes muestras de bovinos, con o sin sintomatología clínica, provenientes de ocho rodeos ubicados en las provincias de Santa Fe, Córdoba, Buenos Aires y San Luis. Mediante el uso de reacciones en cadena de la polimerasa (PCR) específicas de especie se identificaron Mycoplasma bovigenitalum, Mycoplasma alkalescens, Mycoplasma bovirhinis y U. diversum, y mediante la amplificación y posterior secuenciación del espacio intergénico 16-23S ARNr se identificaron Mycoplasma arginini y M. californicum. La identificación de estas especies por primera vez en nuestro país es un hecho de relevancia, que representa un importante avance en el conocimiento para incluir estos patógenos en el diagnóstico diferencial de determinadas entidades clínico-patológicas de los bovinos de Argentina.
Several species of Mycoplasma and Ureaplasma diversum can cause diseases in dairy cattle, which can be associated or not with clinical manifestations. In our country, the presence of Mycoplasma bovis, Mycoplasma californicum and Mycoplasma canadense has been detected, being the only mycoplasma species identified so far. The objective of this study was to identify other species of the Mycoplasmataceae family. Thirty-five Mycoplasma spp.-like isolates obtained from different samples from cattle, with or without clinical symptoms, from eight herds located in the provinces of Santa Fe, Cordoba, Buenos Aires and San Luis were utilized in the present study. Through the use of species-specific polymerase chain reactions (PCR) Mycoplasma bovigenitalium, Mycoplasma alkalescens, Mycoplasma bovirhinis and U. diversum were identified and through amplification and further sequencing of the 16-23S rRNA intergenic spacer regions, Mycoplasma arginine and M. californicum were identified. The identification of these species represents an important advance in knowledge in order to include these pathogens in the differential diagnosis of certain clinical and pathological entities of cattle from Argentina.
La familia Mycoplasmataceae comprende dos géneros de significación como patógenos para el hombre y los animales domésticos, que son Mycoplasma y Ureaplasma17. Algunos miembros de esta familia pueden causar en el ganado bovino lechero enfermedades con manifestaciones clínicas como neumonías, conjuntivitis, poliartritis, otitis, mastitis, agalactia, aborto e infertilidad14.
Si bien Mycoplasma bovis es el agente responsable más importante de brotes de mastitis causados por Mycoplasma, otras especies como Mycoplasma arginini, Mycoplasma bovigenitalum, Mycoplasma bovirhinis, Mycoplasma alkalescens, Mycoplasma canadense y Mycoplasma californicum también han sido aisladas de leche5 y asociadas con algunas de las enfermedades mencionadas14. En nuestro país, se informó la presencia de M. bovis en un brote de mastitis en la provincia de Buenos Aires4 y posteriormente se detectó M. californicum y M. canadense a partir de muestras de leche de tanque de enfriado en dos tambos de esa misma provincia19; estas son, hasta el momento, las únicas especies de Mycoplasma informadas.
El otro miembro de la familia Mycoplasmataceae que puede afectar al ganado bovino lechero es Ureaplasma diversum. Inicialmente se la consideró una especie no patógena, dado que es un comensal de la mucosa genital y su aislamiento no es prueba suficiente de enfermedad; sin embargo, varios estudios han demostrado que puede causar fallas reproductivas8,11. En nuestro país no ha sido informada su presencia hasta el momento.
El conocimiento de la presencia y la circulación de estos patógenos en rodeos lecheros de nuestro país es importante para los profesionales veterinarios encargados de controlar su sanidad, y también para los laboratorios de diagnóstico, que deberían ofrecer alternativas para la detección certera de estos agentes, con el objeto de contribuir a optimizar el tratamiento y el control de las enfermedades que estos organismos producen.
El objetivo del presente estudio fue identificar a nivel de especie 35 aislamientos compatibles con Mycoplasma spp. obtenidos de muestras de bovinos con o sin sintomatología clínica.
Materiales y métodosEste trabajo fue realizado en el Laboratorio de Patología Animal de la Facultad de Agronomía y Veterinaria (UNRC, Río Cuarto, Córdoba, Argentina), de acuerdo con las normas internacionales del Consejo Internacional de Organizaciones de las Ciencias Médicas (CIOMS).
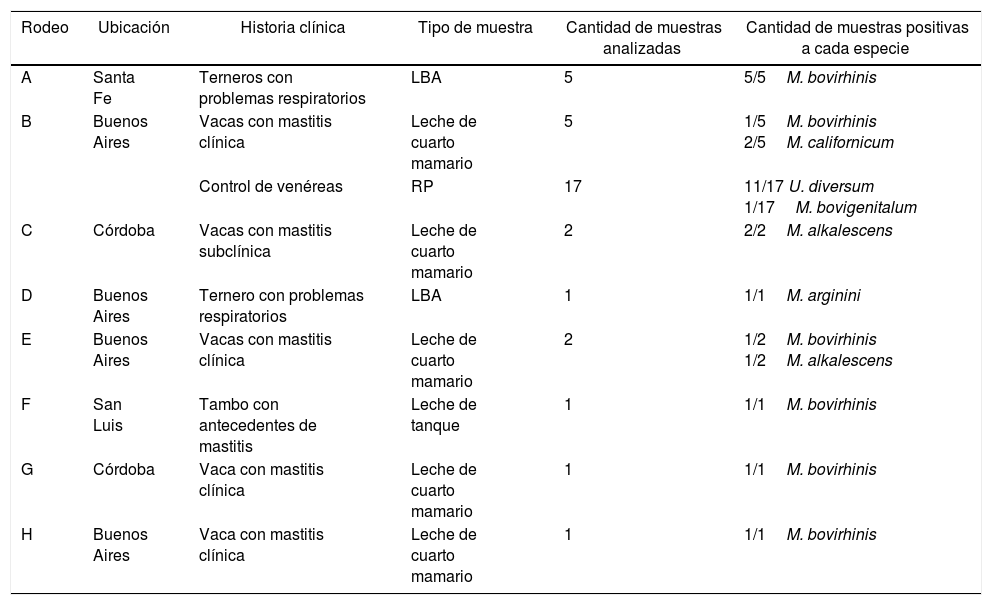
Origen de los aislamientosUn total de 35 aislamientos provenientes de vacas, terneros y toros de 8 rodeos lecheros ubicados en las provincias de Córdoba, San Luis, Santa Fe y Buenos Aires fueron utilizados en el presente estudio. Los aislamientos habían sido obtenidos a partir de los siguientes tipos de muestras: 5 muestras de lavado bronquio-alveolar (LBA) de terneros con problemas respiratorios del rodeo A; 5 muestras de leche de cuartos mamarios de vacas con mastitis clínica y 17 muestras de raspado prepucial (RP) de toros sanos del rodeo B; 2 muestras de leche de vacas con mastitis subclínica del rodeo C; una muestra de LBA de ternero con problemas respiratorios del rodeo D; 2 muestras de leche de cuartos mamarios de vacas con mastitis clínica del rodeo E; una muestra de leche de tanque del rodeo F; una muestra de leche de cuarto mamario de vaca con mastitis clínica del rodeo G, y una muestra de leche de cuarto mamario de vaca con mastitis clínica del rodeo H (tabla 1).
Cantidad de muestras positivas a cada especie de Mycoplasma y U. diversum según tipo de muestra, historia clínica de los animales y ubicación de los establecimientos
| Rodeo | Ubicación | Historia clínica | Tipo de muestra | Cantidad de muestras analizadas | Cantidad de muestras positivas a cada especie |
|---|---|---|---|---|---|
| A | Santa Fe | Terneros con problemas respiratorios | LBA | 5 | 5/5M. bovirhinis |
| B | Buenos Aires | Vacas con mastitis clínica | Leche de cuarto mamario | 5 | 1/5M. bovirhinis 2/5M. californicum |
| Control de venéreas | RP | 17 | 11/17 U. diversum 1/17M. bovigenitalum | ||
| C | Córdoba | Vacas con mastitis subclínica | Leche de cuarto mamario | 2 | 2/2M. alkalescens |
| D | Buenos Aires | Ternero con problemas respiratorios | LBA | 1 | 1/1M. arginini |
| E | Buenos Aires | Vacas con mastitis clínica | Leche de cuarto mamario | 2 | 1/2M. bovirhinis 1/2M. alkalescens |
| F | San Luis | Tambo con antecedentes de mastitis | Leche de tanque | 1 | 1/1M. bovirhinis |
| G | Córdoba | Vaca con mastitis clínica | Leche de cuarto mamario | 1 | 1/1M. bovirhinis |
| H | Buenos Aires | Vaca con mastitis clínica | Leche de cuarto mamario | 1 | 1/1M. bovirhinis |
Los aislamientos se obtuvieron cultivando las muestras en placas de medio de Hayflick modificado durante al menos 7 días a 36 (± 1) °C con 10% CO2. Las colonias sospechosas fueron identificadas por examen diario de las placas utilizando lupa estereoscópica. Finalmente, las colonias fueron repicadas en caldo de Hayflick modificado e incubadas por 48h bajo iguales condiciones.
Para la extracción del ADN, 1ml del caldo fue centrifugado a 8.000g durante 10min, el sedimento fue lavado 2 veces con solución salina bufferada, luego resuspendido en 150μl de agua bidestilada estéril, hervido en bloque seco a 100°C durante 10min y centrifugado a 12.000g por 2min. Se reservaron alícuotas de 100μl de sobrenadante para conservar a –20°C hasta el procesamiento.
En primera instancia, se comenzó con la identificación de M. bovigenitalum y M. bovirhinis, de modo que el ADN de los 35 aislamientos fue procesado utilizando cebadores y condiciones previamente descriptas9. Dado que 4 aislamientos del rodeo B, los 2 del rodeo C, el único del rodeo D y uno del rodeo E no fueron identificados como M. bovigenitalum ni como M. bovirhinis, el ADN de esos aislamientos fue amplificado en una segunda instancia bajo las condiciones previamente descriptas, pero utilizando los cebadores correspondientes a M. alkalescens9.
Debido a que 2 aislamientos obtenidos de leche de vacas con mastitis clínica del rodeo B y el único aislamiento del rodeo D tampoco fueron identificados como M. alkalescens, el ADN de esos aislamientos fue amplificado en una tercera instancia utilizando los cebadores y condiciones descriptas por Harasawa et al.7, para su posterior identificación mediante la secuenciación del espacio intergénico 16S-23S ARNr. Una vez amplificados, los productos de PCR fueron purificados (Puriprep-GP Kit, Inbio Highway, Tandil, Argentina), cuantificados y secuenciados (ABI 3130xl; Applied Biosystems, Foster City, California, EE. UU.) utilizando los cebadores descriptos previamente7. Las secuencias fueron visualizadas utilizando el programa BioEdit6 y luego alineadas contra la base de datos BLAST1. Para eliminar las regiones flanqueantes correspondientes a las secuencias parciales de los genes 16S ARNr y 23S ARNr, las secuencias fueron alineadas usando el programa ClustalW12.
Solamente a las muestras de ADN correspondientes a los 17 aislamientos obtenidos de RP de toros del rodeo B se les realizó la PCR anidada previamente descripta20 para la identificación de U. diversum.
ResultadosVeintiocho de los 35 aislamientos fueron positivos a alguna especie de Mycoplasma o a U. diversum. Mediante la utilización de las PCR específicas de especie, 10 aislamientos resultaron positivos para M. bovirhinis, en 6 rodeos (A, B, E, F, G y H) de las 4 provincias, por lo que resultó el Mycoplasma más frecuentemente encontrado. Tres aislamientos de los rodeos C y E, de Córdoba y Buenos Aires, respectivamente, resultaron positivos a M. alkalescens, mientras que 11 de los 17 aislamientos obtenidos de muestras de RP de toros del rodeo B (Buenos Aires) resultaron positivas a U. diversum (tabla 1).
Respecto de las muestras que fueron secuenciadas, la secuencia del espacio intergénico 16S-23S ARNr de la muestra de la granja D (Buenos Aires) mostró un 100% de similitud con la misma región de las siguientes cepas de M. arginini: HAZ145_1 (AP014657.1); ATCC 23243 (JN935883.1); EF-Hungary (HM179556.1) y M. arginini (AY737013.1; AF443604.1). Las secuencias del mismo segmento de las dos muestras del rodeo B mostraron 100% de similitud con la misma región de las siguientes cepas de M. californicum: ST6 (DQ847428.1) y ATCC 33461 (AY736031.1).
En un aislamiento de RP de toros del rodeo B se identificó U. diversum y M. bovigenitalum (tabla 1).
DiscusiónEn el presente estudio se identificaron 4 especies de Mycoplasma y U. diversum mediante técnicas moleculares a partir del cultivo de muestras provenientes de 8 rodeos lecheros de las provincias de Córdoba, Santa Fe, San Luis y Buenos Aires, y dado que la presencia de algunas de ellas no había sido informada antes, este dato genera un precedente que debería ser considerado en el momento del diagnóstico diferencial de patologías donde puedan estar involucrados estos agentes etiológicos.
M. bovirhinis fue la especie más encontrada en el presente estudio, ya que se identificó en 6 rodeos ubicados en las 4 provincias. Los aislamientos en los que se identificó M. bovirhinis fueron obtenidos de terneros con problemas respiratorios y de leche de vacas con mastitis clínica, en coincidencia con lo que informan estudios realizados en otras partes del mundo14. En este sentido, sería muy oportuno realizar estudios para estimar la prevalencia de este agente, a fin de determinar el impacto de las enfermedades que produce.
M. alkalescens fue detectado a partir de muestras de leche de vacas con mastitis clínica y subclínica. Cabe señalar que, según datos previos, es este un importante agente causal de mastitis5, pero su aislamiento también se ha asociado con casos graves de artritis2 y de neumonía en terneros10.
La detección de M. arginini se enmarca en un escenario controvertido, ya que a pesar de estar asociado con patologías de los ruminates14 se conoce poco de su patogenicidad y de su implicancia real en el desarrollo clínico-patológico de la enfermedad. Es preciso recalcar que se ha informado zoonosis por M. arginini en pacientes inmunocoprometidos21,22, por lo que este microorganismo debería ser especialmente considerado en el diagnóstico.
En el presente estudio se identificó M. bovigenitalum en una de las muestras de RP tomadas del rodeo B, de toros aparentemente sanos. El papel de esta especie en enfermedades reproductivas no es del todo claro, puesto que se la ha asociado a epididimitis, orquitis, uretritis y vesiculitis seminal en toros, y a vulvitis granulomatosa en terneras14, así como también con neumonía, artritis y mastitis. En este sentido, cabe destacar que aparentemente su distribución se limita a Europa, África del Norte y Estados Unidos de América14, por lo que este constituiría el primer informe de su presencia en Latinoamérica. Respecto de la identificación de M. californicum en las muestras de leche del rodeo B, este agente había sido detectado recientemente en un tambo de la misma provincia19.
En el presente trabajo se informa la presencia de M. arginini, M. bovigenitalum, M. bovirhinis, M. alkalescens y M. californicum. Dado que M. bovis, M. canadense y M. californicum ya habían sido identificados antes4,19, puede afirmarse ahora que en nuestro país están presentes las 7 principales especies de Mycoplasma causantes de mastitis en bovinos. Si bien en la práctica diaria y ante casos sospechosos de mastitis causadas por Mycoplasmas, no es necesario determinar la especie involucrada, puesto que con la sola identificación del género pueden tomarse las medidas pertinentes para el control de la enfermedad, la determinación de especie es importante para analizar los potenciales cuadros clínicos que diferentes especies de Mycoplasma pueden causar.
En los últimos años se han realizado estudios moleculares tendientes a asociar la presencia de determinadas especies o genotipos de Mycoplasma con la presentación de diferentes cuadros clínico-patológicos3,15,16,18; no obstante, existe escasa información en lo referente a la evolución de tales cuadros clínicos. En este sentido, la determinación de la especie de Mycoplasma constituiría un paso necesario para profundizar en ese tipo de estudios.
Finalmente, cabe señalar que U. diversum fue identificado en muestras que provenían de toros sanos. En este sentido, ya se ha señalado que la infección de los toros es frecuentemente asintomática13. Debido a que la presencia de U. diversum ha sido asociada con infertilidad en vacas, placentitis, aborto y terneros nacidos débiles8,11, es importante reconocer la presencia de este agente en el país, puesto que debería ser considerado en el diagnóstico diferencial de otras enfermedades venéreas, como infecciones por Campylobacter spp., Trichomonas y Herpesvirus bovino tipo 1. Lamentablemente, no se obtuvieron datos reproductivos del rodeo del cual provenían las muestras; sin embargo, revelar su presencia es importante. El aislamiento de U. diversum fue un hallazgo afortunado, ya que existen medios de cultivo específicos, adicionados con urea, que favorecen el crecimiento del microorganismo. Sin embargo, el aislamiento de este agente ha sido informado también en medios no específicos, con y sin urea14.
El hecho de haber identificado distintas especies de la familia Mycoplasmataceae, algunas de ellas asociadas a patologías del ganado lechero, representa un importante avance en el conocimiento, de gran relevancia para investigadores y profesionales veterinarios. Este hallazgo debería llevar a incluir a estas especies en el diagnóstico diferencial de determinados cuadros clínico-patológicos. Esto, sumado al hecho de que las especies identificadas podrían afectar también a los bovinos de carne, a otras especies de animales domésticos e incluso al hombre, debería conducir a la intensificación de esfuerzos tendientes a estudiar la circulación de estos patógenos en los rodeos del país y de su eventual impacto productivo que ocasionan para así mejorar las medidas de control de las enfermedades que estos producen.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datosLos autores declaran que en este artículo no aparecen datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores declaran que en este artículo no aparecen datos de pacientes.
FinanciaciónEste trabajo fue parcialmente financiado por los PPI 2016- 2018 18/A349 y 18/A354 SeCyT-UNRC. C. Sosa posee una beca de Ayudantía de Investigación, SeCyT-UNRC.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.