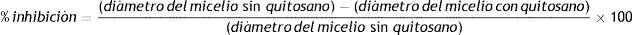Phytophtora capsici es un patógeno que incide sobre cultivos de la familia de las solanáceas causando pérdidas económicas en cultivos de pimientos, tomates, berenjenas y curcubitáceas. En este trabajo evaluamos el efecto del quitosano de bajo grado de polimerización (QBP) sobre el crecimiento de P. capsici y sobre la regulación génica de este fitopatógeno a nivel transcripcional. A una concentración de 0,4mg/l de QBP se obtuvo un 88% de inhibición en el crecimiento; concentraciones superiores a 1,6mg/l inhibieron el crecimiento en un 100%. Mediante ensayos de cambio en la movilidad electroforética de ácidos nucleicos se comprobó que el quitosano interactúa con el ADN y el ARN del hongo frente a concentraciones entre 2 y 4 mg/l de ADN y entre 0,5 y 3 mg/l de ARN. Además, se efectuó un análisis de despliegue diferencial de los productos de amplificación por RT-PCR de los ARN mensajeros de P. capsici obtenidos en presencia o ausencia de QBP; este mostró cambios en el perfil de expresión inducidos por el tratamiento con quitosano. El análisis bioinformático de las secuencias de los transcritos expresados diferencialmente sugiere que el QBP afectó la regulación génica de elementos involucrados en la síntesis de quitina y de proteínas de unión a hidratos de carbono.
Phytophthora blight of peppers, caused by oomycete Phytophthora capsici, currently causes economic losses in crops such as peppers, tomatoes, eggplant and cucurbits. In this work, we evaluated the effect of chitosan with low degree of polymerization (LDP) on growth and gene expression of P. capsici cultures. LDP chitosan inhibited 88% of P. capsici mycelial growth at concentrations up to 0,4 mg/l, whereas at concentrations higher than 1,6 mg/l it completely inhibit growth. Gel mobility shift assays demonstrated that chitosan interacts with DNA and RNA of the fungus at concentrations ranging from 2 to 4mg/l for DNA and 0,5 to 3mg/l for RNA. The differential display analysis of RT-PCR-amplification products of P. capsici messenger RNA revealed changes in gene expression profiles after the chitosan treatment. Bioinformatic analysis of sequences from selected differentially-expressed bands showed the gene regulation of elements involved in chitin synthesis and carbohydrate-binding proteins.
Las plantas, en su medio natural, pueden ser atacadas por microorganismos patógenos. El oomiceto Phytophthora capsici es un patógeno que causa una grave enfermedad en plantas de la familia de las solanáceas, entre ellas el chile habanero, la papa, el tomate y la calabaza15. La infección por este fitopatógeno se previene mediante la rotación de cultivos, el manejo adecuado del riego y la aplicación de agentes químicos sintéticos20. Sin embargo, el uso de pesticidas tiene efectos adversos sobre el medio ambiente.
El quitosano, un compuesto no tóxico para las plantas, puede inducir o incrementar la resistencia a los microorganismos patógenos cuando es aplicado a semillas22,37, frutos6 y hojas41; este efecto se basa en la inducción de enzimas relacionadas con la defensa4,24. En este sentido, se ha evidenciado que el tratamiento con quitosano incrementa la actividad de quitinasas y β-1,3 glucanasas en frutos de naranja y fresas4,9,45. Por otra parte, en tomate se observó que la aplicación de quitosano incrementó la actividad de polioxidasas, lo que dio como resultado un incremento en la resistencia a Botrytis cinerea5,26.
Se ha demostrado que el quitosano tiene un amplio espectro de actividad antimicrobiana1,17,25,29,31,38,46, al inhibir el crecimiento de microorganismos como Escherichia coli, Pseudomonas aeruginosa, Candida albicans, Candida glabrata, Fusarium oxysporum, Alternaria kikuchiana y Physalospora piricola, así como de otros agentes que pueden encontrarse en frutos almacenados en la etapa de poscosecha4,6,16,40. Tal es el caso de Alternaria alternata, Colletrotichum gloeosporioides, Rhizopus stolonifer y miembros del género Penicillium, entre otros.
Algunos compuestos derivados de la hidrólisis del quitosano (quitooligosacáridos) han demostrado tener actividad antifúngica y han evitado la penetración del patógeno a través de la membrana celular del hospedante43.
Derivados del quitosano fueron capaces de penetrar en la célula de P. capsici y de inhibir este microorganismo, por lo que se hipotetiza que el quitosano de bajo grado de polimerización (QBP) sería capaz de inhibir su crecimiento, por unirse a los ácidos nucleicos y modificar así la expresión génica.
El objetivo del presente trabajo fue evaluar la capacidad del QBP de inhibir el crecimiento de P. capsici, de unirse a su ADN y ARN y de incidir sobre la regulación génica de dicho fitopatógeno.
Materiales y métodosEnsayos de inhibición del crecimiento de P. capsici en placaLa cepa de P. capsici empleada en este estudio fue donada por el Dr. José Juan Zúñiga Aguilar (Departamento de Biología Molecular, Centro de Investigación Científica de Yucatán, México). Para su multiplicación, P. capsici fue sembrada en cajas de Petri que contenían PDA (glucosa-papa-agar) e incubada por 6días a 27°C. Se empleó QBP (PM 40.000- 60.000), con un grado de acetilación entre el 75 y el 85% (Sigma-Aldrich, St. Louis, MO, EE. UU.). Las soluciones de uso de QBP fueron preparadas según la metodología descrita por Benhamou y Thériault7, a partir de una solución madre de quitosano (2g/l) en HCl 0,25M pH 5,5 (ajustado con NaOH). Se adicionaron distintas alícuotas de solución de quitosano en el medio PDA estéril a 50°C, antes de su vertido en las cajas Petri, para obtener las concentraciones de ensayo (0,4-4mg/l). Las placas así preparadas se dejaron gelificar a temperatura ambiente y se incubaron a 27°C durante 24h para verificar su esterilidad. Luego se almacenaron en oscuridad hasta su uso.
Para evaluar si se producía inhibición del crecimiento, discos de micelio (6mm de diámetro) fueron transferidos a placas con PDA que contenían diferentes concentraciones de quitosano. Tras una incubación en oscuridad a 27°C durante 7 días, se midió el crecimiento radial del oomiceto. El porcentaje de inhibición del crecimiento fue calculado a partir de la siguiente fórmula:
El experimento fue realizado por triplicado. Los resultados fueron tratados mediante un análisis de varianza (ANOVA) y prueba de Tukey, con una p ≤ 0,05.
Extracción de ARN total de micelios y ensayo de despliegue diferencialMuestras de micelio desarrollado en placas con PDA con o sin quitosano fueron colectadas en tubos Eppendorf estériles y almacenadas a −80°C. La extracción de ARN se realizó utilizando el reactivo TRIZOL (Invitrogen, cat. N.° 15596026), siguiendo las recomendaciones del fabricante. Los extractos de ARN fueron cuantificados mediante espectrofotometría a 260nm.
Se sintetizó el ADN complementario a partir de 2μg de ARN total (de las muestras tratadas con 0,4mg de QBP/l) mediante retrotranscripción, usando el sistema de transcriptasa reversa ImProm-II (Promega, cat. N.° A3800). La reacción se realizó durante 60min a 42°C, haciendo uso del cebador oligo(dT)12-18 del sistema. Posteriormente, se inactivó la transcriptasa reversa por calentamiento durante 5min a 70°C. La reacción en cadena de la polimerasa (PCR) se llevó a cabo utilizando una alícuota de 1μl del producto de la retrotranscripción y las siguientes combinaciones de cebadores directo (T) y reverso (P) del kit comercial Clontech Delta Differential Display28:
- •
Combinación 1:
- ∘
T10: 5’-ATTAACCCTCACTAAAGCACCGTCC-3’.
- ∘
P6: 5’- CATTATGCTGAGTGATATCTTTTTTTTTCG-3’.
- •
Combinación 2:
- ∘
T2: 5’-CATTATGCTGAGTGATATCTTTTTTTTTAC-3’
- ∘
P7: 5’-ATTAACCCTCACTAAATGCTGTATG-3’
- •
Combinación 3:
- ∘
T6: 5’-CATTATGCTGAGTGATATCTTTTTTTTTCG-3’.
- ∘
P1: 5’-ATTAACCCTCACTAAATGCTGGGGA-3’.
- •
Combinación 4:
- ∘
T7: 5’-CATTATGCTGAGTGATATCTTTTTTTTTGA-3’.
- ∘
P3: 5’-ATTAACCCTCACTAAATGCTGGTGG-3’).
El programa de amplificación fue el siguiente:
- •
Etapa 1: 94°C por 5min, 40°C por 5min, 68°C por 5min, 1 ciclo.
- •
Etapa 2: 94°C por 2min, 40°C por 5min, 68°C por 5min, 2 ciclos.
- •
Etapa 3: 94°C por 1min, 60°C por 1min, 68°C por 2min, 25 ciclos.
Para la comparación de las muestras, 5μl de los productos de la PCR fueron fraccionados en geles de secuenciación desnaturalizantes al 5% de poliacrilamida/8M de urea en amortiguador TBE 0,5×70 V por 2,5h. Los geles fueron teñidos con nitrato de plata, de acuerdo con la metodología descrita por Bassam et al.3. Las bandas de ADNc diferenciales fueron escindidas del gel, purificadas y reamplificadas con el programa descrito anteriormente. Los productos de amplificación fueron clonados mediante el sistema de clonación pGEM-T-Easy (Promega, cat. N.° A1360). Los clones recombinantes fueron cultivados en caldo LB y tratados mediante extracción del plásmido recombinante haciendo uso del sistema PureYield™ Plasmid Miniprep (Promega, cat. N.° A1222). Los plásmidos recombinantes fueron secuenciados en el Instituto de Biotecnología-UNAM. Las secuencias obtenidas de cada banda fueron analizadas mediante el algoritmo BLAST2 para su comparación con los registros de secuencias en la base de datos del NCBI.
Ensayos de cambio de movilidad electroforética de ácidos nucleicosSe extrajeron los ácidos nucleicos (ADN y ARN) de micelios de P. capsici crecido en medio PDA sin quitosano. El ADN genómico fue extraído de acuerdo con la metodología descrita por Goodwin et al.11, mientras que el ARN total se extrajo haciendo uso de TRIZOL, como ya fue descrito. Los extractos de ARN fueron cuantificados mediante espectrofotometría a 260nm.
Los ensayos de cambio de movilidad electroforética se llevaron a cabo mezclando 0,5μg de QBP en solución 0,25M de HCl (pH 5,5) con 7μg de ARN total o 4,3μg de ADN genómico para un volumen total de 10μl. La mezcla de reacción fue incubada 30 min a 25°C. Posteriormente, se adicionaron 2μl de amortiguador de carga para ADN 6X (Thermo Scientific, cat. N.° R0611). Las muestras de ácidos nucleicos fueron cargadas en gel de agarosa al 1% en amortiguador TBE (ARN) o al 0,5% en amortiguador TAE (ADN) y tratadas mediante una diferencia de potencial de 100V durante 40min. Los fragmentos de ácidos nucleicos fueron revelados haciendo uso de un baño del fluorocromo intercalador SYBRSafe (Thermo Scientific, cat. N.° S33102) y visualizados en un fotodocumentador UV.
Resultados y discusiónEl quitosano puede inhibir el crecimiento de un importante número de microorganismos patógenos y esta inhibición depende de la concentración y del microorganismo8,21,35. En el presente estudio se evaluó el efecto del QBP sobre el crecimiento de P. capsici (tabla 1).
Inhibición del crecimiento micelial de Phytophtora capsici en placa en presencia de diferentes concentraciones quitosano de bajo grado de polimerización
| Concentración de quitosano (mg/l) | Inhibición micelial (%) de P. capsici |
|---|---|
| 0 | 0 a |
| 0,4 | 88 b |
| 0,8 | 92 b |
| 1,6 | 99 c |
| 2,4 | 100 c |
| 3,2 | 100 c |
| 4 | 100 c |
Letras distintas indican diferencias significativas entre tratamientos (p ≤ 0,05).
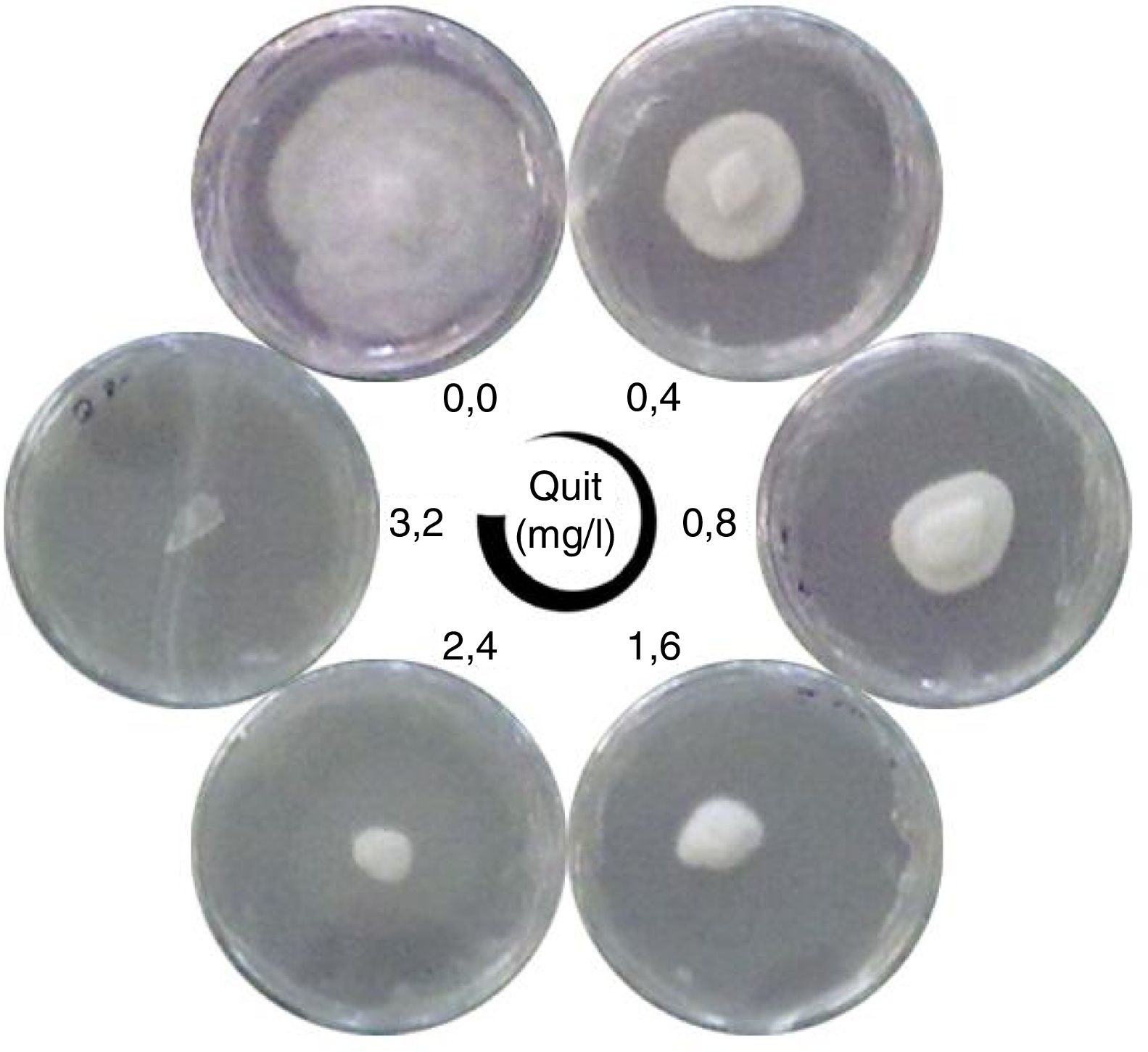
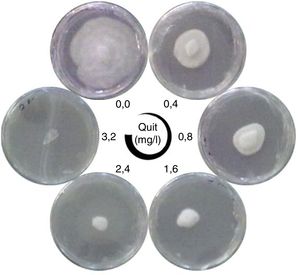
En la figura 1 se observa el efecto del QBP sobre el crecimiento del micelio de P. capsici. Mientras que concentraciones bajas de este compuesto (0,4mg/l) ocasionaron un 88% de inhibición del crecimiento del oomiceto, concentraciones superiores a 0,8mg/l inhibieron completamente su crecimiento. Las concentraciones de quitosano empleadas en este estudio son inferiores a las evaluadas por Mukul López et al.30, quienes analizaron el efecto del quitosano de alto grado de polimerización sobre P. capsici. Nuestros resultados corroboran la observación de que el grado de polimerización es un factor que influye sobre el efecto antimicrobiano de este compuesto18,42.
Efecto del quitosano de bajo grado de polimerización (QBP) sobre el crecimiento de Phytophtora capsici en placa. Discos inoculados con P. capsici fueron incubados durante 7 días en placas de medio PDA que contenían diferentes concentraciones de QBP (Quit). Se muestran placas representativas de cada tratamiento.
Varios estudios indican que el quitosano neutraliza las cargas electronegativas en la superficie celular y cambia la permeabilidad de la célula fúngica, lo que causa la fuga de los electrolitos intracelulares y de material proteico12. Asimismo, se ha demostrado que el quitosano provoca la liberación de aminoácidos y proteínas en células de R. stolonifer8. Además, se ha reportado que el tratamiento con quitosano causa cambios en la integridad de la membrana de esporas y liberación de proteínas intracelulares, y que este efecto es dependiente del microorganismo, el grado de polimerización y la concentración del quitosano10. Se ha determinado que existe una relación directa entre el contenido de ácidos grasos poliinsaturados de la membrana plasmática microbiana y el grado de permeabilización celular inducida por el tratamiento con quitosano34.
Otro modo de acción del quitosano ocurre intracelularmente, aunque son pocos los reportes que demuestran que el quitosano puede introducirse en la célula12,14,19. En estudios recientes se comprobó que el quitosano penetra en conidios de F. oxysporum y causa desorganización del citoplasma y pérdida del contenido intracelular33,34. También se observó que el oligoquitosano penetra en la célula y causa una disrupción en el sistema endomembranal de P. capsici43, además de asociarse a los ácidos nucleicos44.
Con el fin de evaluar si el quitosano se puede unir a los ácidos nucleicos, en este estudio se realizaron ensayos de movilidad electroforética en gel de agarosa del ADN genómico y del ARN total de P. capsici. Se observó la retención del ADN en los pocillos cuando las muestras se incubaron con 0,5mg/l o más de quitosano (fig. 1A, material suplementario), por lo que se sugiere que la asociación del quitosano a las moléculas de ADN impide su entrada en el gel. Un efecto similar fue observado en las muestras de ARN total (fig. 1B, material suplementario), en donde el incremento de la concentración de quitosano en la muestra disminuye la cantidad de ARN en el gel, hasta el punto de disminuir su entrada y provocar su acumulación en el pozo de carga (a partir de 1mg/l de quitosano).
Respecto al mecanismo de unión del quitosano a los ácidos nucleicos, debemos descartar la asociación electrostática debido al pH alcalino del amortiguador de corrida (TBE y TAE), que confiere cargas negativas tanto para los grupos funcionales del quitosano como para el esqueleto de fosfatos de los ácidos nucleicos.
Novoa-Carballal et al. demostraron que la disminución del grado de polimerización y acetilación de los polímeros de quitosano favorece la formación de dominios hidrofóbicos en la molécula32. Dichos dominios tienen efectos importantes en la reología de las soluciones acuosas del quitosano, así como en su interacción con otras macromoléculas. Específicamente, se ha demostrado que la interacción de moléculas que poseen dominios hidrofóbicos con el ADN es a través de su asociación con el surco menor27. Considerando lo anterior, es probable que el QBP interactúe a través de dichos dominios con los ácidos nucleicos de P. capsici, y que estos formen aglomerados voluminosos que son retenidos en el pozo de siembra en los geles de agarosa, lo que explicaría lo observado en los ensayos de cambio de movilidad electroforética.
Se ha propuesto que los derivados del quitosano se unen al ADN, lo cual ocasionaría la inhibición de la síntesis de ARNm y, por consiguiente, la síntesis de proteínas, dado que estos compuestos penetran hacia el interior de la célula hasta el núcleo de los microorganismos13,39.
Con el fin de explorar si el tratamiento con QBP modifica la abundancia de transcritos en P. capsici, se recurrió a la técnica del despliegue diferencial23. Las muestras de ARN fueron extraídas del tratamiento con 0,4mg/l de quitosano; los cambios ocasionados por dicho compuesto fueron evaluados con 4juegos de cebadores P-T. En la figura 2 se muestra un gel representativo del experimento, llevado a cabo con la combinación de cebadores P6-T10. Se puede observar que la mayoría de las bandas se mantienen sin cambios; sin embargo, hay algunos genes que son sobreexpresados o reprimidos (fig. 2). Se seleccionaron 13 bandas para su purificación y secuenciación, con base en el criterio del nivel de intensidad de la banda (sobreexpresión o represión). Los productos de amplificación correspondientes fueron recuperados de las bandas escindidas del gel, reamplificados por PCR y clonados en plásmidos vectores. Los plásmidos recombinantes fueron secuenciados y las secuencias obtenidas fueron analizadas mediante el algoritmo BLAST2. Este análisis mostró que el inserto del clon RST-4 (que se sobreexpresa en el tratamiento con quitosano) presentó una homología del 94% con la secuencia de glucosaminil (N-acetil) transferasa, que cataliza la síntesis de quitina (n.° de acceso en GenBank AK317729.1). El inserto del clon RIC1 (que se reprime en el tratamiento con quitosano) presentó homología del 98% con una secuencia de Phytophtora sojae, cuya función está relacionada con la unión a hidratos de carbono (n.° de acceso en GenBank AAQY00000000.2). En el clon RIC-8 (reprimido por el tratamiento), el inserto presentó una homología del 99% con la proteína hipotética PITG, cuya función no ha sido establecida (N°. de acceso en GenBank XM_002907859.1). Los clones restantes no presentaron homología con ningún gen anotado para hongos.
Despliegue diferencial de genes transcritos en micelios de Phytophtora capsici desarrollados en ausencia o en presencia de quitosano de bajo grado de polimerización (0,4mg/l). Se muestra el producto de la retrotranscripción amplificado con la combinación de cebadores 1 (T10-P6) del kit comercial Clontech Delta Differential Display. La abundancia de transcritos fue revelada por tinción con nitrato de plata. Las flechas ubicadas a la derecha señalan los transcritos sobreexpresados en el tratamiento con quitosano; las ubicadas a la izquierda, los reprimidos.
La regulación génica inducida por el quitosano en microorganismos ha sido demostrada al menos en Staphylococcus aureus SG511, en donde se observó efecto sobre genes involucrados en la síntesis de proteínas y de ARN, así como en el metabolismo de hidratos de carbono, aminoácidos, lípidos y coenzimas36.
Los resultados de este trabajo demuestran que el QBP a la concentración de 0,4mg/l ejerce un efecto de inhibición sobre el crecimiento de P. capsici de al menos del 88%. Los ensayos de cambio de movilidad electroforética demostraron que el QBP es capaz de unirse a moléculas como el ADN y el ARN. Por otra parte, los ensayos de despliegue diferencial demostraron que el quitosano induce cambios a nivel transcripcional, activando o reprimiendo genes. Entre las secuencias obtenidas se encontraron algunas con homología con aquellas que codifican transportadores y receptores de hidratos de carbono; se ha informado que estas moléculas son reguladas por quitosano13.
El análisis global de estos resultados demuestra que el QBP y a bajas concentraciones ejerce un efecto inhibitorio sobre P. capsici.
FinanciaciónSe agradece al financiamiento otorgado por parte del Programa para el Desarrollo Profesional Docente (PRODEP) al proyecto ITMER-PTC-003.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.