Las bacterias forman biopelículas de manera ubicua, y esta característica les otorga una flexibilidad que es resultado, en parte, de una matriz compleja construida según las exigencias de las condiciones ambientales. Aunque los estadios de la formación de las biopelículas bacterianas se conocen con detalle, para entender con profundidad la formación de las biopelículas es deseable un conocimiento mayor de los mecanismos de señalización. Las bacterias detectan cambios en la densidad de población por regulación del quórum y condiciones específicas, empleando señales como el di-GMPc y el óxido nítrico. La importancia del conocimiento de estas vías de señalización radica en que controlan una variedad de funciones, como la formación de biopelículas y la movilidad, y proporcionan a las bacterias beneficios en la colonización del hospedador, la defensa contra competidores y los cambios adversos del entorno. Por la trascendencia que revisten estos aspectos, revisamos aquí las redes de regulación y la conexión de la señalización entre quorum sensing, di-GMPc y óxido nítrico.
Bacterial biofilms are ubiquitous in nature, and their flexibility is derived in part from a complex extracellular matrix that can be made-to-order to cope with environmental demand. Although common developmental stages leading to biofilm formation have been described, an in-depth knowledge of genetic and signaling is required to understand biofilm formation. Bacteria detect changes in population density by quorum sensing and particular environmental conditions, using signals such as cyclic di-GMP or nitric oxide. The significance of understanding these signaling pathways lies in that they control a broad variety of functions such as biofilm formation, and motility, providing benefits to bacteria as regards host colonization, defense against competitors, and adaptation to changing environments. Due to the importance of these features, we here review the signaling network and regulatory connections among quorum sensing, c-di-GMP and nitric oxide involving biofilm formation.
La formación de biopelícula es una estrategia de vida para la mayoría de las bacterias, ya que esta les brinda estabilidad, desempeña funciones catalíticas, aumenta las posibilidades de transferencia de material genético y la resistencia a los antibióticos, participa en los procesos de comunicación celular y ofrece protección para sobrevivir a las condiciones adversas y variables del medio ambiente; aspectos que contribuyen a una colonización exitosa del hospedador. La biopelícula es un consorcio de microorganismos de una o varias especies, que se encuentra embebido dentro de una matriz polimérica. Esta matriz está constituida por sustancias poliméricas extracelulares (SPE), características de cada microorganismo, y se adhiere a una superficie biótica o abiótica.
La composición de la biopelícula es variable, aunque en general el componente mayoritario es el agua, que representa un 97 % del contenido total. La matriz está formada por exopolisacáridos (EPS), fimbrias, proteínas de adherencia, ADN exógeno (eDNA) y pilus, así como diversos productos procedentes de la lisis de las bacterias. La estructura de la matriz contiene canales que permiten el flujo de agua, nutrientes y oxígeno, incluso hasta las zonas más profundas de la biopelícula. En la biopelícula se encuentran diversos ambientes en los que la concentración de nutrientes, el pH y la disponibilidad de oxígeno son diferentes12,15,17,78.
El desarrollo de la biopelícula consta de seis etapas esenciales: 1) unión inicial a la superficie, en la que participan proteínas de superficie celular (adhesinas), flagelos, pili de tipo I y IV y “tight adherence pilus” (Tad); 2) formación de una monocapa de células: una vez que la bacteria se ha adherido a la superficie comienza a dividirse, y las células hijas se extienden alrededor del sitio de unión formando una microcolonia; 3) desplazamiento y multiplicación de las células para formar multicapas de microcolonias; 4) producción de la matriz extracelular por la síntesis de los EPS; 5) maduración de la biopelícula; y 6) dispersión de la biopelícula para la formación de una nueva37,45,75.
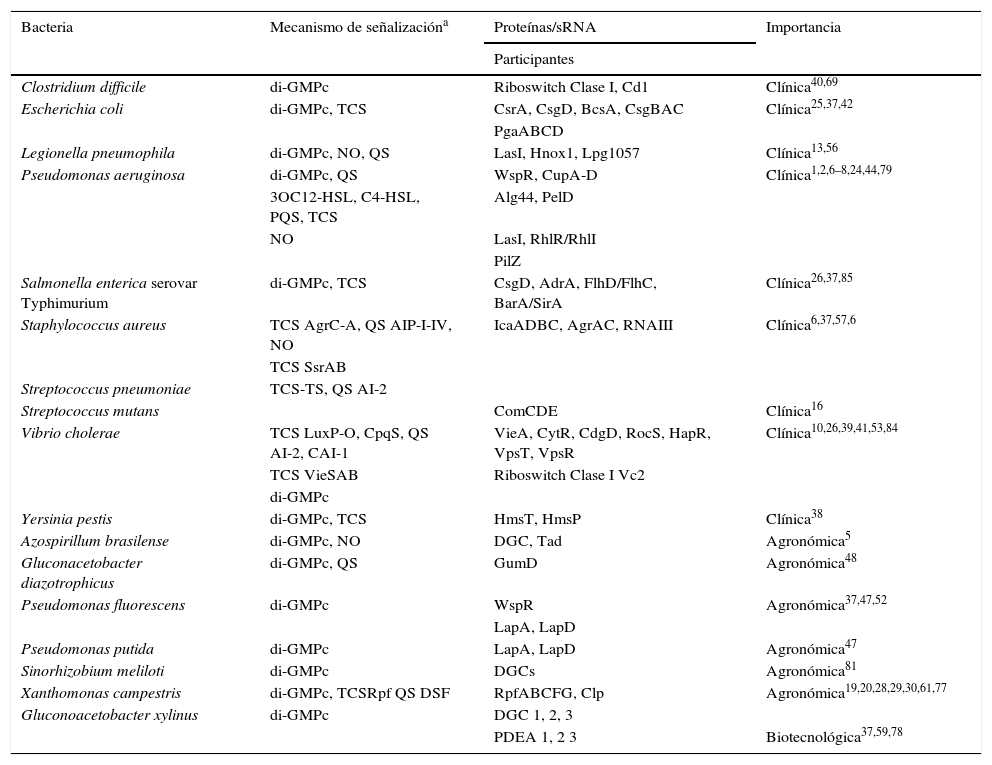
Entre las bacterias que forman biopelícula se incluyen patógenos gram positivos como Staphylococcus epidermidis, Staphylococcus aureus, varias especies de Streptococcus, y, entre los gram negativos, Escherichia coli enteropatógena y uropatógena, especies de Salmonella spp., Yersinia spp., Pseudomonas aeruginosa, Pseudomonas putida y Vibrio cholerae, entre otras. También son formadoras de biopelículas algunas bacterias que se asocian con plantas: Sinorhizobium meliloti, Gluconacetobacter diazotrophicus, Azospirillum brasilense y Pseudomonas fluorescens, las cuales se consideran no patógenas. Y dentro de las bacterias patógenas de plantas un ejemplo es Xhantomonas campestris pv. campestris. El estudio de la formación de las biopelículas es importante, ya que esta estructura representa la forma mayoritaria de vida de una bacteria, es ubicua y afecta directa o indirectamente al hombre (tabla1).
Algunas bacterias que forman biopelícula y sus mecanismos de señalización
| Bacteria | Mecanismo de señalizacióna | Proteínas/sRNA | Importancia |
|---|---|---|---|
| Participantes | |||
| Clostridium difficile | di-GMPc | Riboswitch Clase I, Cd1 | Clínica40,69 |
| Escherichia coli | di-GMPc, TCS | CsrA, CsgD, BcsA, CsgBAC | Clínica25,37,42 |
| PgaABCD | |||
| Legionella pneumophila | di-GMPc, NO, QS | LasI, Hnox1, Lpg1057 | Clínica13,56 |
| Pseudomonas aeruginosa | di-GMPc, QS | WspR, CupA-D | Clínica1,2,6–8,24,44,79 |
| 3OC12-HSL, C4-HSL, PQS, TCS | Alg44, PelD | ||
| NO | LasI, RhlR/RhlI | ||
| PilZ | |||
| Salmonella enterica serovar Typhimurium | di-GMPc, TCS | CsgD, AdrA, FlhD/FlhC, BarA/SirA | Clínica26,37,85 |
| Staphylococcus aureus | TCS AgrC-A, QS AIP-I-IV, NO | IcaADBC, AgrAC, RNAIII | Clínica6,37,57,6 |
| TCS SsrAB | |||
| Streptococcus pneumoniae | TCS-TS, QS AI-2 | ||
| Streptococcus mutans | ComCDE | Clínica16 | |
| Vibrio cholerae | TCS LuxP-O, CpqS, QS AI-2, CAI-1 | VieA, CytR, CdgD, RocS, HapR, VpsT, VpsR | Clínica10,26,39,41,53,84 |
| TCS VieSAB | Riboswitch Clase I Vc2 | ||
| di-GMPc | |||
| Yersinia pestis | di-GMPc, TCS | HmsT, HmsP | Clínica38 |
| Azospirillum brasilense | di-GMPc, NO | DGC, Tad | Agronómica5 |
| Gluconacetobacter diazotrophicus | di-GMPc, QS | GumD | Agronómica48 |
| Pseudomonas fluorescens | di-GMPc | WspR | Agronómica37,47,52 |
| LapA, LapD | |||
| Pseudomonas putida | di-GMPc | LapA, LapD | Agronómica47 |
| Sinorhizobium meliloti | di-GMPc | DGCs | Agronómica81 |
| Xanthomonas campestris | di-GMPc, TCSRpf QS DSF | RpfABCFG, Clp | Agronómica19,20,28,29,30,61,77 |
| Gluconoacetobacter xylinus | di-GMPc | DGC 1, 2, 3 | |
| PDEA 1, 2 3 | Biotecnológica37,59,78 |
Sistema de dos componentes (TCS) y “quorum sensing” (QS) se indican los que participan en la formación/dispersión de biopelículas. CsrA, regulador global de carbono; FlhD/FlhC, reguladores de los genes del flagelo; WspR (DGC); RcsCB; Agr accesory gene regulator, AIP auto-induced peptide; VieSAB sistema dos componentes VieA regulador de respuesta (PDE); IcaADBC intracelular adhesine, SSrAB staphylococcal respiratory response regulator; ComCDE competence genes/biopelícula; HmsT (DGC), HmsP (PDE).
Las bacterias que crecen en biopelícula difieren de su contraparte “planctónica” (que viven como células únicas) en términos morfológicos, fisiológicos y metabólicos. En el caso de especies bacterianas móviles, la célula planctónica móvil transita del estadio de célula móvil al de sedentaria. Estas diferencias fisiológicas de la célula son reflejo de cambios importantes en la expresión genética, como se ha descrito ampliamente en varios trabajos de genómica funcional, en los que se comparan las células cuando crecen en biopelícula con aquellas que lo hacen como planctónicas. La regulación de esta forma de vida depende del microorganismo y de la participación de redes de señalización celular complejas, en las que intervienen diversas proteínas que responden a diferentes señales ambientales. Se incluyen entre estas la norespermidina, el oxígeno, la luz, el óxido nítrico, la arginina, el pH, autoinductores, etcétera5,9,20,35–37,78.
Además de la regulación transcripcional del proceso de formación de biopelícula, numerosos indicios de la existencia de una regulación postranscripcional y postraduccional, algunos de estos serán discutidos en esta revisión. En Gluconacetobacter xylinus, V. cholerae, Yersinia pestis y P. fluorescens se han descrito proteínas implicadas en la síntesis del segundo mensajero, el ácido di-(3′-5′) guanosín-monofosfato (di-GMPc), que podrían afectar la formación de la biopelícula. Ello indica que esta molécula es un transmisor secundario de señal común al proceso de regulación de la producción de EPS en bacterias37–39,72,83.
La formación de las biopelículas se produce en respuesta a las condiciones ambientales. Es por ello que existen sistemas de fosfotransferencia de dos componentes conocidos como TCS (acrónimo de two-component systems), que transmiten la señal ambiental al interior de la bacteria para adecuar la expresión de genes a las situaciones externas fluctuantes, además del mecanismo que detecta la densidad celular denominado quorum sensing (QS), el cual regula la expresión de genes mediante la secreción y detección de señales químicas conocidas como autoinductores34,51,53.
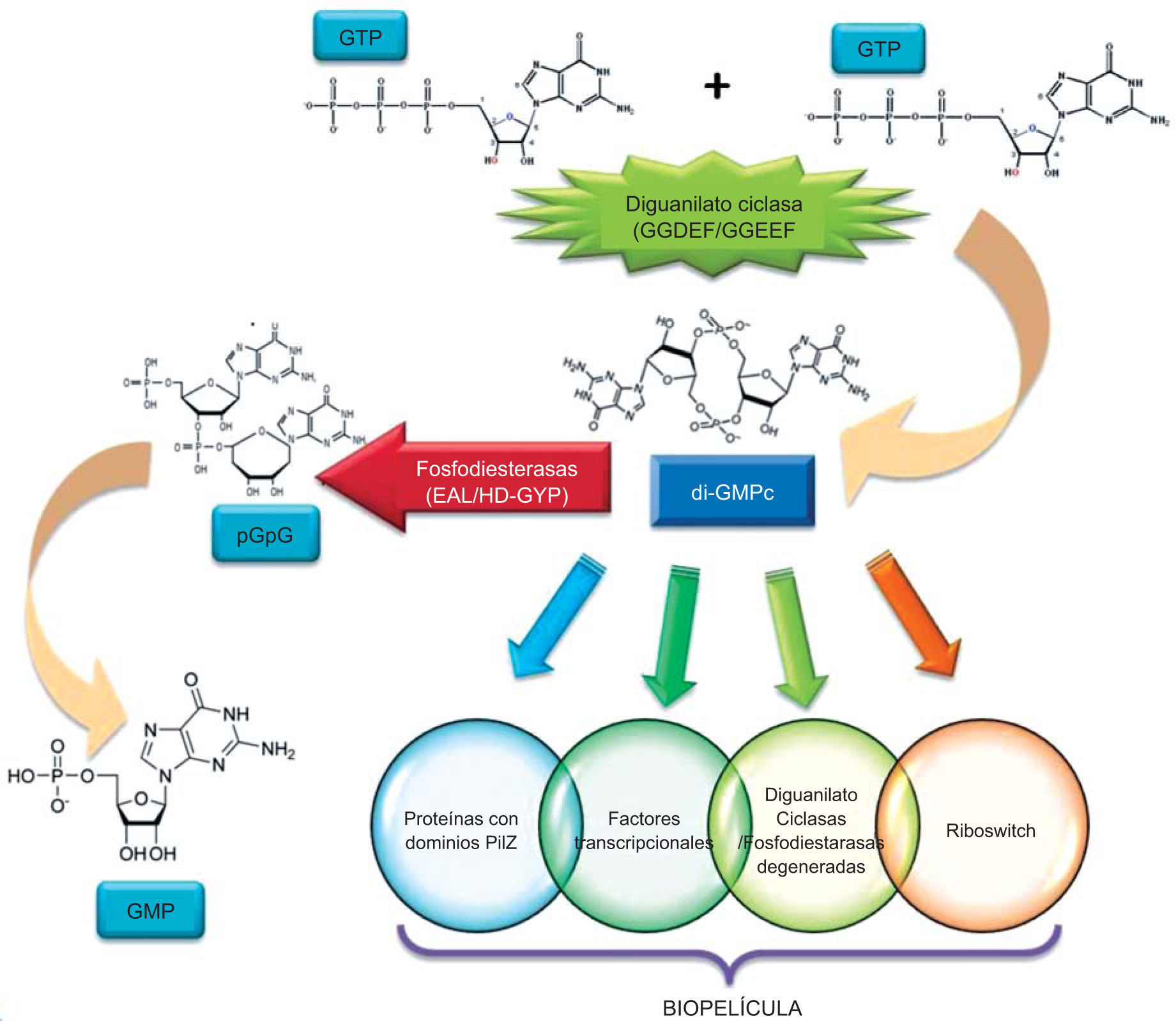
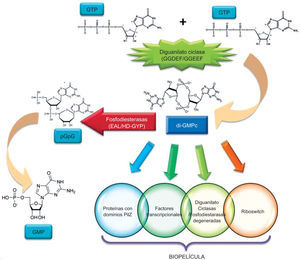
Papel del segundo mensajero ácido di-(3′-5′) guanosín-monofosfato cíclico (di-GMPc), en la regulación de la formación de biopelículaEn la década de los ochenta, se describió el sistema novedoso de señalización celular en bacterias dirigido por el nucleótido cíclico di-GMPc, descubierto como un activador alostérico de la enzima celulosa sintasa en G. xylinus59. Posteriormente, se ha descrito como un mecanismo de regulación generalizado en el proceso de formación de biopelícula, tanto en bacterias gram positivas como gram negativas (fig. 1).
Esquema de vías de señalización en las que interviene el di-GMPc en la formación de biopelícula. Enzimas que lo metabolizan: DGC, y PDE. Se indican los dominios característicos de cada una de ellas. Estructura del segundo mensajero di-GMPc y de los nucleótidos pGpG y GMP derivados de la actividad de PDE. Receptores (blancos) que transfieren la señal para formar la biopelícula.
El di-GMPc es sintetizado a partir de dos moléculas de GTP (guanosín trifosfato); esta reacción es catalizada por un grupo de enzimas denominadas diguanilato ciclasas (DGC). Estas enzimas contienen el dominio GGDEF/GGEEF, que alberga el sitio catalítico de la enzima. Este dominio consta de aproximadamente 170 residuos de aminoácidos y son nombrados así debido al patrón de secuencia de aminoácidos muy conservado23,37,83 (G[Gly]-G[Gly]-D[Asp]-E[Glu]-F[Phe]).
La degradación (hidrólisis) de di-GMPc a GMP (guanosín-monofosfato) es catalizada por fosfodiesterasas (PDE), que contienen los dominios HD-GYP (H[His]-D[Asp]-G[Gly]-Y[Tyr]-P[Pro]) o EAL(E[Glu]-A[Ala]-L[Leu]). Este último consta de aproximadamente 250 residuos de aminoácidos y fue descrito originalmente en la proteína BvgR de Bordetella pertussis, que actúa como reguladora de algunos genes denominados vrg49 (virulence repressed genes). Además, el dominio EAL se descubrió independientemente como un dominio adyacente al dominio GGDEF responsable de la actividad de fosfodiesterasa (PDE) del di-GMPc en la proteína A1 (PDEA1) de A. xylinus59.
Los dominios GGDEF, EAL y HD-GYP están ampliamente representados en proteínas de algunas bacterias, mientras que están ausentes en otras. Se han identificado más de 4200 dominios GGDEF, 2500 dominios EAL y 200 dominios HD-GYP en diferentes genomas secuenciados de bacterias23. Estas proteínas que sintetizan y degradan al diGMPc son consideradas proteínas modulares de señalización debido a que en su estructura están presentes dominios que detectan señales tanto externas como internas, entre ellas luz, gases (NO, O2), FAD, etc., o bien a través de la interacción proteína-proteína37. Participan como reguladores de respuesta en cascadas complejas de regulación, en las que intervienen sistemas de dos componentes y en donde algunas de las proteínas que detectan el estímulo a su vez son reguladas por moléculas tipo autoinductores31. Las DGC y PDE pueden ser activadas por fosforilación, o por su traducción. Algunas DGC contienen un sitio de inhibición alostérica denominado I, el que regula su actividad de manera negativa31,37.
El di-GMPc interviene en la regulación de la formación de la biopelícula por su participación en diferentes procesos incluyendo la expresión de los genes que codifican componentes de la matriz extracelular de la biopelícula, como la síntesis de los EPS, adhesinas tipo pilus y proteínas ancladas a la membrana. Existen también otras etapas que participan en el desarrollo de la biopelícula: muerte celular, dispersión y movilidad. Esta regulación puede ocurrir de modo transcripcional, postranscripcional o postraduccional, con la participación de proteínas con sitios de unión al di-GMPc, como por ejemplo, reguladores transcripcionales, ribonucleasas, glicosiltransferasas, pequeños ARN (sRNA) llamados “riboswitches”40, proteínas con dominios PilZ62,82, proteína estructural del pilus tipo IV2, e incluso las propias DGC y PDE31,73, desprovistas de actividad catalítica por la presencia del dominio degenerado; algunos ejemplos se anotan en la figura 1.
Recientemente se describió un regulador de la transcripción en Klebsiella pneumoniae denominado MrkH, en cuya estructura se localiza el dominio PilZ; este une al di-GMPc y genera MrkH-di-GMPc activo, e inicia la transcripción de los genes que codifican la formación y producción de la fimbria tipo 3 misma, que interviene en la estructura de la matriz de la biopelícula82.
En Salmonella enterica Typhimurium, la concentración intracelular del di-GMPc incrementa la expresión de la fimbria denominada “curli”, un componente de la matriz de la biopelícula; esta regulación es efectuada por el regulador transcripcional central CsgD42,85, que regula de manera positiva la transcripción de los genes que codifican la formación y producción de la la fimbria (csgBAC), adrA, que codifica la DGC AdrA (agfD regulated gene) y su propia trascripción26,85.
El estudio en P. aeruginosa de cinco grupos de genes denominados cup (cupA-E) (chaperon/usher pathway), que codifican la formación y producción de las fimbrias CupA-E, mostró que estas presentan propiedades de adhesión y contribuyen a la formación de la matriz de la biopelícula producida por la bacteria. Los genes de las fimbrias CupA-D son regulados en la etapa transcripcional por el sistema de dos componentes RcsCB vía di-GMPc24,50.
También están implicados en la formación de la biopelícula mecanismos de regulación postranscripcional, en los que participan los sRNA o riboswitches. Estas moléculas de ARN no codificante adoptan una estructura secundaria que permite su unión a otras moléculas (entre ellas, al di-GMPc) y actúan en cis de manera postranscripcional40,74. Dependiendo de su estructura corresponden a dos clases, y frecuentemente son codificados en la región UTR (región 5′ no traducida del ARNm, upstream untranslated region) de los genes que codifican DGC y PDE, o genes que son controlados por el di-GMPc; la unión di-GMPc-riboswitches presenta alta afinidad69,74. En Clostridium difficile se conocen varios, y algunos de ellos participan en la regulación negativa de la movilidad (represión de genes estructurales de la formación del flagelo) y positiva de la producción de biopelícula69.
La regulación postraduccional tipo alostérica de enzimas que realizan la síntesis de los EPS es un mecanismo común observado en varias especies bacterianas, por ejemplo, especies de Rhizobium, E. coli, S. enterica y P. aeruginosa, en las cuales el di-GMPc regula la síntesis de los EPS celulosa, sintetizada por BcsA; P. aeruginosa exopolisacárido locus Pel, sintetizado por la sintasa PelD, y alginato, sintetizado por la alginato sintasa26,37,42.
Otro mecanismo de participación del di-GMPc en la construcción de la matriz de la biopelícula es el observado en términos de interacción proteína-proteína, descrito en E. coli y en especies de Pseudomonas.
La producción y secreción del EPS poli N-acetil glucosamina (PGA) asociado a la formación de biopelícula en E. coli K-12 está mediada por una maquinaria celular que incluye a la enzima β-glicosiltransferasa (PgaC) y a una pequeña proteína integral de la membrana citoplasmática llamada PgaD, así como a la lipoproteína PgaB y a la porina PgaA, ambas ubicadas en la membrana externa. Se demostró que la unión del di-GMPc a las proteínas PgaC y PgaD de la membrana citoplasmática favorece su interacción, a la vez que se estimula la activación de PgaC; esta interacción también propicia la creación de un poro en la membrana interna y permite que la bacteria secrete el PGA al espacio periplásmico37.
P. putida y P. fluorescens, bacterias colonizadoras de plantas, forman biopelícula en asociación con la planta hospedadora. La adhesina LapA, producida por ambas bacterias, es esencial para la construcción de la matriz, ya que estabiliza e impide la dispersión de la biopelícula52. La localización de la adhesina en la membrana externa es modulada por un mecanismo novedoso, en este participa la proteasa LapG, que degrada a la adhesina LapA. A alta concentración de fosfatos, el di-GMPc se acumula en la célula; la proteína LapD contiene los dominios GGDEF-EAL carentes de actividad catalítica; sin embargo, une al di-GMPc y secuestra a LapG, de esta forma LapA se mantiene en la superficie de la membrana externa, apta para contribuir a la formación de la matriz. Si la concentración de di-GMPc disminuye, el complejo LapD-di-GMPc-LapG se disocia, se activa la proteasa que degrada la adhesina LapA y su ausencia hace que la estructura de la biopelícula sea laxa y se disperse52.
El genoma de S. meliloti Rm1021 contiene un repertorio importante de genes que codifican DGC y PDE. Se comprobó que mutaciones en los genes que codifican enzimas DGC, se asociaron con una baja producción de EPS y, por consiguiente, con la alteración en la formación de biopelícula; esto condujo a una menor colonización de la raíz de la planta hospedadora81.
Conexión entre quorum sensing, di-GMPc y biopelículaEl sistema de señalización QS es un tipo de comunicación que presentan las bacterias para detectar la densidad de su población; este fue descrito por primera vez en Vibrio fischeri, bacteria productora de bioluminiscencia en alta densidad celular19,21,34,68.
Esta comunicación ocurre debido a la producción y secreción de moléculas denominadas “autoinductores”, compuestos que funcionan como señales específicas. Cuando la concentración de la molécula señal alcanza un determinado valor, se dispara el mecanismo de detección de quórum y la célula bacteriana activa genes que expresan funciones de manera cooperativa, y se favorece la adaptación de la bacteria al entorno20,68,70,71.
Dependiendo del entorno específico y de su estilo de vida, las bacterias controlan de manera definida los fenotipos asociados a la densidad de población. Por ejemplo, los genes que participan en la formación de biopelícula en V. cholerae se expresan a baja densidad de población27,60, mientras que lo contrario sucede de manera parcial en P. aeruginosa, ya que los genes que intervienen en la formación de la biopelícula en esta bacteria se expresan en condiciones de alta densidad de población18. En P. aeruginosa, el autoinductor 3-oxo-C12 HSL (acrónimo de N- acil homoserino lactona), sintetizado por LasI, es necesario para la formación de biopelícula, ya que las mutantes lasI forman biopelícula laxa e indiferenciada18; en tanto que el sistema RhlI/RhlR44 (responsable de la síntesis de N-butanoil-homoserino lactona y del receptor, respectivamente) colabora durante el proceso de maduración63. Además, participa en la constitución de la matriz al promover la secreción del eDNA vía HSL (3-oxo-C12 HSL y C4-HSL) y por la quinolona PQS1,45 (acrónimo de Pseudomonas quinolone signal). No obstante, de manera similar a lo que sucede en V. cholerae, existe un punto de regulación negativa por QS vía HSL al activar la transcripción de una fosfatasa TpbA, que inactiva la enzima DGC y, consecuentemente, disminuye la concentración celular del di-GMPc, así como la producción de EPS y de biopelícula79.
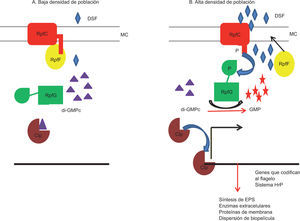
Se han descrito en la literatura varios modelos en los que se integra la producción de autoinductores y la generación intracelular del di-GMPc, uno de ellos es el estudiado en X. campestris pv. campestris, bacteria patógena de varias plantas. Xanthomonas campestris produce el autoinductor denominado factor difusible de señalización o DSF (acrónimo de diffusible signal factor), cuya estructura corresponde al ácido cis-11-metil-2-dodecenoico28,80. El QS en condiciones de alta densidad de población regula de manera positiva factores de virulencia de la bacteria: producción del EPS xantano, secreción de enzimas hidrolíticas y formación de la biopelícula30.
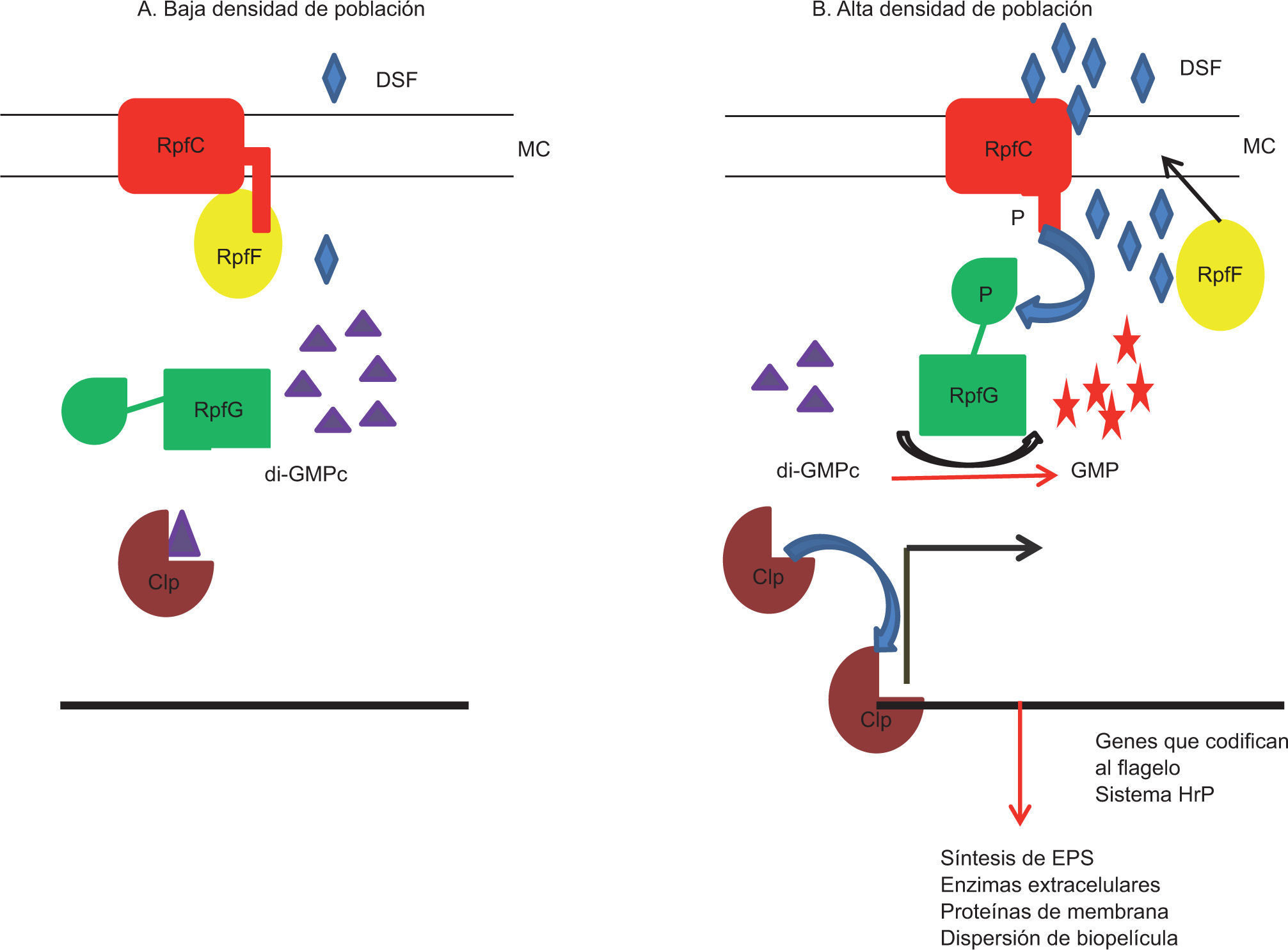
Los genes implicados en la producción de DSF y la señalización son designados rpfA-G por su participación en la regulación de los factores de patogenicidad28,29. La cascada de señalización es compleja: en ella intervienen las proteínas RpfB y RpfF, las cuales participan en la síntesis del autoinductor DSF; el sistema de dos componentes constituido por la quinasa histidínica RpfC y su regulador de respuesta (RR), la proteína RpfG. Esta proteína contiene dos dominios: HD-GYP (característico de las enzimas PDE que hidrolizan al di-GMPc) y REC. El DSF se une a la quinasa histidínica RpfC, esta a su vez fosforila al regulador de respuesta RpfG (en el domino REC), unión que favorece la actividad de PDE29,30. El modelo descrito reveló que a baja densidad de la población (concentración de DSF baja), la RpfC está unida a RpfF (DSF sintasa). Bajo estas condiciones el regulador RpfG se encuentra inactivo, y como resultado, el nivel de di-GMPc celular es elevado. Cuando esto ocurre, el di-GMPc se encuentra unido al factor de transcripción Clp, lo cual impide su unión al ADN blanco. A medida que aumenta la población, asimismo, se incrementa la concentración de DSF; esto propicia la liberación de la sintasa RpfF, cuya actividad acrecienta la concentración de DSF, por tanto, la quinasa RpfC participa también en la regulación de la molécula señal29. Por otra parte, el cambio conformacional que sufre RpfC luego de su unión al DSF favorece su activación y se fosforila; así, RpfC~P transfiere el grupo fosfato a la fosfodiesterasa RpfG para que convierta al di-GMPc en GMP. La disminución intracelular del di-GMPc tiene como consecuencia la activación del factor de transcripción Clp7, esto conduce a la expresión de los genes responsables de virulencia y producción de EPS, según se muestra en la figura 2. Por análisis de transcriptoma se demostró que el autoinductor DSF regula varios genes implicados en la producción del EPS, la biosíntesis del flagelo, el transporte de Fe y la resistencia a antibióticos. En concordancia, la mutación de los genes rpfC y rpfF reduce la virulencia de X. campestris en las plantas hospedadoras30.
Regulación en X. campestris pv. campestris de la concentración celular de di-GMPc por la molécula señal DSF producida por QS, (A) baja densidad celular, (B) alta densidad celular. A. La quinasa RpfC localizada en la membrana secuestra a la sintasa RpfF, esta unión disminuye su actividad catalítica. En esta condición, RpfG (PDE, defosforilada) es inactiva, por lo tanto, el nivel intracelular del di-GMPc es alto (triángulos violetas) y el di-GMPc une a Clp. B. Una vez que la concentración de la molécula señal DSF (rombos azules) aumenta, RpfC detecta a DSF y se autofosforila. RpfC~P activa a RpfG por fosforilación, esto conduce a la disminución del segundo mensajero di-GMPc, transformándolo en GMP (estrellas rojas). Como consecuencia se activa el regulador transcripcional Clp. RpfG también se une a otras proteínas con dominios GGDEF (DGC). Permiso: American Society of Microbiology, Srivastava y Waters70.
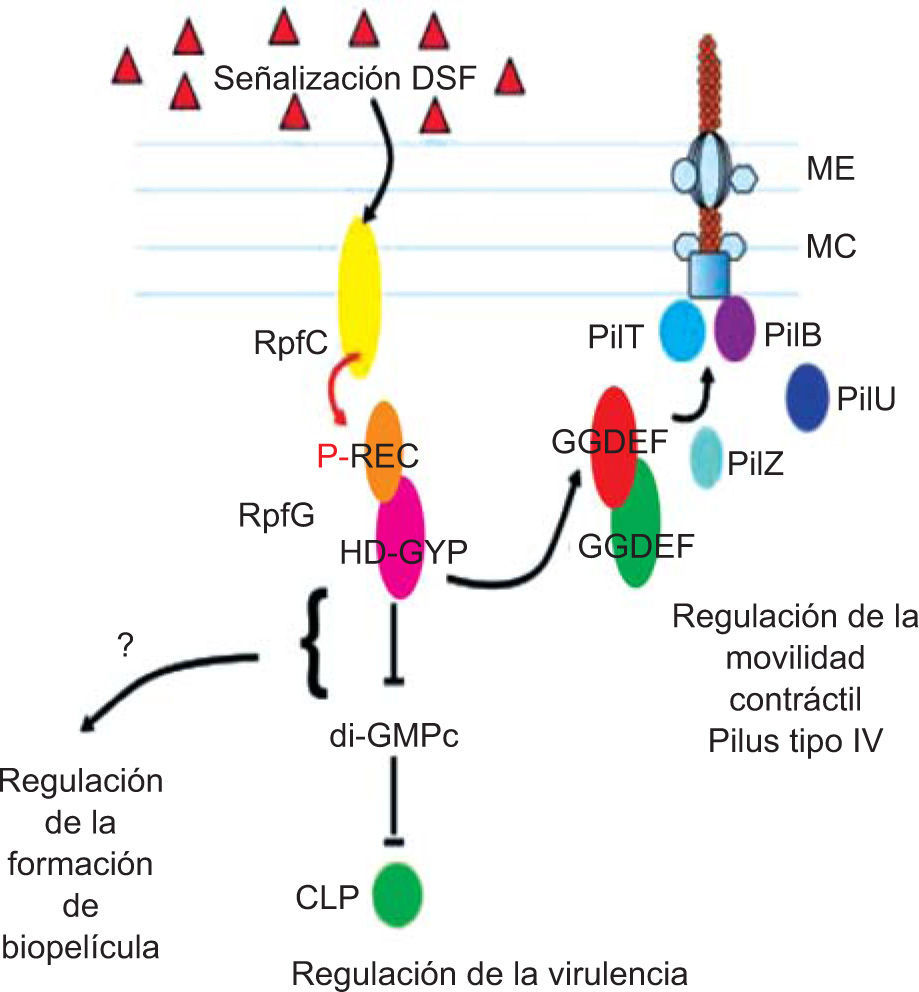
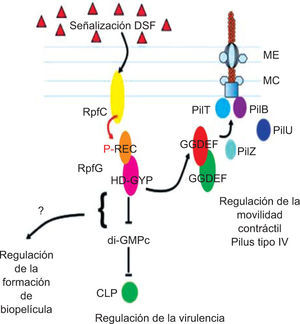
Por otra parte, RpfG (PDE) regula la movilidad contráctil mediada por el pilus tipo IV de manera independiente de su actividad enzimática a través de la interacción proteína-proteína. La evidencia inicial del papel de RpfG en la interacción con varias DGC se describió en el patógeno Xanthomonas axonopodis pv. citri4. Los estudios realizados en X. campestris pv. campestris tienden también a mostrar la interacción de RpfG con DGC, unión que ocurre durante la señalización por DSF. Esto correlaciona con la relocalización de RpfG en los polos de la célula, donde tanto la quinasa RpfC como DSF se ubican62. El complejo formado por las proteínas RpfG-GGDEF recluta a la proteína XC_224, la cual contiene un dominio PilZ (que une di-GMPc). Este complejo tripartito se une a PilU y PilT (proteínas que intervienen en la retracción del pilus) para modular su contracción y, por tanto, la movilidad contráctil62 (fig. 3). En conclusión, en la red de regulación interviene la señalización por QS, TCS (Rpf/DSF) y di-GMPc para promover la activación de genes cuyos productos actuarán en la síntesis de factores de virulencia y, como resultado final, contribuirán en la patogenicidad de la bacteria62.
La señalización de DSF regula la movilidad generada por el pilus de X. campestris pv. campestris. Esto ocurre por medio de la formación de un complejo proteico tripartito en el que intervienen RpfG-DGC XC_0249, XC_0420 + proteína XC_2249, que contiene el dominio PilZ. En la interacción intervienen GYP del motivo HD-GYP de RpfG (PDE) y el motivo DxD del dominio GGDEF de las DGC. Permiso: Microbiology, Ryan62.
Otra vía de regulación y señalización referida recientemente es aquella en la que intervienen XC_0250, que codifica una guanilato ciclasa (síntesis de GMPc), y XC_0249, que codifica una proteína que contiene el dominio de GGDEF (DGC) XC_0250; esta es responsable de la síntesis del di-GMPc al activar a XC_0250. La mutación de ambos genes conduce a la disminución de la virulencia y a la formación de biopelícula in vitro. Este nuevo efector XC_0250 representa una conexión directa entre el GMPc y el di-GMPc3.
Un sistema parecido al de X. campestris se describió también en Burkholderia cenocepacia, que produce el autoinductor ácido cis-2-dodecenoico (BDSF), cuya estructura es similar a la molécula señal DSF producida por X. campestris, y una HSL (N-octanoil homoserino lactona, C8-HSL). La mutación generada en los genes rpfFBc y rpfGBc, los cuales codifican la RpfFBc (sintasa, ortólogo de RpfF) y el receptor RpfR (PDE, ortólogo de RpfG), controlan los fenotipos de movilidad de superficie tipo “swarming”, la formación de biopelícula y la producción de proteasas. RpfR es una proteína de señalización modular que contiene los dominios PAS-GGDEF-EAL19. La mutante BDSF (rpfFBc minus) es rescatada por RpfG (de forma heteróloga) o su dominio EAL, que funciona como PDE; cuando está unida al autoinductor BDSF se produce la disminución de los niveles celulares del segundo mensajero di-GMPc y la producción de HSL. La expresión in trans del dominio GGDEF de RpfR en la mutante RpfFBc minus disminuye la movilidad tipo swarming y la actividad de proteasa, mientras que cuando el dominio EAL de RpfR se incorpora a la mutante in trans, aumenta: la producción de la molécula señal HSL, la movilidad y la actividad de proteasa, lo que sugiere que ambos dominios son activos. Los datos obtenidos apuntan a que el sistema BDSF controla la producción de la señal HSL y tiene una influencia en la fisiología de la bacteria vía modulación de la concentración intracelular del di-GMPc19.
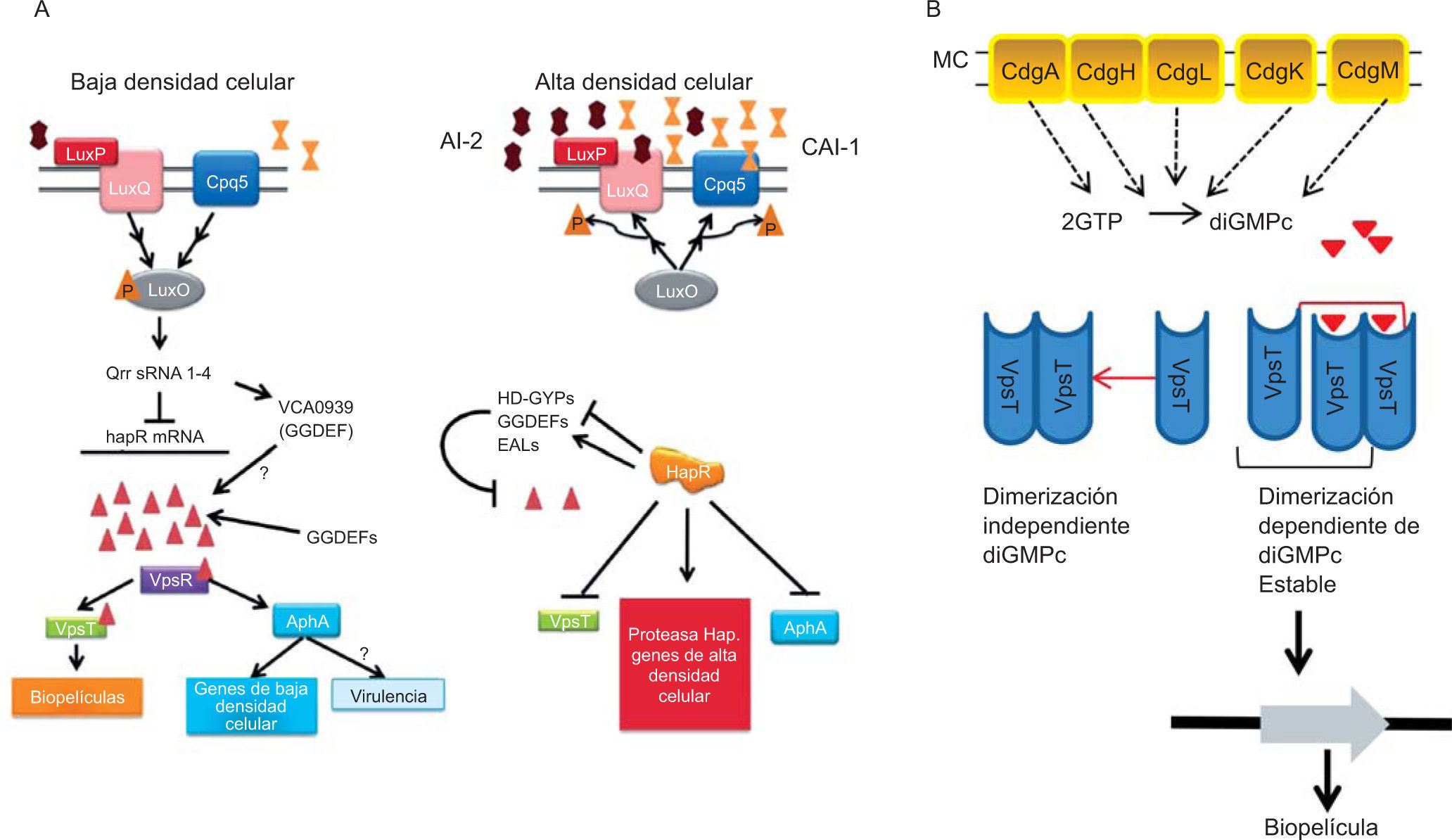
La formación de biopelícula en V. cholerae es un factor clave para que este microorganismo pueda crecer en vida libre en ambientes acuáticos y colonizar a su hospedador. La biopelícula le permite a la bacteria la colonización del intestino delgado. Los genes que participan en la síntesis del EPS son regulados por dos factores de transcripción: VpsR y VpsT; en la cascada de señalización intervienen cinco DGC: CdgA, CdgH, CdgL, CdgK, y CdgM, cuyas actividades aumentan la concentración celular del di-GMPc. La unión del di-GMPc a VpsT propicia la activación de VpsT67 (fig. 4B). VpsT, a su vez, es regulado por QS, autoinductores y TCSs10.
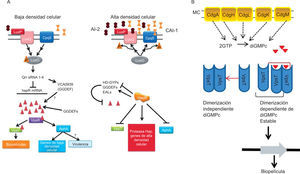
Participación de las moléculas señal producidas por QS y del di-GMPc en la patogenia de V. cholerae. A. En baja densidad de población, la concentración de AI-2 y CA-1 es baja. Las quinasas se encuentran activas y transfieren su grupo fosfato al regulador Lux O. LuxO~P activa la transcripción de los qrr sRNA. Se impide la traducción de HapR, se activa la traducción de la proteína Vac0939 (GGDEF) y se incrementa la concentración intracelular del di-GMPc. La actividad enzimática de cinco DGC aumenta la concentración intracelular del diGMPc, y se activan los reguladores transcripcionales VpsT y VpsR. Si la densidad de población se incrementa, la concentración de AI-2 y CA-1 aumenta y la situación se revierte. En esta condición, la traducción del regulador maestro HapR promueve la activación de PDE y disminuye la concentración celular del di-GMPc. B. Al aumentar la concentración intracelular de diGMPc, se une a VpsT, regulador de los genes de la producción de los EPS, y se promueve la producción de biopelícula. Triángulos rojos: di-GMPc. Permiso: American Society of Microbiology, Srivastava y Waters70.
El sistema de QS es complejo, ya que posee dos circuitos sensores paralelos que responden a dos moléculas autoinductoras: el diéster furanosil fosfato, denominado AI-2, y la cetona-alquil-hidroxilada, denominada CAI-I33. En esta bacteria, el mecanismo de QS regula varios fenotipos: la producción de biopelícula, la expresión de factores de virulencia (producción de proteasa) y la competencia. El primero de dichos fenotipos se expresa en condiciones de baja densidad celular; mientras que la virulencia, la producción de proteasa y la competencia se observan bajo condiciones de alta densidad53 (fig. 4A). En el periplasma de la bacteria se localizan dos quinasas histidínicas receptoras de los autoinductores, denominadas LuxP-LuxQ y CpqS. A baja densidad celular, las moléculas autoinductoras AI-2 y CAI-1 se encuentran en baja concentración; en estas condiciones ambas quinasas están activas y trasfieren su grupo P al regulador de respuesta y factor de transcripción LuxO. LuxO~P activo promueve la trascripción de un grupo de cuatro sRNA no codificantes denominados Qrr1-4; bajo estas condiciones estos sRNA activan la traducción de la enzima DGC (Vac0939) y se estimula la producción del di-GMPc41. El aumento de la concentración intracelular del segundo mensajero tiene como resultado final la expresión de los genes asociados a la síntesis de los EPS que forman la matriz de la biopelícula, a través de la activación de VpsT10,67 (fig. 4A y 4B). Más aún, el di-GMPc reprime la transcripción de los genes de la síntesis del flagelo y los factores de virulencia, aunque no han sido definidos los mecanismos por medio de los cuales se efectúa la represión11,39. Por otra parte, los Qrr1-4, a través de un mecanismo de regulación postranscripcional, inhiben la traducción del ARNm de HapR, el cual es un regulador transcripcional global que reprime la expresión de genes que participan en la síntesis de los EPS41,51. Así, los Qrr1-4 tienen una intervención dual por su interacción directa con: el ARNm de la DGC Vac0939, para promover la formación de biopelícula dependiente del di-GMPc, y por la inhibición de la traducción del ARNm de HapR60,84.
Bajo condiciones de alta densidad de población bacteriana se incrementa la concentración de los autoinductores. La unión de estos a las quinasas promueve que cambien su actividad a fosfatasas76, por tanto, la situación se revierte y LuxO~P sufre una desfosforilación, y detiene la producción de los Qrr1-4. Se permite así la traducción del ARNm de HapR, regulador de la expresión de genes que codifican la síntesis de enzimas PDE y proteasas, cuyas actividades se incrementan, mientras que la concentración celular del di-GMPc disminuye y decae la formación de biopelícula (fig. 4B). Entonces, en V. cholerae dos sistemas de QS convergen en un circuito único en el cual intervienen cuatro sRNA no codificantes, que probablemente regulan de manera fina y diferencial la expresión de virulencia y la formación de biopelícula25.
Participación del óxido nítrico y el di-GMPc en la regulación de la biopelículaEl óxido nítrico (nitric oxide, NO) es una molécula gaseosa lipofílica y también un radical libre, que actúa como molécula de señalización en células de mamíferos. El papel del NO se ha documentado ampliamente como un modulador de la neurotransmisión y un efector de la respuesta inmunitaria65. El NO es producido por los macrófagos en áreas localizadas a concentraciones citotóxicas y funciona como un mecanismo de defensa del hospedador46. Los biólogos de plantas también comunicaron tiempo después su participación como molécula de señalización en varios procesos vegetales14. Las bacterias están expuestas al NO en concentraciones altas (μM, como en los macrófagos) o bajas (nM). Algunas son capaces de producir NO por la vía de la desnitrificación como un intermediario5–7,65. El NO como molécula señal tiene una participación dual, ya que puede intervenir en la formación o bien en la dispersión de la biopelícula, esto dependiendo del estilo de vida de la bacteria y de la concentración fisiológica acumulada dentro de la célula55,56.
A concentración baja (nM), el NO es un factor que regula la dispersión de la biopelícula en Legionella pneumophila13, P. aeruginosa y S. aureus6,7,64. S. aureus ha desarrollado una respuesta al estrés nitroso necesaria para desencadenar la respuesta inmunitaria innata; así, en esta bacteria, la detoxificación y posibilidad de evitar exponerse a la concentración de NO derivada del hospedador es una estrategia para escapar del sistema inmunitario innato57. En concentración subletal (25nM), propicia la dispersión de la biopelícula producida por P. aeruginosa en pacientes con fibrosis quística7,8. Sin embargo, en Neisseria gonorrhoeae el NO funciona como una molécula señal para aumentar la formación de biopelícula; en efecto, se describió que el NO a concentraciones de 15–22nM aumenta la expresión de los genes que codifican a las enzimas nitrato reductasa y nitrito reductasa y fomenta la formación de la biopelícula en la uretra22. Por otra parte, en Nitrosomonas europea el NO a concentración de 30μM conduce a la formación de la biopelícula, y menos de 5μM inducen su dispersión66. Azospirillum brasilense produce NO como intermediario de la ruta de desnitrificación por la actividad concertada de tres enzimas: nitrato reductasa periplásmica (codificada por el operón nap) y dos nitrito reductasas (NirK1 y NirK2). Se estudió la capacidad de formación de biopelícula bajo condiciones de desnitrificación en una mutante nap::Tn5, la cual produce solo un 5 % de NO comparada con la cepa silvestre. La mutante deficiente en nitrato reductasa formó menor cantidad de biopelícula que la cepa silvestre, pero la producción se recuperó de manera parcial a los cinco días de crecimiento, lo que sugiere que el NO favorece la formación de biopelícula en el estadio inicial5.
Varios estudios realizados en bacterias patógenas de humano muestran que la biopelícula aumenta la resistencia a los antimicrobianos y que, como consecuencia de la dispersión de la biopelícula regulada por el NO, disminuye tanto la biopelícula como la resistencia a los antimicrobianos. De allí el interés en los estudios tendientes a identificar moléculas que dispersen las biopelículas producidas por los patógenos, pues estas podrían representar una estrategia importante para el tratamiento de infecciones producidas por estos microorganismos7.
A pesar de estas observaciones, los datos sobre las vías de señalización en las que participa el NO para regular la formación o producir la dispersión de la biopelícula aún son escasos. Sin embargo, se ha descrito recientemente un grupo de proteínas que funcionan como sensores del NO56. La familia de proteínas que contienen el dominio H-NOX (acrónimo de heme nitric oxide/oxygen) está compuesta por Fe-hemoproteínas que unen NO. Estas proteínas no funcionan como reguladores de la transcripción de los genes implicados en la dispersión, unen al NO de manera específica y participan en cascadas sofisticadas de regulación. Algunos ejemplos se describen a continuación.
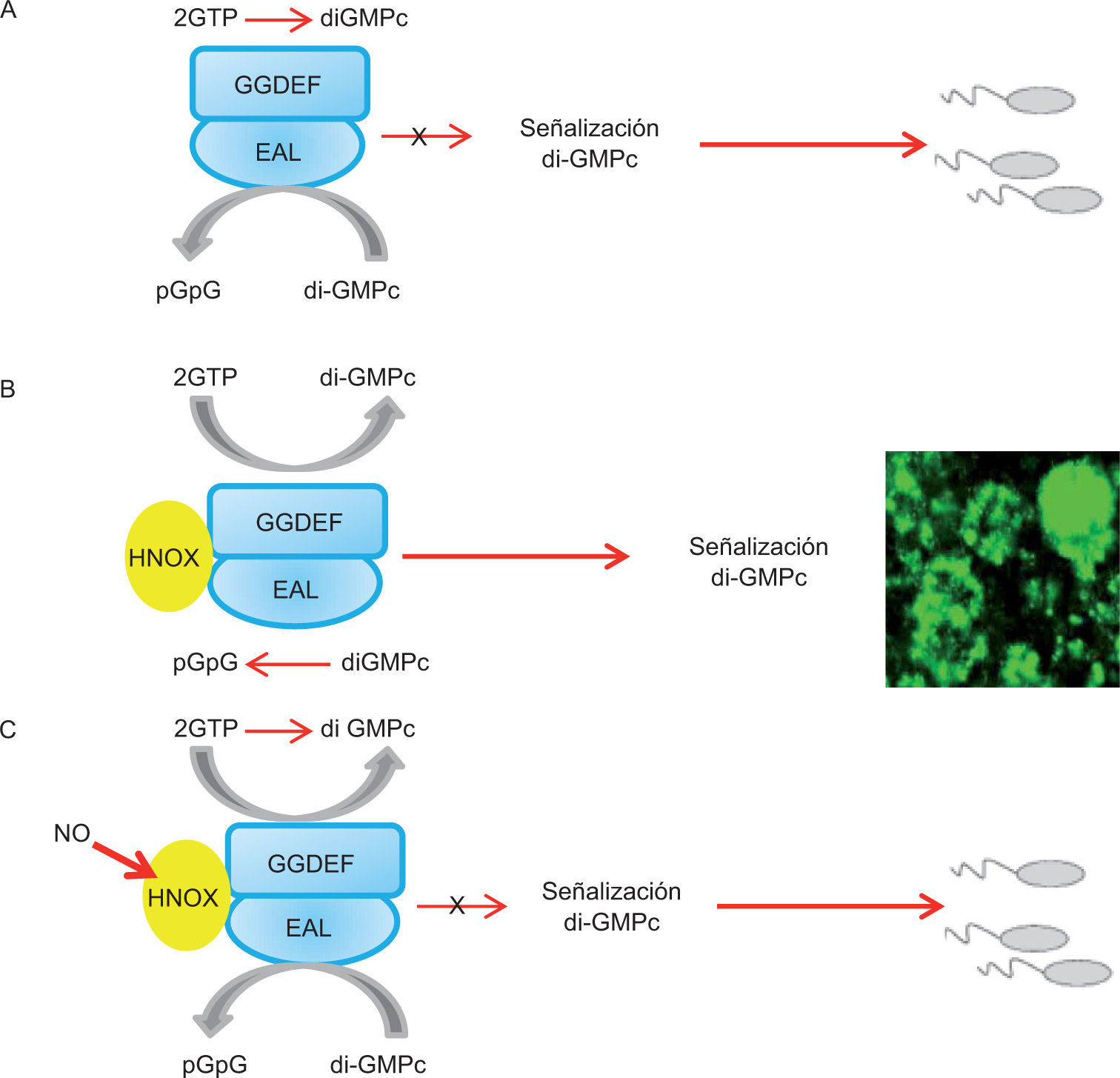
Un papel directo de la participación de estas proteínas en la señalización es el descrito en L. pneumophila. En el genoma de esta bacteria están presentes dos genes codificantes de proteínas que contienen el dominio H-NOX; uno de estos genes se localiza adyacente a otro gen (lpg1057), el cual codifica la síntesis de una proteína con dominios GGDEF-EAL. Esta proteína presentó actividad de DGC in vitro. La mutación del gen Δhnox1 produce un aumento en la producción de biopelícula. La actividad de DGC fue inhibida cuando se adicionó la proteína Hnox1 en la forma de Fe(II)-NO, lo que sugiere que ambas proteínas forman un heterodímero. En coincidencia con lo citado, la mutación del gen lpg1057 en la mutante Δhnox1 conduce a un fenotipo de hiperproducción de biopelícula. Los datos apuntan a la participación del NO en la regulación negativa de la actividad de DGC, así como en la disminución de la concentración intracelular del di-GMPc y, consecuentemente, de la formación de biopelícula13.
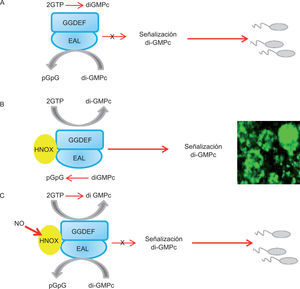
Otro ejemplo de la contribución de estas proteínas es el descrito en Shewanella woodyi MS3243. En este modelo la exposición al NO (60–80nM) disminuye la producción de biopelícula a través de la regulación directa de la actividad de DGC y el recambio del di-GMPc. En el proceso participan las proteínas SwDGC (Sw00_2750), la que presenta los dominios GGDEF-EAL, y SwHNOX (Sw00_2751), hemoproteína cuyo dominio liga NO. Ambas proteínas interactúan directamente. En ausencia de NO, SwDGC/SwH-NOX funcionan como DGC y regulan de manera positiva la producción de biopelícula (fig. 5). En la mutante Δhnox, SwDGC presenta actividad de PDE, lo que disminuye la concentración intracelular del di-GMPc y la formación de la biopelícula. Además, H-NOX-NO regula la actividad enzimática de SwDGC; el parámetro de eficiencia de la enzima (Kcat/Km) aumenta 13 veces en su actividad de PDE. Cuando el NO está presente y ligado a SwH-NOX, los resultados muestran el aumento de la actividad de PDE y una caída de la actividad de la DGC. En el modelo, la molécula señal NO contribuye a la regulación negativa de la formación de la biopelícula (fig. 5), por lo que se propone que el NO induce una rápida transición del estilo de vida sésil al planctónico43.
Regulación de la formación de biopelícula por NO en S. woondyi. A. La proteína SwDGC tiene actividad dual (dominios GGDEF y EAL). Como consecuencia de la actividad basal de PDE, la concentración celular del di-GMPc es baja y la célula es móvil. B. En ausencia del NO, SwH-NOX y SwDGC forman un heterodímero, este complejo presenta actividad de PDE basal y alta actividad de DGC, aumenta la concentración celular de di-GMPc y la formación de biopelícula. C. En presencia de NO (80nM), disminuye la actividad de DGC y aumenta la actividad de PDE, por lo tanto, disminuye el di-GMPc y la formación de biopelícula. Permiso: Biochemistry, modificado, de Liu et al.43.
En Shewanella oneidensis funciona un mecanismo más complejo, en el que participan la quinasa histidínica (hnoK, SO2145) y la proteína HNOX (hnoX, SO2144), así como tres RR de HnoK: HnoB-PDE, HnoC y HnoD-HD-GYP. Esta última proteína posee un dominio PDE degenerado que funciona como un inhibidor alostérico (une al di-GMPc). La caracterización bioquímica del sistema mostró que cuando el NO se une a su receptor (HNOX-NO), se inhibe la autofosforilación, mientras que cuando los RR están fosforilados (ausencia de NO), HnoB~P estimula la actividad de hidrólisis del di-GMPc; el heterodímero HnoB~P + HnoD~P aumenta la concentración intracelular del di-GMPc, con el resultado de aumentar a su vez la biopelícula. Adicionalmente, la proteína HnoC~P controla la transcripción de los elementos participantes en la red de regulación. En resumen, la inactivación de la quinasa HnoK por la presencia del NO regula finamente la concentración intracelular del di-GMPc55, lo que sugiere que el efecto del NO sobre la formación o dispersión de la biopelícula es un fenómeno especie-específico y depende de la transición del estilo sésil al planctónico de la bacteria estudiada.
El sistema “sencillo” en el cual participa un componente lleva a la dispersión de la película en respuesta a la concentración del NO, mientras que en el sistema más “sofisticado”, como es el caso de S. oneidensis, las señales que participan tienen efectos opuestos: aumentan la adhesión y la biopelícula en respuesta al NO como un mecanismo de protección frente a la concentración citotóxica de este compuesto32, de manera similar a lo que se observa en Neisseria meningitidis, aunque en esta bacteria no se ha descrito aún el mecanismo molecular subyacente22.
ConclusionesGeneralmente se forma biopelícula en respuesta a varios factores ambientales (temperatura, pH, oxígeno, nutrientes, etc.) y, en algunos casos, a la presencia de antibióticos a concentraciones subinhibitorias. Las bacterias detectan y responden a las variaciones del entorno para controlar procesos de su desarrollo, cambios en la densidad de población por regulación del quórum y condiciones locales, con señales como autoinductores, di-GMPc y óxido nítrico.
En años recientes, el di-GMPc emerge como un regulador global central y clave en la fisiología bacteriana, al intervenir en vías de transducción de señales en las bacterias y controlando, en células móviles, la transición del estado planctónico al sésil. Además, en general controla la formación de biopelícula, la virulencia, la señalización célula-célula y la regulación del ciclo celular. A pesar de la importante información de la que disponemos, el conocimiento de las vías de señalización mediada por el di-GMPc es parcial: únicamente la descripción de algunos circuitos es completa y, en la mayoría de los casos, se conocen solo algunos de los componentes que intervienen. En efecto, los ejemplos aquí incluidos ilustran que los mecanismos por los cuales el di-GMPc ejerce su papel regulador en las bacterias involucran diferentes señales, efectores, enzimas, factores de transcripción, receptores y blancos mismos que frecuentemente se desconocen. Aquellos de los que se tiene un mejor panorama ilustran que las redes de regulación son complejas y que pueden actuar de manera independiente, o bien en conjunto, para modular la fisiología de las bacterias de manera fina y eficaz. Quizás uno de los sistemas mejor conocidos es el de V. cholerae, que comprende dos quinasas histídinicas/fosfatasas reguladas por QS, cuyas señales son canalizadas hacia un único circuito que activa o reprime la expresión de los sRNA mismos, que regularán los genes específicos de virulencia o biopelícula. En conjunto con el di-GMPc, el QS controla a baja densidad de población la formación de biopelícula, y en alta densidad se reprime y se lleva a cabo la dispersión de la misma, esto propicia una colonización eficiente del hospedador y permite la expresión de los factores de virulencia. En tanto que en condiciones de baja densidad de población se realiza la represión de los factores de virulencia. Los autores sugieren que se propicia un escape eficiente del hospedador. No obstante, esta hipótesis no ha sido probada experimentalmente60,84. Esta fina regulación ofrece ventajas competitivas y flexibilidad en las interacciones: hospedador-patógeno y microbio-microbio.
Por otra parte, el conocimiento de los mecanismos moleculares implicados en la formación de la biopelícula puede proporcionar estrategias en la clínica, particularmente para el control de infecciones crónicas y otros problemas relacionados con presencia de biopelículas. En efecto, recientemente se describió el compuesto meta-bromo-tiolactona que funciona como un inhibidor del sistema de QS de P. aeruginosa, el cual es capaz de inhibir la formación de biopelícula y la expresión de factores de virulencia54,58,68.
La biopelícula no es una estructura inmutable, la etapa final de esta es la dispersión de las células de la comunidad. La capacidad de inhibir la formación de la biopelícula y disociarse de la matriz es activamente inducida por varias señales, entre las que se incluyen QS, NO y secreción de proteínas como nucleasas y glucosil hidrolasas79,45.
La respuesta de las bacterias al NO, como se anotó, es género-específica y especie-específica, y modula de manera directa o indirecta la actividad de las DGC13,22,43. Ello le permite a la célula bacteriana acceder a otros nichos favorables para su desarrollo; esto es de suma importancia en bacterias que se asocian con plantas y producen efectos benéficos en ellas: la dispersión de la biopelícula puede proporcionar a la bacteria una ventaja competitiva al acceder a un nuevo nicho y continuar colonizando al hospedador5.
Sin embargo, a pesar del enorme esfuerzo realizado en los últimos años para incrementar el conocimiento sobre el repertorio de las funciones implicadas en la formación de la biopelícula, todavía se está en la etapa inicial, por lo que es muy probable que en el futuro, cuando otros modelos bacterianos sean caracterizados, se comprenda mejor este estilo de vida. No hay duda de que futuros estudios revelarán e identificarán circuitos de regulación más elaborados que participan en la construcción, maduración y dispersión de la biopelícula.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datosLos autores declaran que en este artículo no aparecen datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores han obtenido el consentimiento informado de los pacientes y/o sujetos referidos en el artículo. Este documento obra en poder del autor de correspondencia.
El trabajo realizado en el laboratorio de la interacción bacteria-planta fue apoyado por el Consejo Nacional de Ciencia y Tecnología (Ref. CB-2010-1-154914), el Programa de Mejora al Profesorado (Ref. 103.5/13/8869) y la Vicerrectoría de Investigación y Estudios de Posgrado de la Universidad Autónoma de Puebla. Agradecemos al Dr. José Emilio Salceda, por la revisión del manuscrito.