El estudio de las variaciones de las secuencias de ADN y ARN en relación con la respuesta a diferentes fármacos, se ha convertido en un área de estudio particularmente prometedora para la aplicación en genómica clínica y estudios de genomas personalizados. Medicamentos de uso diario en el tratamiento de enfermedades cardiovasculares han demostrado variaciones en la respuesta en función de las variantes genéticas de los individuos. Dos fármacos han concentrado el interés mundial: la warfarina, un anticoagulante oral, y el clopidogrel, un antiagregante plaquetario, los cuales actúan alterando diferentes vías que conforman la cascada de la coagulación, ya sea limitando directamente la producción de trombina o bloqueando otros activadores de la ruta. Los cambios genéticos que se han asociado a la reducción de la actividad enzimática de estos fármacos ocurren en los genes, CYP2C19 para clopidogrel y CYP2C9 y VKORC1 para warfarina. Las variaciones genéticas identificadas para estos genes se relacionan con perfiles genotípicos que determinan la dosis requerida para el paciente. Es allí donde ciencias como la farmacogenómica tienen como fin brindar una ayuda diagnóstica más objetiva al optimizar tiempo y recursos, así como disminuir el riesgo del paciente a sufrir complicaciones que comprometan su vida.
The study of the variations in DNA and RNA sequencing as regards the response to different drugs has become a particularly promising area for their application in clinical genomics and personalised genome studies. Drugs of daily use in the treatment of cardiovascular diseases have shown variations in the response depending on the genetic variations of the individuals. Two drugs have gathered worldwide interest: warfarin, an oral anticoagulant, and clopidogrel, an antiplatelet drug, which act by altering different pathways that constitute the clotting cascade either by directly limiting the production of thrombin, or by blocking other activators of the pathway. The genetic changes that have been associated with the reduction in the enzyme activity of these drugs occur in the genes, CYP2C19 for clopidogrel, and the genes, CYP2C9 and VKORC1 for warfarin. The genetic variations identified for these genes are associated with genotype profiles that determine the dose required by the patient. It is from there, sciences like pharmacogenomics have as their aim to provide a more objective diagnostic aid in order to optimise time and resources, as well as to reduce the risk of the patient suffering complications that may compromise their life.
La mayoría de los fármacos en sus dosificaciones convencionales no generan efectos uniformes en todos los pacientes que reciben la misma terapia. Se estima que en promedio entre el 30 y el 60% de los pacientes no responden de manera correcta al tratamiento1. La efectividad de un agente farmacológico puede estar influenciada por factores endógenos como la constitución genética de cada individuo, y exógenos como edad, sexo, talla y dieta2.
Los factores genéticos se han asociado como la variable más significativa en el metabolismo de los fármacos. Las alteraciones en el genoma explican entre un 20 y un 95% la variabilidad fenotípica que puede mostrar un grupo de individuos en la respuesta a un tratamiento farmacológico3. Genes que codifican proteínas metabolizadoras, transportadoras o receptoras, son considerados altamente polimórficos en los humanos. Muchas de las diferencias genéticas que existen entre las diversas poblaciones, e incluso entre individuos de la misma población, van desde cambios de un sólo nucleótido conocidos como SNP (sigla del inglés single nucleotide polymorphisms), repeticiones de secuencias cortas en tándem STR (sigla del inglés short tandem repeat), hasta variaciones en el número de copias de un gen CNV (sigla del inglés copy number variation) e inserciones y deleciones (indels)4–6. Gracias a la secuenciación del genoma completo y tras la realización del proyecto HapMap se ha logrado establecer que existen más de 10 millones de SNP dispersos en todo el genoma humano1,2, algunos de los cuales se han asociado con cambios en la cinética o la dinámica de los fármacos, por lo que se han convertido en un blanco habitual de análisis farmacogenómicos.
Algunos estudios se han enfocado en la identificación de cambios nucleotídicos que causen mutaciones que interfieran con la acción y la respuesta fenotípica de fármacos. Con el fin de conocer el valor predictivo de estos cambios y optimizar el uso de los mismos, dentro de la farmacogenómica se genera una clasificación del fenotipo metabolizador para cada paciente antes de iniciar algún tratamiento. Esto permite de manera individual y basada en la genómica personalizada de cada individuo, prescribir de forma más eficaz y optimizar tiempo y recursos.
En este artículo se revisan los aspectos más relevantes de la farmacogenómica de la warfarina y el clopidogrel, la relación entre las variables genéticas y la respuesta metabólica a estos fármacos, buscando consolidar información que sea de gran utilidad para el ejercicio de los clínicos, debido a que vienen ejecutándose y validándose avances en este tema con el fin de disponer de pruebas moleculares sencillas, de bajo costo y al alcance de los clínicos e individuos que las requieran e impactar de manera positiva la práctica clínica local.
Variabilidad enzimática (fenotipo metabolizador)Tras la administración de un fármaco, el organismo procede a la biotransformación de este, la cual se genera en diferentes tejidos como el hígado, el intestino, los pulmones, los riñones, el cerebro, el plasma o la piel7. Para llevar a cabo este proceso existen varias familias de enzimas metabolizadoras que presentan polimorfismos genéticos causantes de cambios funcionales en la proteína codificada.
Los determinantes genéticos pueden agruparse en dos categorías, los que afectan la farmacodinámica y aquellos que afectan la farmacocinética. El primero se refiere al efecto del fármaco en el sitio de acción, o a la interacción fármaco-receptor, mientras el segundo se relaciona con los factores que determinan la manera en que los fármacos son metabolizados y la disponibilidad de estos en su sitio de acción8,9. Los genes que codifican las proteínas encargadas de metabolizar diferentes fármacos presentan alta variabilidad genética, la cual puede llegar a afectar la expresión, el nivel de actividad o la función enzimática que da origen a distintos fenotipos, conocer acerca de la presencia de estos polimorfismos responsables del metabolismo de fármacos, puede ayudar a individualizar el tratamiento terapéutico, asegurando la elección del mismo, adaptando su dosificación y disminuyendo la aparición de reacciones adversas10.
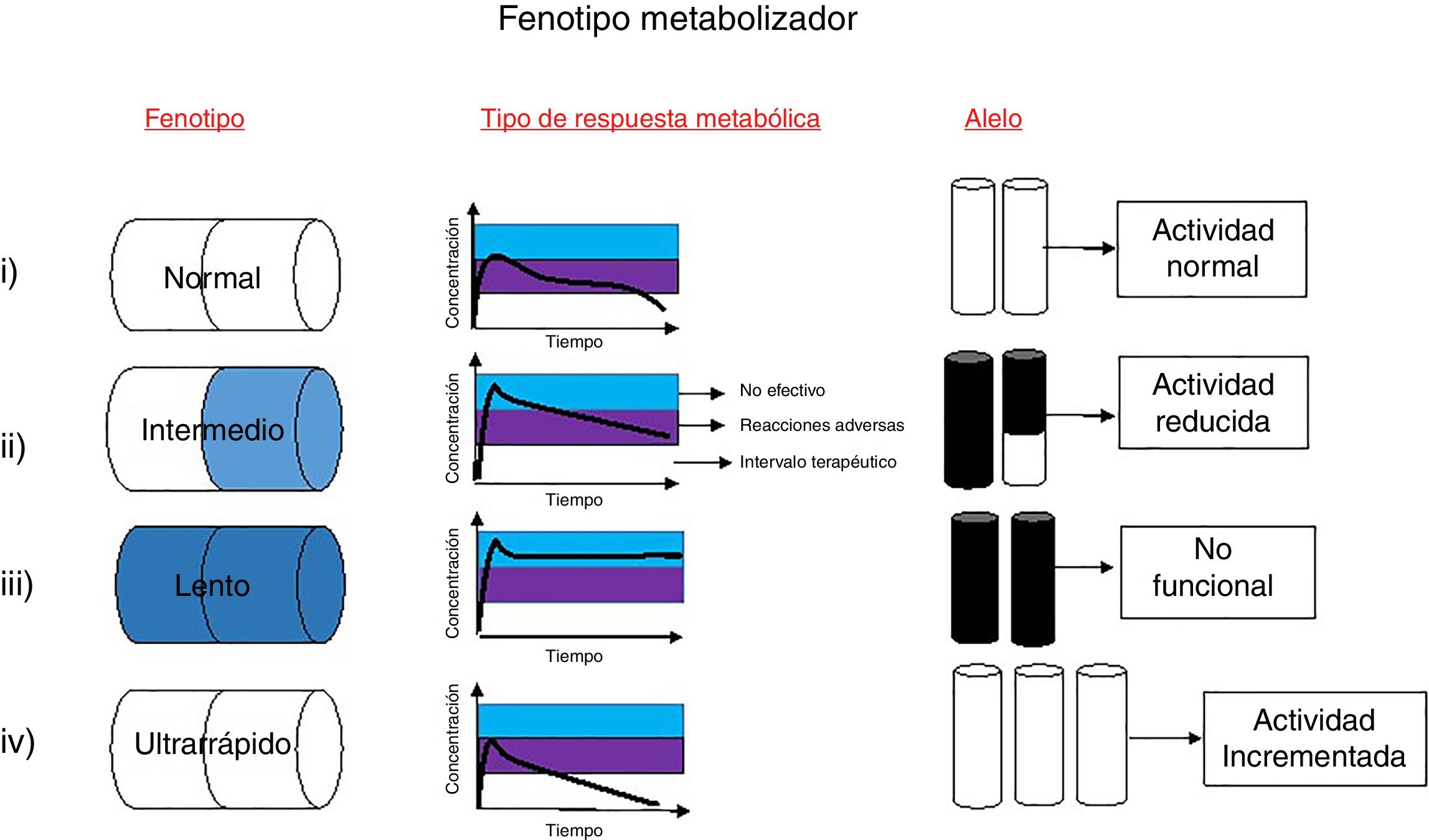
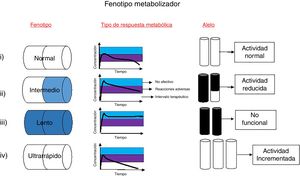
El término fenotipo metabolizador describe la respuesta de una persona al procesamiento de ciertos fármacos (fig. 1). La determinación de éste se basa en el número y tipo de alelos funcionales presentes en los genes involucrados en las rutas metabólicas y de su respuesta. Los genes de la familia del citocromo P450 (CYP450) codifican enzimas encargadas del metabolismo de más del 80% de los fármacos existentes11. Con frecuencia, en los pacientes que presentan variaciones a nivel estructural en estos genes, se observan tasas metabólicas reducidas comparadas con el promedio de las personas que no tienen ninguna variación genética en esta familia de genes12. Se han determinado cuatro posibles fenotipos en función de la actividad metabolizadora de las enzimas CYP. A continuación, se realizará la relación a cada fenotipo11.
Fenotipo metabolizador. Existen cuatro fenotipos metabolizadores relacionados con cambios en la respuesta de un paciente frente a terapias farmacológicas: i) Metabolizadores normales: estos metabolizan los fármacos de manera normal, es decir, procesan los fármacos en tiempo y forma adecuada. Su genotipo es homocigoto para alelos de actividad normal y son conocidos como *1/*1. ii) Metabolizadores intermedios: metabolizan los fármacos de manera lenta comparada con el fenotipo normal; estos presentan un genotipo heterocigoto con un alelo de actividad reducida, junto con un alelo no funcional. iii) Metabolizadores lentos: poseen deficiencia para metabolizar los fármacos; este tipo de fenotipo requiere menores dosis del medicamento, y poseen dos copias iguales del alelo no funcional. iv) Metabolizadores ultrarrápidos: se caracterizan por tener mayor capacidad metabólica. La dosis media del fármaco es metabolizada de forma inmediata y como resultado su concentración en plasma disminuye, estando por debajo de los niveles terapéuticos, por lo que es necesario aumentar su dosis. La selección de un fenotipo se basa en el número de copias y tipo de cambio que puedan presentar en los genes que codifican las enzimas metabolizadoras.
a. Metabolizadores pobres (PM, poor metabolizer): los individuos con este fenotipo no son capaces de realizar la conversión del fármaco activo en metabolitos inactivos, lo que lleva a un riesgo de toxicidad. Los PM corresponden a homocigotos para los alelos no funcionales, lo cual genera una proteína ausente o sin función12.
b. Metabolizadores normales (EM, extensive metabolizer): corresponden a individuos homocigotos para los alelos de función normal. Los individuos con este fenotipo no experimentan reacciones adversas frente al tratamiento12.
c. Metabolizadores intermedios (IM, intermediate metabolizer): los individuos con este fenotipo son heterocigotos, presentando un alelo funcional y otro con actividad reducida. Este tipo de metabolizadores, al igual que en el fenotipo PM presentan una reducción en la capacidad de transformar el fármaco activo a metabolitos inactivos, además de que aumenta el riesgo de tener niveles séricos más altos del fármaco activo en la sangre, causando toxicidad en dichos individuos13.
d. Metabolizadores ultrarrápidos (UM, ultrarapid metabolizer): los UM son individuos que transforman los fármacos muy rápidamente. Portan más de dos copias de los alelos funcionales. En el caso de las enzimas CYP2C19 corresponde a un homocigoto con dos alelos de función incrementada o un heterocigótico con un alelo de función incrementada con otro de función normal13.
Farmacogenómica para medicamentos de uso en enfermedades cardiovascularesLas enfermedades cardiovasculares son una de las principales causas de morbilidad y mortalidad en el mundo, reportando cerca de 17,5 millones de muertes por año3. Medicamentos cardiovasculares comúnmente utilizados tales como antiarrítmicos, hipolipemiantes y anticoagulantes han demostrado tener un amplio rango de respuesta metabólica en función de variantes genéticas. Con la necesidad mundial de mejorar la atención en las enfermedades cardiovasculares, se han buscado diferentes herramientas que faciliten la determinación de la dosis de los respectivos medicamentos impactando en el tiempo de ensayo y error y aumentando la especificidad y sensibilidad en el tratamiento. Es así como actualmente se registra un incremento en la investigación y desarrollo de métodos moleculares que puedan ser implementados como un examen de rutina en la consulta clínica13, minimizando la incidencia de eventos adversos de tipo hemorrágico que ponen en riesgo la vida del paciente.
Los estudios de farmacogenómica en el área cardiovascular se han concentrado, en gran medida, en los medicamentos que ya tienen licencia y que son de uso clínico generalizado. Proyectos como el CIPIC (Clinical Pharmacogenetics Implementation Consortium) y PharmGKB (The Pharmacogenomics Knowledgebase)13 han desarrollado y publicado guías que contienen recomendaciones de dosis terapéuticas basadas en el genotipo de los biomarcadores. Teniendo en cuenta esta información, la FDA (Food and Drugs Administration) ha elaborado una lista con diferentes fármacos que presentan una efectividad terapéutica reducida14. El ejemplo más conocido es el del anticoagulante oral warfarina, medicamento empleado para la prevención y el tratamiento de eventos tromboembolíticos. La etiqueta comercial de este contiene información farmacogenética basada en los genes CYP2C9 y VKORC1. Dentro de la lista también se encuentra otro fármaco con relevancia cardiovascular, el antiagragante plaquetario clopidogrel, utilizado para el tratamiento de eventos aterotrombóticos y cardíacos, cuya dosificación depende del biomarcador CYP2C1915–17.
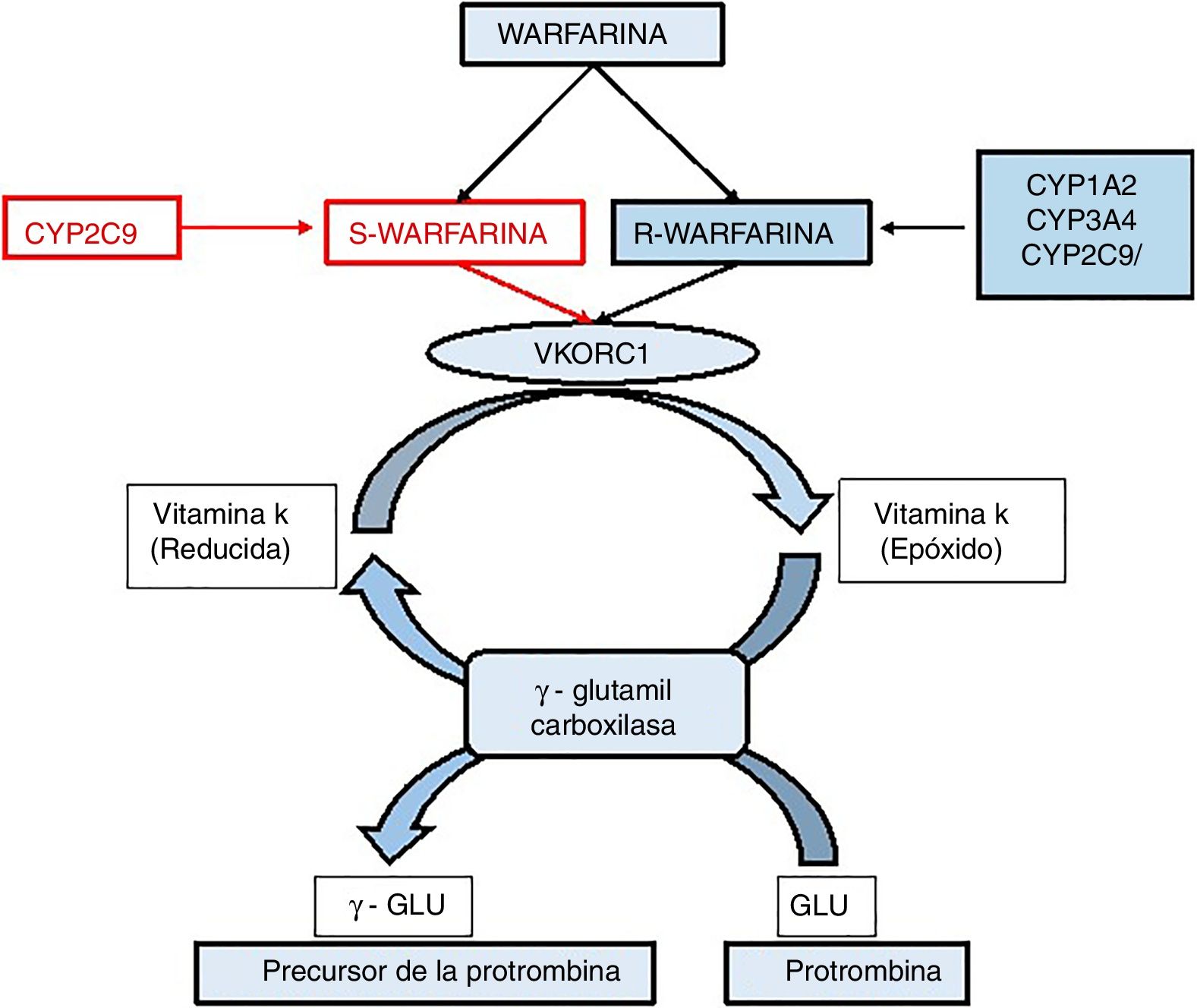
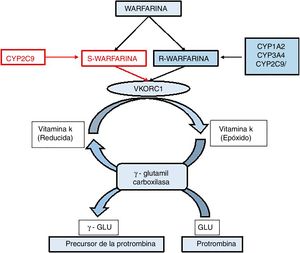
WarfarinaFue descubierta en los inicios del siglo xx y hoy en día es el anticoagulante oral más utilizado en el mundo. En la práctica clínica se destina principalmente para pacientes con tromboembolia venosa, fibrilación auricular y válvulas mecánicas cardíacas18; sin embargo, su respuesta tiene un índice terapéutico estrecho y una amplia variabilidad interindividual. En cuanto a la farmacodinámica, este es absorbido rápidamente en el tracto gastrointestinal logrando en pocas horas su concentración máxima en plasma. La warfarina pertenece al grupo de las 4-hidroxicumarinas, tiene un centro quiral y su síntesis industrial origina una mezcla racémica, presentando el enantiomero S con una actividad cinco veces más que la del enantiomero R19. La figura 2 ilustra la vía metabólica de la warfarina, la cual actúa inhibiendo la vitamina K reductasa, lo que desencadena un agotamiento de la forma reducida de dicha vitamina. La vitamina K es un cofactor de la carboxilación de los residuos de glutamato presentes en el extremo N terminal de las proteínas dependientes de esta. Mediante la administración de warfarina se limita la gamma-carboxilación y posterior activación de las proteínas coagulantes dependientes de la vitamina K. Es decir, se inhibe la síntesis de los factores II, VII, IX y X de la cascada de coagulación, así como las proteínas anticoagulantes C y S. La falta de tres de los cuatro factores coagulantes dependientes de vitamina K, desencadenan un descenso en los niveles de protrombina, lo que hace que se reduzca la trombogenicidad de los coágulos20.
Vía metabólica de la warfarina. El gen diana de la warfarina, VKORC1, codifica el complejo 1 de la vitamina K epóxido reductasa que es un cofactor clave en la carboxilación de ciertos residuos de glutamato, los cuales son esenciales para el correcto funcionamiento de los factores de coagulación II, VII, IX y X, en este proceso la vitamina K es oxidada. La acción de la warfarina inhibe la enzima VKOR lo que impide la activación de los factores de la coagulación.
Los polimorfismos de un solo nucleótido (SNP), han sido involucrados como responsables de la variabilidad de la respuesta a los fármacos observada entre individuos. Particularmente se han estudiado SNP relacionados con la familia de enzimas citocromo P450 (CYP). Un caso puntual es el gen CYP2C9, miembro de esta familia, que tiene una gran importancia en el metabolismo e inactivación del enantíomero-S de la warfarina. Los polimorfismos en esta enzima dan lugar a proteínas con diferente actividad catalítica, donde el alelo salvaje (actividad enzimática de 100%) se ha designado como CYP2C9*1 y las otras dos variantes las cuales son las más comunes en diferentes poblaciones en el mundo, CYP2C9*2 con el cambio (Arg144Cys) y CYP2C9*3 (Ile359Leu)21, donde al alelo *2 se le atribuye una disminución del 30% de la actividad enzimática. A los pacientes con este genotipo se les debe disminuir la dosis diaria en un 17%22. Por otro lado, la variante *3 ocasiona una deficiencia en el metabolismo de la warfarina en un 95% comparada con la actividad enzimática del alelo normal23. Estos polimorfismos determinan el fenotipo metabolizador lento. Los portadores de estos alelos requieren menores dosis de warfarina para lograr la anticoagulación. A dosis muy altas, están expuestos a mayor riesgo de hemorragia durante el tratamiento24.
Dentro de la ruta metabólica de las cumarinas existe una molécula diana llamada epóxido reductasa de la vitamina K, (VKOR) la cual es altamente polimórfica y está asociada a los cambios fenotípicos de la warfarina. Esta enzima hace parte del ciclo de la vitamina K y es responsable de la regeneración de la hidroquinona de vitamina K, a partir de la 2,3-epóxido vitamina K. Esta funciona como un cofactor y es esencial para la activación postraduccional. El gen codificante para la enzima es VKORC1, el cual contiene diferentes SNP que se encuentran en cercanía espacial que por lo general suelen presentarse juntos, o en disequilibrio de ligamiento. Las variantes con importancia funcional dentro del promotor y de la región intrónica del gen son 1639G→A, 497 T→G, 1173 C→T, 1542 G→C, 2255 C→T. Dada su herencia combinada, la identificación de cualquiera de estos polimorfismos permite la caracterización del paciente, teniendo en cuenta estos SNP25. Rider et al. crearon dos grupos de haplotipos: uno denominado de dosis bajas (A) y el otro de dosis altas (B). Las variaciones de las dosis son 2,7±0,2mg/día para un individuo con haplotipo A/A, 9±0,2mg/día para un haplotipo A/B, y 6,2±0,3mg/día para el haplotipo B/B20. La variación en la dosis se representa en la tabla 1 donde se muestra la correlación entre los haplotipos de VKORC1 y los SNP del gen CYP2C9. La asignación de estos índices terapéuticos se hizo respecto a la formulación de algoritmos que tienen en cuenta no solo factores genéticos sino también diferentes variables externas tales como edad, dieta, sexo, entre otras3. El mayor beneficio de la inclusión de la constitución genética del individuo en la dosificación de la warfarina se ha generado en pacientes que requieren menos de 21mg/semana o más de 49mg/semana, lo que constituye más del 40% de todos los pacientes que consumen el fármaco26.
Recomendación en la dosis (mg/día) para warfarina basada en los genotipos de los genes CYP2C9 y VKORC1
| VKORC1 -1639 G>A | CYP2C9 *1/*1 | CYP2C9 *1/*2 | CYP2C9 *1/*3 | CYP2C9 *2/*2 | CYP2C9 *2/*3 | CYP2C9 *3/*3 |
|---|---|---|---|---|---|---|
| AA | 5-7 | 5-7 | 3-4 | 3-4 | 3-4 | 0,5-2 |
| AB | 5-7 | 3-4 | 3-4 | 3-4 | 0,5-2 | 0,5-2 |
| BB | 3-4 | 3-4 | 0,5-2 | 0,5-2 | 0,5-2 | 0,5-2 |
Es un agente antiplaquetario que pertenece a la familia de las ticlopidinas. En la práctica clínica se prescribe para inhibir la formación de coágulos de sangre en las arterias coronarias, periféricas y cerebrales. Además, se administra como tratamiento estándar en casos de síndrome coronario agudo, y en pacientes que han sufrido infarto al miocardio, ataques carebrovasculares recientes o para la prevención de trombosis después de una intervención coronaria percutánea con implante de endoprótesis coronaria3.
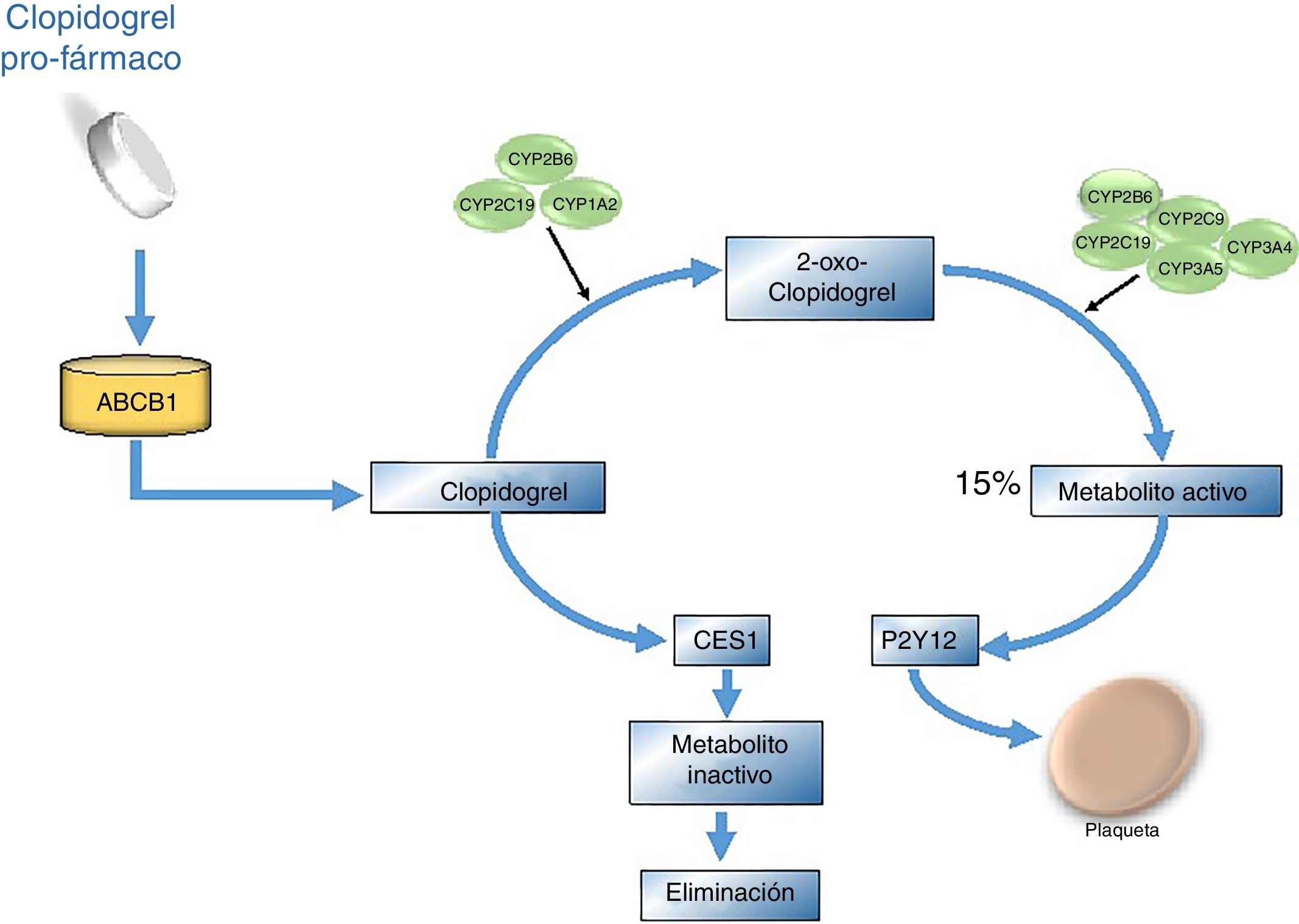
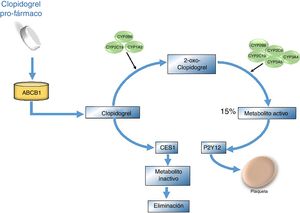
La activación del clopidogrel, que se cataloga como un profármaco, se produce por medio de una enzima de la familia citocromo P450 oxidasa llamada CYP2C19. Algunas variantes de estas enzimas han sido implicadas en la diversidad del efecto clínico del mismo27. Para cumplir su ruta metabólica y proceso de absorción en el organismo, este requiere la presencia de la glicoproteína P, codificada por el gen de multirresistencia a drogas (MDR1) o ATP (ABCB1). Una vez absorbida, la molécula debe transformarse a su metabolito activo a través de dos pasos; en el primero de ellos se produce el 2-oxo-clopidogrel por medio de las enzimas CYP2C19, 2B6 y 1A2, mientras que la producción del tiol activo depende de la actividad de CYP2C19, 3A4/5, 2B6 y 2C9. Finalmente, este compuesto inhibe el receptor P2Y12 de adenosín difosfato (ADP) expresado en las plaquetas, el cual normalmente facilita la degranulación de las mismas y la expresión del receptor GP IIb/IIIa en su superficie. El 15% del fármaco es convertido a metabolito activo y el resto es hidrolizado por esterasas (fig. 3)28. Existe una variabilidad sustancial relacionada con la función anti-plaquetaria del clopidogrel, ya que esta cambia ampliamente entre individuos, reportándose falta de respuesta que va desde un 4% a un 30% posterior a 24 horas de administrado el medicamento29.
Vía metabólica del clopidogrel. El clopidogrel se administra en forma inactiva, es decir requiere ser activado una vez se encuentre dentro del organismo. Su absorción se efectúa en el intestino por medio de la glicoproteína P codificada por el gen ABCB1. Durante la biotransformación del clopidogrel, primero se da la formación de 2-oxo-clopidogrel y de este pasa a su metabolito activo por medio de las enzimas CYP2C19, 3A4/5,2B6 y 2C9; finalmente este proceso inhibe el receptor plaquetario de adenosín difosfato (P2Y12) y de este modo se impide la agregación plaquetaria. El 15% del clopidogrel es transformado al metabolito activo y el resto es hidrolizado por esterasas como CES1.
La enzima CYP2C19 es un factor clave en la transformación del clopidogrel; actúa en la primera fase del metabolismo y es responsable aproximadamente del 45% de la conversión del clopidogrel a 2-oxo-clopidogrel y alrededor de un 20% del paso final en la generación del metabolito activo30. Se han identificado varios polimorfismos asociados al gen que generan un cambio en la respuesta del paciente al fármaco.
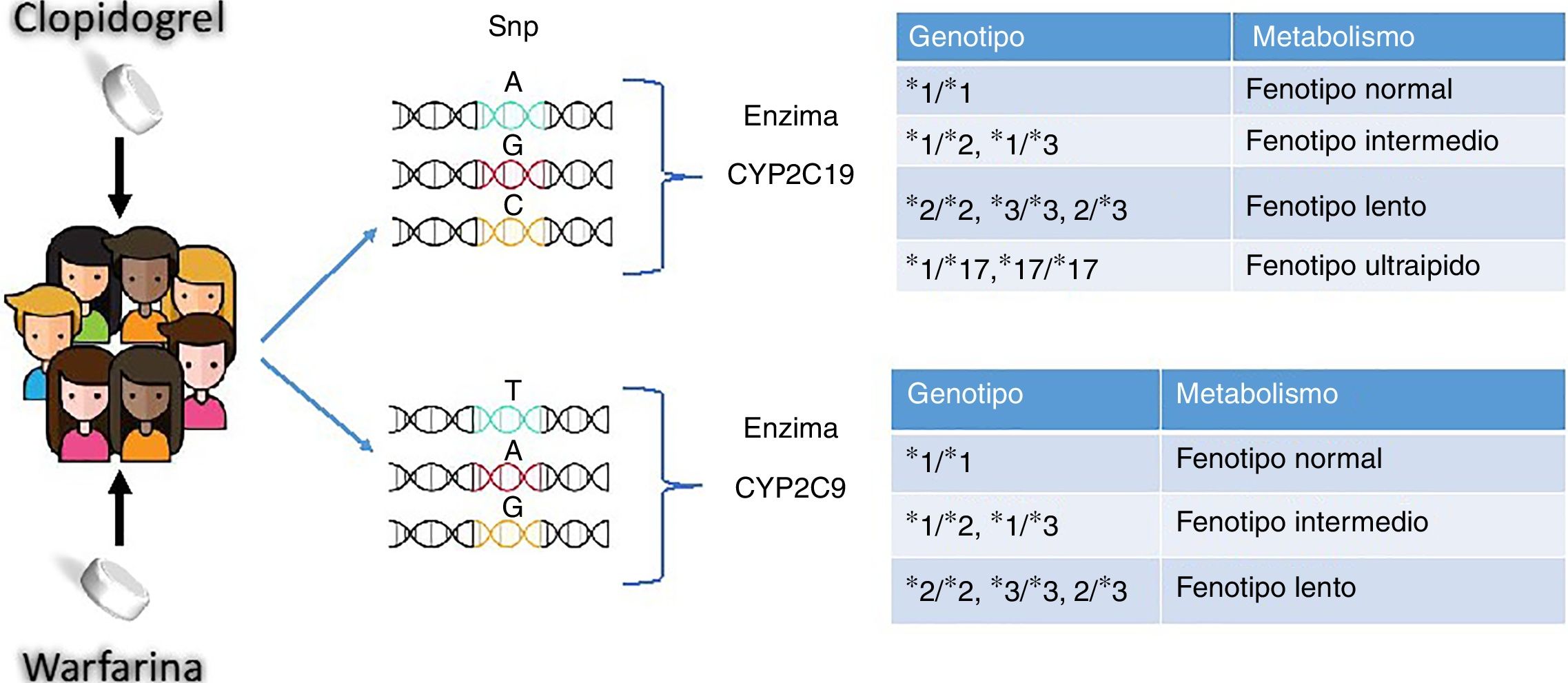
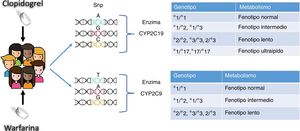
Se han reportado cerca de 30 variaciones alélicas para este gen30. Las variantes con mayores índices de escasa actividad enzimática son CYP2C19*2, CYP2C19*3 y CYP2C19*17. El alelo que codifica para la enzima normal o tipo salvaje se conoce como CYP2C19*1. El cambio reportado como CYP2C19 *2 es el más común, cuya mutación se encuentra en el exón 5 con un cambio en el nucleótido (G>A), dando como resultado un sitio de corte y empalme aberrante. Los portadores de este cambio tienen menores niveles de clopidogrel como metabolito activo en sangre. La inhibición plaquetaria se ve reducida, lo que aumenta el riesgo de eventos cardiovasculares31. Por su parte, la variante CYP2C19*3 se produce por una mutación en el exón 4, el cual genera un codón de parada prematuro. Estas dos variaciones son las responsables del fenotipo metabolizador lento32. El alelo CYP2C19*17 (C>T) se encuentra en una región no codificante y clasifica a los individuos de fenotipo metabolizador rápido quienes tienen mayores niveles del metabolito activo en la sangre, lo que aumenta el riesgo de hemorragias33. En la figura 4 se observan los principales genotipos para los fármacos warfarina y clopidogrel con sus respectivos fenotipos metabolizadores.
Variaciones genéticas en la respuesta individual a un tratamiento. El enfoque tradicional por el cual se efectuaba la prescripción de fármacos propone que todos los individuos con la misma patología reciban la misma dosis de tratamiento; sin embargo por estudios farmacogenómicos se han identificado los cambios genéticos como un factordecisivo a la hora de la prescripción de un fármaco en la práctica clínica. La estrategia del tratamiento basado en el perfil genético de cada individuo permite que este sea clasificado según su genotipo y fenotipo. Dentro de los fármacos más comunes en los tratamientos para condiciones cardiológicas, se incluyen el antiagregante plaquetario clopidogrel el cual presenta cuatro cambios genotípicos importantes y a su vez la warfarina que presenta dos cambios considerados con importancia clínica.
La monitorización terapéutica de fármacos (MTF), se basa en pruebas bioquímicas de rutina, tales como concentraciones séricas de protrombina para la warfarina o pruebas de agregometría plaquetaria para el clopidogrel34. Este tipo de exámenes se realiza para dar seguimiento y verificar que el paciente esté respondiendo a la dosis que se le está suministrando. Sin embargo, no pueden predecir las posibles reacciones adversas que puede llegar a desarrollar un paciente antes de iniciar una terapia farmacológica, contrario a las pruebas farmacogenómicas que funcionan como un modelo predictivo para la dosis del fármaco. Estas se basan en la identificación de los polimorfismos de genes encargados de sintetizar enzimas metabolizadoras. Este tipo de pruebas se hacen justo antes de comenzar una terapia farmacológica y se incluyen en los algoritmos de predicción de dosis que en la actualidad han sido aprobados por organizaciones como la FDA35. Por ejemplo, para el año 2007 dicha organización recomendó que antes de iniciar el uso de anticoagulantes como la warfarina, se debe considerar la determinación de los cambios genéticos relacionados con los genes CYP2C9 y VKORC1, para así poder ajustar la dosis del fármaco. Adicionalmente, para el año 2009 se agregó una advertencia en la información de prescripción del clopidogrel para resaltar el impacto en los genotipos metabolizadores deficientes causados por las variantes del gen CYP2C1935.
La justificación más importante de las pruebas farmacogenómicas se centra en su capacidad de predicción, ya que estas buscan generar tratamientos más eficaces y seguros, a la vez que aportan alternativas a los pacientes no respondedores y los que padecerán efectos adversos tales como hemorragias, tromboembolia o accidentes cerebrovasculares ocasionados por tratamientos no efectivos36. Se estima que aproximadamente 1 de cada 3 pacientes padecen falta de eficiencia o no responden al fármaco, lo que causa entre el 4 al 7% de los ingresos hospitalarios36. A pesar de toda la información conocida hasta el momento, la aceptación de los estudios farmacogenómicos y su aplicación en la práctica clínica ha sido gradual, dado que diferentes argumentos han impedido su rápida aplicación, desde la falta de laboratorios clínicos disponibles para realizar estas pruebas, hasta la escasez de profesionales de la salud que pueden interpretar los resultados de las pruebas y la farmacología asociada.
ComentariosLa medicina personalizada consiste en proporcionar el tratamiento adecuado que cubra las necesidades individuales de los pacientes. Esta busca mediante el uso de pruebas diagnósticas específicas, que los médicos puedan predecir de qué forma responderá un paciente al tratamiento en algunas patologías. Para generar un tratamiento único es importante comprender las bases moleculares de la enfermedad y así poder desarrollar fármacos más específicos de la mano de herramientas adecuadas de diagnóstico1,2. La necesidad mundial de reducir la incidencia de casos fatales causados por las diferentes reacciones que puede presentar un individuo frente a un fármaco, impulsa la búsqueda de diferentes métodos moleculares que faciliten la prescripción de los mismos donde se reduzca el tiempo, y se aumente la especificidad y sensibilidad en el tratamiento. Por esta razón en la actualidad se registra un incremento en la investigación y el desarrollo de métodos moleculares que puedan ser implementados en la consulta clínica, minimizando la incidencia de eventos adversos en la respuesta a fármacos que ponen en riesgo la vida del paciente37.
No obstante, pese a los múltiples beneficios que implica la incursión de esta nueva ciencia a la práctica clínica, las pruebas de diagnóstico molecular para detectar la susceptibilidad de un paciente a la respuesta de un fármaco siguen siendo ofrecidas por un número exclusivo de laboratorios pioneros en el mundo38. Para el caso puntual de Colombia, algunos laboratorios ofrecen ser intermediaros para la realización de estas puesto que no cuentan con recursos como equipos y personal entrenado, además de los altos costos operativos que hacen que sea más difícil la implementación de éstas en la práctica clínica. Sin embargo, el desarrollo de pruebas moleculares para genes específicos implicados en el fenotipo metabolizador de los individuos, se enfoca en disminuir el tiempo y los costos operativos, lo que a su vez abre nuevas oportunidades para el diagnóstico clínico haciendo de ellas pruebas accesibles y de uso rutinario en la práctica clínica. Con certeza la creación de vínculos interdisciplinarios entre médicos, genetistas y biólogos moleculares, será fundamental para la introducción de las pruebas a la práctica médica local, al igual que para la ejecución, interpretación y evaluación del impacto de estas en la calidad de vida del paciente39.
La investigación en medicina personalizada y la innovación en pruebas para diagnóstico clínico, es un mercado en constante crecimiento y que se diversifica rápidamente. Para 2015 se estimó que se introdujeron alrededor de 4.000 pruebas de diagnóstico nuevas al mercado mundial39. Lo mismo puede decirse para el mercado de la farmacogenómica; de hecho, el 28% de todos los fármacos aprobados por la FDA de los Estados Unidos fueron personalizados para el mismo año39. La creación de medicamentos basados en el perfil genético de los individuos lleva a terapias más específicas y a fármacos más seguros, por lo cual se verá sustancialmente reducida la probabilidad de reacciones adversas, escenario en el cual toma importancia la implementación de la farmacogenómica en la práctica clínica impactando los costos operacionales de la atención médica. Por ejemplo, para el caso de las enfermedades cardiovasculares en Colombia, el costo promedio de los servicios médicos ocasionados por un paciente es aproximadamente 12,8 millones de pesos colombianos, de modo que el costo total de la atención de todos los pacientes con enfermedad de origen cardiovascular es de 13,2 billones de pesos40. En la práctica cardiovascular la identificación de fármacos más efectivos puede llegar a minimizar los costos de tratamiento ya que tener un conocimiento temprano y preciso del tipo de respuesta metabólica que puede llegar a presentar un individuo ante un fármaco, constituye un gran beneficio para la salud del paciente y para el sistema de salud en general41.
ConclusionesCon los avances que en la última década se han logrado en la farmacogenómica, se ha demostrado que es posible establecer una relación con las variables genéticas y las respuestas metabólicas a un determinado fármaco. La repercusión que tiene esta información genética en la práctica clínica es de gran utilidad, ya que la aplicación de este conocimiento está determinando el futuro de las prescripciones de fármacos e incluso hasta el desarrollo de los mismos. La posibilidad de poder catalogar un paciente antes de que este empiece un tratamiento beneficia no solo a este sino al sistema de salud. Sin embargo, la implementación de este tipo de pruebas aun es pobremente explorada por el personal de la salud, lo que representa un obstáculo adicional, sumado a que la prescripción tradicional de ensayo y error sigue siendo el método más empleado. Finalmente, es necesario mencionar que el componente genético es uno de los múltiples factores que influyen en la respuesta farmacológica; por ello se hace necesaria la creación de algoritmos de dosificación, que ayuden a evitar la fluctuación en la dosis de los medicamentos usados para el tratamiento de enfermedades cardiovasculares.
Conflicto de interésNinguno.