La asociación genética más importante en artritis reumatoide (AR) se presenta con algunos alelos del gen HLA DRB1 que codifican el epítope compartido (EC).
ObjetivosAplicar los métodos de clasificación de EC de Gregersen et al., de Vries et al., Raychaudhuri et al., Mattey et al., y Tezenas du Montcel et al., en un grupo de pacientes colombianos con AR, y determinar los alelos HLA DRB1 más frecuentes en esta población.
MétodosDiagnóstico para AR, estudio genético de la región HLA DRB1 por tecnología Luminex® de 50 sujetos AR y 50 sanos. Para análisis comparativos de clasificaciones EC, se aplicaron las pruebas test exacto de Fisher y Chi-cuadrado y se realizaron tablas de conteos para los alelos relacionados con AR. Se estimó la razón de odds para determinar el riesgo entre la presencia de EC y los anticuerpos antipéptidos cíclicos citrulinados (anti-PCC).
ResultadosLos métodos de Gregersen et al. y de Vries et al. fueron adecuados para la caracterización de AR en esta población (p=0,006). Los alelos HLA DRB1 más prevalentes en el grupo AR fueron 14:02, 04:04, 08:02, 04:05 y 10:01. Se encontraron altas frecuencias de los alelos HLA DRB1 07:01, 03:01, 13:02, 01:02 y 12:01 en población sana. Alelos HLA DRB1 con distribución similar en ambas poblaciones fueron: 04:07, 15:01, 11:01, 16:02 y 01:01. Se observó alta frecuencia de individuos EC+ en el grupo AR anti-PCC+ (63,15%); no obstante, sin asociación estadística (OR: 2,4 [0,63-9,01]; p=0,19).
ConclusiónLos métodos de clasificación para EC de Gregersen et al. y de Vries et al. fueron adecuados caracterizando AR en un grupo de población colombiana. Se corroboró equivalencia del 100% entre los alelos de susceptibilidad definidos por de Vries y los alelos asignados como EC según Gregersen et al.
The most important genetic association in rheumatoid arthritis (RA) is presented with some alleles from the HLA-DRB1 gene that encode the shared epitope (SE).
ObjectivesTo apply the SE classification methods of Gregersen, de Vries, Raychaudhuri, Mattey, and Tezenas du Montcel in a group of Colombian patients with RA and determine the most common HLA-DRB1 alleles in the population.
MethodsRA diagnosis, genetic study of the HLA-DRB1 region using Luminex® technology in 50 RA and 50 healthy subjects. For the classification analysis, Fisher's exact test and chi-squared test were applied. Tables were created to count the RA-related alleles. We used odds ratio to determine the risk between the presence of the shared epitope (SE) and anti-cyclic citrullinated peptides (Anti-CCP).
ResultsGregersen et al. and de Vries et al. methods were suitable for the characterization of RA in this population (P=.006). The most prevalent HLA-DRB1 alleles in the RA group were 14:02, 04:04, 08:02, 04:05, and 10:01. High frequencies of the 07:01, 03:01, 13:02, 01:02, and 12:01 HLA-DRB1 alleles were found in the healthy population. HLA-DRB1 alleles with similar distribution in both populations were 04:07, 15:01, 11:01, 16:02, and 01:01. A high frequency of SE+ was observed in Anti-CCP+ individuals (63.15%); however, this was not statistically significant (OR 2.4 [.63–9.01]; P=.19).
ConclusionThe SE classification methods of Gregersen et al. and de Vries et al. were adequate in characterizing RA in a Colombian population group. An equivalence of 100% was verified between the susceptibility alleles defined by de Vries and the alleles assigned as SE according to Gregersen et al..
El locus HLA DRB1 es el factor de riesgo genético con mayor significancia asociado con artritis reumatoide (AR)1,2. De la década de 1970 datan estudios sobre esta relación, en los cuales se observó que el subtipo HLA DR4 tenía una mayor frecuencia en pacientes con AR, comparado con la población control3. Otros alelos HLA DR, por ejemplo, HLA DR14 en nativos americanos4 o HLA DR1 y DR10 en poblaciones caucásicas5, también se relacionan con la enfermedad. El análisis de la secuencia de los alelos HLA DRB1 asociados con AR mostró que la mayoría de los pacientes con AR comparte unas secuencias conservadas de aminoácidos en la tercera región hipervariable (HV3) de la molécula HLA DRβ, las cuales han sido denominadas epítope compartido (EC)6,7. Los alelos que codifican para el EC se asocian con susceptibilidad para AR6–9 y también con la severidad de la enfermedad10,11; adicionalmente, se han asociado con la producción de anticuerpos antipéptidos cíclicos citrulinados (anti-PCC)12,13. Aunque hay acuerdo, en general, acerca de la predisposición de los alelos con EC para AR, no todos conllevan el mismo riesgo5,7,14,15, además hay discrepancias relacionadas con los alelos precisos que determinan esta susceptibilidad12,16.
Se han propuesto gran número de modificaciones con respecto a la clasificación del EC en diferentes poblaciones analizadas, en relación con variaciones en las posiciones de los aminoácidos dentro de la cadena HLA DRβ. El objetivo de este estudio fue determinar los alelos HLA DRB1 en un grupo de individuos colombianos con y sin AR, y correlacionar los métodos de clasificación de EC de Gregersen et al.6, de Vrieset al.5, Raychaudhuri et al.14, Mattey et al.7 y Tezenas du Montcel et al.15 en esta población.
Clasificación Gregersen et al.La clasificación clásica de EC se basa en secuencias conservadas de tres variantes de aminoácidos homólogos en las posiciones 70-74 de la molécula HLA DRβ16,7. Estas secuencias corresponden a los aminoácidos QKRAA (alelos 04:01, 04:11, 04:09), QRRAA (alelos 01:01, 01:02, 01:04, 01:05, 04:04, 04:05, 04:08, 14:02) y RRRAA (alelos 10:01 y 10:02).
Clasificación Tezenas du Montcel et al.Tezenas du Montcel et al.15 sugirieron que el riesgo representado por la presencia de la secuencia RAA en las posiciones 72 a 74 es modulado por los aminoácidos en las posiciones 70 y 71 de la cadena HLA DRβ1. El estudio fue realizado en pacientes franceses con AR, y los alelos se dividieron en dos grupos, de acuerdo con la presencia o ausencia de la secuencia RAA en las posiciones 72-74, representados como alelos S o X, respectivamente. Los alelos S se dividieron subsecuentemente de acuerdo con el aminoácido en la posición 71: S1 para ARAA y ERAA, S2 para KRAA y S3 para RRAA. Los alelos S3 se subdividieron adicionalmente de acuerdo con el aminoácido en la posición 70, de modo tal que S3D correspondió a la secuencia DRRAA y S3P a las secuencias QRRAA o RRRAA. Comparaciones entre alelos mostraron que el riesgo asociado con S1, S3D y X no difirió significativamente y que estos alelos representaron bajo riesgo, por lo cual fueron agrupados como un solo alelo denominado L. Se estableció la siguiente jerarquía de riesgo: significativamente mayor para S2 con respecto a S3P (p<0,002), el cual a su vez fue mayor que el asignado a L. El genotipo de mayor riesgo fue el S2/S3P; este riesgo fue 6,6 veces mayor con respecto al genotipo L/L, seguido por S2/S2, S3P/S3P, S2/L y S3P/L15.
Clasificación De Vries et al.Estudios en población caucásica consideraron el EC según las posiciones de aminoácidos 67, 70, 71, 73 y 74, correspondientes a las secuencias LQKAA (DRB1*04:01 o 04:09), LQRAA (DRB1*01:01, 01:02, 04:04, 04:05, 04:08, 14:02) o LRRAA (DRB1*10:01), y los denominaron alelos de susceptibilidad (S). Adicionalmente, clasificaron los alelos que codifican isoleucina en la posición 67 o ácido aspártico en la posición 70 HLA DRβ1 como protectores (P) y a todos los demás alelos como neutrales (N)5.
Clasificación Mattey et al.En estudios realizados en poblaciones del Reino Unido y España estratificaron los alelos HLA DRB1 en cinco grupos, dependiendo del aminoácido en la posición 70 y si formaban parte o no de la secuencia clásica del EC, y se establecieron las siguientes jerarquías de riesgo: Q70EC+>Q70EC+>R70EC+>R70EC+>D70EC+7.
Clasificación Raychaudhuri et al.En este caso se usaron datos de polimorfismos de nucleótido único (SNP) de genoma completo, a partir de un metaanálisis GWAS de AR para HLA-A, B, C, DPA1, DPB1, DQA1, DQB1 y DRB1 en el complejo mayor de histocompatibilidad (CMH). Los análisis de haplotipos revelaron que los aminoácidos en las posiciones 11 (o 13) 71 y 74 HLA DRβ1 se relacionaron con riesgo para AR, y se definieron 16 haplotipos de riesgo14.
Pacientes y métodosConsideraciones éticasLos pacientes incluidos en el estudio acudieron a consulta en el Hospital Militar Central, ubicado en Bogotá (Colombia), durante el periodo de mayo del 2012 a mayo del 2014, y aprobaron su participación en el estudio firmando un consentimiento informado. Los comités de ética de las instituciones participantes, según los principios de la Asamblea Médica Mundial expuestos en su Declaración de Helsinki, aprobaron la investigación: Universidad Nacional de Colombia (24/10/2013-62), Hospital Militar Central (2014-4337).
Se incluyeron 50 individuos con diagnóstico de AR, según criterios clasificatorios del Colegio Americano de Reumatología de 198717 o del 201018, y 50 individuos sin AR que no cumplieran estos criterios diagnósticos ni presentaran antecedentes familiares de autoinmunidad. Con respecto a la ascendencia, para toda la población que perteneció a esta investigación se tuvo en cuenta que al menos tres líneas generacionales de los individuos participantes hubiesen nacido en territorio colombiano; la muestra se consideró constituida por mestizos colombianos. La población sana fue seleccionada en el mismo hospital, en el sitio de trabajo, o fueron vecinos de los pacientes, con condiciones ambientales y socioeconómicas similares, y se parearon por edad y sexo, excepto por un individuo de sexo masculino en el grupo sin AR.
Criterios de exclusiónLos participantes de este estudio lo fueron también de una investigación alterna (resultados en proceso de publicación), y se tuvieron en cuenta los siguientes criterios de exclusión: infección actual o diagnóstico de neoplasia, presencia de otras enfermedades autoinmunes, tratamiento con antibióticos en los últimos tres meses, tener menos de seis dientes en la boca, tratamiento periodontal u ortodóntico en los últimos seis meses, mujeres en embarazo o lactancia, hábito de fumar actual o pasado.
Anticuerpos antipéptidos cíclicos citrulinados IgG/IgASe utilizó el kit Quanta Lite CCP3.1® IgG/IgA ELISA, según instrucciones del fabricante. El resultado final se obtuvo en unidades ELISA/ml, y se consideró valor negativo <20UI.
Caracterización de alelos HLA DRB1La extracción de ADN se llevó a cabo con el método de Salting Out, kit CorpoGen®. La tpificación de alelos HLA DRB1 se realizó en una plataforma Luminex® 100/200, utilizando el kit Inmucor, Lifecodes® HLA-DRB1 SSO Typing Kit, 628923, Stamford, CT, EE. UU., siguiendo las instrucciones del fabricante; los datos se analizaron empleando el software LifeMatch DNA.
Análisis estadísticoSe usaron los programas SPSS® versión 20.0 y R versión 3.2.2, se aplicaron las pruebas Chi-cuadrado o Fisher, una u otra seleccionada según la cantidad de eventos dentro de las categorías para cada método de clasificación. Se realizaron tablas de conteos para los alelos relacionados con AR, con sanos y con aquellos con similar distribución en la población. Se estimó la razón de odds para determinar el riesgo entre la presencia del EC y los anti-PCC.
ResultadosCaracterísticas clínicas, relación entre anticuerpos antipéptidos cíclicos citrulinados y epítope compartidoLa población de estudio estuvo compuesta por 100 individuos: varones y mujeres colombianos no fumadores, entre 18 y 65 años; 50 con diagnóstico de AR y 50 individuos sanos. La presencia de anti-PCC fue exclusiva de población con AR (p=0,001), con una positividad del 76%. La distribución de la enfermedad con respecto al sexo reflejó la mayor ocurrencia de AR en las mujeres, siendo el 84% de sexo femenino (tabla 1). Se observó alta frecuencia de individuos EC+ (EC+/EC+ y EC+EC−) en el grupo AR anti-PCC+ (63,15% de la población anti-PCC), teniendo en cuenta la categorización clásica de EC (según Gregersen et al.). Las diferencias para los grupos anti-PCC+ y anti-PCC− relacionadas con la presencia del EC no fueron estadísticamente significativas (OR: 2,4 [0,63-9,01]; p=0,19) (tabla 2).
Características de la población con AR y sanos
| ARn (%)50 (100,00) | Sanosn (%)50 (100,00) | Valor de p | |
|---|---|---|---|
| Edad (promedio en años) | 47,83 | 49,06 | NA |
| Mujeres | 42 (84,00%) | 41 (82,00%) | NA |
| Anti-PCC | 38 (76,00%) | 0 | 0,001* |
Anti-PCC: anticuerpos frente a péptidos cíclicos citrulinados; AR: artritis reumatoide; NA: no aplicable (individuos pareados por edad y sexo, excepto por un individuo de sexo masculino en el grupo sin AR).
Relación entre anti-PCC y EC según Gregersen en individuos con AR
| EC | Anti-PCC+n (%)38 (76) | Anti-PCC−n (%)12 (24) | OR (IC 95%) | Valor de p |
|---|---|---|---|---|
| EC+ (%) | 24 (63,15) | 5 (41,66) | 2,4 (0,63-9,01) | 0,19 |
| EC− (%) | 14 (36,84) | 7 (58,33) |
Datos disponibles para 50 individuos AR.
Anti-PCC: anticuerpos frente a péptidos cíclicos citrulinados; AR: artritis reumatoide; EC: epítope compartido; EC+=EC+/EC+ y EC+/EC−. EC−=EC−/EC−; OR: razón de odds.
Puesto que los resultados se obtuvieron por tecnología Luminex® (de mediana resolución), para los análisis se incluyeron exclusivamente datos no ambiguos, según las secuencias de aminoácidos establecidas en cada uno de los métodos de clasificación, de la siguiente manera: de acuerdo con la clasificación clásica de Gregersen et al., se incluyó información de 97 muestras (50 AR y 47 sanos; 194 alelos). En las clasificaciones de Vries y Mattey et al. los resultados sin ambigüedades comprendieron 50 individuos AR y 45 individuos sanos (190 alelos). Para el análisis según Tezenas du Montcel et al., se contó con 47 individuos AR y 47 individuos sanos (188 alelos). Para el análisis alélico según Raychaudhuri et al., se obtuvieron sin ambigüedad 97 alelos en la población AR y 92 alelos en la población sana. Entre el sistema de clasificación para EC propuesto por Gregersen et al. y el sistema de Vries et al., no se observaron diferencias con respecto a los alelos categorizados como EC según Gregersen et al., definidos por de Vries et al. como alelos S. Los sistemas de clasificación Gregersen et al. y de Vries et al. fueron apropiados para la diferenciación entre sanos y AR en esta población (p=0,006); los otros sistemas de clasificación no obtuvieron significación estadística. Se pudo evidenciar cómo la clasificación de Raychaudhuri et al. impidió hacer una diferenciación de población AR/sanos; con respecto al sistema Tezenas Du Montcel et al., pese a no tener resultados estadísticamente significativos, se observó una mayor frecuencia de alelos L en la población sin AR (65,95%) (tabla 3). De acuerdo con el sistema clasificatorio de Mattey et al., con respecto a la relación entre los alelos Q70EC+DR4 y Q70EC+DR1 para la susceptibilidad de AR, encontramos mayor frecuencia del haplotipo DR4 (datos no mostrados).
Distribución de epítope compartido según métodos de Gregersen et al., de Vrieset al., Matteyet al., Tezenas du Montcel et al. y Raychaudhuri et al., en individuos colombianos sanos y con artritis reumatoide
| Clasificación HLA DRβ 1 | AR | Sanos | Valor de p | ||
|---|---|---|---|---|---|
| n (%) | Frecuencia alélica AR | n (%) | Frecuencia alélica sanos | ||
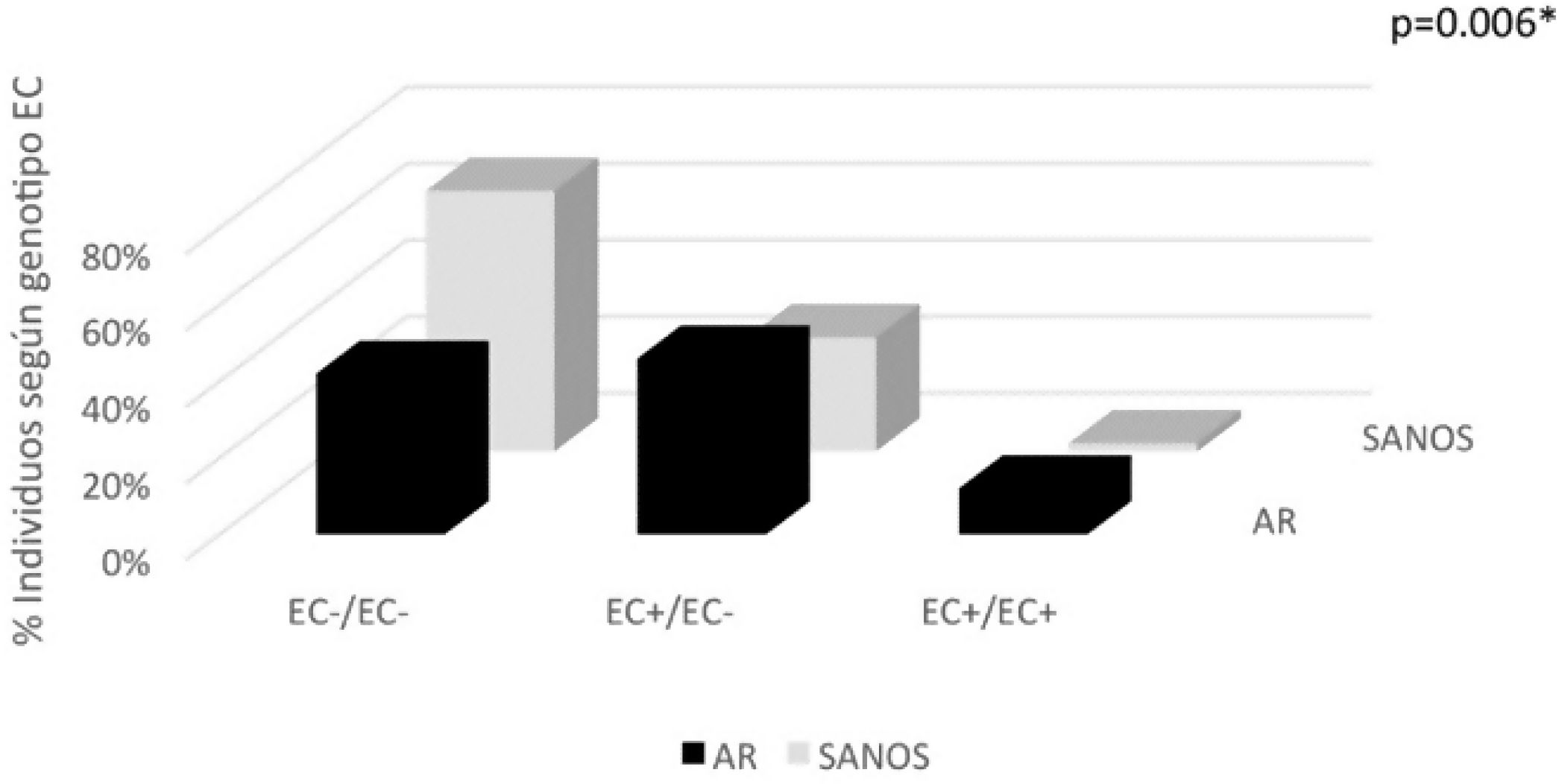
| Genotipo Gregersen | 50 (100) | 100 | 47 (100) | 94 | *0,006 |
| EC−/EC− | 21 (42) | 42 | 32 (68,1) | 64 | |
| EC+/EC− | 23 (46) | 23(EC+)/23(EC−) | 14 (29,8) | 14(EC+)/14(EC−) | |
| EC+/EC+ | 6 (12) | 12 | 1 (2,1) | 2 | |
| ≥1 EC+ | 29 (58) | 35 | 15 (32) | 16 | |
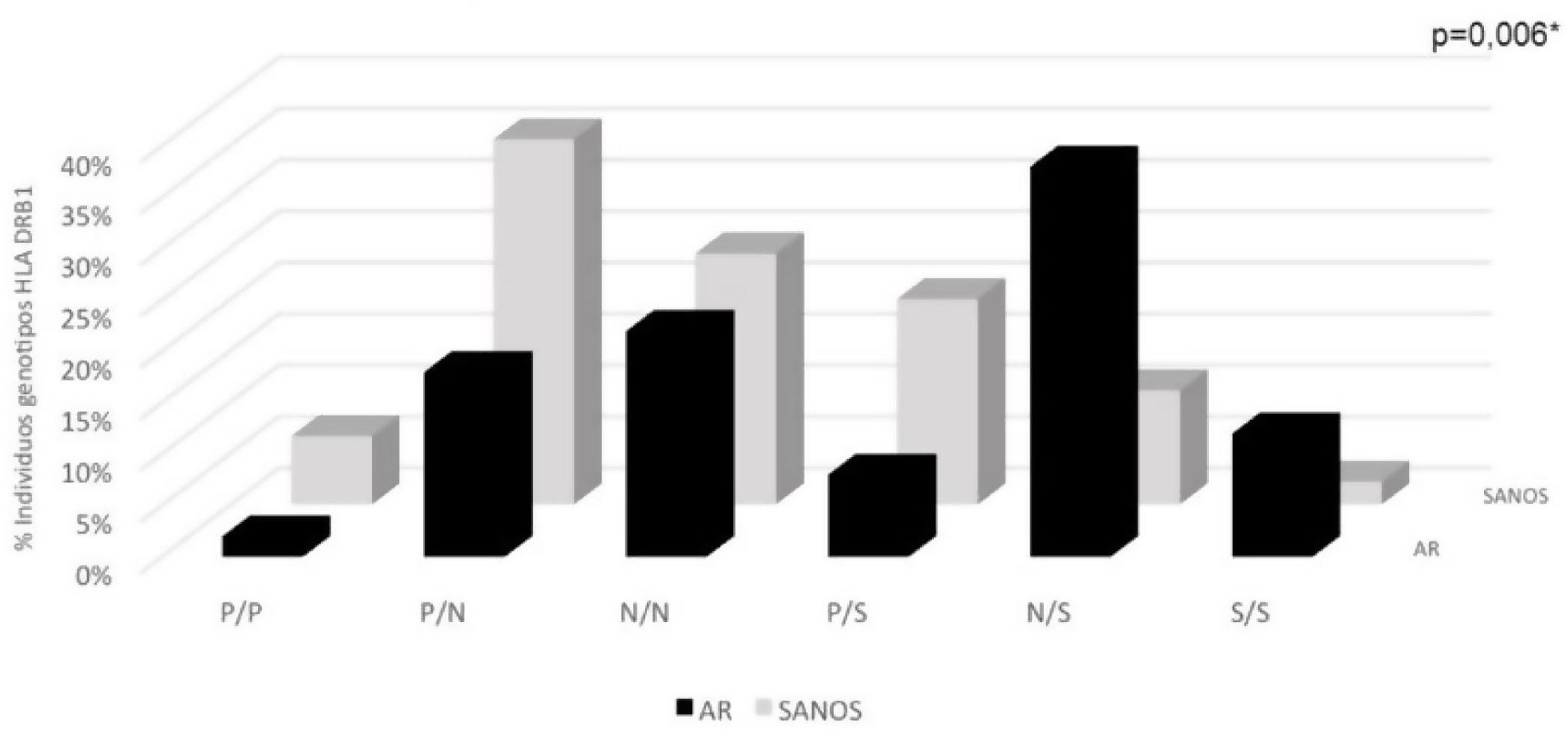
| Genotipo de Vries | 50 (100) | 100 | 45 (100) | 90 | *0,006 |
| P/P | 1 (2,00) | 2 | 3 (6,66) | 6 | |
| P/N | 9 (18,00) | 9(P)/9(N) | 16 (35,55) | 16(P)/16(N) | |
| N/N | 11 (22,00) | 22 | 11 (24,44) | 22 | |
| P/S | 4 (8,00) | 4(P)/4(S) | 9 (20,02) | 9(P)/9(S) | |
| N/S | 19 (38,00) | 19(N)/19(S) | 5 (11,11) | 5(N)/5(S) | |
| S/S | 6 (12,00) | 12 | 1 (2,22) | 2 | |
| Genotipo Mattey | 50 (100) | 100 | 45 (100) | 90 | 0,62 |
| D70EC−/D70EC− | 7 (14,00) | 14 | 12 (26,70) | 24 | |
| Q70EC−/D70EC− | 10 (20,00) | 10(Q70EC)/10(D70EC−) | 9 (20,00) | 9 (Q70EC−)/9(D70EC−) | |
| Q70EC−/Q70EC− | 4 (8,00) | 8 | 6 (13,33) | 12 | |
| Q70EC+/D70EC− | 9 (18,00) | 9(Q70EC+)/9(D70EC−) | 6 (13,33) | 6(Q70EC+)/6(D70EC−) | |
| Q70EC+/Q70EC− | 11 (22,00) | 11(Q70EC+)/11(Q70EC−) | 7 (15,55) | 7(Q70EC+)/7(Q70EC−) | |
| Q70EC+/Q70EC+ | 3 (6,00) | 6 | 1 (2,22) | 2 | |
| Otros | 6 (12,00) | 12 | 4 (8,88) | 8 | |
| Genotipo Tezenas du Montcel | 47 (100) | 94 | 47 (100) | 94 | 0,19 |
| L/L | 21 (44,68) | 42 | 31 (65,95) | 62 | |
| S3P/L | 20 (42,55) | 20(S3P)/20(L) | 12 (25,53) | 12(S3P)/12(L) | |
| S2/L | 2 (4,27) | 2(S2)/2(L) | 3 (6,38) | 3(S2)/3(L) | |
| S3P/S3P | 3 (6,38) | 6 | 1 (2,12) | 2 | |
| S2/S2 | 0 | 0 | 0 | 0 | |
| S2/S3P | 1 (2,12) | 1(S2)/1(S3P) | 0 | 0 | |
| Clasificaciónde riesgo Raychaudhuri | AR 97 alelos | Sanos 92 alelos | 0,18 | ||
|---|---|---|---|---|---|
| n | % | n | % | ||
| (1) SSEA | 3 | 3,09 | 7 | 7,62 | |
| (2) SSKR | 3 | 3,09 | 7 | 7,62 | |
| (3) SGRL | 11 | 11,34 | 7 | 7,62 | |
| (4) LFEA | 2 | 2,06 | 3 | 3,10 | |
| (5) SSRE | 1 | 1,03 | 1 | 1,10 | |
| (6) SGRA/SSRA | 19 | 19,58 | 11 | 11,95 | |
| (7) GYRQ | 2 | 2,06 | 8 | 8,70 | |
| (8) PRAA | 6 | 6,18 | 8 | 8,70 | |
| (9) SSKA | 1 | 1,03 | 0 | 0,00 | |
| (10) VHEA | 1 | 1,03 | 1 | 1,08 | |
| (11) DFRE | 2 | 2,06 | 2 | 2,19 | |
| (12) VHRE | 18 | 18,57 | 17 | 18,50 | |
| (13) PRRA | 7 | 7,22 | 6 | 6,56 | |
| (14) LFRA | 4 | 4,14 | 7 | 7,62 | |
| (15) VFRA/VHRA | 15 | 15,46 | 5 | 5,45 | |
| (16) VHKA | 2 | 2,06 | 2 | 2,19 | |
AR: artritis reumatoide.
* p < 0,05 significativa por las pruebas Test exacto de Fisher o Chi cuadrado.
Con referencia a la clasificación clásica EC (Gregersen et al.), en el grupo con AR el mayor porcentaje (46%) tuvo genotipo EC+/EC−, seguido por un 42% de negativos para EC, en tanto que el 12% presentó genotipo EC+/EC+. En total, en la población AR el 58% tuvo al menos un alelo positivo para EC (frecuencia alélica de EC+ en AR=35). En contraste, la población sana en su mayoría fue negativa para EC (68,1%); el 29,8% tuvo genotipo EC+/EC−, siendo un sujeto sano EC+/EC+; el 32% de la población sana tuvo al menos un alelo positivo para EC (frecuencia alélica de EC+ en sanos=16); estas diferencias fueron estadísticamente significativas (p=0,006) (tabla 3, fig. 1).
La clasificación de Vries et al. según los genotipos S/S, N/S, P/S, N/N, P/N y P/P evidenció diferencias entre población AR y población sana: el 12% de la población AR tuvo dos alelos de susceptibilidad, comparado con el 2% en la población sana. El 6,66% de la población sana tuvo dos alelos protectores, mientras que de la población AR los tuvo solo el 2%. El genotipo N/S tuvo una mayor frecuencia en población con AR, a su vez, el genotipo P/N fue más frecuente en población sin AR; la frecuencia del genotipo N/N fue similar en ambos grupos poblacionales. Estos hallazgos fueron estadísticamente significativos (p=0,006) (tabla 3, fig. 2).
Distribución de genotipos HLA DRB1 en AR y sanos según clasificación de Vries et al. Aminoácidos en posiciones HLA DRβ1 67, 70, 71, 73 y 74
genotipos P/P: protector/protector; P/N: protector/neutro; N/N: neutro/neutro; P/S: protector/susceptibilidad; N/S: neutro/susceptibilidad: S/S susceptibilidad/susceptibilidad.
*p<0,05 significativa por las pruebas test exacto de Fisher y Chi-cuadrado.
Para la tipificación de los alelos, no se encontraron ambigüedades en 188 alelos (97 AR y 91 sanos) (tabla 4). Los alelos más prevalentes en el grupo AR con respecto a la población sana fueron 14:02, 04:04, 08:02, 04:05 y 10:01. Se encontraron altas frecuencias de los alelos 07:01, 03:01, 13:02, 01:02 y 12:01 en población sana, en comparación con el grupo AR. Los alelos que acompañaron el genotipo en los individuos sanos portadores del alelo 01:02 (cinco individuos) fueron EC− (clasificación Gregersen et al.), (01:02/04:07, 01:02/03:01, 01:02/16:02, 01:02/13:02, 01:02/15:01). El 50% de los individuos que portaron el alelo 08:02 tuvo en su genotipo el alelo 14:02 (08:02/15:01, 08:02/04:07, 08:02/14:02, 08:02/14:02, 08:02/08:02, 08:02/14:02).
Frecuencia de alelos HLA DRB1 en individuos colombianos con AR y sanos
| Alelo | AR | Sanos |
|---|---|---|
| Alelos HLA DRB1 más frecuentes en individuos con AR | ||
| 14:02 | 10 | 2 |
| 04:04 | 8 | 2 |
| 08:02 | 7 | 4 |
| 04:05 | 4 | 1 |
| 10:01 | 4 | 1 |
| 04:03 | 3 | 1 |
| 16:01 | 3 | 1 |
| 04:11 | 2 | 0 |
| 11:02 | 1 | 0 |
| 12:07 | 1 | 0 |
| 13:03 | 1 | 0 |
| Alelos HLA DRB1 más frecuentes en indiviuos sanos | ||
| 07:01 | 2 | 8 |
| 03:01 | 3 | 6 |
| 13:02 | 0 | 5 |
| 01:02 | 1 | 5 |
| 12:01 | 1 | 4 |
| 03:02 | 0 | 1 |
| 15:03 | 0 | 1 |
| Alelos HLA DRB1 con similar frecuencia en individuos con AR y sanos | ||
| 04:07 | 12 | 16 |
| 15:01 | 5 | 5 |
| 11:01 | 5 | 3 |
| 16:02 | 4 | 5 |
| 01:01 | 3 | 2 |
| 01:03 | 2 | 3 |
| 04:01 | 2 | 2 |
| 08:04 | 2 | 2 |
| 09:01 | 2 | 2 |
| 11:04 | 2 | 2 |
| 13:01 | 2 | 2 |
| 08:01 | 2 | 1 |
| 15:02 | 1 | 2 |
| 04:02 | 1 | 1 |
| 14:01 | 1 | 1 |
AR: artritis reumatoide.
Los alelos 04:07, 15:01, 11:01, 16:02 y 01:01 tuvieron representación similar en población AR y sanos (tabla 4).
DiscusiónEste es el primer estudio que compara diferentes métodos de clasificación de alelos HLA DRB1 relacionados con AR en población colombiana. Muchos análisis se han enfocado en la región de aminoácidos referida como EC y su influencia en la patogénesis de AR, incluidas la susceptibilidad, la severidad y la respuesta al tratamiento. Se han propuesto varias modificaciones a la propuesta inicial, restringida a los aminoácidos 70 a 74 de la tercera región hipervariable HLA DRB119. De los cinco sistemas de clasificación HLA DRB1 utilizados en este estudio, los de Gregersen et al. y de Vries et al. fueron adecuados para la caracterización AR en esta población. La clasificación clásica de EC (Gregersen et al.)6 fue efectiva, sencilla y significativa en la diferenciación entre AR y sanos. Los alelos relacionados con EC según este sistema han sido un referente histórico para una adecuada determinación de riesgo para AR relacionado con HLA DR en la mayoría de los estudios y para diversos grupos étnicos, y se han empleado antes de las emergentes propuestas de los nuevos sistemas de clasificación8–10,20,21.
La investigación de Nick de Vries et al.5 se realizó en una cohorte de sujetos caucásicos holandeses y fue adecuada para la asignación de riesgo de AR en la población de este estudio; nuestros hallazgos con respecto al sistema de Vries et al. son los primeros reportados en población mestiza. Este sistema fue aplicado por Morgan et al.16 en caucásicos del Reino Unido y se determinó un efecto protector similar de los alelos N y P, que fueron considerados parte de un mismo grupo. Asimismo, en el análisis de los genotipos se negó la hipótesis de protección contra AR propuesta por alelos P.
De manera análoga a los hallazgos de Tezenas du Montcel et al.15, nuestros resultados no demostraron relación de las posiciones 72 a 74 con riesgo para AR; sin embargo, no hallamos relación de la enfermedad con los genotipos de riesgo. El método Tezenas du Montcel et al. ha sido adecuado en otras poblaciones caucásicas según lo encontrado por Morgan et al.16 al asociar alelos S2 y S3P con RA en residentes del Reino Unido, pero no en este grupo de población colombiana.
El método propuesto por Mattey et al.7 involucró población española y del Reino Unido. En nuestra muestra, la clasificación de Mattey no fue adecuada en la caracterización de población AR. De acuerdo con los resultados de esta investigación, tampoco fue posible confirmar el efecto dominante protector del aminoácido D70 asociado con EC−, relacionado por los autores cuando estuvo incorporado al genotipo Q70EC+/D70EC. Encontramos mayor frecuencia del haplotipo DR4 en individuos con AR, de manera similar a los hallazgos de los autores7, quienes reportaron esta relación mayor para el Reino Unido con respecto a España; sin embargo, entre los españoles había una alta frecuencia de Q70EC+DR4. Morgan et al.16 encontraron adecuado el sistema de clasificación Mattey cuando fue aplicado en caucásicos residentes en el Reino Unido.
La clasificación de Raychaudhuri et al. no fue adecuada en la diferenciación de población AR/sanos en nuestra esta población, lo cual podría deberse al que existan 16 grupos de haplotipos para la definición de riesgo de AR, en tanto que en este estudio había un número poblacional limitado. Asimismo, el alelo HLA-DRB1*14:02, positivo para EC+, se relacionó con AR en esta y otras investigaciones en diversos grupos poblacionales6,7,15; sin embargo, Raychaudhuri et al. lo consideraron de riesgo bajo.
Otro punto importante que analizar es la frecuencia y el significado de los alelos HLA DRB1 en las poblaciones AR y sanos. Dentro de los alelos más prevalentes en el grupo AR estuvieron HLA-DRB1*14:02, 04:04, 04:05 y 10:01, todos portadores del EC22. De la misma manera, Spinel et al.8 encontraron el alelo HLA-DRB1*14:02 dos veces más frecuente en AR que en controles en población colombiana; el alelo HLA-DRB1*04:04 ha sido característico en caucásicos, asociado con riesgo para la enfermedad9,23. Se ha reportado, riesgo para AR con la presencia del alelo HLA-DRB1*10:01 en diversas poblaciones5,14,15, que fue igualmente encontrado con alta frecuencia en población AR colombiana, según los resultados de este estudio. Contrariamente, Morgan et al.16 en su estudio con población caucásica no encontraron asociación entre el alelo HLA-DRB1*10:01 y la enfermedad16. Según Mattey et al., este alelo se clasifica como riesgo intermedio7.
Gilbert et al.24 relacionaron el alelo HLA-DRB1*08:02 con un efecto protector (pero no significativo) para AR, en tanto que de Vries et al.5 lo consideraron de efecto neutral. Aunque en la actual investigación el alelo DRB1*08:02 tuvo una mayor frecuencia en pacientes AR que en pacientes sanos, el 50% de los pacientes que tuvieron este alelo y AR portó en su genotipo el alelo DRB1*14:02, lo cual pudo influir en una mayor representación en población con AR, siendo el alelo DRB1*08:02 característico de poblaciones amerindias colombianas25.
Se encontraron altas frecuencias de los alelos HLA-DRB1*07:01, 03:01, 01:02 y 13:02 en población sana, comparado con el grupo AR. Los alelos HLA-DRB1*07:01, 03:01 y 13:02 son EC−, a diferencia del alelo 01:02, el cual es EC+. El efecto protector del alelo HLA-DRB1*07:01 fue confirmado por de Vries et al.5 y Raychaudhuri et al.14, en tanto que el alelo HLA-DRB1*01:02, así como el alelo HLA-DRB1*01:01, se han relacionado con susceptibilidad para AR en diversos grupos poblacionales, codifican para EC y se han asociado con parte del riesgo genético más importante en el desarrollo de la enfermedad6,7,15. En esta población, de manera particular, el alelo HLA-DRB1*01:02 tuvo más representatividad en población sana, mientras que el alelo HLA-DRB1*01:01, característico de riesgo en diferentes poblaciones, presentó similar frecuencia en AR y en sanos. La población sana portadora del alelo HLA-DRB1*01:02, sin embargo, no presentó en su genotipo otro alelo de susceptibilidad.
Uno de los individuos sanos portó el genotipo HLA-DRB1*01:02/13:02; siendo el alelo HLA-DRB1*13:02 exclusivo de población sana en este estudio. El efecto protector del alelo HLA-DRB1*13:02 ha sido confirmado en varios estudios, como el de Gibert et al.24, quienes asociaron al alelo HLA-DRB1*13:02 con protección contra AR en población francesa, debido a la carga negativa en el bolsillo P4; los autores reportaron este efecto pese a la relación con alelos de riesgo dentro del genotipo. Otro individuo sano positivo para el alelo HLA-DRB1*01:02 presentó en su genotipo el alelo HLA-DRB1*15:01. En el análisis de la relación del alelo HLA-DRB1*15:01, De Vries et al.5 asociaron este alelo con efectos protectores significativos, atribuidos a la presencia de isoleucina en la posición 67 HLA DRβ1. Adicionalmente, otro genotipo presente en población sana con el alelo HLA-DRB1*01:02 fue acompañado por el alelo HLA-DRB1*03:01; entre los individuos no AR de este estudio hubo alta prevalencia de este último alelo, el cual comúnmente se ha considerado protector7,14,15. Contrariamente a ello, de Vries et al. asociaron el alelo HLA-DRB1*03:01 con riesgo para AR en población caucásica5.
Los alelos HLA-DRB1*04:07, 15:01 y 16:02 tuvieron frecuencias similares en población sana y AR. El alelo HLA-DRB1*04:07 (EC−) fue de «efecto neutral» según de Vries et al.5; así mismo lo clasificó el modelo Revirón et al., debido a la carga eléctrica positiva en el bolsillo P426, lo que coincide con los hallazgos sugeridos en el actual estudio5. Raychaudhuri et al.14, por su parte, lo clasificaron en la categoría de riesgo. El alelo HLA-DRB1*16:02 es negativo para EC de acuerdo con la clasificación clásica de Gregersen6, en tanto que se clasifica como neutro de acuerdo con de Vries et al.5 y el modelo Revirón et al.26, de manera similar a nuestra investigación, y es categorizado como de bajo riesgo por los demás autores7,15. Con respecto al alelo HLA-DRB1*15:01, observamos la misma frecuencia en población sana y AR, con tendencia a ser un alelo neutral, clasificación igualmente asignada por Raychaudhuri et al. (OR=1); no obstante, de Vries et al.5 lo reportaron como un alelo protector.
Con respecto a la presencia de anti-PCC, esta fue exclusiva de población con AR en esta investigación; las pruebas positivas anti-PCC predicen el desarrollo de AR, con frecuencia años antes de la confirmación clínica de la enfermedad18. Existe evidencia de la alta afinidad en la interacción entre los péptidos citrulinados y las moléculas que comparten el EC22. En este estudio se observó una tendencia a la presencia de al menos un alelo EC+ en la producción de anti-PCC (63% de los sujetos anti-PCC con EC+); no obstante, hubo un porcentaje representativo de individuos AR anti-PCC+ negativos para EC, de manera similar a los hallazgos de Balsa et al.9 en su estudio en población española.
La posibilidad de generar anti-PCC por alelos diferentes al EC puede deberse en parte a que aun cuando los alelos EC+ se relacionan con proteínas HLA DRβ1 con carga altamente positiva en el bolsillo P4, los péptidos citrulinados también pueden unirse a otras secuencias de anclaje de péptidos, más allá de aquellas que portan la secuencia del EC27. Adicionalmente, otros genes fuera del CMH se han asociado con la autoinmunidad inducida por citrulina. Morgan et al.28 encontraron el gen PTPN22 asociado fundamentalmente con anti-PCC; de la misma manera, la presentación de péptidos citrulinados se ha identificado con alelos HLA DQ29,30.
Dentro de las fortalezas de la actual investigación, nuestros hallazgos con respecto a métodos de clasificación de EC son los primeros reportados en población mestiza y, adicionalmente, se aporta información relacionada con los alelos de mayor frecuencia en individuos colombianos con AR. Consideramos que las debilidades asociadas con la investigación se debieron principalmente a la reducción en más de un 30% del número de muestra, debido a que se tuvo en cuenta el hábito de fumar actual o en el pasado, siendo una variable que se debe controlar en el modelo de la AR y el EC.
ConclusionesNuestros hallazgos demuestran que mientras las clasificaciones alternativas de EC parecen ser útiles en las poblaciones en las cuales han sido inicialmente probadas, no son completamente aplicables a todos los grupos poblacionales; existe una diversidad metodológica entre los estudios para la determinación de los alelos HLA DRB1, los cuales representan además un sistema altamente polimórfico. Los sistemas de Gregersen et al. y de Vries et al. fueron adecuados para la caracterización de AR en este grupo de población colombiana; nuestros resultados tienen relevancia clínica, teniendo en cuenta las diferencias genéticas de nuestra población en relación con los hallazgos en otras latitudes. De igual forma, esta investigación aporta información relacionada con los alelos de mayor frecuencia en individuos colombianos que presentan esta patología; lo anterior, en el contexto de múltiples factores de riesgo, no solo medioambientales sino también genéticos. Finalmente, consideramos que nuestros resultados podrían ser herramientas de apoyo clínico complementarias, útiles para estratificar el riesgo individual en nuestra población y en individuos con antecedentes familiares de AR; no obstante, se considera pertinente la reproducción del actual estudio en un grupo poblacional mayor, así como en otras poblaciones mestizas en las cuales aún no han sido probados.
FinanciaciónEste estudio fue apoyado por la División de Investigación de Bogotá, Universidad Nacional de Colombia (Código Hermes: 20166), el Departamento Administrativo de Ciencia, Tecnología e Innovación Colciencias (N.o 130854531734-2011), Hospital Militar Central (2014-4337), Universidad El Bosque (UB-293-2011), Bogotá, Colombia.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Agradecemos al Dr. Víctor Hidalgo por los análisis estadísticos, al Dr. Wilson Bautista por sus aportes y la orientación en la redacción desde el área de reumatología, al Dr. Mauricio Rodríguez por el enfoque metodológico, a la Universidad el Bosque y al Dr. Rafael Valle por facilitar los espacios para el desarrollo del proyecto en el Servicio de Reumatología del Hospital Militar Central.