La variabilidad genética individual favorece que la capacidad de respuesta y toxicidad a los fármacos sea diferente en cada persona. En la artritis reumatoide se reportan índices de respuesta a los medicamentos etanercept, infliximab, adalimumab y metotrexato cercanos al 60%. Esta variabilidad puede explicarse por polimorfismos genéticos característicos de cada paciente.
ObjetivoIdentificar polimorfismos genéticos reportados en artículos científicos que pueden afectar la farmacocinética y la farmacodinámica de etanercept, infliximab, adalimumab y metotrexato, y su respuesta en pacientes con artritis reumatoide.
Materiales y métodoSe realizó una búsqueda sistemática en PubMed/Medline, con los términos clave: «rheumatoid arthritis» and «pharmacogenomic» and «polymorphisms» and «metotrexato» and «infliximab» and «adalimumab» and «etanercept» obteniendo 164 artículos, 117 no duplicados y 19 artículos que cumplieron los criterios de inclusión.
ResultadosDe los 19 artículos, 2 reportaron polimorfismos que afectan la farmacocinética de infliximab, adalimumab, etanercept y metotrexato, y 17, la farmacodinámica. En los 19 artículos se identificaron 23 polimorfismos de relevancia clínica en población europea, japonesa, jordana e india.
ConclusionesSe identifican 23 polimorfismos de relevancia clínica, los cuales podrían ser el soporte para el diseño de un test de secuenciación específica en pacientes con artritis reumatoide, en los que se considere la utilización de infliximab, adalimumab, etanercept o metotrexato. La utilidad práctica de este tipo de estrategia requiere ser evidencia en estudios clínicos específicos, relacionados con una prescripción orientada por test genéticos y personalizada, y su efecto sobre la efectividad y seguridad de la farmacoterapia con estos medicamentos.
Individual genetic variability favours the capacity of response and toxicity to the drugs is different in each person. Rheumatoid arthritis reported rates of response to the drugs etanercept, infliximab, adalimumab and methotrexate is close to 60%. This variability can be explained by genetic polymorphisms characteristic of each patient.
ObjectiveTo identify genetic polymorphisms reported in scientific articles that may affect the pharmacokinetics and pharmacodynamics of etanercept, infliximab, adalimumab, and methotrexate, and their response in patients with rheumatoid arthritis.
Materials and methodA systematic search was performed in PubMed and Medline, with the key terms: “rheumatoid arthritis” and “pharmacogenomic” and “polymorphisms” and “methotrexate” and “infliximab” and “adalimumab” and “etanercept”, obtaining 164 articles, 117 non-duplicates, and 19 articles that met the inclusion criteria.
ResultsOf the 19 articles, 2 reported polymorphisms affecting the pharmacokinetics of infliximab, adalimumab, etanercept, methotrexate, and 17, pharmacodynamics. In the 19 articles, 23 polymorphisms of clinical relevance were identified in European, Japanese, Jordanian, and Indian populations.
ConclusionsA total of 23 polymorphisms with clinical relevance were identified, which could be the basis for the design of a specific test sequencing in rheumatoid arthritis patients being considered for treatment with infliximab, adalimumab, etanercept, or methotrexate. The practical usefulness of this strategy requires evidence in specific clinical studies, associated with a targeted and personalised genetic test, and its effect on the effectiveness and safety of drug therapy with these drugs prescription.
Los seres humanos presentan variación genética asociada a diferencias en su secuencia de ADN, lo que conlleva que la capacidad de respuesta a los efectos terapéuticos y tóxicos a los fármacos pueda ser diferente en cada individuo1,2. La disciplina que estudia dicha variabilidad genética, a nivel individual, en respuesta a un fármaco es la farmacogenética; por su parte, la que, además, incluye las variables ambientales en la misma es la farmacogenómica3-5. Estas disciplinas tienen utilidad en la práctica clínica para predecir la respuesta individual de los pacientes a los diferentes fármacos, en especial para el tratamiento de las enfermedades autoinmunes6. La artritis reumatoide (AR) es una enfermedad autoinmune, heterogénea, crónica, que produce la inflamación en la membrana sinovial, el cartílago y el hueso; con el tiempo genera deformidad, discapacidad y deterioro de la calidad de vida7,8. Entre los medicamentos utilizados en su tratamiento se encuentran los inhibidores del factor de necrosis tumoral alfa (TNF-α) etanercept, infliximab, adalimumab, los cuales reportan un índice de respuesta del 40%, y el metotrexato (MTX), del 50%8. Esta variabilidad puede presentarse por polimorfismos genéticos de un solo nucleótido que afectan la farmacocinética y la farmacodinámica de estos medicamentos.
Una revisión sistemática del International Journal of Rheumatology en 2013 reporta que aproximadamente el 50% de los pacientes con AR en Europa interrumpen su inhibidor de TNF-α durante los primeros 5años debido a problemas de efectividad o de seguridad del respectivo medicamento9. De forma similar, un estudio en Boston, el Brigham and Women's Hospital Rheumatoid Arthritis Sequential Study (BRASS), reporta que el 42% de los pacientes con AR informaron del abandono de su tratamiento anti-TNF-α10. En este contexto de un elevado número de pacientes que suspenden el tratamiento con anti-TNF-α, la generación de información que mejore la capacidad de predecir los pacientes que responderán a las terapias específicas sería una contribución significativa en el manejo adecuado de la AR.
Los problemas de efectividad y seguridad mencionados podrían estar asociados a la variabilidad genética de cada paciente, incluyendo las variables ambientales, las cuales se expresan por polimorfismos genéticos. De forma general, un polimorfismo genético es una variación en una secuencia determinada de ADN presente en más del 1% de los individuos de una población11. Según el Instituto Canario de Investigación del Cáncer «se clasifican en: polimorfismos en el número de repeticiones en tándem (VNTR), polimorfismos de longitud de fragmentos de restricción (RFLP) y polimorfismos de nucleótido simple (SNP, por sus iniciales en inglés)»12. En este contexto, la variabilidad genética se asocia en un 90% a los SNP, debido a la actividad de los genes dependientes de enzimas implicadas en la farmacocinética o la farmacodinámica de los fármacos utilizados en el tratamiento de la AR, afectando así su eficacia y su seguridad13.
La AR constituye un importante problema de salud pública; en los últimos años se han logrado mejores resultados de salud con la incorporación de medicamentos modificadores de la enfermedad sintéticos y biológicos. Sin embargo, se reportan problemas de variabilidad en la respuesta, lo que conlleva inefectividad y reacciones adversas en el 30-40% de los pacientes14. En este sentido, la farmacogenómica, a través del estudio de variantes genéticas de proteínas involucradas en la farmacocinética y la farmacodinamia de los medicamentos, se convierte en una forma de maximizar la eficacia y la seguridad de la farmacoterapia. En este aspecto, la farmacogenómica favorece una comprensión mejor de los mecanismos biológicos y, con ello, el desarrollo de modelos clínicos que predigan el fármaco antirreumático con mayor beneficio en cada paciente.
Se estima que, a los 10años de evolución de la AR, casi el 80% de los pacientes presentan algún grado de limitación. El pronóstico es incierto y las lesiones, que se producen en los primeros años de enfermedad, pueden continuar a pesar de la mejoría clínica, ya que la patogénesis de estas lesiones puede diferir de la simple inflamación aguda articular15.
En Colombia se han encontrado factores de riesgo comunes con respecto al desarrollo de la AR, tales como el tabaquismo, el sexo femenino, la presencia del HLA DRB1 y el nivel educativo16.
Datos epidemiológicosEn Canadá y Estados Unidos la prevalencia oscila entre el 0,8 y el 1,1%17,18. De forma similar, en Europa los datos de prevalencia varían entre el 0,5 y el 1%19,20. En Latinoamérica se ha reportado en el 0,45 y el 0,9%21-23. Por su parte, diversos estudios muestran que la incidencia de la AR tiende a disminuir; así, entre 1955 y 1994 la incidencia se habría reducido en un 50%24. En Europa, las tasas de incidencia informadas de AR varían entre 0,009-0,045%; en Norteamérica varían entre 0,024-0,075% en población caucásica y entre 0,09-0,89% en población india americana. Aunque no está claro, se han propuesto varios factores que permitan explicar las diferencias en la incidencia, entre los cuales se encuentran factores genéticos, ambientales y culturales25.
Etiología y patogenia de la artritis reumatoideLa AR se caracteriza por la presencia de factor reumatoide y los anticuerpos anti-péptidos citrulinados. Esto se observa en la fase inicial de la AR e implica la activación de ambas célulasT yB. Las citoquinas proinflamatorias, tales como TNF, IL-1 e IL-17, estimulan la inflamación y la degradación de hueso y cartílago. Sin embargo, no se produce una disminución en el número de células Treg, lo que podría contribuir a la fisiopatología de la enfermedad26.
Susceptibilidad genéticaSe estima que hasta un 66% de los pacientes con AR presentan riesgo genético, el cual se ha asociado con la presencia de ciertos alelos del complejo mayor de histocompatibilidad, especialmente con el tipo HLA-DR4. En este sentido, se acepta que la enfermedad se asocia a una carga genética determinada que genera una mayor susceptibilidad a la respuesta inmune frente a un antígeno (Ag) y, con ello, se genera la inflamación de la membrana sinovial y la destrucción osteoarticular27.
De forma global, los individuos responden diferente a la farmacoterapia y ningún medicamento es 100% eficaz en todos los pacientes28, lo cual puede presentarse por una alteración en la farmacocinética y farmacodinámica de los medicamentos asociada a condiciones genético-ambientales29. En este contexto, el estudio de genes candidatos farmacogenómicos, comparado con el de genes candidatos de la enfermedad, ha sido más exitoso en la identificación y explicación de la variación de la respuesta farmacológica30, lo que ha favorecido la prescripción personalizada31. En este marco, es necesario disponer de información sistemática de la farmacogenómica de la AR, identificando polimorfismos de relevancia clínica en la respuesta y la toxicidad de inhibidores del TNF-α y metotrexato. Este tipo de información podía contribuir al desarrollo de herramientas genéticas para apoyar la decisión farmacoterapéutica y mejorar la respuesta al tratamiento de esta enfermedad. Por ello, el objetivo de este trabajo fue identificar los polimorfismos para ser utilizados en el diseño del test de secuenciación específica en pacientes con AR antes de la prescripción de infliximab, adalimumab, etanercept y metotrexato.
MétodosRevisión sistemática en PubMed/Medline. Para identificar los estudios se realizó una búsqueda en PubMed/Medline de septiembre de 2011 a septiembre de 2016, sin límite de idioma y tipo de estudio, utilizando los términos: «rheumatoid arthritis» and «pharmacogenomic» and «polymorphisms» and «metotrexato» and «infliximab» and «adalimumab» and «etanercept». De forma específica, se contempló la combinación de palabras clave y filtros: «rheumatoid arthritis» [MeSH Terms] AND «pharmacogenomic» [MeSH Terms] AND «polymorphisms» [MeSH Terms] AND «metotrexate» [MeSH Terms] AND «infliximab» [MeSH Terms] AND «adalimumab» [MeSH Terms] and «etanercept» [MeSH Terms]. Como criterios de inclusión se definieron estudios que reportaran polimorfismos de un solo nucleótido y que afectaran la eficacia y la seguridad de infliximab, adalimumab, etanercept y metotrexato.
Extracción de datosLos artículos que cumplieron los criterios de inclusión fueron analizados para la extracción de datos. Se creó un formato de recolección de datos que incluyó: el autor y referencia del artículo que reportaba el polimorfismo, el o los medicamentos estudiados, el tipo de estudio, el año de publicación, la población, el polimorfismo de interés, su manifestación y el reporte o no de variabilidad farmacodinámica o farmacocinética. Finalmente, cada polimorfismo identificado se buscó en Hap Map, con el fin de identificar el gen al que pertenece, junto con sus alias, el organismo, linaje, localización y exón.
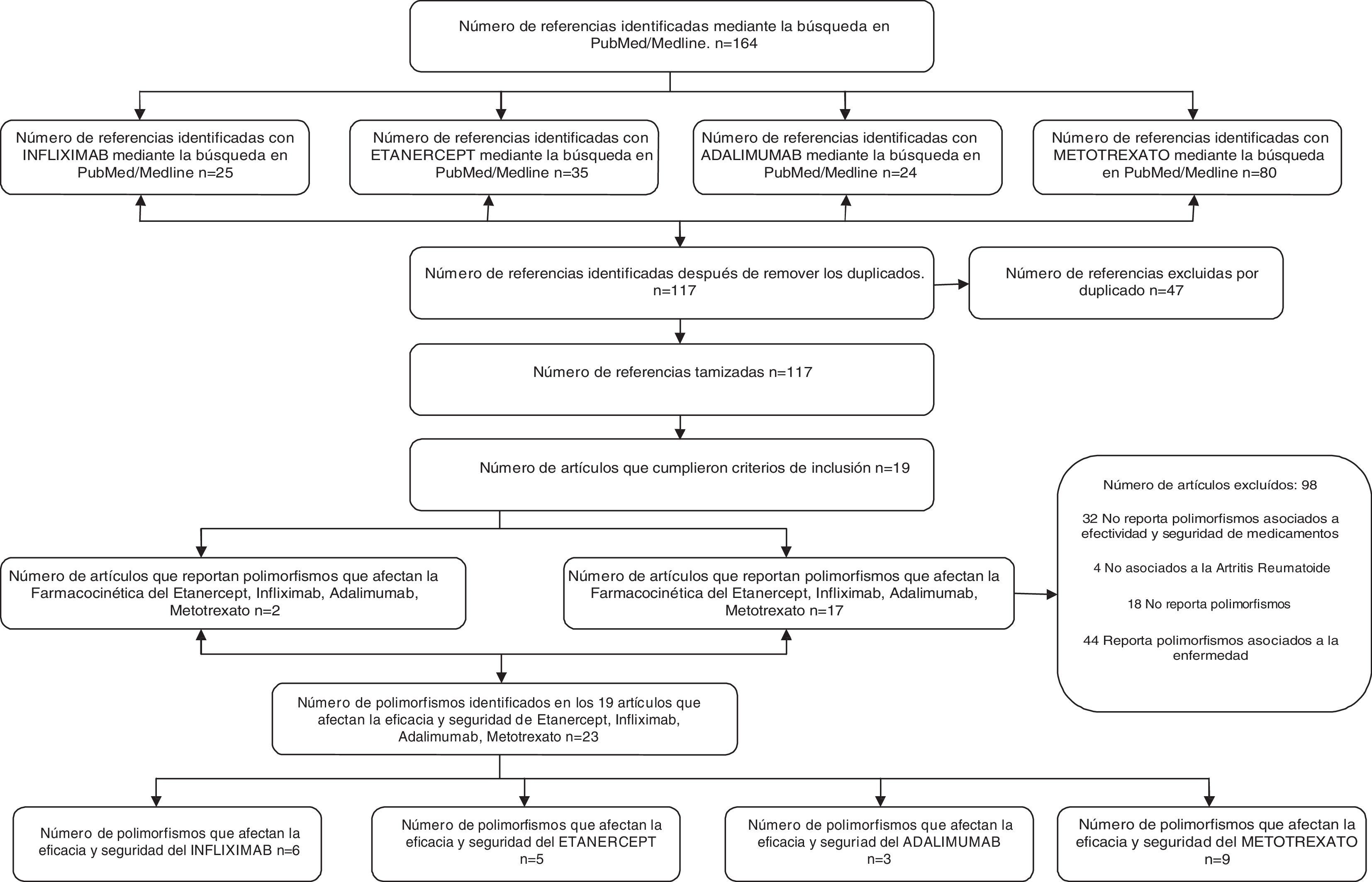
ResultadosAplicando los criterios de búsqueda definidos se encontraron 164 artículos (25 relacionados con infliximab, 24 con adalimumab, 35 con etanercept y 80 con metotrexato), de los que 47 eran repetidos, quedando 117. De este grupo de 117 artículos, 19 cumplieron con los criterios de inclusión (fig. 1). De los 19 artículos, dos reportaron polimorfismos que afectan la farmacocinética de los medicamentos infliximab, adalimumab, etanercept y metotrexato, los cuales están asociados al aumento de las concentraciones plasmáticas, mientras que 17 artículos identificaron polimorfismos que afectan la farmacodinámica de los mismos, lo que se asocia a bloqueo de receptores y se manifiesta por inefectividad de los medicamentos (tabla 1).
Polimorfismos de un solo nucleótido asociados a variabilidad en la farmacocinética y farmacodinamia de metotrexato e inhibidores del factor de necrosis tumoral: infliximab, etanercept, adalimumab
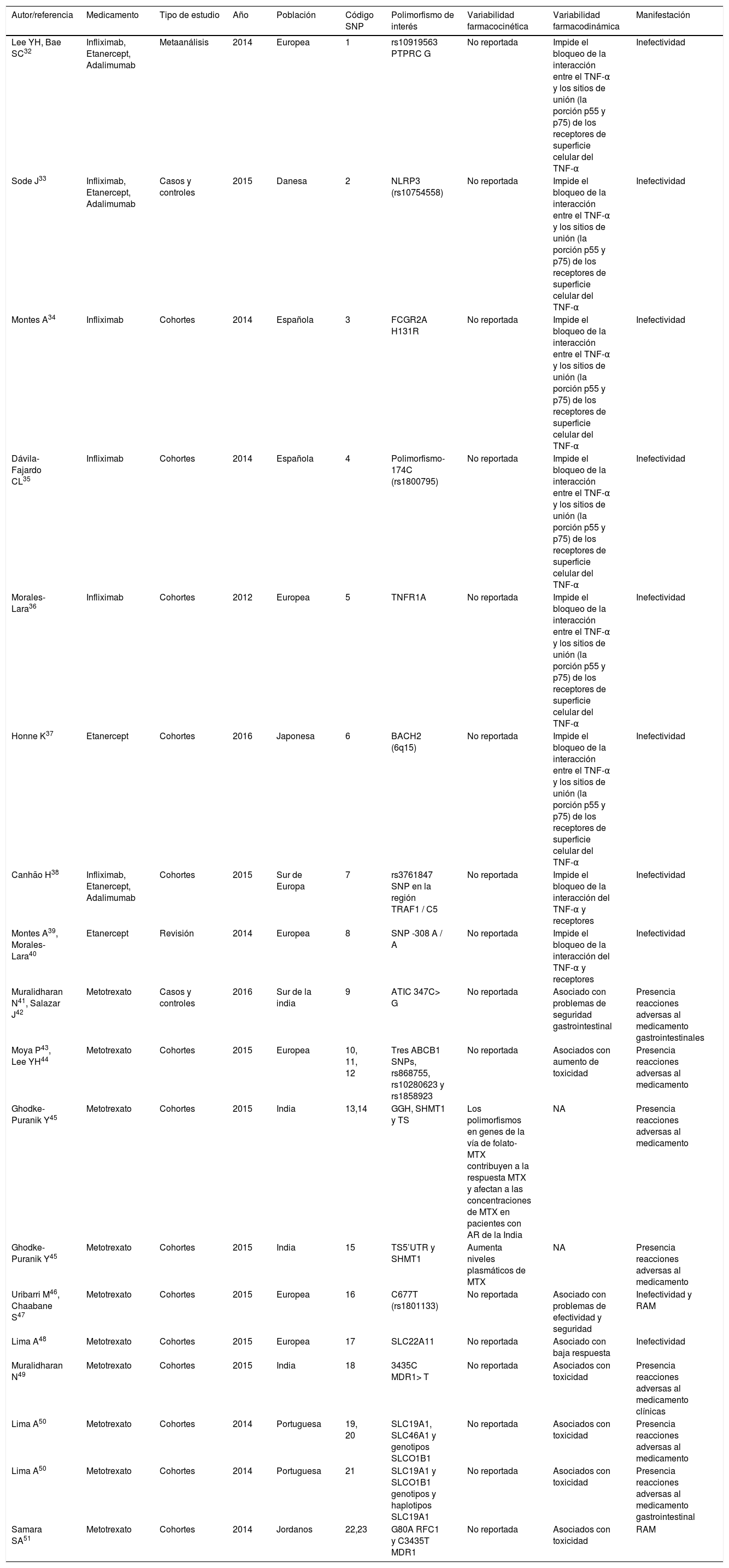
| Autor/referencia | Medicamento | Tipo de estudio | Año | Población | Código SNP | Polimorfismo de interés | Variabilidad farmacocinética | Variabilidad farmacodinámica | Manifestación |
|---|---|---|---|---|---|---|---|---|---|
| Lee YH, Bae SC32 | Infliximab, Etanercept, Adalimumab | Metaanálisis | 2014 | Europea | 1 | rs10919563 PTPRC G | No reportada | Impide el bloqueo de la interacción entre el TNF-α y los sitios de unión (la porción p55 y p75) de los receptores de superficie celular del TNF-α | Inefectividad |
| Sode J33 | Infliximab, Etanercept, Adalimumab | Casos y controles | 2015 | Danesa | 2 | NLRP3 (rs10754558) | No reportada | Impide el bloqueo de la interacción entre el TNF-α y los sitios de unión (la porción p55 y p75) de los receptores de superficie celular del TNF-α | Inefectividad |
| Montes A34 | Infliximab | Cohortes | 2014 | Española | 3 | FCGR2A H131R | No reportada | Impide el bloqueo de la interacción entre el TNF-α y los sitios de unión (la porción p55 y p75) de los receptores de superficie celular del TNF-α | Inefectividad |
| Dávila-Fajardo CL35 | Infliximab | Cohortes | 2014 | Española | 4 | Polimorfismo-174C (rs1800795) | No reportada | Impide el bloqueo de la interacción entre el TNF-α y los sitios de unión (la porción p55 y p75) de los receptores de superficie celular del TNF-α | Inefectividad |
| Morales-Lara36 | Infliximab | Cohortes | 2012 | Europea | 5 | TNFR1A | No reportada | Impide el bloqueo de la interacción entre el TNF-α y los sitios de unión (la porción p55 y p75) de los receptores de superficie celular del TNF-α | Inefectividad |
| Honne K37 | Etanercept | Cohortes | 2016 | Japonesa | 6 | BACH2 (6q15) | No reportada | Impide el bloqueo de la interacción entre el TNF-α y los sitios de unión (la porción p55 y p75) de los receptores de superficie celular del TNF-α | Inefectividad |
| Canhão H38 | Infliximab, Etanercept, Adalimumab | Cohortes | 2015 | Sur de Europa | 7 | rs3761847 SNP en la región TRAF1 / C5 | No reportada | Impide el bloqueo de la interacción del TNF-α y receptores | Inefectividad |
| Montes A39, Morales-Lara40 | Etanercept | Revisión | 2014 | Europea | 8 | SNP -308 A / A | No reportada | Impide el bloqueo de la interacción del TNF-α y receptores | Inefectividad |
| Muralidharan N41, Salazar J42 | Metotrexato | Casos y controles | 2016 | Sur de la india | 9 | ATIC 347C> G | No reportada | Asociado con problemas de seguridad gastrointestinal | Presencia reacciones adversas al medicamento gastrointestinales |
| Moya P43, Lee YH44 | Metotrexato | Cohortes | 2015 | Europea | 10, 11, 12 | Tres ABCB1 SNPs, rs868755, rs10280623 y rs1858923 | No reportada | Asociados con aumento de toxicidad | Presencia reacciones adversas al medicamento |
| Ghodke-Puranik Y45 | Metotrexato | Cohortes | 2015 | India | 13,14 | GGH, SHMT1 y TS | Los polimorfismos en genes de la vía de folato-MTX contribuyen a la respuesta MTX y afectan a las concentraciones de MTX en pacientes con AR de la India | NA | Presencia reacciones adversas al medicamento |
| Ghodke-Puranik Y45 | Metotrexato | Cohortes | 2015 | India | 15 | TS5’UTR y SHMT1 | Aumenta niveles plasmáticos de MTX | NA | Presencia reacciones adversas al medicamento |
| Uribarri M46, Chaabane S47 | Metotrexato | Cohortes | 2015 | Europea | 16 | C677T (rs1801133) | No reportada | Asociado con problemas de efectividad y seguridad | Inefectividad y RAM |
| Lima A48 | Metotrexato | Cohortes | 2015 | Europea | 17 | SLC22A11 | No reportada | Asociado con baja respuesta | Inefectividad |
| Muralidharan N49 | Metotrexato | Cohortes | 2015 | India | 18 | 3435C MDR1> T | No reportada | Asociados con toxicidad | Presencia reacciones adversas al medicamento clínicas |
| Lima A50 | Metotrexato | Cohortes | 2014 | Portuguesa | 19, 20 | SLC19A1, SLC46A1 y genotipos SLCO1B1 | No reportada | Asociados con toxicidad | Presencia reacciones adversas al medicamento |
| Lima A50 | Metotrexato | Cohortes | 2014 | Portuguesa | 21 | SLC19A1 y SLCO1B1 genotipos y haplotipos SLC19A1 | No reportada | Asociados con toxicidad | Presencia reacciones adversas al medicamento gastrointestinal |
| Samara SA51 | Metotrexato | Cohortes | 2014 | Jordanos | 22,23 | G80A RFC1 y C3435T MDR1 | No reportada | Asociados con toxicidad | RAM |
Los 19 artículos seleccionados corresponden a población europea, japonesa, jordana e india y reportan los siguientes 23 polimorfismos de un solo nucleótido: rs10919563 PTPRC G, NLRP3 (rs10754558), FCGR2A H131R, 174C (rs1800795), TNFR1A, BACH2 (6q15), rs3761847 SNP en la región TRAF1 / C5, SNP -308 A / A, ATIC 347C> G, ABCB1 rs868755, rs10280623, rs1858923, GGH, SHMT1 y TS, TS5’UTR, C677T (rs1801133), SLC22A11, 3435C MDR1> T, SLC19A1, SLC46A1 y genotipos SLCO1B1, G80A RFC1 y C3435T MDR1 (tabla 1). De forma específica, de estos 23 polimorfismos se encontraron 6 asociados a infliximab, 5 a etanercept, 3 a adalimumab y 9 a metotrexato (fig. 1).
La búsqueda en la base de datos HapMap de los 23 polimorfismos identificados permitió registrar, para cada uno de ellos, el gen que lo codifica, el organismo en el que se manifiesta, su linaje, su localización o locus y el exón. En este sentido, de los 23 polimorfismos identificados se destacó (tabla 2):
- •
Diecinueve (82,6%) corresponden a la especie Homo sapiens.
- •
En la secuencia del polipéptido, las regiones que se traducen a aminoácidos provienen principalmente del exón 10 (5 polimorfismos), del exón 29 (3 polimorfismos) y del exón 17 (2 polimorfismos).
- •
El gen SLC (de diferentes familias genéticas) se encontró asociado a 4 polimorfismos, mientras que el ABCB1 (ATP binding cassette subfamily B member 1) se encontró asociado a 3 de los 23 polimorfismos y el gen MDR1 a 2 polimorfismos.
Localización y exones de genes identificados con polimorfismo de un solo nucleótido que afectan la efectividad y la seguridad de infliximab, etanercept, adalimumab y metotrexato
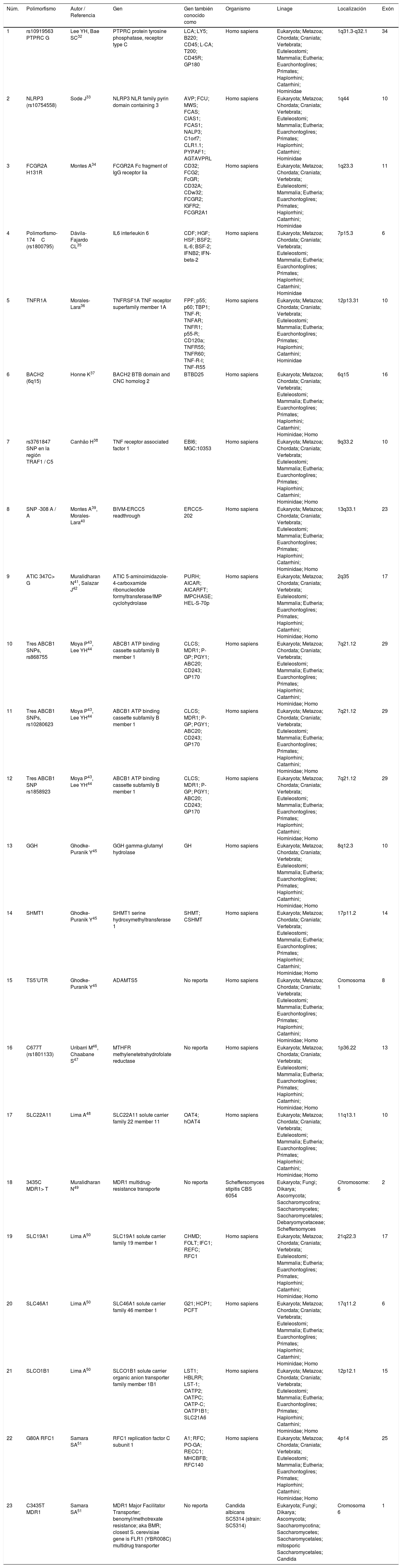
| Núm. | Polimorfismo | Autor / Referencia | Gen | Gen también conocido como | Organismo | Linage | Localización | Exón |
|---|---|---|---|---|---|---|---|---|
| 1 | rs10919563 PTPRC G | Lee YH, Bae SC32 | PTPRC protein tyrosine phosphatase, receptor type C | LCA; LY5; B220; CD45; L-CA; T200; CD45R; GP180 | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae | 1q31.3-q32.1 | 34 |
| 2 | NLRP3 (rs10754558) | Sode J33 | NLRP3 NLR family pyrin domain containing 3 | AVP; FCU; MWS; FCAS; CIAS1; FCAS1; NALP3; C1orf7; CLR1.1; PYPAF1; AGTAVPRL | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae | 1q44 | 10 |
| 3 | FCGR2A H131R | Montes A34 | FCGR2A Fc fragment of IgG receptor Iia | CD32; FCG2; FcGR; CD32A; CDw32; FCGR2; IGFR2; FCGR2A1 | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae | 1q23.3 | 11 |
| 4 | Polimorfismo-174C (rs1800795) | Dávila-Fajardo CL35 | IL6 interleukin 6 | CDF; HGF; HSF; BSF2; IL-6; BSF-2; IFNB2; IFN-beta-2 | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae | 7p15.3 | 6 |
| 5 | TNFR1A | Morales-Lara36 | TNFRSF1A TNF receptor superfamily member 1A | FPF; p55; p60; TBP1; TNF-R; TNFAR; TNFR1; p55-R; CD120a; TNFR55; TNFR60; TNF-R-I; TNF-R55 | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae | 12p13.31 | 10 |
| 6 | BACH2 (6q15) | Honne K37 | BACH2 BTB domain and CNC homolog 2 | BTBD25 | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae; Homo | 6q15 | 16 |
| 7 | rs3761847 SNP en la región TRAF1 / C5 | Canhão H38 | TNF receptor associated factor 1 | EBI6; MGC:10353 | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae; Homo | 9q33.2 | 10 |
| 8 | SNP -308 A / A | Montes A39, Morales-Lara40 | BIVM-ERCC5 readthrough | ERCC5-202 | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae; Homo | 13q33.1 | 23 |
| 9 | ATIC 347C> G | Muralidharan N41, Salazar J42 | ATIC 5-aminoimidazole-4-carboxamide ribonucleotide formyltransferase/IMP cyclohydrolase | PURH; AICAR; AICARFT; IMPCHASE; HEL-S-70p | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae; Homo | 2q35 | 17 |
| 10 | Tres ABCB1 SNPs, rs868755 | Moya P43, Lee YH44 | ABCB1 ATP binding cassette subfamily B member 1 | CLCS; MDR1; P-GP; PGY1; ABC20; CD243; GP170 | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae; Homo | 7q21.12 | 29 |
| 11 | Tres ABCB1 SNPs, rs10280623 | Moya P43, Lee YH44 | ABCB1 ATP binding cassette subfamily B member 1 | CLCS; MDR1; P-GP; PGY1; ABC20; CD243; GP170 | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae; Homo | 7q21.12 | 29 |
| 12 | Tres ABCB1 SNP rs1858923 | Moya P43, Lee YH44 | ABCB1 ATP binding cassette subfamily B member 1 | CLCS; MDR1; P-GP; PGY1; ABC20; CD243; GP170 | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae; Homo | 7q21.12 | 29 |
| 13 | GGH | Ghodke-Puranik Y45 | GGH gamma-glutamyl hydrolase | GH | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae; Homo | 8q12.3 | 10 |
| 14 | SHMT1 | Ghodke-Puranik Y45 | SHMT1 serine hydroxymethyltransferase 1 | SHMT; CSHMT | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae; Homo | 17p11.2 | 14 |
| 15 | TS5’UTR | Ghodke-Puranik Y45 | ADAMTS5 | No reporta | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae; Homo | Cromosoma 1 | 8 |
| 16 | C677T (rs1801133) | Uribarri M46, Chaabane S47 | MTHFR methylenetetrahydrofolate reductase | No reporta | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae; Homo | 1p36.22 | 13 |
| 17 | SLC22A11 | Lima A48 | SLC22A11 solute carrier family 22 member 11 | OAT4; hOAT4 | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae; Homo | 11q13.1 | 10 |
| 18 | 3435C MDR1> T | Muralidharan N49 | MDR1 multidrug-resistance transporte | No reporta | Scheffersomyces stipitis CBS 6054 | Eukaryota; Fungi; Dikarya; Ascomycota; Saccharomycotina; Saccharomycetes; Saccharomycetales; Debaryomycetaceae; Scheffersomyces | Chromosome: 6 | 2 |
| 19 | SLC19A1 | Lima A50 | SLC19A1 solute carrier family 19 member 1 | CHMD; FOLT; IFC1; REFC; RFC1 | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae; Homo | 21q22.3 | 17 |
| 20 | SLC46A1 | Lima A50 | SLC46A1 solute carrier family 46 member 1 | G21; HCP1; PCFT | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae; Homo | 17q11.2 | 6 |
| 21 | SLCO1B1 | Lima A50 | SLCO1B1 solute carrier organic anion transporter family member 1B1 | LST1; HBLRR; LST-1; OATP2; OATPC; OATP-C; OATP1B1; SLC21A6 | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae; Homo | 12p12.1 | 15 |
| 22 | G80A RFC1 | Samara SA51 | RFC1 replication factor C subunit 1 | A1; RFC; PO-GA; RECC1; MHCBFB; RFC140 | Homo sapiens | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae; Homo | 4p14 | 25 |
| 23 | C3435T MDR1 | Samara SA51 | MDR1 Major Facilitator Transporter; benomyl/methotrexate resistance; aka BMR; closest S. cerevisiae gene is FLR1 (YBR008C) multidrug transporter | No reporta | Candida albicans SC5314 (strain: SC5314) | Eukaryota; Fungi; Dikarya; Ascomycota; Saccharomycotina; Saccharomycetes; Saccharomycetales; mitosporic Saccharomycetales; Candida | Cromosoma 6 | 1 |
En la AR, medicamentos como el metotrexato y los inhibidores del TNF-α como infliximab, adalimumab, etanercept son muy importantes para lograr la remisión de los pacientes; sin embargo, la variabilidad genética de los individuos conlleva que la capacidad de respuesta y toxicidad a estos medicamentos sea diferente y que se reporten índices inferiores al 60%8. La farmacogenómica trata las posibles asociaciones de polimorfismos genéticos con respuestas a los fármacos, es una rama de la ciencia médica que está aún en su fase inicial y requiere más tiempo para demostrar resultados positivos, así que depende de una buena articulación entre el sector investigativo, la legislación, las farmacéuticas y las instituciones de salud para que en un futuro las medicinas se desarrollen en beneficio de mejorar las condiciones de salud y los costes de tratamientos en pequeños grupos objetivo; de igual manera, no se debe dejar de lado el valor de las variables no-genéticas en el progreso de una enfermedad, como la edad, el género, la dieta y el estilo de vida, que también son claves en la respuesta a una terapia.
La relevancia de este estudio consiste en brindar la posibilidad de aplicar la investigación de genes candidatos seleccionados por su importancia biológica, ya sea en la cinética o por su relación en la acción farmacológica, en la identificación de individuos con riesgo de experimentar efectos adversos o con probabilidad de ser resistentes al tratamiento. Por ello, se espera que la información generada sea susceptible de ser utilizada en el diseño de test de secuenciación específica, contribuyendo a identificar la mejor opción terapéutica (mayor efectividad y seguridad) en pacientes con AR18.
En el tratamiento de la AR, la posibilidad de asociar ciertos polimorfismos con la eficiencia del tratamiento, reduciendo los efectos adversos del uso de un medicamento, puede ser de gran valor tanto para el paciente como para la práctica médica y para el aspecto económico de esta1. Los resultados obtenidos con la presente revisión sistemática son una base para el diseño del test de secuenciación específica en pacientes con AR antes de la prescripción de infliximab, adalimumab, etanercept y metotrexato; de aquí la importancia de diseñar y validar con estudios multicéntricos test genómicos específicos que contribuyan a una prescripción personalizada para mejorar la efectividad y la seguridad de los tratamientos con estos medicamentos y lograr disminuir costos en la atención en salud. La identificación de los 23 polimorfismos de relevancia clínica obtenidos con la revisión son muy importantes para el diseño de test genómicos; sin embargo, es necesario incluir en los estudios multicéntricos, durante la secuenciación exómica, otros genes antes de validar y estandarizar un test de uso en la práctica clínica.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Universidad de Antioquia, Colciencias, Hospital Universitario Pablo Tobón Uribe.