La metilación del ácido desoxirribonucleico (ADN) es una rama de la epigenética que puede ser útil para la identificación, incluso precoz, del cáncer de pulmón. El objetivo del estudio fue evaluar la sensibilidad, especificidad y rendimiento diagnóstico de un panel de metilación formado por los genes APC (adenomatous polyposis coli), DAPK (death associated protein kinase) y RASSF1A (Ras association domain familiy 1A) asociado a la citología habitual en broncoaspirados (BAS) de pacientes con sospecha de cáncer de pulmón.
Material y métodosSe seleccionaron 39 BAS, 24 positivos y 15 negativos, para cáncer de pulmón de diferentes tipos y estadios. Las citosinas de las muestras se transformaron en uracilos con bisulfito sódico, se efectuó una doble reacción en cadena de la polimerasa (PCR) (bisulfite conversion specific-methylation specific PCR) y se identificó el estado de metilación mediante electroforesis.
ResultadosAPC resultó metilado en 2 de los 24 tumores pulmonares (8%), DAPK en 0 de 24 (0%) y RASSF1A en 9 de 23 (39%). La sensibilidad total del panel fue del 37,5% (9 de 24). No se detectó metilación en ninguna de las muestras libres de cáncer de pulmón (0 de 15), por lo que la especificidad fue del 100%. Además, el panel detectó metilación en una de las 7 muestras de cáncer primario de pulmón, cuya prueba citológica había sido negativa (14%) y en 3 de las 5 muestras con cáncer de pulmón con citologías meramente sospechosas (60%). Su contribución al diagnóstico correcto ante pruebas citológicas sospechosas o discordantes con el diagnóstico final fue del 33% (4 de 12).
ConclusionesLa metilación del ADN puede ser un arma útil en el diagnóstico del cáncer de pulmón. Hasta el momento, la sensibilidad en el panel escogido depende exclusivamente de RASSF1A. Nuevos estudios son necesarios para garantizar la reproducibilidad y optimizar su sensibilidad.
DNA methylation is a part of epigenetics that can be useful for lung cancer detection even at early stages. The aim of this study was to evaluate sensitivity, specificity and diagnostic capacity of a methylation panel including APC, DAPK and RASSF1A genes, together with routine cytology tests in patients suspected with lung cancer.
Material and methodsWe selected 39 bronchoaspirates, 24 positive and 15 negative for lung cancer of different histological types and stages. Samples were transformed with sodium bisulphite. A double PCR (BS-MSP) was performed and methylation status was identified by electrophoresis.
ResultsAPC was found to be methylated in 2 out of 24 lung tumours (8%), DAPK in 0 out of 24 (0%) and RASSF1A in 9 out of 23 (39%). Panel sensibility was 37.5% (9/24). No methylation was detected in any of the negative lung cancer samples (0/15), so specificity was 100%. Moreover, the panel detected methylation in 1 of the 7 primary lung cancer tumour samples in which the cytology test was negative (14%) and in 3 of 5 lung cancer tumour samples in which cytology test was only suspicious (60%). Its contribution to the correct diagnosis when cytology tests were suspicious or discordant with final diagnosis was 33% (4/12).
ConclusionsDNA methylation can be a useful tool in lung cancer diagnosis. The chosen panel sensibility depends exclusively on RASSF1A to date. Consequently, new studies are required to guarantee reproducibility and optimize sensitivity.
El cáncer de pulmón es la neoplasia más frecuente y con mayor tasa de mortalidad en los países occidentales1. Clínicamente, se divide en 2 subtipos: carcinoma de pulmón de células pequeñas (el más grave, y que supone aproximadamente un 20% de los casos) y carcinoma de pulmón de célula no pequeña. Éste representa el 80% restante y se clasifica nuevamente en 4 categorías: adenocarcinoma (del 35 al 40%), carcinoma escamoso (30%), carcinoma de células grandes (10%), y otros tipos histológicos, como los tumores neuroendocrinos (del 0 al 5%). De forma global, la supervivencia a los 5 años no supera el 15%, lo que supone una tasa entre 4 y 6 veces inferior a la que presentan otros tumores frecuentes, como los de mama, colon o próstata. Este hecho se debe, en gran parte, a que tan sólo el 15% de los tumores malignos de pulmón se encuentra en estadios precoces en el momento del diagnóstico2. En este sentido, el beneficio que supone una detección precoz se refleja en que los pacientes que presentan un tumor en estadio inicial (≤1cm), donde la resección quirúrgica suele ser el tratamiento indicado, presentan una tasa de recurrencia a los 5 años inferior al 50%3. Por este motivo, se buscan permanentemente nuevas técnicas diagnósticas que mejoren la identificación tumoral; algunas de ellas son el estudio de las mutaciones en los genes K-ras y p53, la alteración de microsatélites, el estudio de marcadores séricos como el Cyfra 21.1 o la tomografía computarizada helicoidal en bajas dosis. A pesar de que algunas de estas técnicas ofrecen resultados prometedores, ninguna de ellas ha supuesto una mejora sustancial2, por lo que siguen siendo necesarias nuevas herramientas que incrementen el rendimiento diagnóstico.
La epigenética se define como el conjunto de cambios heredables en la expresión génica que no van acompañados de cambios en la secuencia de ácido desoxirribonucleico (ADN). Éstos incluyen la hipermetilación de promotores, la metilación o la acetilación de histonas, o el ácido ribonucleico de interferencia o de transferencia; y, en general, suponen una inhibición de la transcripción génica4. La metilación se produce exclusivamente en los nucleótidos de citosina que van seguidos de guanina (dinucleótidos CpG [cytosine-phospho-guanine]). Esta característica ha permitido desarrollar técnicas que permiten detectar cuantitativa o cualitativamente el estado de metilación (uso de enzimas de restricción, MSP [methylation specific PCR {polymerase chain reaction ‘reacción en cadena de la polimerasa’} específica de metilación], qMSP [quantitative MSP], o técnicas basadas en curvas Melting, entre otras). La mayor parte de ellas se basan en una transformación de la muestra con bisulfito, de tal manera que las citosinas no metiladas se convierten en uracilos, mientras que las metiladas mantienen la secuencia original. Este cambio es el que permite la posterior identificación a través del uso, por ejemplo, de cebadores específicos para muestras metiladas o no metiladas.
La metilación aberrante en los promotores de los genes se ha relacionado con numerosas enfermedades, especialmente con el cáncer, y son numerosas las ventajas potenciales que parece ofrecer: a) la metilación de determinados genes es un episodio temprano en el desarrollo tumoral y, por tanto, puede servir como herramienta para la detección precoz5; b) diferentes tipos tumorales presentan diferencias en los genes que tienden a estar metilados, por lo que se pueden desarrollar patrones relativamente específicos para el diagnóstico6, y c) existe la posibilidad de usar material obtenido mediante técnicas no invasivas o mínimamente invasivas. En el caso del cáncer de pulmón, por ejemplo, se ha estudiado con muestras de biopsias, pero también en broncoaspirados (BAS), exhalados, saliva, esputo o sangre. En este sentido, las pruebas citológicas obtenidas mediante lavados broncoalveolares (LBA) o BAS son un paso fundamental para la confirmación del diagnóstico de cáncer de pulmón. Su limitada sensibilidad, sin embargo, obliga en muchas ocasiones a añadir pruebas más invasivas, como la toma de biopsias directamente del tejido tumoral, con el riesgo añadido de neumotórax, hemorragia o infección que esto supone.
El objetivo del presente estudio ha sido analizar la metilación de los genes APC (adenomatous polyposis coli), DAPK (death associated protein kinase) y RASSF1A (Ras association domain familiy 1A) en BAS de pacientes con sospecha de cáncer de pulmón, y determinar así su sensibilidad y especificidad. Se ha estudiado también su rendimiento diagnóstico frente al que ofrecen las pruebas citológicas, y las ventajas que podría ofrecer un análisis combinado citomolecular en este tipo de muestras.
Material y métodosPacientes y muestrasSe seleccionaron 39 BAS de pacientes con sospecha de cáncer de pulmón recogidos en el Servicio de Neumología del Hospital de la Santa Creu i Sant Pau de Barcelona como parte del proceso diagnóstico y según los requerimientos éticos establecidos en esta institución. La muestra obtenida se dividió en 2 partes: una de ellas se envió al servicio de anatomía patológica para su estudio citológico y la otra se envió al servicio de bioquímica para su estudio molecular.
En la elección de las muestras se incluyó un número representativo de casos de cáncer primario de pulmón y de controles, en los que se había descartado la enfermedad, con la finalidad de determinar la correspondiente sensibilidad y especificidad. De las 39 muestras seleccionadas, 24 presentaron un diagnóstico final positivo para cáncer de pulmón (establecido definitivamente mediante estudio patológico, por imagen o por exploración quirúrgica) y 15 fueron negativas para cáncer primario de pulmón. Entre las primeras, 2 correspondían a carcinomas de células pequeñas, 15 a carcinomas escamosos, 4 a carcinomas de células grandes, una a adenocarcinoma, una a un tumor carcinoide y una última muestra presentaba un diagnóstico histológico sin determinar. Las muestras con diagnóstico negativo para cáncer de pulmón incluían procesos benignos no tumorales o procesos tumorales no originarios de pulmón (un linfoma hodgkiniano y 2 tumores metastásicos, uno renal y otro vesical) (tabla 1). En cuanto a la estadificación, 12 se encontraban en estadio IV, 6 en estadio III–B, 2 en estadio III–A y 2 en estadio I–B (tabla 2). Las muestras correspondientes a carcinomas de células pequeñas se consideraron de estadio IV debido a su elevada gravedad. Sin embargo, por su diferente clasificación y abordaje no se incluyen en los recuentos por estadios.
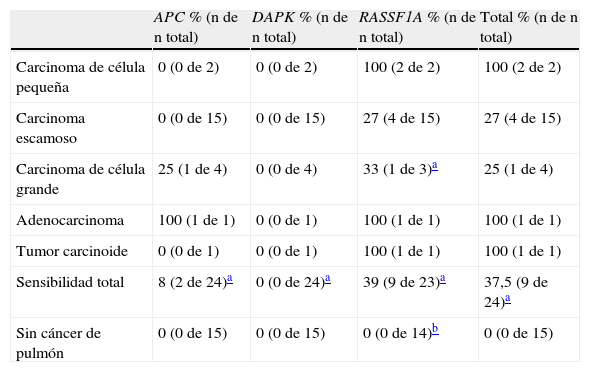
Frecuencia de metilación en los broncoaspirados de cada uno de los tipos de tumores estudiados y en muestras libres de cáncer de pulmón
| APC % (n de n total) | DAPK % (n de n total) | RASSF1A % (n de n total) | Total % (n de n total) | |
| Carcinoma de célula pequeña | 0 (0 de 2) | 0 (0 de 2) | 100 (2 de 2) | 100 (2 de 2) |
| Carcinoma escamoso | 0 (0 de 15) | 0 (0 de 15) | 27 (4 de 15) | 27 (4 de 15) |
| Carcinoma de célula grande | 25 (1 de 4) | 0 (0 de 4) | 33 (1 de 3)a | 25 (1 de 4) |
| Adenocarcinoma | 100 (1 de 1) | 0 (0 de 1) | 100 (1 de 1) | 100 (1 de 1) |
| Tumor carcinoide | 0 (0 de 1) | 0 (0 de 1) | 100 (1 de 1) | 100 (1 de 1) |
| Sensibilidad total | 8 (2 de 24)a | 0 (0 de 24)a | 39 (9 de 23)a | 37,5 (9 de 24)a |
| Sin cáncer de pulmón | 0 (0 de 15) | 0 (0 de 15) | 0 (0 de 14)b | 0 (0 de 15) |
APC: adenomatous polyposis coli; DAPK: death associated protein kinase; RASSF1A: Ras association domain familiy 1A.
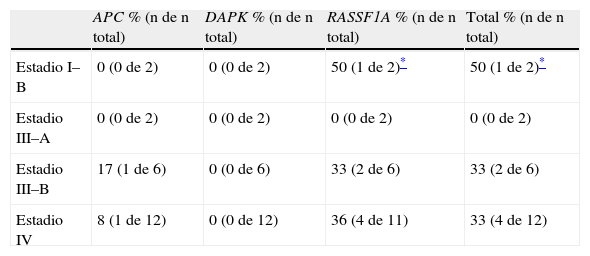
Frecuencia de metilación en cada uno de los diferentes estadios tumorales estudiados
| APC % (n de n total) | DAPK % (n de n total) | RASSF1A % (n de n total) | Total % (n de n total) | |
| Estadio I–B | 0 (0 de 2) | 0 (0 de 2) | 50 (1 de 2)* | 50 (1 de 2)* |
| Estadio III–A | 0 (0 de 2) | 0 (0 de 2) | 0 (0 de 2) | 0 (0 de 2) |
| Estadio III–B | 17 (1 de 6) | 0 (0 de 6) | 33 (2 de 6) | 33 (2 de 6) |
| Estadio IV | 8 (1 de 12) | 0 (0 de 12) | 36 (4 de 11) | 33 (4 de 12) |
APC: adenomatous polyposis coli; DAPK: death associated protein kinase; RASSF1A: Ras association domain familiy 1A.
No se incluyen los carcinomas de célula pequeña.
El ADN de las muestras se extrajo mediante el método estándar de fenol-cloroformo-alcohol isoamílico; se partió del pellet celular obtenido en un volumen de 2 a 3ml de BAS y de la posterior precipitación con isopropanol. El ADN obtenido se resuspendió en 25 a 50μl de agua autoclavada.
Se emplearon 500ng de ADN de cada muestra para transformar las citosinas no metiladas en uracilos mediante el EZ-DNA-methylation-Gold-kit (Zymo Research Orange, CA. EE. UU.) y se recuperaron mediante 20μl de tampón de elución. Como control positivo se usó ADN procedente de linfocitos o líneas celulares tumorales, en ambos casos tratados in vitro con SssI methyltransferase (New England Biolabs Inc., Beverly, MA, EE. UU.). Además, como control negativo para garantizar la completa y correcta transformación de las muestras, se usó ADN procedente de linfocitos al que se le realizó un procedimiento de Whole Genome Amplification (Qiagen, Basel, Suiza); así, se aseguró mediante un elevado número de copias la ausencia de metilación en el ADN tratado.
Tras la transformación de las citosinas en uracilos se efectuó una doble PCR (BS-MSP [bisulfite conversion specific-MSP]) y se identificó el estado de metilación mediante electroforesis.
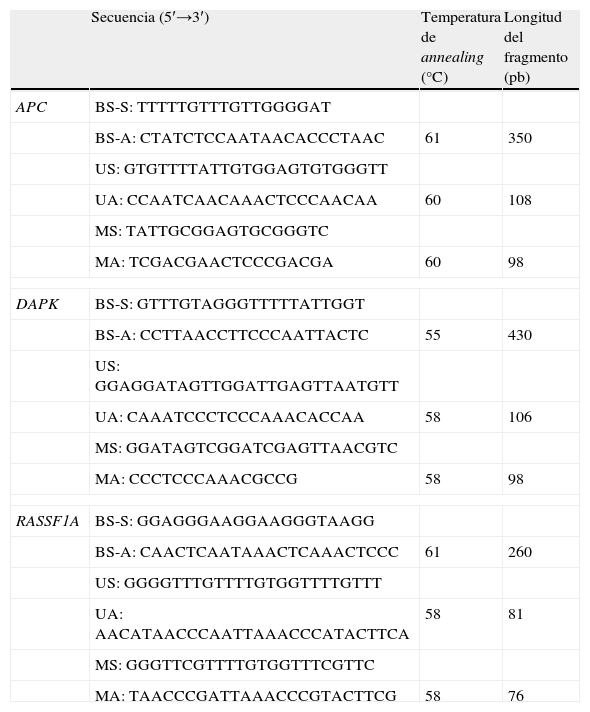
Análisis de metilación del ácido desoxirribonucleico: reacción en cadena de la polimerasa específica de metilaciónSe estudió la metilación de los promotores de los genes APC, DAPK y RASSF1A mediante MSP, un método que permite distinguir entre los alelos metilados y no metilados de un determinado gen7. Después del tratamiento con bisulfito y la transformación de los residuos de citosina no metilados en uracilo, el ADN se amplificó mediante una doble PCR (nested PCR). Esta técnica permite incrementar la sensibilidad al aumentar la cantidad de muestra inicial: teóricamente, una única reacción de MSP detecta un alelo metilado entre 10.000 no metilados, mientras que la técnica nested permite captar uno entre 50.0002. Además, un correcto diseño de los cebadores para la primera reacción (técnica de BS-MSP) consigue incrementar la especificidad analítica al evitar la amplificación de muestras en las que la transformación haya sido incompleta y que darían lugar a falsos positivos, como ha sido descrito8. Se debe señalar que los cebadores de la primera reacción (tabla 3) reconocen una secuencia común a todas las muestras, por lo que no permiten determinar un diagnóstico diferencial de metilación. Esta primera PCR incluyó: 1μl de ADN convertido; 0,06μl (5U/μl) de EcoStart Polymerase (Qiagen); 0,04μl de cada cebador (100μm); 0,65μl de MgCl2 (50mm); 1,5μl de tampón 10x (Qiagen), y 1,5μl de dNTP (deoxyribonucleotide triphosphate) (2mm) para un volumen total de reacción de 16μl (completado con agua autoclavada). Las condiciones de la reacción fueron las siguientes: 7min a 95°C; 20 ciclos de 95°C durante 30s; 61°C durante 30s para APC y RASSF1A, o 55°C durante 30s para DAPK y 30s a 72°C y 15min a 72°C como período de extensión (tabla 3).
Secuencias de los cebadores correspondientes a cada una de las reacciones, temperatura de annealing y longitud del fragmento correspondiente
| Secuencia (5′→3′) | Temperatura de annealing (°C) | Longitud del fragmento (pb) | |
| APC | BS-S: TTTTTGTTTGTTGGGGAT | ||
| BS-A: CTATCTCCAATAACACCCTAAC | 61 | 350 | |
| US: GTGTTTTATTGTGGAGTGTGGGTT | |||
| UA: CCAATCAACAAACTCCCAACAA | 60 | 108 | |
| MS: TATTGCGGAGTGCGGGTC | |||
| MA: TCGACGAACTCCCGACGA | 60 | 98 | |
| DAPK | BS-S: GTTTGTAGGGTTTTTATTGGT | ||
| BS-A: CCTTAACCTTCCCAATTACTC | 55 | 430 | |
| US: GGAGGATAGTTGGATTGAGTTAATGTT | |||
| UA: CAAATCCCTCCCAAACACCAA | 58 | 106 | |
| MS: GGATAGTCGGATCGAGTTAACGTC | |||
| MA: CCCTCCCAAACGCCG | 58 | 98 | |
| RASSF1A | BS-S: GGAGGGAAGGAAGGGTAAGG | ||
| BS-A: CAACTCAATAAACTCAAACTCCC | 61 | 260 | |
| US: GGGGTTTGTTTTGTGGTTTTGTTT | |||
| UA: AACATAACCCAATTAAACCCATACTTCA | 58 | 81 | |
| MS: GGGTTCGTTTTGTGGTTTCGTTC | |||
| MA: TAACCCGATTAAACCCGTACTTCG | 58 | 76 | |
APC: adenomatous polyposis coli; BS-A: bisulfite conversion specific-cebador externo antisense; BS-S: bisulfite conversion specific -cebador externo sense; DAPK: death associated protein kinase; MA: cebador methylated antisense; MS: cebador methylated sense; RASSF1A: Ras association domain familiy 1A; UA: cebador unmethylated antisense; US: cebador unmethylated sense.
En la segunda reacción se emplearon 2 parejas de cebadores (tabla 3), una de ellas específica para secuencias metiladas y la otra para secuencias no metiladas. Esta segunda PCR incluyó: 1μl de ADN procedente de la primera reacción; 0,06μl (5U/μl) de Hot Start Polymerase (Qiagen); 0,04μl de cada cebador (100μm); 0,65μl de MgCl2 (50mM); 1,5μl de tampón 10x (Qiagen); y 1,5μl de dNTP (2mm) para un volumen total de reacción de 16μl (completado con agua autoclavada). Las condiciones para cada uno de los 3 genes fueron las siguientes: 7min a 95°C; 35 ciclos de 95°C durante 1min; 60°C durante 30s para APC o 58°C durante 30s para DAPK y RASSF1A y 30s a 72°C, y 15min a 72°C como período de extensión (tabla 3).
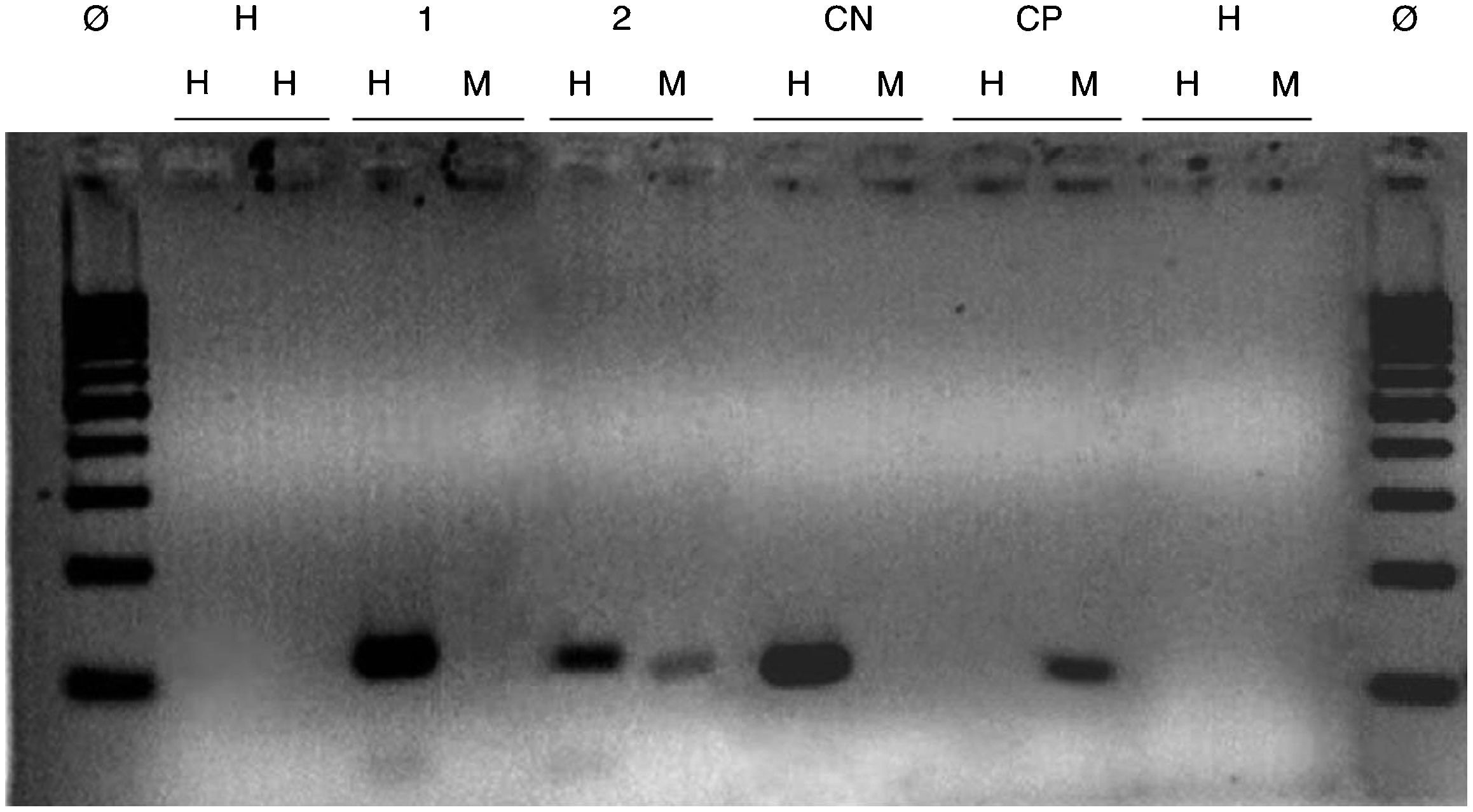
Los productos amplificados se visualizaron mediante un gel de agarosa al 2,5% y una posterior tinción con bromuro de etidio. En cada secuencia de reacciones se incluyeron controles tanto positivos como negativos para garantizar la validez de los resultados (fig. 1).
ResultadosFrecuencia de metilación en broncoaspirados según tipo tumoral o ausencia de tumor pulmonarTras el proceso de identificación mediante MSP, las frecuencias de muestras metiladas en cada gen fueron las siguientes (tabla 1): APC se detectó metilado en el 25% de los carcinomas de célula grande (uno de 4) y en el único adenocarcinoma (100%). Sin embargo, no se detectó metilación en ninguno de los carcinomas de célula pequeña, escamosos o en el tumor carcinoide. La sensibilidad total de APC fue de un 8% (2 de 24) y la especificidad del 100% (0 de 15), ya que ninguna de las muestras libres de tumor primario pulmonar resultó metilada. En cuanto a DAPK, ninguna de las muestras, tanto procedentes de tumores pulmonares como libres de él, se encontró metilada en BAS. Respecto a RASSF1A, se encontró metilado el 100% de los carcinomas de células pequeñas (2 de 2), el 27% de los carcinomas escamosos (4 de 15), el 33% de los carcinomas de célula grande (uno de 3, si se tiene en cuenta que una de las muestras en las que se habían analizado APC y DAPK debió excluirse para RASSF1A por cantidad insuficiente), el único adenocarcinoma (100%) y el tumor carcinoide (100%). La sensibilidad total de RASSF1A fue del 39% (9 de 23) y la especificidad del 100% (0 de 14). Si se tiene en cuenta el panel completo, incluidos los 3 genes analizados, y si se considera como resultado positivo la metilación en al menos uno de ellos, la sensibilidad total del panel de metilación fue del 37,5% (9 de 24). La sensibilidad según cada tipo tumoral fue del 100% para carcinoma de células pequeñas (2 de 2), 27% para carcinoma escamoso (4 de 15), 25% para carcinoma de célula grande (uno de 4), 100% para adenocarcinoma (uno de uno) y 100% para tumor carcinoide (uno de uno). La especificidad total del panel de metilación fue del 100% (0 de 15).
Frecuencia de metilación en broncoaspirados según estadio tumoralLa frecuencia de muestras metiladas (tabla 2) para el gen APC fue del 17% en estadio III–B (1 de 6) y del 8% en estadio IV (1 de 12). No se detectó metilación en ninguna de las muestras en estadios III–A y I–B (0 de 2, en ambos casos). DAPK no se encontró metilado en ninguna de las muestras de BAS. RASSF1A resultó positivo para metilación en el 50% de las muestras en estadio I–A (1 de 2), en el 33% de las muestras en estadio III–B (2 de 6) y en el 36% de las muestras en estadio IV (4 de 11). No se detectó metilación en los 2 tumores con estadio III–A. La sensibilidad total del panel de metilación fue del 50% para estadios I–B (1 de 2), 0% para estadios III–A (0 de 2), 33% para estadios III–B (2 de 6) y 33% para estadios IV (4 de 12).
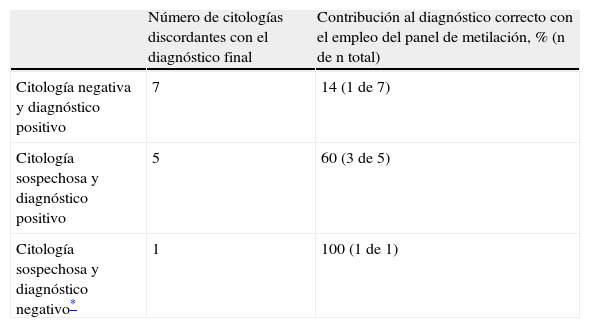
Contribución del panel de metilación en broncoaspirados al diagnóstico correcto en citologías sospechosas o discordantes con el diagnóstico finalEn la tabla 4 se recoge el número de citologías en BAS sospechosas o discordantes con el diagnóstico final y la contribución al diagnóstico correcto al emplear el panel de metilación. Se encontraron 7 citologías negativas en muestras que acabaron teniendo un diagnóstico de cáncer de pulmón. Una de estas muestras (14%) se detectó por el panel de metilación. De los 5 pacientes que presentaban citologías sospechosas con diagnóstico final positivo, 3 se detectaron por el panel (60%). Por tanto, se puede decir que ante la presencia de citologías no concluyentes o discordantes con el diagnóstico final, la sensibilidad del panel fue del 33% (4 de 12). Por otro lado, en la única muestra en la que la citología era sospechosa y el diagnóstico final excluyó el cáncer de pulmón, el panel descartó su presencia (0 de una), por lo que la especificidad fue del 100%.
Número de citologías sospechosas o discordantes con el diagnóstico final y contribución al diagnóstico correcto al emplear el panel de metilación
| Número de citologías discordantes con el diagnóstico final | Contribución al diagnóstico correcto con el empleo del panel de metilación, % (n de n total) | |
| Citología negativa y diagnóstico positivo | 7 | 14 (1 de 7) |
| Citología sospechosa y diagnóstico positivo | 5 | 60 (3 de 5) |
| Citología sospechosa y diagnóstico negativo* | 1 | 100 (1 de 1) |
La metilación en los promotores de los genes se presenta como un arma potencialmente útil para la detección de diferentes tipos tumorales. Entre sus ventajas se encuentra la posibilidad de anteceder a la presentación clínica y el poder emplear diferentes tipos de muestras más o menos invasivas2. En el caso del cáncer de pulmón, no hay aún un gran número de trabajos que determinen el papel que podría tener en la práctica clínica directa de pacientes con sospecha de enfermedad en relación con las pruebas citológicas9–13. Por esto, en este estudio se analizó el beneficio que supondría la aplicación de un panel de metilación compuesto por 3 genes en BAS añadido a las correspondientes pruebas citológicas.
Uno de los principales problemas en las técnicas de detección de muestras metiladas es la elevada tasa de falsos positivos que pueden presentar, lo que supone un grave obstáculo para el diagnóstico de enfermedades como el cáncer. Una transformación incompleta con bisulfito llevaría a la amplificación por unión de los cebadores específicos de metilación (complementarios de la secuencia original) y a una pérdida de especificidad. Nuevas técnicas, como la qMSP, permiten cuantificar el grado de metilación de la secuencia amplificada y establecer un umbral de decisión, lo que permite aumentar en gran medida la sensibilidad y la especificidad. Otra forma de mejorarlas consiste en introducir una reacción previa a la MSP (nested MSP); en este caso, esta reacción incorpora cebadores diseñados con el fin de que sólo las secuencias completamente transformadas se amplifiquen8.
APC es un gen supresor tumoral relacionado con la vía Wnt, cuya metilación se ha visto implicada fundamentalmente en la tumorogénesis del carcinoma colorrectal, pero también en el cáncer de pulmón14. Otros estudios en LBA y BAS han detectado frecuencias de metilación globales de APC medidas por qMSP del 29% y del 16%, respectivamente9,11, especialmente en adenocarcinomas. En este caso, APC detectó únicamente un 8%, incluido el único adenocarcinoma presente, lo que se podría explicar por la menor sensibilidad de la técnica empleada.
DAPK es un gen importante en la vía de la apoptosis celular y se ha encontrado inactivado en muchos tipos tumorales15,16. La frecuencia de metilación de DAPK se ha cifrado hasta en un 44% en tejidos de tumores pulmonares17, mientras que en LBA ha sido hasta del 35%13, en ambos casos mediante MSP. En este estudio, y en contra de lo esperado, ninguna de las muestras tumorales en BAS se encontró metilada, lo que se podría explicar por una mayor especificidad analítica de la técnica (nested MSP) y los cebadores empleados a costa de una reducción en su sensibilidad. Sería necesario, sin embargo, un mayor número de muestras para analizar y, así, determinar su verdadera sensibilidad con las condiciones empleadas.
El silenciamiento de RASSF1A es capaz principalmente de bloquear la apoptosis mediada por RAS2. La frecuencia de metilación descrita oscila entre un 18% en LBA de carcinomas de célula no pequeña mediante MSP18, hasta un 28%19 o un 30%9 en BAS de carcinomas de célula no pequeña, y un 83%9 o un 89%19 en BAS de carcinomas de células pequeñas, en ambos casos mediante qMSP. En este estudio se encontraron metilados los 2 únicos carcinomas de células pequeñas (100%) y 7 de los 20 carcinomas de célula no pequeña analizados (35%), lo que está en consonancia con la literatura médica, a pesar de la menor sensibilidad de la técnica empleada.
Las frecuencias de metilación de los 3 genes estudiados difieren de las de otros estudios recientes, como el de Brock et al, que las sitúa en un 36% para APC y DAPK y en un 50% para RASSF1A20, o el de Wang et al, que obtiene un 68% para APC, un 50% para DAPK y un 21% para RASSF1A21. Se debe tener en cuenta, sin embargo, que ambos estudios se realizaron a partir de piezas tumorales de carcinomas de célula no pequeña, en estadio I en el primer caso, y mediante una técnica diferente, como es el caso de los microarrays, en el segundo.
La sensibilidad del panel de metilación empleado fue del 37,5%. La gran proporción existente de carcinomas escamosos entre las muestras analizadas (15 de 24, un 62,5%) frente al 30% teórico, y el hecho de que sea uno de los tumores que ofrece una menor sensibilidad hacen suponer que ésta sería mayor al aumentar el tamaño muestral. DAPK no detectó ninguno de los casos, y los 2 tumores captados por APC fueron también positivos para RASSF1A. Estos hechos llevarían a pensar en la conveniencia de buscar nuevas combinaciones de marcadores, incluido este último y descartados APC y DAPK. Sin embargo, los datos previos recogidos en la literatura médica apoyan su utilidad y evidencian que posiblemente se debería aumentar el tamaño muestral para determinar con mayor exactitud su verdadera sensibilidad con las condiciones empleadas.
Los resultados muestran que ninguna de las 15 muestras libres de tumor primario pulmonar presentó metilación en alguno de los 3 genes. Aunque sería necesario un mayor número de muestras para precisar la verdadera especificidad, los datos apuntan a que la técnica es útil a la hora de evitar falsos positivos.
Como ha sido mencionado, una de las ventajas teóricas de este tipo de diagnóstico es la capacidad de anteceder a la fase sintomática del tumor, lo que se ha demostrado en algunos trabajos22,23. En el caso del cáncer de pulmón, dada su gravedad y tardía presentación clínica, la detección precoz es de especial relevancia. En este estudio se seleccionaron muestras de diferentes estadios tumorales, aunque la dificultad para encontrar muestras en fases tempranas de la enfermedad hizo que la mayoría perteneciera a estadios III–B y IV. La sensibilidad global del panel fue similar en estos 2 estadios (2 de 6 y 4 de 12, respectivamente). Por otra parte, de los 2 tumores en estadio I–B, uno sí se identificó, pero su diagnóstico quedó por determinar tras el fallecimiento del paciente, ya que pudo haberse tratado de una metástasis de tumor otorrinolaringológico. Sería necesario, por tanto, un mayor número de muestras en estadios tempranos para determinar la capacidad del panel en la detección precoz.
La aplicación directa del panel en la práctica clínica se analizó determinando la contribución que ofrecía al diagnóstico correcto cuando las pruebas citológicas en BAS eran meramente sospechosas o discordantes con el diagnóstico final (tabla 4). Estudios de revisión24 han cifrado la sensibilidad de las biopsias y los BAS con cepillado bronquial en un 74% y un 59%, respectivamente, para lesiones centrales y del 46% y del 52% para lesiones periféricas; sensibilidad que disminuye cuando las lesiones son inferiores a 2cm. Trabajos previos ya han tratado de evaluar la aplicación de un panel de metilación como sustitución o complemento de las pruebas citológicas. Chan et al13 observaron que la metilación de los genes p16, RARβ (retinoic acid receptor β), DAPK y MGMT (O6-methylguanine-DNA [deoxyribonucleic acid]-methyltransferase) mediante MSP en LBA concordaba en 15 de 17 (88%) muestras con cáncer de pulmón y citología positiva y, además, detectaba 32 de 39 tumores (82%), incluso en estadios precoces con citología negativa. Sin embargo, también observaron 7 de 39 (18%) falsos positivos. Schmiemann et al9 analizaron la contribución al diagnóstico de un panel de metilación compuesto por los genes APC, p16 y RASSF1A medidos por qMSP. El panel detectó 20 de los 44 tumores (45%) en los que la citología era falsamente negativa, y uno de 4 (25%) en los que había sido dudosa. Además, su aplicación mejoró especialmente la detección de tumores periféricos, menos accesibles. Los resultados del presente trabajo están en consonancia con estos resultados. El panel detectó uno de los 7 tumores con citología negativa (14%) y 3 de los 5 con citología sospechosa (60%). La sensibilidad global fue, por tanto, del 33%. Además, en la muestra con diagnóstico final negativo para cáncer primario de pulmón en la que la citología había sido sospechosa, el panel resultó negativo, conforme a la elevada especificidad que presentaba en las muestras analizadas.
En conclusión, el uso de marcadores epigenéticos de metilación se presenta como un arma potencialmente útil para el diagnóstico del cáncer de pulmón. Este estudio en BAS muestra que, en las muestras analizadas y con las técnicas empleadas, el panel escogido presenta una sensibilidad moderada y una gran especificidad. Además, su rendimiento, junto con las pruebas citológicas, permite considerarlo como una prueba complementaria que podría ser eficaz para su detección, lo que evitaría, por otro lado, la aplicación de técnicas más cruentas o invasivas. Sin embargo, se precisan más estudios y un mayor número de muestras para confirmar su eficacia y reproducibilidad. Por último, el hecho de que hasta el momento, y con las técnicas empleadas, la sensibilidad dependa exclusivamente de RASSF1A, el gran número de carcinomas escamosos presentes y la escasa proporción de muestras en estadios precoces de la enfermedad abren la puerta a nuevos estudios y combinaciones destinados a incrementar su utilidad.
Marc Gallegos Marmolejo ha colaborado en el diseño de los cebadores y en la supervisión técnica.
Este trabajo corresponde a una comunicación científica presentada y premiada en el II Congreso Nacional del Laboratorio Clínico celebrado en La Coruña del 4 al 7 de junio de 2008.