Los programas de cribado de cromosomopatías requieren la utilización de una aplicación informática para calcular el riesgo prenatal en cada una de las gestaciones cribadas. Este software de cálculo incorpora las diferentes fórmulas y parámetros estadísticos necesarios para obtener el riesgo, en términos de probabilidad. A continuación, se describe la aplicación de un simulador de cálculo de riesgo de anomalías cromosómicas fetales en el primer trimestre de gestación, así como la evaluación del rendimiento obtenido con el mismo utilizando diferentes parámetros estadísticos poblacionales.
Chromosomal abnormality screening programs require the use of computer software to calculate the prenatal risk in pregnancy. This software incorporates the different formulas and statistical parameters needed to calculate the risk in terms of probability. Here, we describe the application of a simulator for calculating the risk of chromosomal abnormalities in the first trimester of pregnancy, and the evaluation of the obtained performance by using different population statistical parameters.
El cribado combinado de primer trimestre se basa en la determinación de dos marcadores bioquímicos en suero materno, la fracción β libre de la gonadotropina coriónica humana (fβ-hCG) y la proteína A plasmática asociada al embarazo (PAPP-A), entre las semanas 8 y 13+6 de gestación1 y un marcador ecográfico, la translucencia nucal (TN), entre las semanas 11 y 13+62. Los resultados de estos marcadores, expresados como múltiplos de la mediana (MoM), se modifican con diversos factores de corrección (ej.: peso materno, etnia, hábito tabáquico, gestante diabética insulino-dependiente, reproducción asistida), se normalizan mediante transformación logarítmica y truncaje de valores extremos, y posteriormente se calcula la razón de probabilidad (likelihood ratio, LR) que aportan al embarazo concreto. Finalmente, dicha LR se combina con el riesgo a priori de la gestante (correspondiente a la edad materna), obteniéndose un riesgo de alteración cromosómica fetal, en términos de probabilidad3. Las aneuploidías habitualmente cribadas son la trisomía 21 (T21), o síndrome de Down, y la trisomía 18 (T18), o síndrome de Edwards, ya que son las anomalías cromosómicas fetales con mayor prevalencia4. No obstante, los algoritmos de cálculo de riesgo para estas aneuploidías también pueden aportar elevadas tasas de detección en los casos de síndrome de Turner5, trisomía 136 y en las triploidías7.
Para obtener el riesgo combinado de cromosomopatía se utiliza una aplicación informática, que incorpora las fórmulas matemáticas y parámetros estadísticos en un algoritmo de cálculo. Estos parámetros se estiman a partir de estudios epidemiológicos que recogen datos de un número importante de gestaciones portadoras de fetos afectados y sanos.
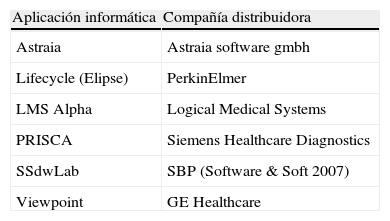
Sin embargo, los programas disponibles comercialmente (tabla 1) suelen ser cerrados y no permiten evaluar objetivamente el impacto de incorporar modificaciones al motor de cálculo. Por este motivo, el objetivo de este trabajo fue desarrollar un simulador de cálculo de riesgo de cromosomopatía con las mismas bases matemáticas que los programas comercialmente disponibles, para evaluar el impacto de la utilización de los parámetros poblacionales publicados en varios estudios epidemiológicos, en el rendimiento de un programa asistencial de cribado. Además, el simulador permitió estudiar la LR que aporta cada uno de los marcadores fβ-hCG, PAPP-A y TN sobre el riesgo a priori, identificando los truncajes más adecuados para que valores extremos de los MoM no produzcan un error en el cálculo. De forma complementaria, dado que se han descrito niveles más elevados de fβ-hCG en gestaciones portadoras de fetos femeninos8,9, se evaluó la utilidad práctica de incorporar un factor de corrección para los valores de fβ-hCG en función del sexo fetal.
Aplicaciones informáticas más utilizadas (en sus diferentes versiones) por los laboratorios participantes en el programa de evaluación externa de la calidad UKNEQAS para el cribado de la trisomía 21 en el primer trimestre de gestación
| Aplicación informática | Compañía distribuidora |
| Astraia | Astraia software gmbh |
| Lifecycle (Elipse) | PerkinElmer |
| LMS Alpha | Logical Medical Systems |
| PRISCA | Siemens Healthcare Diagnostics |
| SSdwLab | SBP (Software & Soft 2007) |
| Viewpoint | GE Healthcare |
Se han descrito previamente comparaciones en cuanto al rendimiento de diferentes estrategias y programas comerciales de cribado10,11. Sin embargo, según nuestro conocimiento, este es el primer estudio en el que se aplica un simulador de cálculo con el objetivo de incrementar el rendimiento de un programa de cribado evaluando parámetros estadísticos de diferentes estudios poblacionales.
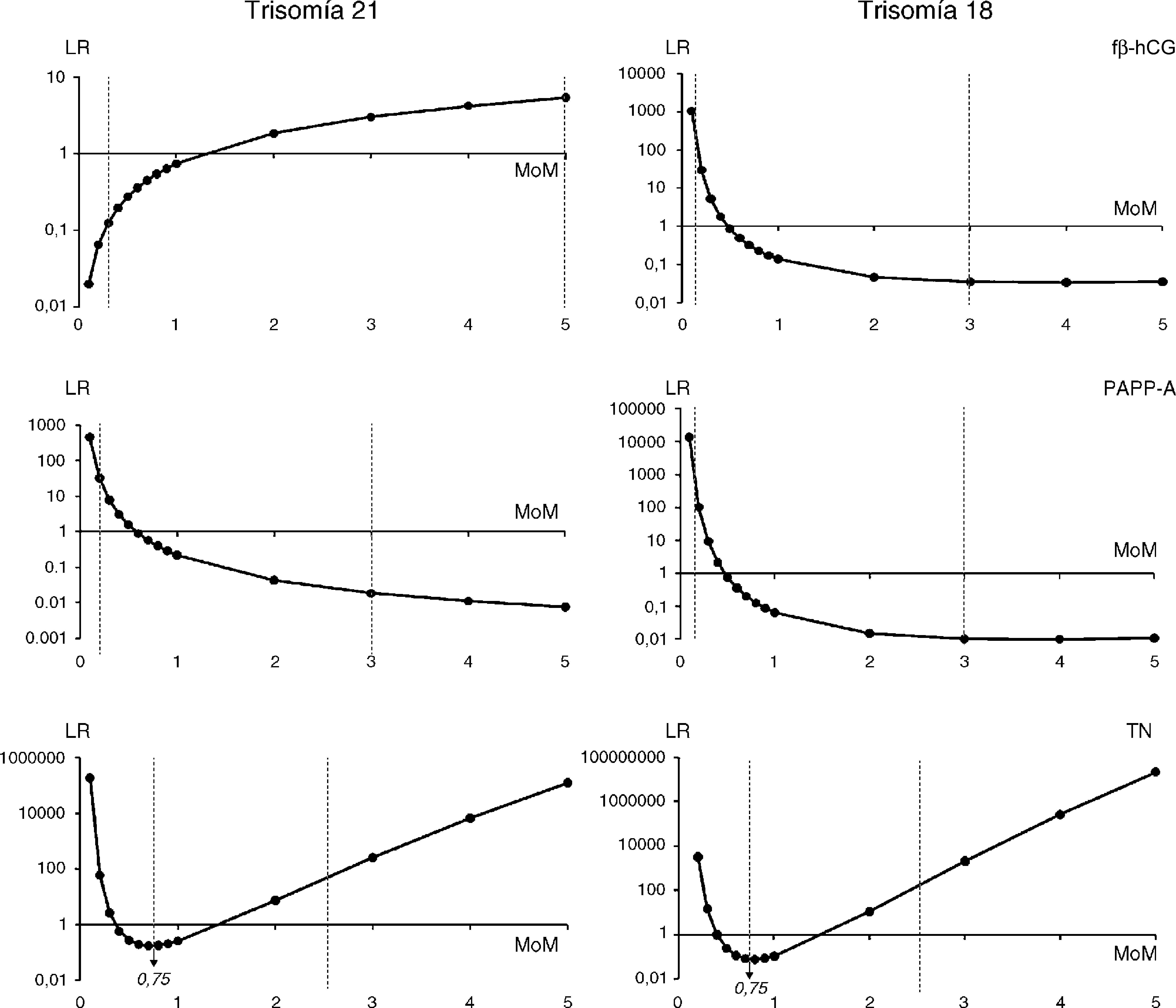
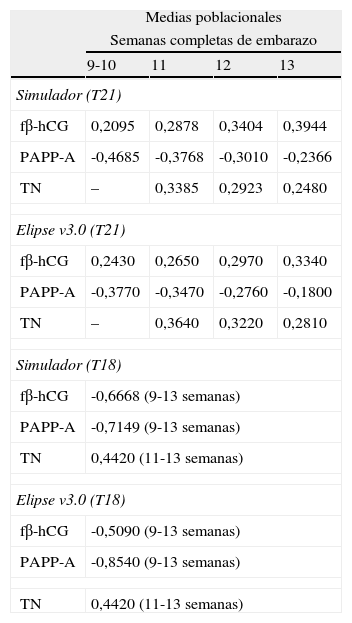
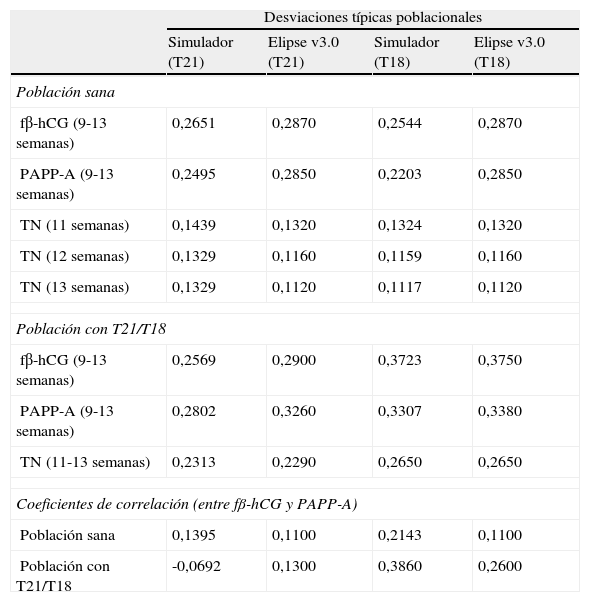
Materiales y métodosSe desarrolló un simulador de cálculo de riesgo de T21 y T18 siguiendo los fundamentos descritos por Reynolds y Penney3, en los cuales se basa el software de cálculo (Elipse v3.0, Perkin Elmer) utilizado asistencialmente en la Unidad Multicéntrica de Cribado de cromosomopatías de Asturias (Servicio de Bioquímica Clínica, Laboratorio de Medicina, Hospital Universitario Central de Asturias). Para ello, se emplearon los programas Microsoft Office Excel 2007 y SPSS 15.0. Para el algoritmo de T21, los parámetros estadísticos poblacionales incorporados al simulador (medias, desviaciones típicas y coeficientes de correlación) fueron los descritos en el estudio SURUSS12,13, mientras que para el algoritmo de la T18, se utilizaron los parámetros bioquímicos de un estudio de la Fetal Medicine Foundation14 (manteniendo los parámetros de Elipse v3.0 para la TN). Entre los parámetros bioquímicos del estudio SURUSS13 se seleccionaron las desviaciones típicas y coeficientes de correlación para fβ-hCG y PAPP-A, con corrección por peso y datación ecográfica. No se utilizaron coeficientes de correlación entre la TN y los marcadores bioquímicos al no ser estadísticamente significativos15. Los parámetros poblacionales utilizados por Elipse v3.0, motor de cálculo comercial que sirvió de comparación en el presente estudio, proceden de dos estudios de Cuckle et al.16, si bien los parámetros utilizados para la T18 por el programa no han sido previamente publicados (tablas 2 y 3). Además, se revisaron los truncajes aplicados a los valores extremos de los MoM de fβ-hCG, PAPP-A y TN, siguiendo el procedimiento descrito por Morris y Wald17 Para ello, se representó la LR obtenida para la trisomía considerada frente a los MoM de cada marcador.
Medias poblacionales (expresadas como log MoM) para fβ-hCG, PAPP-A y TN, entre 9 y 13 semanas de gestación, utilizadas para las trisomías 21 y 18 en el simulador y en Elipse v3.0
| Medias poblacionales | ||||
| Semanas completas de embarazo | ||||
| 9-10 | 11 | 12 | 13 | |
| Simulador (T21) | ||||
| fβ-hCG | 0,2095 | 0,2878 | 0,3404 | 0,3944 |
| PAPP-A | -0,4685 | -0,3768 | -0,3010 | -0,2366 |
| TN | – | 0,3385 | 0,2923 | 0,2480 |
| Elipse v3.0 (T21) | ||||
| fβ-hCG | 0,2430 | 0,2650 | 0,2970 | 0,3340 |
| PAPP-A | -0,3770 | -0,3470 | -0,2760 | -0,1800 |
| TN | – | 0,3640 | 0,3220 | 0,2810 |
| Simulador (T18) | ||||
| fβ-hCG | -0,6668 (9-13 semanas) | |||
| PAPP-A | -0,7149 (9-13 semanas) | |||
| TN | 0,4420 (11-13 semanas) | |||
| Elipse v3.0 (T18) | ||||
| fβ-hCG | -0,5090 (9-13 semanas) | |||
| PAPP-A | -0,8540 (9-13 semanas) | |||
| TN | 0,4420 (11-13 semanas) | |||
Desviaciones típicas (expresadas como log) para fβ-hCG, PAPP-A y TN, entre 9 y 13 semanas de gestación, y coeficientes de correlación entre fβ-hCG y PAPP-A utilizados para las trisomías 21 y 18 en el simulador y en Elipse v3.0
| Desviaciones típicas poblacionales | ||||
| Simulador (T21) | Elipse v3.0 (T21) | Simulador (T18) | Elipse v3.0 (T18) | |
| Población sana | ||||
| fβ-hCG (9-13 semanas) | 0,2651 | 0,2870 | 0,2544 | 0,2870 |
| PAPP-A (9-13 semanas) | 0,2495 | 0,2850 | 0,2203 | 0,2850 |
| TN (11 semanas) | 0,1439 | 0,1320 | 0,1324 | 0,1320 |
| TN (12 semanas) | 0,1329 | 0,1160 | 0,1159 | 0,1160 |
| TN (13 semanas) | 0,1329 | 0,1120 | 0,1117 | 0,1120 |
| Población con T21/T18 | ||||
| fβ-hCG (9-13 semanas) | 0,2569 | 0,2900 | 0,3723 | 0,3750 |
| PAPP-A (9-13 semanas) | 0,2802 | 0,3260 | 0,3307 | 0,3380 |
| TN (11-13 semanas) | 0,2313 | 0,2290 | 0,2650 | 0,2650 |
| Coeficientes de correlación (entre fβ-hCG y PAPP-A) | ||||
| Población sana | 0,1395 | 0,1100 | 0,2143 | 0,1100 |
| Población con T21/T18 | -0,0692 | 0,1300 | 0,3860 | 0,2600 |
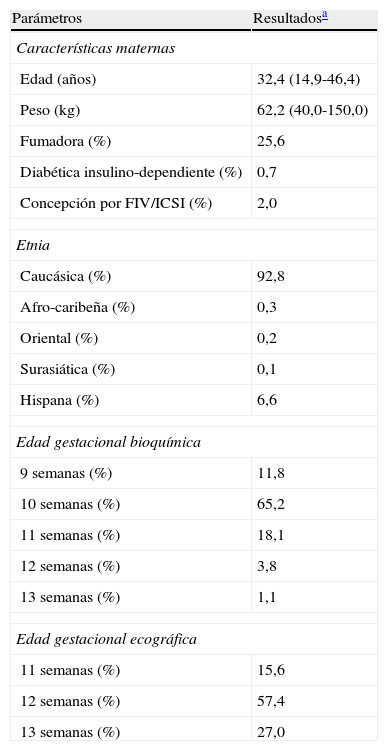
Se realizó un estudio retrospectivo con los resultados correspondientes a las gestantes que participaron en nuestra Unidad Multicéntrica de Cribado Combinado entre el 13 de julio de 2006 y el 31 de diciembre de 2009 (n=15.470), excluyendo las gestaciones gemelares (1,8%; n=279), los casos con medida de TN anterior a la semana 11 (0,3%; n=44) y aquellos en que se desconocía el desenlace del embarazo (3%; n=469). Las principales características de estas gestantes se describen en la tabla 4. Para evaluar el rendimiento obtenido con cada simulador, se incorporaron al cálculo los MoM de fβ-hCG, PAPP-A y TN, así como el riesgo a priori, obtenidos con el software de cribado prenatal Lifecycle (versión 3.0, Perkin Elmer). En el periodo de estudio se diagnosticaron 59 casos de T21, 11 casos de T18 y 22 casos de otras aneuploidías diferentes.
Características de la población a estudio
| Parámetros | Resultadosa |
| Características maternas | |
| Edad (años) | 32,4 (14,9-46,4) |
| Peso (kg) | 62,2 (40,0-150,0) |
| Fumadora (%) | 25,6 |
| Diabética insulino-dependiente (%) | 0,7 |
| Concepción por FIV/ICSI (%) | 2,0 |
| Etnia | |
| Caucásica (%) | 92,8 |
| Afro-caribeña (%) | 0,3 |
| Oriental (%) | 0,2 |
| Surasiática (%) | 0,1 |
| Hispana (%) | 6,6 |
| Edad gestacional bioquímica | |
| 9 semanas (%) | 11,8 |
| 10 semanas (%) | 65,2 |
| 11 semanas (%) | 18,1 |
| 12 semanas (%) | 3,8 |
| 13 semanas (%) | 1,1 |
| Edad gestacional ecográfica | |
| 11 semanas (%) | 15,6 |
| 12 semanas (%) | 57,4 |
| 13 semanas (%) | 27,0 |
Para el estudio de la influencia del sexo fetal en los valores de fβ-hCG y, por lo tanto, en el rendimiento del programa de cribado, se calcularon la sensibilidad y porcentaje de falsos positivos (FP) obtenidos en función del sexo fetal en el cribado de la T21. Además, se utilizó el simulador para aplicar un factor de corrección a los MoM de fβ-hCG de los fetos femeninos y se calculó el impacto de dicha modificación en el rendimiento del cribado.
ResultadosMediante la representación gráfica de la LR en función del valor de los MoM de cada marcador (fig. 1), se identificaron los valores extremos a partir de los cuales el riesgo aportado por el marcador (en términos de LR) no sigue un comportamiento normal. La LR representa el incremento o reducción del riesgo a priori que genera cada marcador en función del valor del MoM. En el caso de la TN, por ejemplo, se observó que tanto para T21 como T18, a valores inferiores a 0,75 MoM se produce una inversión del riesgo generado, lo que indica la necesidad del truncaje en ese valor. En la T18, para los marcadores bioquímicos, se seleccionó un truncaje inferior (0,15 MoM) que no limitara la sensibilidad alcanzada con fβ-hCG y PAPP-A y un truncaje superior de 3 MoM, ya que a valores superiores el riesgo obtenido es constante. Así pues, a partir de la figura 1 se dedujeron los truncajes considerados como más adecuados para cada marcador, que en el caso de la T21 fueron: 0,3 y 5 MoM para fβ-hCG, 0,2 y 3 MoM para PAPP-A y 0,75 y 2,5 MoM para TN. En el caso de la T18, los truncajes seleccionados fueron: 0,15 y 3 MoM para fβ-hCG y PAPP-A y los mismos que para la T21 en el caso de la TN (0,75 y 2,5 MoM).
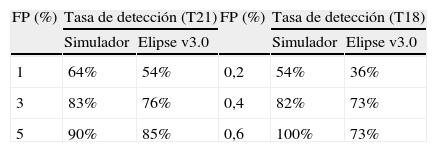
Empleando los truncajes propuestos, y utilizando un punto de corte de 1:250 para considerar un resultado de riesgo combinado como positivo, el simulador generó una mayor tasa de detección en todos los grupos de cromosomopatías. Así, para la T21, Elipse v3.0 detectó 45 de los 59 casos (76%) para un 3,4% de FP, y el simulador detectó 51 casos de T21 (86%) para un 3,7% de FP. Para la T18, Elipse v3.0 detectó 7 de los 11 casos diagnosticados (64%) para un 0,30% de FP, y el simulador detectó todos los casos (100%) para un 0,57% de FP. En cuanto al resto de aneuploidías analizadas, el simulador incrementó la tasa de detección en los casos de síndrome de Turner, pasando de un 75% (6 de 7 casos detectados) a un 88% y además, detectó la única triploidía diagnosticada en el periodo de estudio, la cual había generado un riesgo bajo con Elipse v3.0.
Para confirmar el incremento observado en la sensibilidad con los nuevos algoritmos utilizados en el simulador, se ajustó el porcentaje de FP al 1, 3 y 5% (para la T21) y al 0,2, 0,4 y 0,6% (para la T18) y se calculó la tasa detección obtenida, siendo esta siempre superior al utilizar el algoritmo de cálculo del simulador (tabla 5).
En cuanto al sexo fetal, el 49,7% de las gestantes participantes portaban un feto de sexo femenino. Estratificando los porcentajes de detección y de FP obtenidos con el simulador en función del sexo fetal, se observó una sensibilidad del 84% (27 de 32 casos de T21) para un 3,6% de FP, cuando el feto era masculino, y un 89% (24 de 27 casos de T21) para un 3,8% de FP, cuando el feto era femenino. La mediana de los MoM de fβ-hCG obtenida fue 1,02 (global), 0,91 (en los fetos masculinos) y 1,17 (en los fetos femeninos) con diferencias estadísticamente significativas en función del sexo fetal, según el prueba U de Mann-Whitney (p<0,001). Por tanto, el factor de corrección que habría que aplicar a los valores de fβ-hCG para las gestaciones cuyo feto fuera femenino sería igual a 1,29. Aplicando dicho valor mediante el simulador, el rendimiento global del programa de cribado pasó de tener una sensibilidad del 86% (51 de 59 casos detectados) a un 85%, ya que se detectó un caso menos (50 de 59 casos detectados), con una reducción del porcentaje de FP de un 0,3%.
DiscusiónEl desarrollo de un simulador de cálculo de riesgo de cromosomopatías ha constituido una valiosa herramienta para la evaluación de algunas piezas clave que componen el algoritmo de cálculo, ya que los programas comerciales generan resultados similares pero no idénticos a la hora de calcular el riesgo individual para cada gestante. Estas diferencias cobran importancia en aquellos casos cuyos marcadores no presentan un valor claramente patológico, obteniéndose resultados discrepantes según el programa empleado.
Los truncajes aplicados a los MoM cumplen dos funciones principales: por un lado, evitar que el comportamiento anómalo de un único marcador (valor extremo) provoque un riesgo elevado de trisomía, cuando los otros dos marcadores tienen un valor normal y, por otro, normalizar la distribución del riesgo generada por cada marcador, desde el punto de vista estadístico17,18. Nuestro estudio ha permitido seleccionar truncajes que cumplan estas premisas, con especial relevancia en el algoritmo de la T18, ya que la modificación del truncaje inferior de los marcadores bioquímicos supuso un incremento considerable de la sensibilidad obtenida. Esto se debe probablemente a que los truncajes utilizados por Elipse v3.0 (0,3 y 5 MoM para fβ-hCG, 0,2 y 3 MoM para PAPP-A y 0,5 y 2,5 MoM para TN) son los mismos tanto para la T21 como para la T18, y en los extremos inferiores resultaron menos adecuados que los seleccionados en este trabajo.
En la bibliografía más relevante relativa a programas de cribado combinado, se ha descrito que esta estrategia es capaz de detectar en torno al 90% de los casos de T211,19 y más del 95% de los casos de T1814, para un porcentaje de FP del 5%. Nuestro programa de cribado oficial aún dista de conseguir este rendimiento, si bien el análisis de las medianas de los MoM ha revelado como punto débil la dificultad de homogeneizar y optimizar la práctica de la medida ecográfica de la TN. Esta dificultad ha sido ampliamente descrita, principalmente en programas multicéntricos, donde para alcanzar la eficacia referida en la bibliografía es esencial un adecuado entrenamiento y alta motivación de los ecografistas implicados20. En este contexto, los resultados obtenidos en el presente estudio evidencian que, al margen de esta dificultad inherente a nuestro programa, que será susceptible de mejora con la implantación de estrategias formativas ya en marcha, un algoritmo de cálculo que incorpore las modificaciones aquí descritas habría alcanzado resultados muy similares a los esperados para esta estrategia de cribado, con tasas de detección del 86% y 100% para las T21 y T18, respectivamente, y un porcentaje de FP combinado cercano al 4%.
Según los resultados del presente estudio, el uso de un factor de corrección para los MoM de fβ-hCG cuando el sexo fetal es femenino genera, para el cribado de la T21, una pequeña reducción del porcentaje de FP (0,3%) pero conlleva el riesgo de una pequeña reducción en la sensibilidad obtenida en el cribado. Dado que además implicaría la incorporación de un análisis de sexo fetal en sangre materna, sería imprescindible realizar un análisis de coste-efectividad para poder evaluar si la utilización de este factor corrector comportaría algún beneficio real al cribado de cromosomopatías, como se ha sugerido en estudios previos8,9.
Por otro lado, para valorar adecuadamente los resultados obtenidos en un programa de cribado prenatal es necesario realizar un seguimiento exhaustivo que permita conocer el desenlace del embarazo, así como el estado de salud fetal y/o neonatal. Este aspecto constituye un punto fuerte en nuestro estudio, puesto que en nuestra unidad de cribado se conoce el resultado del parto en el 97% de las gestaciones cribadas hasta ahora.
Como conclusión, la utilización de los parámetros poblacionales generados en dos importantes estudios epidemiológicos junto con unos truncajes adecuados, mejora notablemente el rendimiento de nuestro programa de cribado combinado. Consideramos interesante que cada centro revise estos aspectos de sus programas de cribado para conocer sus limitaciones y puntos fuertes.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Este trabajo corresponde a una comunicación científica presentada y premiada con accésit en el IV Congreso Nacional del Laboratorio Clínico celebrado en Zaragoza del 20 al 22 de octubre de 2010.