El síndrome de inversión duplicación del cromosoma 15 se refiere a un conjunto de características clínicas entre las que se encuentran hipotonía central desde el nacimiento, retraso psicomotor, epilepsia o trastorno del espectro autista. Una invdup(15) resulta de la tetrasomía parcial de 15q y generalmente está implicada la región del Síndrome de Prader-Willi (SPW). Se evalúan tres casos remitidos a Genética por hipotonía y retraso psicomotor.
Material y métodosCultivo de linfocitos de sangre periférica, cariotipo de alta resolución, FISH, extracción de ADN de linfocitos de sangre periférica, MS-MLPA de SPW y estudio de microsatélites.
ResultadosEl primer caso presentó un cariotipo 47,XY+der(15)(q13;p11.2)(pter->q13::p11.2->pter) y un cariotipo molecular arr 15q12.1q13(18,432,358-26,658,490)x3∼4 con ganancia de 8,23Mb implicando a genes sometidos a imprinting de la región causante de los síndromes de PWS y Angelman (SA). En el segundo caso se obtuvo una fórmula cromosómica 47, XX, + mar.ish idic (15)(q13)(Acro p-arm ++, D15Z1 ++, D15S10 ++, PML-) y cariotipo molecular arr 15q11.2q13.3(18,432,358-30,230,511)x3, con duplicación de aproximadamente 12Mb. En el tercer caso la paciente resultó ser portadora de una doble línea celular en mosaico 47,XX+ der (15) inv (15)(q11;p11.2) [40%] / 46,XX [60%]. En los tres casos se analizó mediante MLPA la región del SPW encontrándose un patrón de metilación alterado y la causa genética resultó ser un síndrome invdup(15) «de novo».
DiscusiónA pesar de la dificultad para establecer una correlación fenotipo-genotipo en los casos con invdup(15) las técnicas genéticas más recientes pueden aportar información para el diagnóstico clínico de estos pacientes.
The chromosome 15 inversion duplication syndrome refers to distinctive clinical findings, such as, early central hypotonia, developmental delay, epilepsy and autistic behaviour. Invdup(15) results from partial 15q tetrasomy and the Prader-Willi syndrome(PWS) region is generally involved. We have analyzed three clinical cases in a Genetics Unit diagnosed with hypotonia and developmental delay.
Material and methodsLymphocyte cultures from peripheral blood samples, high resolution karyotype, FISH, DNA isolation from peripheral blood leukocytes, PWS MS-MLPA and microsatellites study.
ResultsThe first case showed a karyotype 47,XY+der(15)(q13;p11.2)(pter->q13::p11.2->pter) and a molecular karyotype arr 15q12.1q13(18,432,358-26,658,490)x3 ∼ 4 with an extra 8.23Mb genetic material involving imprinted genes from SPW and Angelman (SA) syndromes region. In the second case there was a karyotype 47, XX, + mar.ish idic (15)(q13)(Acro p-arm ++, D15Z1 ++, D15S10 ++, PML-) and a molecular karyotype arr 15q11.2q13.3(18,432,358-30,230,511)x3 with an approximately 12Mb duplication. The third patient was a carrier of a mosaic double line cell with karyotype 47,XX+ der (15) inv (15)(q11;p11.2) [40%] / 46,XX [60%]. In the three cases the SPW region was analysed using a modified methylation pattern and all resulted from a invdup(15) «de novo» genetic defect.
DiscussionAlthough it is difficult to establish a phenotype-genotype correlation in invdup (15) cases, most recent genetic techniques should provide information for the clinical diagnosis in these patients.
Las inversiones duplicaciones del cromosoma 15, también llamadas invdup(15), cromosoma 15 isodicéntrico o idic(15) son cromosomas marcadores morfológicamente heterogéneos, que están constituídos por el brazo corto del cromosoma 15 y secuencias proximales 15q. Este reordenamiento genético se ha denominado síndrome invdup(15) y está incluído en la clasificación MIM (www.ncbi.nlm.nih.gov; Mendelian Inheritance in Man; Johns Hopkins University), junto con otras alteraciones cromosómicas que implican una duplicación de la región 15q11-q13 como síndrome duplicación 15q11-q13 (MIM #608636). Han sido descritas diversas manifestaciones clínicas en pacientes con invdup(15), desde la ausencia de síntomas en individuos aparentemente no afectos hasta casos más severos con hipotonía central temprana, retraso en el desarrollo psicomotor y discapacidad intelectual, epilepsia y trastornos del espectro autista1-3. Presenta una prevalencia estimada de 1 por cada 30.000 nacidos con una razón de sexos de 1:14. La invdup(15) es el cromosoma marcador más frecuente. La frecuencia estimada para los cromosomas marcadores es del 0,07% en individuos normales y del 0,3% en individuos con retraso mental, presentándose la invdup(15) en el 0,02%5,6. En pacientes diagnosticados de síndrome de Prader Willi (SPW; MIM #176270) se ha observado la invdup(15) con una alta frecuencia (1/40), a diferencia de lo que ocurre en el Síndrome de Angelman (SA; MIM #105830).
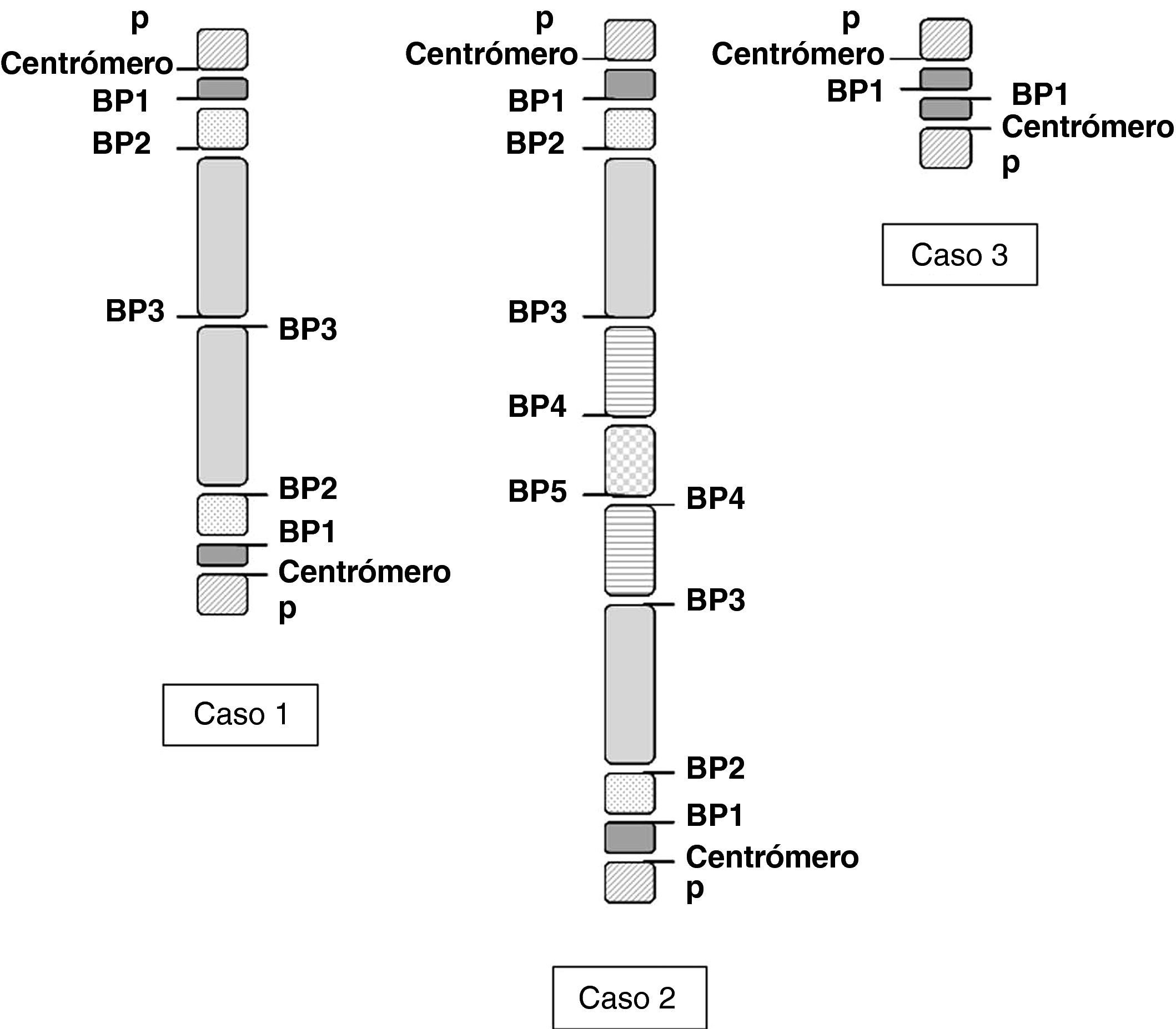
La mayoría de los cromosomas invdup(15) son dicéntricos, con un área de eucromatina entre ellos y pueden incluir o no la región 15q11-q13, relacionada con los síndromes de SPW y SA. En la región 15q11-q13 se han podido caracterizar tres puntos calientes de rotura, que se denominan BP1, BP2 y BP3 (del inglés breakpoints) y en una región más distal se encuentran BP4 (15q13.1) y BP5 (15q13.3, muy próximo a 15q14). Se considera que estos puntos de rotura, formados por duplicones de 50-200kb o LCR (del inglés low copy repeats) están implicados en la mayoría de las reorganizaciones cromosómicas que se producen en el cromosoma 157–9. Se ha demostrado que estos duplicones presentan un número de copias variable en la población normal (CNV) y se considera que algunas variantes polimórficas podrían estar relacionadas con una predisposición a la génesis de los reordenamientos. La presencia de segmentos duplicados en 15q proximal proporciona un mecanismo para la recombinación homóloga, cuyo resultado dependerá del tamaño, la homología, la orientación y la disposición relativa de estos LCR, junto con el tipo de intercambios que puedan ocurrir entre ellos10. La formación de las invdup(15) puede ser por recombinación entre repeticiones invertidas a través de un intercambio intracromosómico, por un intercambio intercromosómico o a través de un intercambio tipo U entre homólogos implicando a BP3 o BP410. Los fenotipos asociados a estos reordenamientos son distintos en función del origen parental de los mismos.
En este trabajo se evalúan tres casos remitidos a la consulta de Genética con diagnóstico clínico de hipotonía y retraso psicomotor.
Materiales y métodosSe presenta el estudio de los siguientes pacientes:
Caso 1: Niño de 3 años. Hijo de padres sanos y hermana de 4 años sana. Parto por cesárea por no progresión. EG:39+6 semanas. PC: 34cm. PRN: 2.870. Apgar: 9:10. Screening metabólico normal. En la revisión pediátrica de los dos meses se objetivó falta de sonrisa social, fijación y seguimiento visual bastante errático y discontinuo e hipotonía axial. Evolucionó con hipotonía y retraso psicomotor, con inicio de sedestación autónoma a los dos años e inicio de la deambulación a los tres. Ausencia de lenguaje hasta los dos años de edad y con tres años dice 4 palabras y presenta estereotipias y un contacto social intermitente compatible con trastorno del espectro autista y deficiencia cognitiva. Desarrollo pondoestatural y fenotipo morfológico normales. Potenciales evocados tronculares: hipoacusia bilateral leve, más acusada en oído izquierdo. ECO transfontanelar, fondo de ojo, EEG de sueño y potenciales evocados visuales, hemograma, bioquímica, amonio, láctico, aminoácidos, ácidos orgánicos, homocisteína, test de toluidina, ácidos grasos de cadena larga y hormonas tiroideas normales. Resonancia magnética cerebral realizada a los 5 meses de vida: inespecífico discreto aumento de espacios pericerebrales frontotemporales.
Caso 2: Niña de 7 años, segunda hija de padres sanos no consanguíneos de origen rumano. Embarazo normal con parto eutócico a término. PRN: 2.500. Periodo neonatal sin incidencias. Presentó importante retraso psicomotor con sedestación a los 15-16 meses y marcha autónoma a los 20 meses; escaso lenguaje: con 7 años máximo junta 3 palabras sin verbos. Perímetro cefálico en la – 2DS, con aplanamiento occipital; resto de exploración normal. Muy dispersa y pobre contacto social, compatible con trastorno del espectro autista y déficit cognitivo. EEG, potenciales evocados auditivos tronculares gases, iones, bioquímica general, hemograma, CK, amonio, láctico, aminoácidos, hormonas tiroideas, 3 hidroxibutirato y ácidos grasos libres normales. Resonancia magnética cerebral a los 4 años y 8 meses normal.
Caso 3: Niña de 8 años remitida a Genética por retraso psicomotor, hipotonía e hiperfagia. Antecedentes de embarazo normal y parto eutócico a término. Periodo neonatal sin incidencias. Screening neonatal normal. Pruebas bioquímicas incluyendo 17-beta estradiol, insulina, somatomedina e IGF-BP-3 y pruebas hematológicas normales. Se había informado de la presencia de un cromosoma marcador no identificado en mosaico en líquido amniótico materno y corroborado en sangre periférica de la paciente.
En los tres casos se informó a los padres sobre el contenido del estudio a realizar en la consulta de Genética y se obtuvo el consentimiento informado tanto para el análisis en ellos como en sus hijos, los cuales se incluyeron en las historias clínicas respectivas.
Estudio citogenéticoSe utilizó el protocolo de Euroclone S.P.A. modificado para el cultivo, sincronización celular y sacrificio de linfocitos en sangre periférica. La técnica de bandeo GTG se realizó según procedimientos estándar con la obtención de 850 bandas correspondientes al cariotipo de alta resolución y la hibridación in situ fluorescente (FISH) se llevó a cabo según las instrucciones del fabricante. Para la técnica FISH se trató una muestra cromosómica preparada en una porta con 10μl de solución de hibridación formada por 7μl de tampón (Vysis, Abbott), 2μl de agua desionizada estéril y 1μl de la sonda específica (Vysis, Abott). En los casos combinados de dos sondas se añadieron 0,5μl de cada una. Se utilizaron las sondas LSI D15S10 (Vysis, Abbott) de la región SPW/SA que incluye D15Z1 como control 15p11.2 y PML como control 15q22 y la sonda acro p-arm (Vysis). Los portas se incubaron durante toda la noche en un baño termostatizado equilibrado (Hybrite, Vysis) a 37°C. Tras la incubación se realizaron dos lavados, dos minutos a 73°C con solución de lavado 0,4X SSC/0.3% NP-40 pH 7,0-7,5 y un minuto con solución de lavado 2X SSC/0,1% NP-40 pH 7,0-7,5 a temperatura ambiente. Como marcador de fluorescencia se utilizó DAPI, 10μl para cada zona de hibridación y se visualizó en un microscopio de fluorescencia Nikon Eclipse 90i a los 30 minutos, analizando 50 metafases en cada caso. El análisis se realizó con el software Ikaros karyotyping system v.5.3 (Metasystems, GMBH, Alemania).
Estudio molecularA partir de sangre periférica extraída con EDTA como anticoagulante se obtuvo ADN de los pacientes mediante extracción automatizada (Robot EZ1, Qiagen). El estudio de SPW se llevó a cabo mediante la técnica Methylation Specific MLPA (MS-MLPA), según instrucciones del fabricante (MRC Holland, Amsterdam, The Netherlands). El kit comercial MS-MLPA P028 (PWS/AS) (MRC Holland, Ámsterdam, Países Bajos) permite el cribado de grandes reordenamientos genéticos en los genes de la región 15q11-q13 mediante dos reacciones de PCR, así como el estudio de metilación del gen SNRPN mediante digestión con la enzima HhaI. Los fragmentos resultantes se analizaron en un secuenciador ABI PRISM 310 mediante el software Genescan (Applied Biosystems) y los datos fueron normalizados en una hoja Excel. Posteriormente, en los casos con alteración en la metilación, se estudiaron los microsatélites D15S10, D15S11, D15S113, D15S128, GABRA3 y GABRB 5 mediante amplificación por PCR según protocolos descritos, electroforesis capilar en un secuenciador ABI310 (Applied Biosystems) y análisis utilizando el software Genescan (Applied Biosystems). El análisis de hibridación genómica comparada mediante microarray (CGH array) se realizó en QGenomics (Barcelona, España). La hibridación se realizó sobre un chip que contiene aproximadamente 60.000 sondas oligonucleotídicas (qChip Post [8x60K]) repartidas por todo el genoma, con mayor cobertura en regiones pericentroméricas, subteloméricas e implicadas en trastornos genómicos recurrentes. Tras la hibridación y lavados, se procedió al escaneado del chip y a la obtención de la imagen del chip, que fue procesada y analizada utilizando el software Genomic Workbench 5.0 de Agilent.
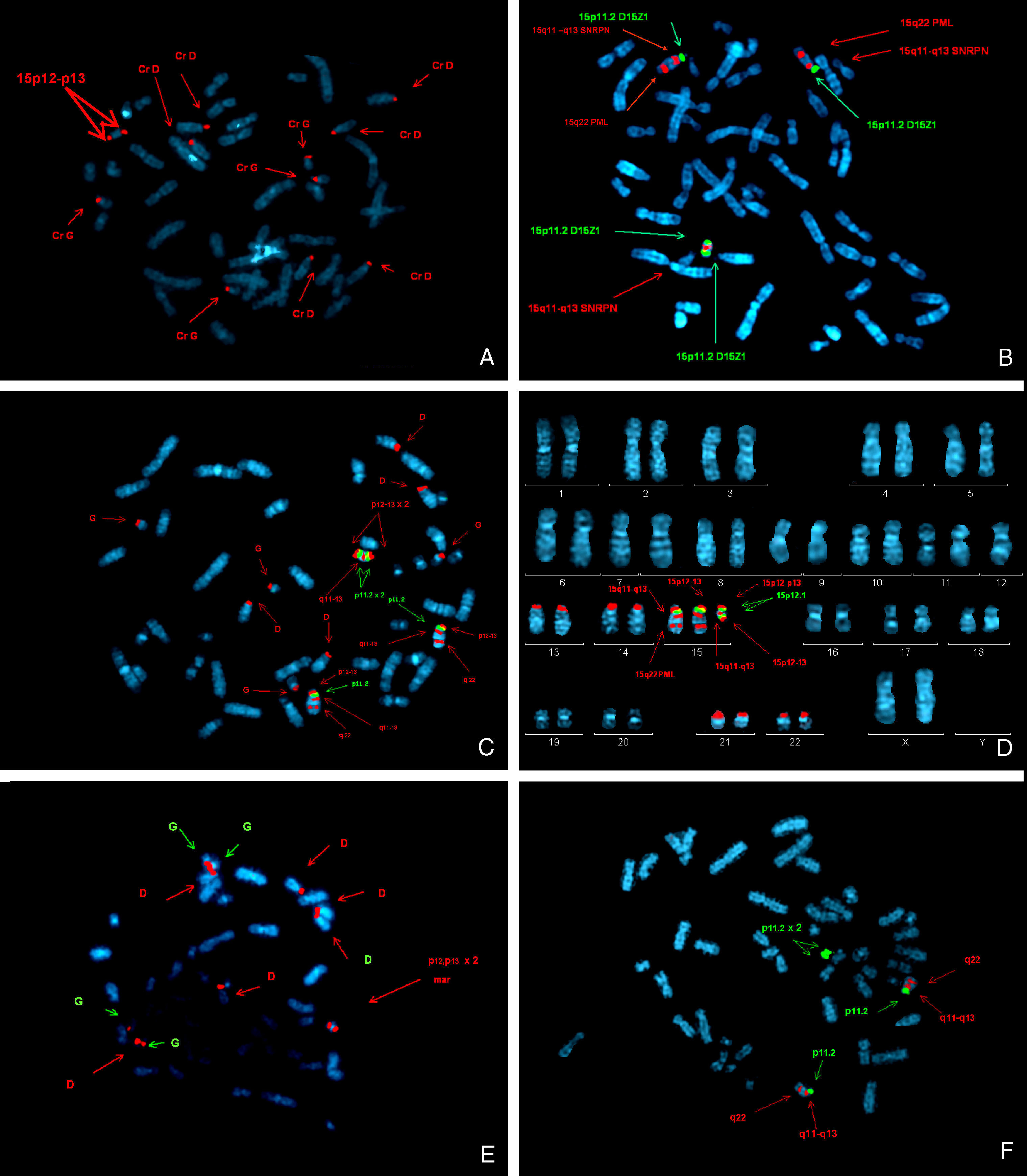
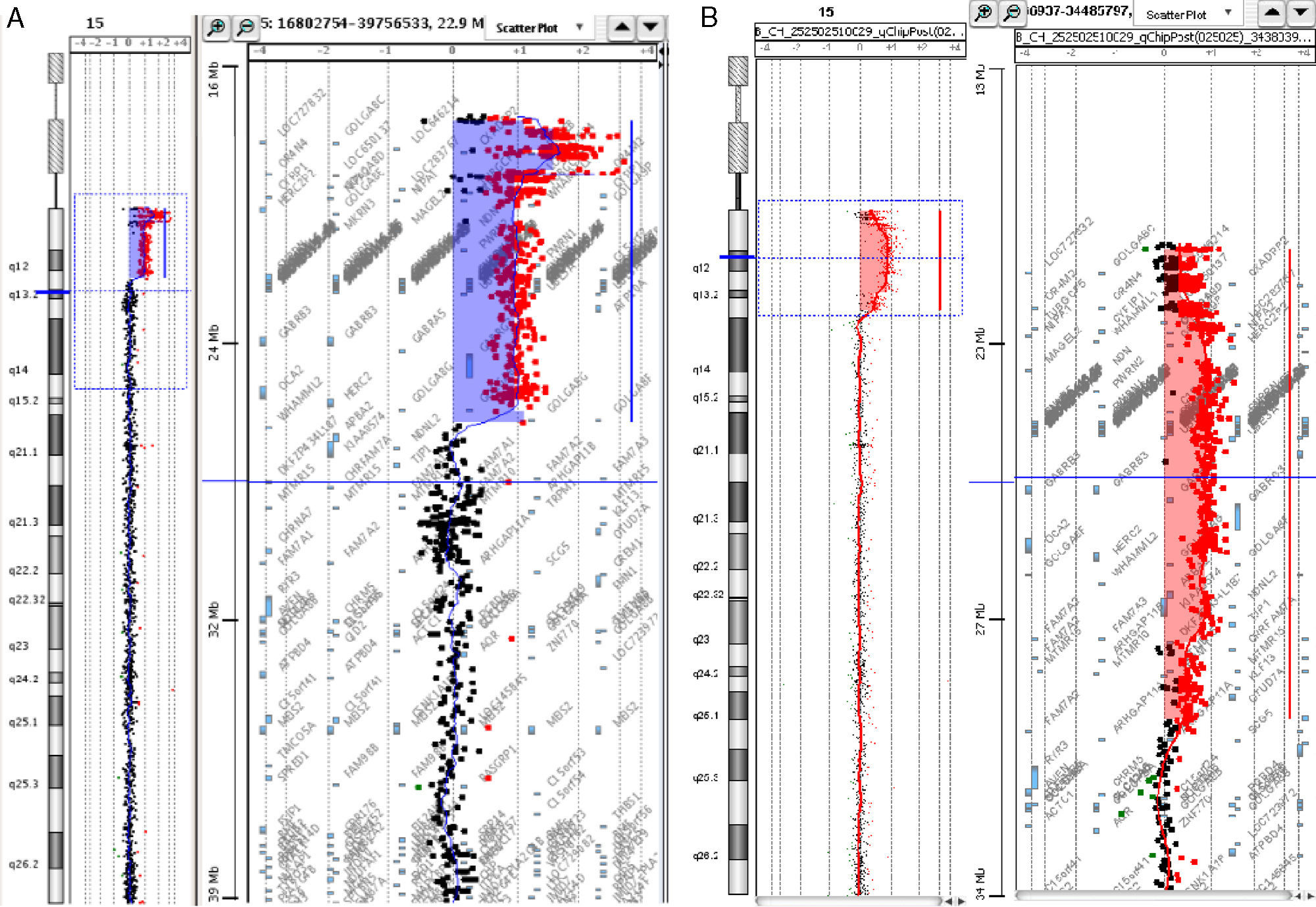
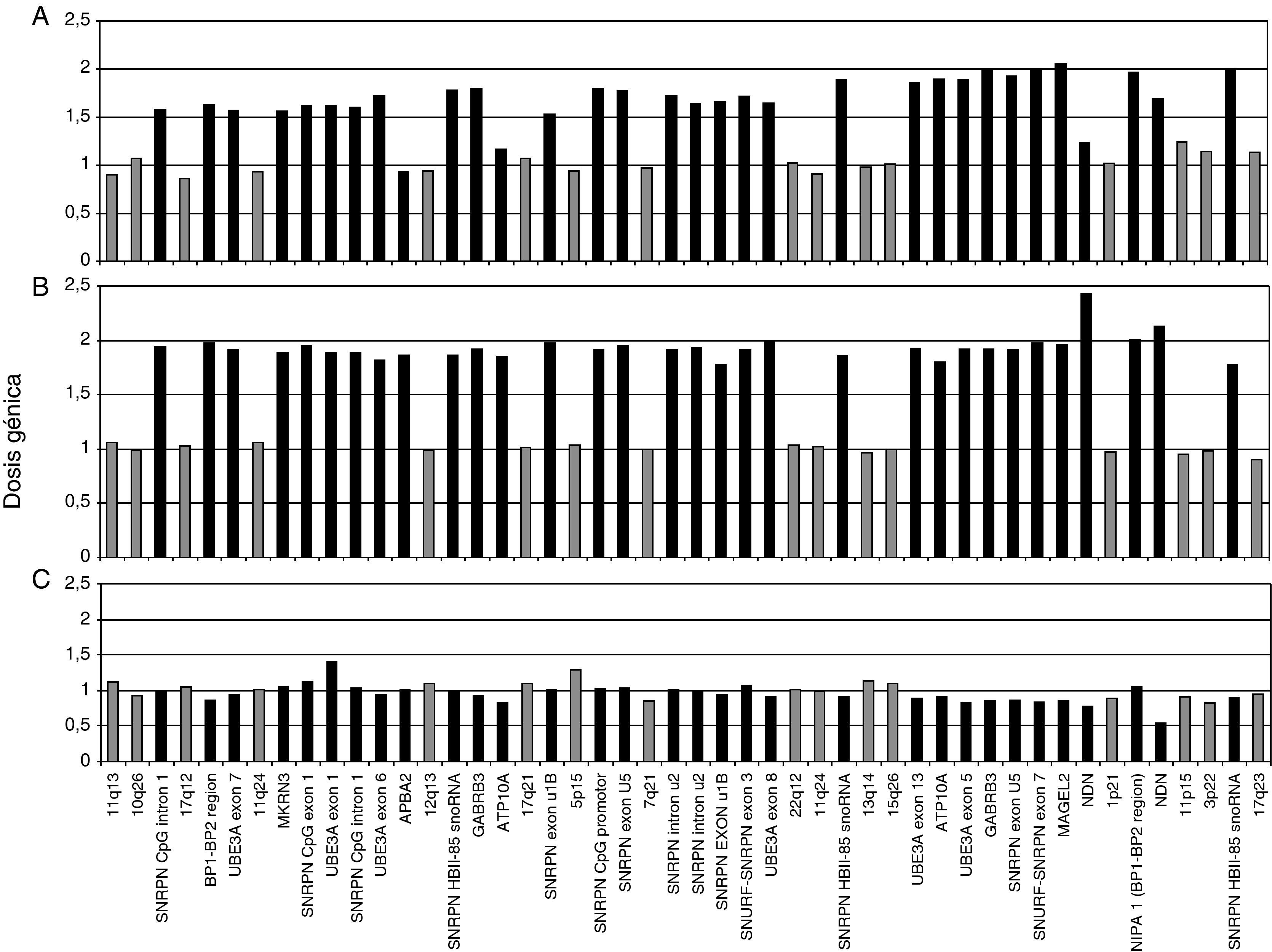
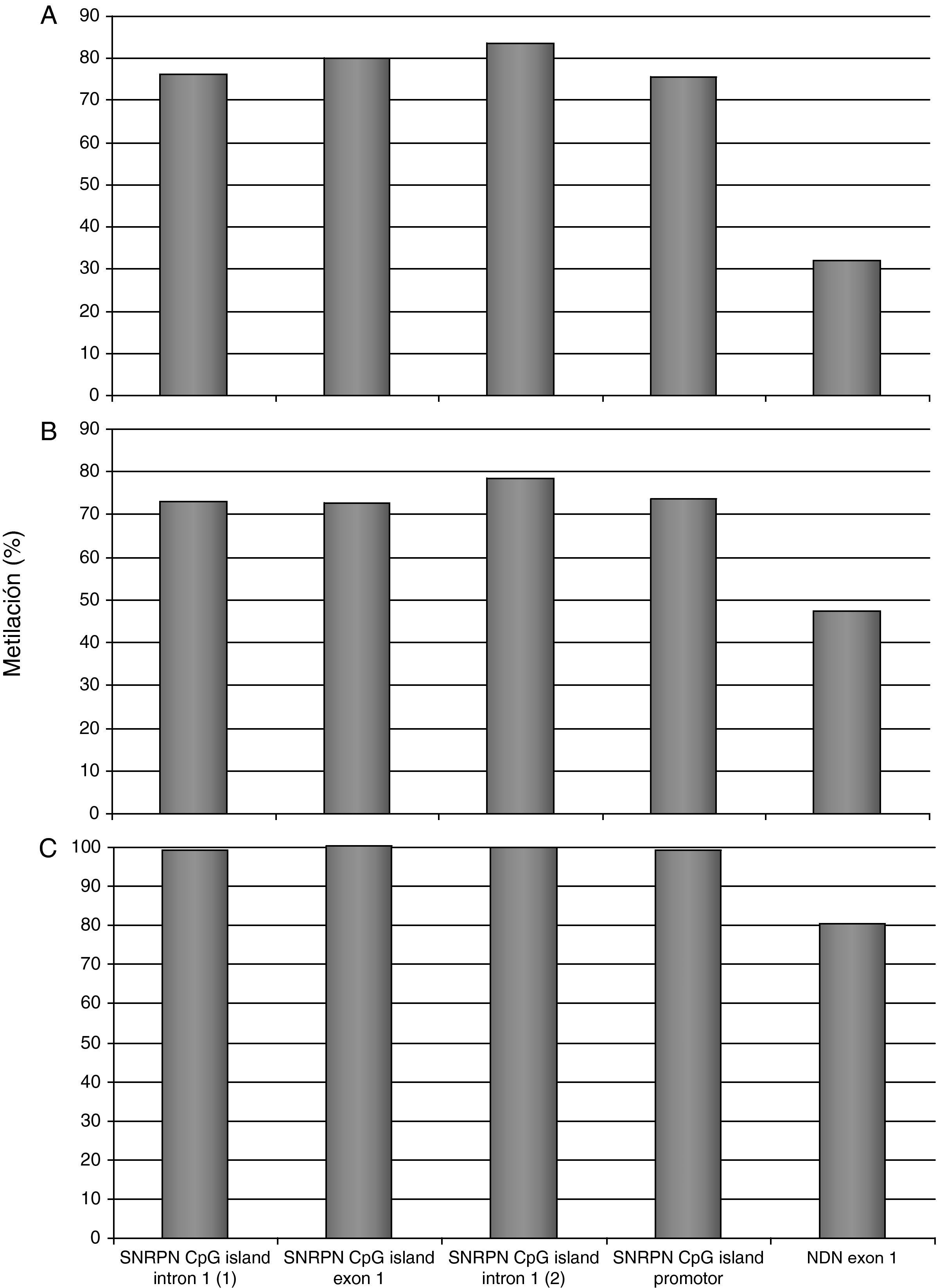
ResultadosCaso 1La fórmula cromosómica obtenida fue 47,XY + der (15)(q13;p11,2)(pter→q13:: p11,2→pter) (fig. 2A y B) y el cariotipo molecular: arr 15q11.2q13.1(18,432,358-26,658,490)x3 ∼ 4 (fig. 3A). Según el análisis CGH array la ganancia de material genético se extiende a lo largo de 8,23Mb (hasta el BP3 de la deleción de PW/AS) y afecta a genes sometidos a imprinting de la región causante (por deleción) de los síndromes de SPW (SNRPN) y SA (UBE3A). Este análisis no aporta información sobre la dosis génica comprendida en regiones de heterocromatina centroméricas del cromosoma 15. Las sondas FISH acro-p-arm (15p12-p13) (fig. 2A) y la sonda control D15Z1 (15p11.2) (fig. 2B) mostraron cuatro señales en 15p, revelando la existencia de tetrasomía en el brazo corto del cromosoma 15. El análisis por MLPA corroboró la existencia de duplicación en la región 15q11-q13 sin poder determinar si se trataba de tri o tetrasomía (fig. 4A). La dosis génica de APBA2 situado en 15q13.1, el gen más distal analizado por MLPA, resultó normal, lo cual corrobora que el cromosoma extra está formado por una duplicación del cromosoma 15 con rotura y unión en BP3. El análisis MS-MLPA (PWS) también mostró una alteración en la metilación del gen SNRPN (fig. 5A) y el análisis de microsatélites (informativos para D15S128 y GABRB3) puso de manifiesto la existencia de heterodisomía uniparental materna.
Hibridación in situ fluorescente (FISH) mediante la sonda LSI D15S10 (Vysis, Abbott) de la región SPW/SA (SNRPN en 15q11-q13), en color rojo, que incluye D15Z1 como control 15p11.2 (verde) y PML como control 15q22 (rojo) y marcaje con sondas acro p-arm (Vysis) (rojo). Se identifica en todas las metafases (A y B: caso 1; C y D: caso 2; E y F: caso 3) el cromosoma marcador como una invdup(15). A, C y E: metafases con marcaje FISH mediante la sonda acro p-arm (Vysis) (rojo) de los centrómeros de los cromosomas D y G. Se observa la presencia de dos señales en los extremos del cromosoma marcador identificado como una invdup(15) que se corresponden con la region p12-p13 del cromosoma 15. B, D y F: metafases con marcaje FISH mediante la sonda LSI D15S10 (Vysis, Abbott) de la región SPW/SA (SNRPN en 15q11-q13), en color rojo, que incluye D15Z1 como control 15p11.2 (verde) y PML como control 15q22 (rojo). Las figuras C y D presentan la hibridación conjunta de las sondas LSI D15S10 y acro p-arm. La metafase ordenada correspondiente a los cromosomas 1 al 22 más los cromosomas sexuales se muestra en la figura D. En las figuras E y F se identifica el cromosoma extra formado por material genético del cromosoma 15 que no incluye la región del Prader-Willi.
Análisis de hibridación genómica comparada mediante array (CGH array). A (caso 1): arr 15q11.2q13.1(18,432,358-26,658,490)x3 ∼ 4. La ganancia de material genético se extiende a lo largo de 8,23 Mb desde la región 15q11 hasta 15q13.1 (hasta el BP3 de la deleción de PW/AS) y afecta a genes sometidos a imprinting de la región causante (por deleción) de los síndromes de SPW (SNRPN) y SA (UBE3A). B (caso 2): arr15q11.2q13.3(18,432,358-30,230,511)x3. Se corresponde con una trisomía de aproximadamente 12Mb de material procedente del extremo más proximal del brazo largo del cromosoma 15, desde la región 15q11 a 15q13.3.
Dosis génica (DG) de la región 15q11-q13 y genes adyacentes, obtenida tras la normalización de los datos del análisis MLPA. Genes analizados (¿) y controles internos (¿). A (caso 1), B (caso 2) y C (caso 3). En los casos 1 y 2 los genes analizados presentan DG=2, correspondiente a una tetrasomía. En el caso 3 los genes analizados presentan DG=1, correspondiente a una dosis génica normal (diploide).
Análisis de metilación por MS-MLPA de los genes sometidos a imprinting en la región 15q11-q13. A (caso 1), B (caso 2) y C (caso 3). En los tres casos se observa un patrón de metilación alterado para el gen SNRPN. El grado de metilación normal para el gen SNRPN es el 50%, que se corresponde con la metilación del alelo materno.
La paciente presentó un cariotipo 47, XX, + mar.ish idic (15)(q13)(Acro p-arm ++, D15Z1 ++, D15S10 ++, PML-) (fig. 2C y D). Las sondas FISH acro-p-arm (15p12-p13) y la sonda control D15Z1 (15p11.2), estudiadas conjuntamente en este caso, revelaron cuatro señales en 15p (fig. 2C y D), marcando una tetrasomía en el brazo corto del cromosoma 15 de forma análoga al caso 1. El CGH array informó un cariotipo molecular arr15q11.2q13.3(18,432,358-30,230,511)x3 (fig. 3B) que se corresponde con una trisomía segmentaria de aproximadamente 12Mb de material procedente del extremo más proximal del brazo largo del cromosoma 15 y altera la dosis de más de un centenar de genes, muchos de ellos formando parte de OMIM. El análisis detallado de los perfiles de la duplicación sugiere un distinto grado de amplificación dentro de esta, lo cual es compatible con la tetrasomía del fragmento localizado desde el centrómero hasta BP4 y la trisomía del fragmento entre BP4 y BP5, como ha sido descrito previamente en la literatura8. Este resultado se corresponde con lo observado en el análisis por MLPA (fig. 4B) con tetrasomía parcial del cromosoma 15 que incluye la región 15q11-q13 del SPW y el gen adyacente APBA2 en 15q13.1. En el análisis MLPA no están incluídos los genes entre BP4 y BP5. Además el análisis MS-MLPA del gen SNPRN reveló un grado de metilación alterado en este gen (fig. 5B), lo cual sugiere como causa más probable el origen materno del cromosoma invdup(15), lo cual en este caso no se pudo comprobar por microsatélites ya que resultaron no informativos.
Caso 3El caso clínico 3 se corresponde con una invdup(15) pequeña en mosaico que no incluye la región SPW/SA (fig. 2E y F). Es un pequeño marcador formado prácticamente de heterocromatina y de aspecto metacéntrico. Presenta un punto de ruptura en 15q11 que se corresponde con los puntos de rotura proximales BP1-BP2 de las deleciones SPW/SA11. Se había identificado la presencia de un cromosoma marcador en mosaico (40%), no identificado previamente a este estudio, en cariotipo de líquido amniótico y en cariotipo de sangre periférica tras el nacimiento. Después de realizar un nuevo cariotipo se corroboró que un 40% de las metafases examinadas, tras cultivo de linfocitos, presentaban 47 cromosomas. Tras aplicar bandas convencionales GTG y QFQ se observó un cromosoma extra que se correspondía con un reordenamiento parcial del cromosoma 15. El 60% restante de las metafases presentaron dotación cromosómica normal. La paciente resultó portadora de una doble línea celular en mosaico. En un 40% de las metafases se comprobó la presencia de un cromosoma extra identificado como una duplicación del cromosoma 15 con rotura y reunión en las bandas 15q11 de dicho cromosoma, con la región 15p12-p13 en los extremos más distales, como indica la sonda acro-p-arm (fig. 2E). La formula cromosómica en esta paciente es 47,XX+ der (15) inv (15)(q11;p11,2) [40%]/46,XX [60%] «de novo» (fig. 2E y F). En dicho reordenamiento no está implicada la región del SPW (fig. 2F), lo cual se comprobó mediante la dosis génica obtenida por MLPA (fig. 4C). Sin embargo, se demostró mediante el análisis de metilación por MS-MLPA (fig. 5C) y microsatélites (informativos para D15S128 y GABRB5) (fig. 6) la existencia de disomía uniparental materna. El análisis por MLPA no mostró dosis génica alterada en la región BP1-BP2 (fig. 4C). Esto puede ser debido a que el cromosoma, formado principalmente por heterocromatina, puede haberse generado por el punto de rotura en BP1 y no hay sondas en el análisis por MLPA situadas entre el centrómero y BP1 o bien a que este grado de mosaicismo no se detecta por esta técnica. El CGH array tampoco mostró alteraciones en la dosis génica, debido a limitaciones técnicas similares. En los tres casos se demostró que se trataba de una alteración «de novo» mediante el análisis en muestras parentales.
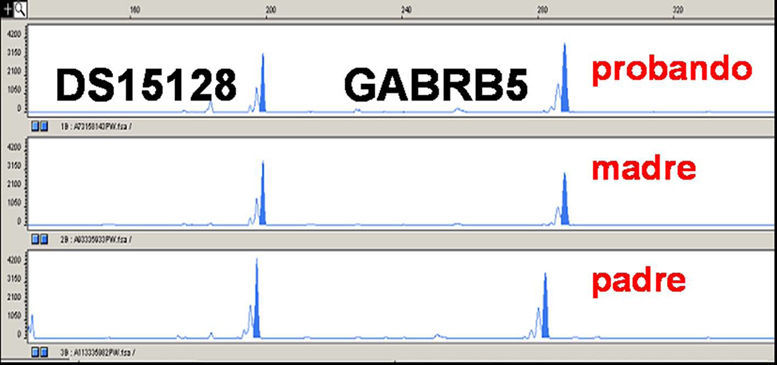
Análisis de microsatélites (caso 3) para la confirmación de la disomía uniparental materna (software Genescan; Applied Biosystems). Se muestra el electroferograma de los microsatélites D15S128 y GABRB5 para la probando y cada progenitor. Se puede observar la ausencia de contribución paterna para ambos microsatélites con presencia de heterodisomía uniparental materna en la probando.
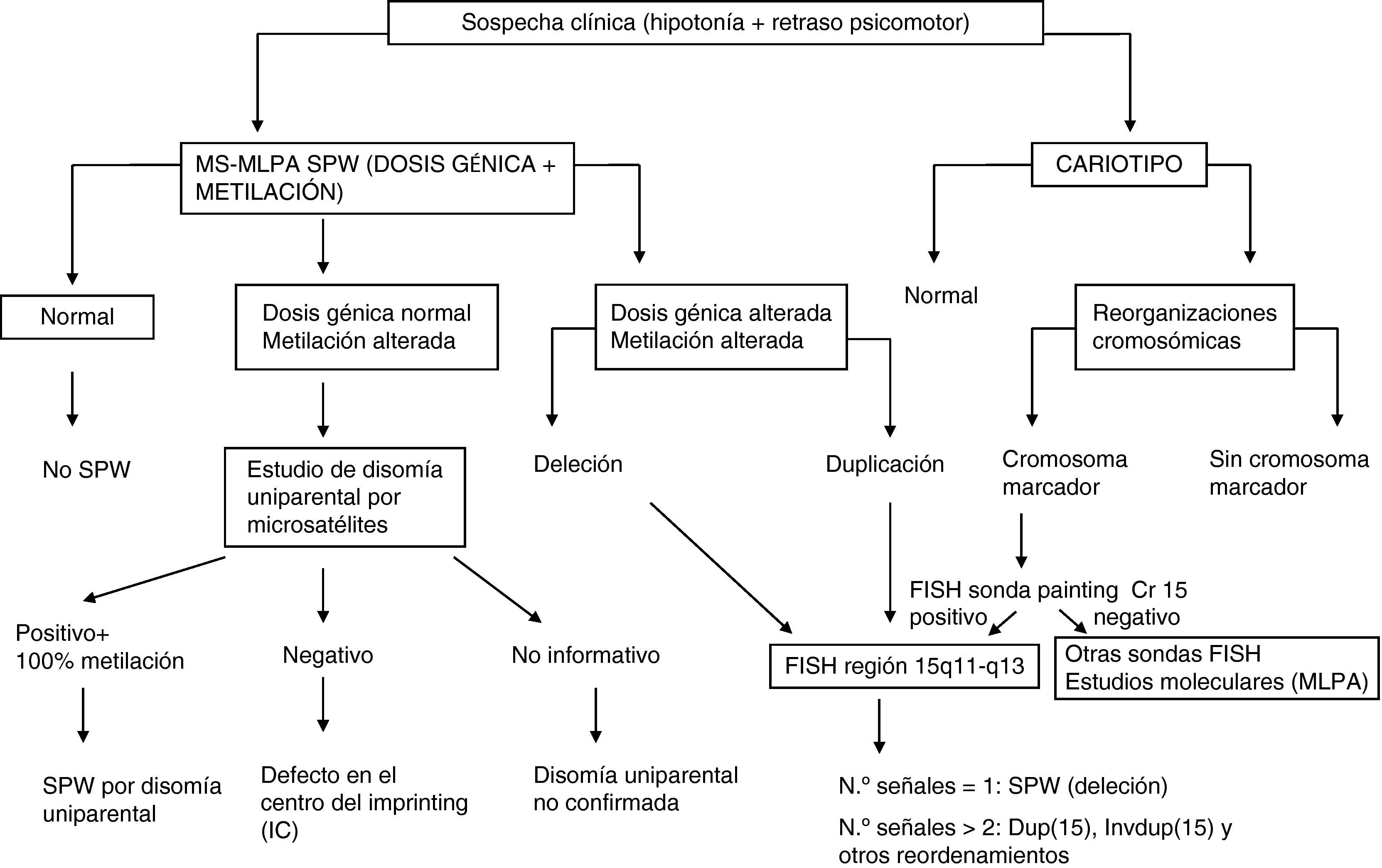
El síndrome invdup(15) es la causa más frecuente de reordenamiento genético con cromosoma extra marcador, representando entre el 35 y el 50% de los casos6,12. Sin embargo, a pesar de su relativa frecuencia, es un síndrome no muy estudiado, con unos criterios clínicos todavía sin definir. Las invdup(15) que incluyen la región SPW/SA son cromosomas que presentan dos centrómeros, cromatina entre ellos y de aspecto submetacéntrico o acrocéntrico, con puntos de rotura en 15q13 (BP3) o 15q14 (BP4). Se han descrito familias con invdup(15) presentes en dos generaciones13 pero en la mayoría de los casos de trata de alteraciones «de novo» y de origen materno. En este grupo estarían incluídos los casos 1 y 2. En el caso 1 el cromosoma extra está formado por una duplicación del cromosoma 15 con rotura y unión en BP3 (fig. 7), a diferencia del caso 2, donde el cromosoma extra estaría formado por dos cromosomas 15 con rotura en BP4 y BP5, respectivamente (fig. 7). Este último es la forma más frecuente de idic(15) formado por recombinación asimétrica entre BP4 y BP58. Es interesante señalar que el punto de rotura del caso 1 se encuentra en la posición del pseudogén HERC2P9 (fig. 1). Este pseudogén pertenece a una subfamilia cuyos miembros han evolucionado a partir del gen HERC2 por duplicación parcial y transposición de una serie de secuencias, las cuales forman parte de los LCR identificados en los puntos de rotura que generan inestabilidad cromosómica en el cromosoma 15. Ambos son casos «de novo» y en ambos el patrón de metilación del gen SNRPN se mostró alterado, lo cual sugiere el origen materno del cromosoma extra. Además, en el caso 1 se pudo demostrar el origen materno mediante análisis por microsatélites. El caso 3 muestra un cromosoma extra en mosaico que estaría formado por dos cromosomas 15 con rotura y reunión de ambos en BP1 (fig. 7). En la literatura hay casos descritos, tanto «de novo» como familiares, portadores de una invdup(15) pequeña sin incluir la región SPW/SA y están asociados tanto a pacientes SPW/SA como a individuos fenotípicamente normales. En los casos de pacientes SPW o SA que presentan estas invdup (15) pequeñas, como es el caso SPW de la paciente del caso clínico 3, se ha demostrado que el fenotipo observado es debido a disomía uniparental del cromosoma 15, de origen materno en la mayoría de los casos, como sucede en el caso clínico 3, o a deleción 15q11-q13 y no a la presencia de la invdup(15)14,15. Además, en este caso al tratarse de una doble línea celular en mosaico (40%), es esperable un fenotipo menos severo16,5. Se puede concluir la importancia del estudio mediante cariotipo y MLPA para los casos con hipotonía y retraso psicomotor, ya que, además de las alteraciones genéticas responsables del síndrome de Prader Willi, pueden presentar una duplicación inversión del cromosoma 15. En la figura 8 se muestra el algoritmo de diagnóstico genético propuesto para pacientes con diagnóstico clínico de hipotonía y retraso psicomotor. Es recomendable el estudio parental, ya que permite valorar si se trata de una alteración heredada o «de novo». Tampoco se puede descartar en los casos «de novo» la existencia de un mosaicismo germinal parental. Estos casos proporcionan, junto con los casos descritos previamente en la literatura, evidencia de la necesidad de analizar la disomía uniparental en los casos con cromosoma marcador supernumerario, en particular cuando se trata del cromosoma 15, en el diagnóstico prenatal y en el posnatal con fenotipo clínico. La ocurrencia simultánea de dos sucesos relativamente raros como son el cromosoma invdup(15) y la disomía uniparental, descrita en casos similares en la literatura, se considera que debe tener una relación causal. Existen teorías sobre la génesis de la disomía uniparental materna y la invdup(15) por errores de segregación en meiosis I y la ausencia de contribución paterna se explica por un gameto nulisómico o por rescate de una aneuploidía17,18, aunque actualmente se considera la posibilidad de un evento tanto pre- como pos- recombinación19. Por otra parte, la extensión del material genético extra determinada por CGH array y el origen parental del mismo puede ayudar al asesoramiento genético, ya que parece existir una correlación entre las características clínicas y la dosis génica de los genes expresados20, muchos de ellos con impronta genómica21. Recientemente se han encontrado ocho nuevos locus relacionados con metilación diferencial en el cromosoma 15, lo cual puede ser útil para valorar el grado de expresión génica en estos casos22. Son muchos los genes en 15q11-q13 cuya sobreexpresión se relaciona con el autismo. Se ha sugerido que la tetrasomía de los genes de las subunidades α5 y β3 del receptor ácido gamma-aminobutírico (GABA) podría alterar la actividad del receptor GABA, uno de los principales mecanismos inhibitorios del sistema nervioso central1,23,24. Sin embargo, no se ha podido establecer una correlación genotipo-fenotipo hasta el momento dada la complejidad y el carácter heterogéneo de este síndrome.
Mapa genético correspondiente al segmento del cromosoma 15 desde el centrómero hasta 15q14 (no a escala). La genes se han distribuído de acuerdo con su posición en el ensamblaje del genoma humano NCBI36/hg18 (www.genome.ucsc.edu) Se muestran los puntos de ruptura BP1 y BP2 (15q11), BP3 (15.13.1) y los puntos más distales BP4 (15q13.3) y BP5 (próximo a 15q14), implicados en los reordenamientos invdup(15) más frecuentes.
Los autores declaran no tener ningún conflicto de intereses.
A los pacientes presentados en este trabajo y a sus padres por su colaboración. A todo el personal del Servicio de Bioquímica Clínica del Hospital Universitario Miguel Servet. Especialmente a los miembros de la Sección de Genética y al Dr. Angel García de Jalón, Jefe del Servicio de Bioquímica Clínica. A los facultativos del Servicio de Pediatría del Hospital Universitario Miguel Servet, especialmente a Neuropediatría. Al Dr. Armengol de la empresa QGenomics.
Este trabajo corresponde a una comunicación científica presentada y premiada con el tercer premio en el IV Congreso Nacional del Laboratorio Clínico celebrado en Zaragoza del 20 al 22 de octubre de 2010.