Desarrollar un modelo que prediga con la mayor exactitud posible el estado vital a los 6meses tras fractura de cadera, utilizando para ello cinco fuentes de datos obtenidas a través del Registro Nacional de Fracturas de Cadera, la Unidad de Gestión Sanitaria y la Dirección Económica.
Material y metodologíaLa población de estudio es una cohorte de pacientes que sufrieron fractura de cadera entre mayo de 2020 y diciembre de 2022. A partir de cinco fuentes diferentes de datos se crea un almacén con las variables necesarias. Se realiza un análisis de valores perdidos y atípicos, así como de desbalanceo de las clases de la variable objetivo («estado vital»). Se entrenan 14 diferentes modelos algorítmicos con los datos de entrenamiento. Se selecciona el modelo que mejor rendimiento obtenga y se realiza una puesta a punto fina. Finalmente se analiza el rendimiento del modelo con datos de test.
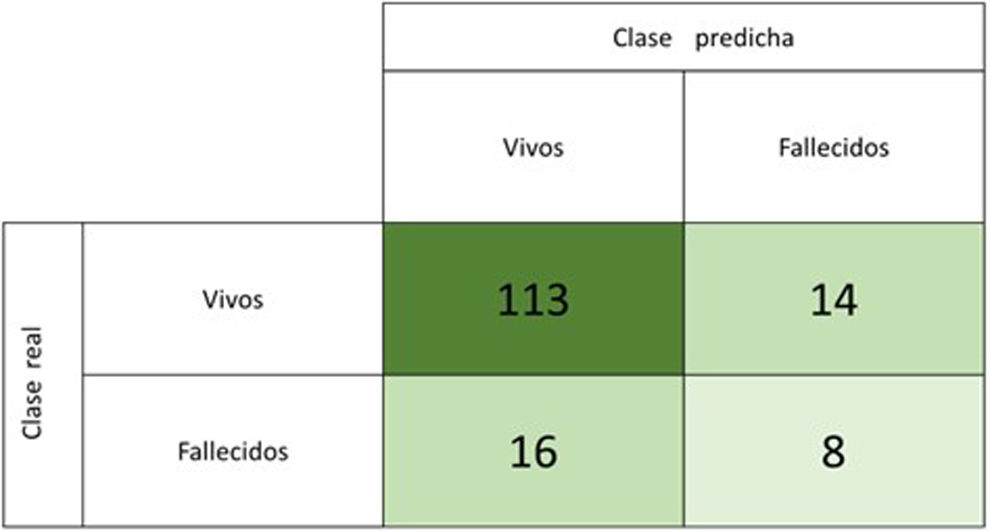
ResultadosSe crea un almacén de datos con 502 pacientes y 144 variables. El modelo con mejor rendimiento es la regresión lineal. Dieciséis de los 24 casos de pacientes fallecidos son clasificados como vivos, y 14 pacientes vivos son clasificados como fallecidos. Se consigue una sensibilidad del 31%, una precisión del 34% y un área bajo la curva de 0,65.
ConclusionesNo se ha conseguido generar un modelo de predicción de éxitus a los 6meses con nuestra cohorte. Sin embargo, creemos que el método utilizado para generar algoritmos basados en aprendizaje automático puede servir de referencia para futuros trabajos.
The objective is to develop a model that predicts vital status six months after fracture as accurately as possible. For this purpose we will use five different data sources obtained through the National Hip Fracture Registry, the Health Management Unit and the Economic Management Department.
Material and methodsThe study population is a cohort of patients over 74 years of age who suffered a hip fracture between May 2020 and December 2022. A warehouse is created from five different data sources with the necessary variables. An analysis of missing values and outliers as well as unbalanced classes of the target variable («vital status») is performed. Fourteen different algorithmic models are trained with the training. The model with the best performance is selected and a fine tuning is performed. Finally, the performance of the selected model is analyzed with test data.
ResultsA data warehouse is created with 502 patients and 144 variables. The best performing model is Linear Regression. Sixteen of the 24 cases of deceased patients are classified as live, and 14 live patients are classified as deceased. A sensitivity of 31%, an accuracy of 34% and an area under the curve of 0.65 is achieved.
ConclusionsWe have not been able to generate a model for the prediction of six-month survival in the current cohort. However, we believe that the method used for the generation of algorithms based on machine learning can serve as a reference for future works.
Las herramientas de aprendizaje automático, o machine learning, no solo han provocado una revolución en tecnologías de la salud1, sino que también sus aplicaciones pueden ser utilizadas sobre los datos médicos para aumentar la potencia de los análisis estadísticos tradicionales2. Por otra parte, en los sistemas de gestión electrónica de datos médicos actuales se cuenta con diferentes fuentes de datos (historias clínicas, analíticas, electrocardiogramas, imágenes radiológicas…). Estas fuentes, tomadas de una en una, ofrecen una excelente información para la gestión del día a día de nuestros pacientes. Pero cuando es posible asociar o fusionar diversas fuentes de datos, se abre la puerta a nuevas posibilidades de análisis e investigación3.
El aprendizaje supervisado es un tipo de aprendizaje automático en el cual los datos que se le ofrecen a la máquina vienen etiquetados (por ejemplo, en el presente trabajo la etiqueta es el estado vital del paciente, vivo/fallecido), de modo que tiene que generar un algoritmo capaz de clasificar correctamente los datos según esas etiquetas. Un modelo supervisado, por lo tanto, es una fórmula matemática, o algoritmo, que utiliza la máquina para tratar de clasificar correctamente los datos. Cabe citar como ejemplos de estos modelos supervisados los más simples, como es la regresión logística, o los más complejos, como puede ser el Gradient Boosting Classifier.
En nuestro caso, realizamos un análisis de diferentes modelos supervisados de aprendizaje automático para conocer su capacidad de predicción del estado vital (vivo/fallecido) de una cohorte de pacientes mayores de 74años a los 6meses tras una fractura de cadera. El objetivo es obtener un modelo que sea capaz de predecir con exactitud el estado vital a los 6meses de la fractura utilizando únicamente datos que se puedan recuperar durante el ingreso hospitalario. Para ello utilizaremos cinco fuentes de datos diferentes obtenidas a través del Registro Nacional de Fracturas de Caderas (RNFC), la Unidad de Gestión Sanitaria (UGS) y la Dirección Económica de nuestro hospital.
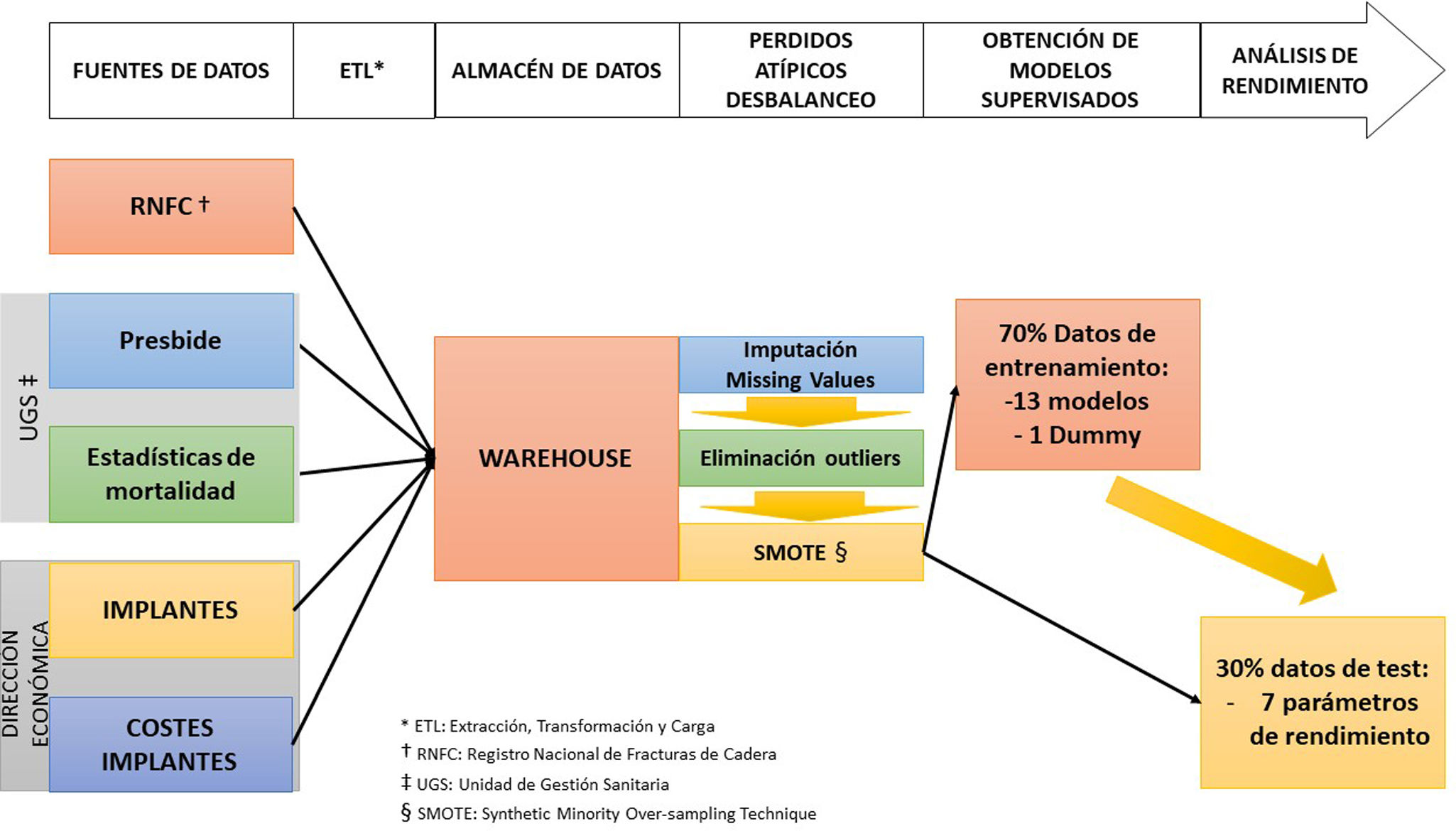
Material y métodosEn la figura 1 se muestra un esquema tanto del proceso de preparación de la base de datos como de la estrategia para la generación de modelos supervisados.
Preparación de la base de datosA continuación se describen las fuentes de datos utilizadas, el método empleado para añadirlas a un almacén de datos, así como el procedimiento utilizado para generar los modelos supervisados de predicción de mortalidad.
Fuentes de datos (Anexo I)Con el fin de comprobar la capacidad de fusión de bases de datos procedentes de diferentes fuentes, se decide seleccionar variables que, a priori, puedan estar relacionadas con el evento estudiado (estado vital a los 6meses) y se generen a lo largo del ingreso hospitalario del paciente con fractura de cadera.
RNFC. RNFC es un registro multicéntrico de las características epidemiológicas, clínicas, funcionales y asistenciales de los pacientes con fractura de cadera y seguimiento al mes del alta hospitalaria en varios hospitales de España4. En el presente trabajo se utiliza una cohorte correspondiente a los casos registrados en nuestro hospital desde mayo de 2020 hasta diciembre de 2022. Se seleccionan aquellos pacientes que son dados de alta con vida tras su ingreso hospitalario por fractura de cadera. De todas las variables recogidas en el registro, se utilizan las 40 que pueden ser obtenidas durante el ingreso hospitalario.
Presbide. Presbide es el sistema de prescripción electrónica del Servicio Vasco de Salud (Osakidetza). Se considera a priori que tanto el número como el tipo de prescripción puedan estar relacionados con la supervivencia del paciente (por ejemplo, se puede suponer menor supervivencia en aquellos pacientes a los que el internista les haya pautado un diurético de asa, como puede serlo la furosemida, ya que suele estar indicado en pacientes con insuficiencias cardiacas congestivas). La UGS suministra información acerca de las prescripciones que son recetadas al alta hospitalaria tras el ingreso por fractura de cadera. Se agrupan las marcas de medicamentos más frecuentes en 17 grupos, y se seleccionan, además, otros medicamentos que, aunque fuera de estos 17 grupos, han sido prescritos diez o más veces. También se crea la variable «Número total de prescripciones», que es el sumatorio de estas variables.
Estado vital a los 6 meses. La UGS proporciona el estado vital (vivo/fallecido) de los pacientes a fecha del 31 de junio de 2023, así como la fecha de fallecimiento, si es que hubiera ocurrido tal evento. Se realiza el cálculo para conocer en qué situación vital se hallaba el paciente a los 6meses tras el ingreso por fractura de cadera.
Implantes. Ante la posibilidad de que el tipo de implante pueda estar relacionado con una mayor o menor supervivencia de los pacientes (por ejemplo, los pacientes a los que se le indica prótesis total de cadera pudieran tener mayor supervivencia que aquellos a los que se les implanta una prótesis bipolar), se solicita a Dirección Económica información acerca de los implantes utilizados durante la intervención quirúrgica de los pacientes con fractura de cadera. Se especifica el subtipo de implante, la familia del implante y el modelo específico del mismo. Se seleccionan todos los subtipos y familias, así como el modelo específico de los implantes que hayan sido implantados en 20 o más ocasiones. Finalmente, se realiza un sumatorio de todos los implantes registrados por cada paciente.
Precio total de los implantes. Desde Dirección Económica también se provee de la información acerca del precio total sin IVA de los materiales implantados a cada paciente durante la cirugía de fractura de cadera, por si pudiera existir alguna relación causal (por ejemplo, a mayor precio del implante, mayor complejidad de la cirugía, y menor supervivencia).
Extracción, transformación y carga (ETL)Teniendo en cuenta la heterogeneidad tanto de los datos como de las fuentes utilizadas, es necesario realizar un procedimiento de extracción de información desde las fuentes. Esta extracción implica la transformación, limpieza, enriquecimiento e integración de los datos, para su posterior carga en un almacén de datos (data warehouse en inglés). Para realizar estos procesos se crea una base de datos NoSQL mediante el sistema de bases en código abierto MongoDB. El producto final es un almacén de datos en el que, además de las variables descritas previamente, se guardan otras que pueden ser de utilidad para análisis posteriores de esta cohorte.
Estrategias de preprocesamiento: manejo de valores perdidos, valores atípicos y desbalanceoSe realiza un análisis de los valores perdidos (missing values) en la base de datos y se elige el modelo de imputación según la naturaleza y las características de los mismos, que puede ir desde la eliminación de casos hasta la imputación con otros valores (un valor constante, una medida de tendencia central como la media, o técnicas más complejas, como lo es la imputación mediante K-vecinos).
Se lleva a cabo una visualización de posibles valores atípicos (outliers) mediante el análisis de componentes principales (PCA). En caso de observarse valores atípicos, se procede a la creación de una base de datos con eliminación de valores atípicos mediante la regla del rango intercuartílico (IQR)5. Siempre que el número de valores atípicos eliminado no exceda del 10% de total de registros, se procede a utilizar esta base de datos transformada.
La variable objetivo de los modelos supervisados analizados es el «estado vital» a los 6meses del ingreso por fractura de cadera, la cual es una variable dicotómica («vivo/fallecido»). En caso de que alguna de las dos clases totalice igual o más del 90% de los casos, se considerará que la base de datos está desbalanceada, por lo que se procederá a balancearla mediante la Synthetic Minority Over-sampling Technique (SMOTE)6, la cual genera ejemplos sintéticos de la clase minoritaria para equilibrar la distribución de clases.
Generación de modelos supervisadosUna vez limpia y preparada, la base de datos es dividida en una proporción 70:30 en dos partes: una que servirá para el entrenamiento de los modelos (datos de entrenamiento, 70%) y otra para probar el rendimiento de estos (datos de test, 30%).
Se entrenan 13 diferentes modelos algorítmicos (tabla 1), además de un «clasificador ficticio» de referencia (Dummy Clasifier), con los datos de entrenamiento siguiendo una estrategia de validación cruzada de 10K-folds (los datos de entrenamiento se dividen en 10 grupos, de modo que los modelos se entrenan 10 veces, tomando en cada iteración 9 de esos 10 grupos). Estos 14 modelos entrenados se prueban con los datos de test y se calculan 7 parámetros de rendimiento (tabla 1).
Listado de modelos y parámetros de rendimiento utilizados
| Modelos supervisados analizados | Parámetros de rendimiento utilizados |
|---|---|
| Ada Boost Classifier | Exactitud |
| Decision Tree Classifier | AUC |
| Dummy Classifier | Sensibilidad |
| Extra Trees Classifier | Precisión |
| Gradient Boosting Classifier | F1 |
| K Neighbors Classifier | Índice kappa |
| Light Gradient Boosting Machine | Coeficiente de correlación de Matthews (MCC) |
| Linear Discriminant Analysis | |
| Naive Bayes | |
| Quadratic Discriminant Analysis | |
| Random Forest Classifier | |
| Ridge Classifier | |
| SVM - Linear Kernel |
Se selecciona el modelo que mejor rendimiento obtenga en el área bajo la curva (AUC) y se realiza una puesta a punto fina (fine tuning) mediante la modificación de sus parámetros, en 10 pruebas de validación cruzada mediante una segmentación de 10K-folds (en total 100 ajustes). En caso de que se obtenga mejor rendimiento, se selecciona el mejor de estos modelos afinados.
Finalmente se genera la curva ROC (Receiver Operating Characteristic) del modelo seleccionado, además de la matriz de confusión, y se analiza el peso de las diferentes variables en la confección del modelo.
Descripción de las herramientas utilizadasLa base de datos NoSQL MongoDB es implementada sobre la herramienta de administración NoSQLBooster y se trabaja en código Python a través del módulo Pymongo.
El proceso ETL se lleva a cabo con la biblioteca de código abierto Pandas.
El preprocesamiento de la base de datos, la generación de modelos supervisados y el análisis del rendimiento se realizan con el módulo Pycaret.
El trabajo en código Python con los módulos Pymongo, Pandas y Pycaret se implementa en la aplicación Jupyter Notebook.
Todo el trabajo de análisis de datos se realiza en entornos locales para evitar el tráfico y la filtración de datos no anonimizados a través de internet.
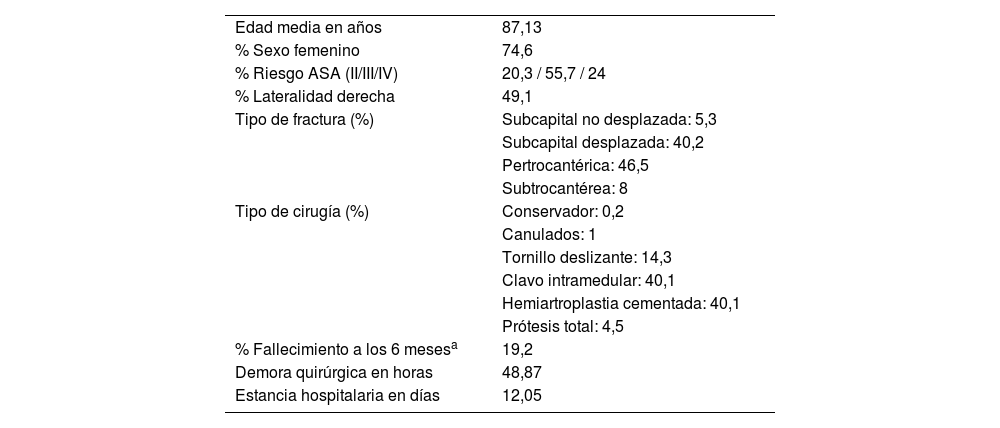
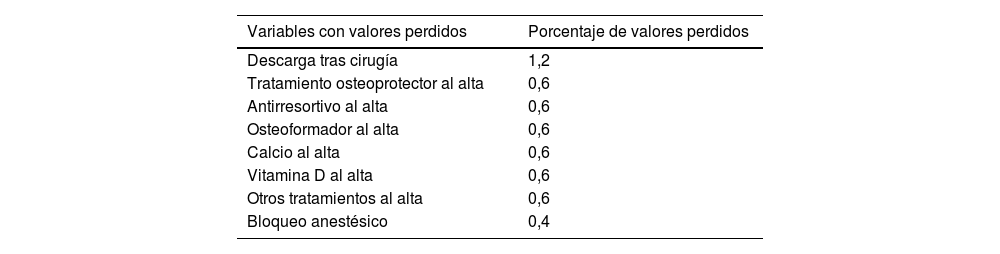
ResultadosSe realiza el proceso ETL, obteniéndose una base de datos de 502 pacientes, cuyas características se reflejan en la tabla 2. Tan solo 9 de las 144 variables utilizadas presentan valores perdidos, en porcentajes inferiores al 1,2% (tabla 3). Se trata sobre todo de casos de pacientes que fueron tratados conservadoramente, por lo que no existen algunos de los datos, como pueden ser los de la intervención quirúrgica. Teniendo en cuenta que se trata de variables cualitativas polinómicas y que en el RNFC se utiliza el código11 en casos en los que la información no puede ser recuperada, se imputa un valor constante, el código12, para estos valores perdidos.
Características de la población a estudio
| Edad media en años | 87,13 |
| % Sexo femenino | 74,6 |
| % Riesgo ASA (II/III/IV) | 20,3 / 55,7 / 24 |
| % Lateralidad derecha | 49,1 |
| Tipo de fractura (%) | Subcapital no desplazada: 5,3 |
| Subcapital desplazada: 40,2 | |
| Pertrocantérica: 46,5 | |
| Subtrocantérea: 8 | |
| Tipo de cirugía (%) | Conservador: 0,2 |
| Canulados: 1 | |
| Tornillo deslizante: 14,3 | |
| Clavo intramedular: 40,1 | |
| Hemiartroplastia cementada: 40,1 | |
| Prótesis total: 4,5 | |
| % Fallecimiento a los 6 mesesa | 19,2 |
| Demora quirúrgica en horas | 48,87 |
| Estancia hospitalaria en días | 12,05 |
Variables con valores perdidos y sus porcentajes (%)
| Variables con valores perdidos | Porcentaje de valores perdidos |
|---|---|
| Descarga tras cirugía | 1,2 |
| Tratamiento osteoprotector al alta | 0,6 |
| Antirresortivo al alta | 0,6 |
| Osteoformador al alta | 0,6 |
| Calcio al alta | 0,6 |
| Vitamina D al alta | 0,6 |
| Otros tratamientos al alta | 0,6 |
| Bloqueo anestésico | 0,4 |
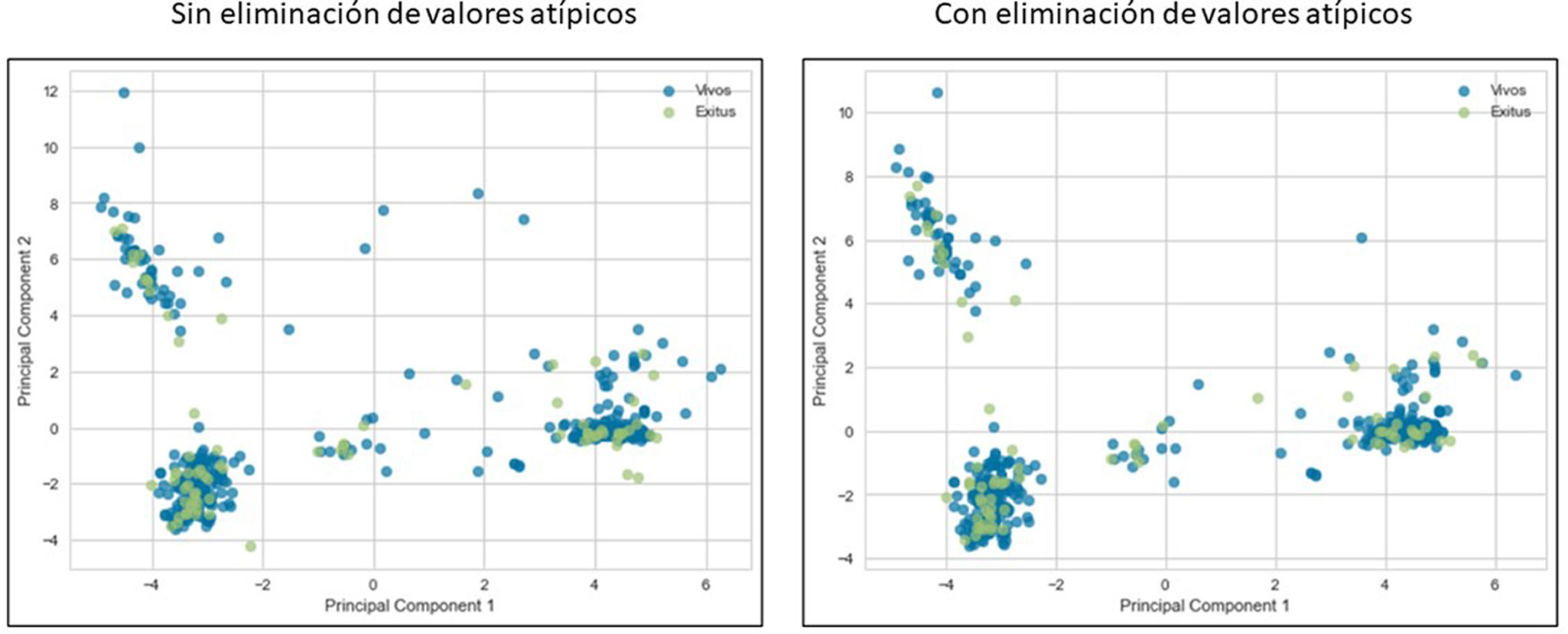
En el análisis PCA se observa la existencia de posibles valores atípicos, sobre todo en casos de pacientes que sobreviven más allá del sexto mes tras ingreso por fractura de cadera (fig. 2). La eliminación de valores atípicos conlleva la pérdida de solo 18 casos (de 502 a 484), que son el 3,58% del total, por lo que se decide realizar el análisis con eliminación de los mismos.
La base de datos resultante tras la eliminación de valores atípicos presenta 76 casos de defunción, frente a 408 de pacientes vivos. A pesar de que existe desbalanceo entre las dos clases (15,7% frente al 84,3%), este no alcanza el 90%, por lo que no se realiza ninguna técnica para corregirlo.
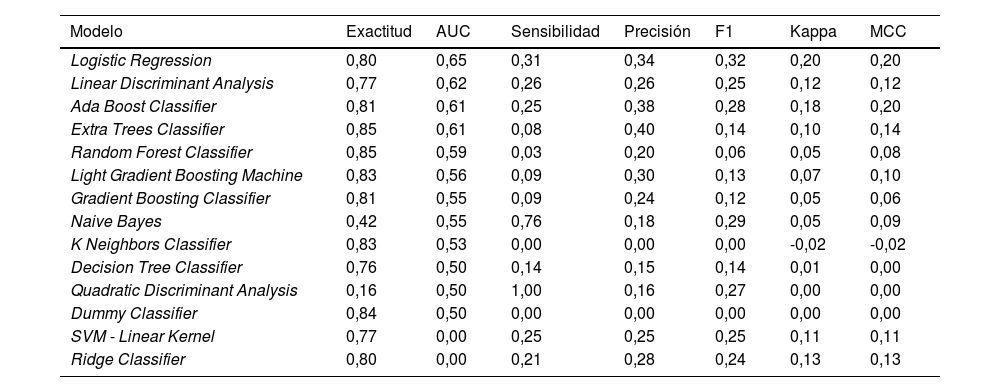
Generación de modelos supervisadosEl modelo más exacto es el Random Forest, con el 85% de casos bien clasificados. En cuanto al parámetro AUC, la regresión lineal obtiene un valor de 0,65. La regresión lineal también obtiene el mejor valor en F1 (0,32), el coeficiente de correlación de Matthews (MCC; 0,2) y el índice kappa (0,2). El análisis discriminante cuadrático presenta un 100% de sensibilidad, aunque los valores del resto de parámetros de rendimiento son pobres (por ejemplo, 50% de exactitud y un AUC de 0,5, lo que significa que está realizando predicciones al azar). El Extra Tree Classifier es el más preciso, con un 40% de pacientes realmente fallecidos bien clasificados entre las predicciones de fallecimiento.
A pesar de que Dummy Classifier presenta una buena exactitud (84%), el AUC y el resto de parámetros indican que sus predicciones son fruto del azar.
En cuanto a los resultados de otros modelos, llama la atención el pobre rendimiento del Gradient Boosting Classifier, con un AUC de 0,55 y un F1 de 0,12 (tabla 4).
Parámetros de rendimiento de los 14 modelos supervisados analizados
| Modelo | Exactitud | AUC | Sensibilidad | Precisión | F1 | Kappa | MCC |
|---|---|---|---|---|---|---|---|
| Logistic Regression | 0,80 | 0,65 | 0,31 | 0,34 | 0,32 | 0,20 | 0,20 |
| Linear Discriminant Analysis | 0,77 | 0,62 | 0,26 | 0,26 | 0,25 | 0,12 | 0,12 |
| Ada Boost Classifier | 0,81 | 0,61 | 0,25 | 0,38 | 0,28 | 0,18 | 0,20 |
| Extra Trees Classifier | 0,85 | 0,61 | 0,08 | 0,40 | 0,14 | 0,10 | 0,14 |
| Random Forest Classifier | 0,85 | 0,59 | 0,03 | 0,20 | 0,06 | 0,05 | 0,08 |
| Light Gradient Boosting Machine | 0,83 | 0,56 | 0,09 | 0,30 | 0,13 | 0,07 | 0,10 |
| Gradient Boosting Classifier | 0,81 | 0,55 | 0,09 | 0,24 | 0,12 | 0,05 | 0,06 |
| Naive Bayes | 0,42 | 0,55 | 0,76 | 0,18 | 0,29 | 0,05 | 0,09 |
| K Neighbors Classifier | 0,83 | 0,53 | 0,00 | 0,00 | 0,00 | -0,02 | -0,02 |
| Decision Tree Classifier | 0,76 | 0,50 | 0,14 | 0,15 | 0,14 | 0,01 | 0,00 |
| Quadratic Discriminant Analysis | 0,16 | 0,50 | 1,00 | 0,16 | 0,27 | 0,00 | 0,00 |
| Dummy Classifier | 0,84 | 0,50 | 0,00 | 0,00 | 0,00 | 0,00 | 0,00 |
| SVM - Linear Kernel | 0,77 | 0,00 | 0,25 | 0,25 | 0,25 | 0,11 | 0,11 |
| Ridge Classifier | 0,80 | 0,00 | 0,21 | 0,28 | 0,24 | 0,13 | 0,13 |
El modelo con mejor rendimiento en el AUC es la regresión lineal. Se realiza una puesta a punto fina del modelo original, pero ninguno de los 100 ajustes analizados obtiene mejores resultados, por lo que se decide seguir trabajando con el modelo original de regresión lineal (ver parámetros en la tabla 5).
Configuración del modelo de regresión lineal seleccionado
| (’trained_model’, | |
| LogisticRegression (C=1.0, class_weight=None, | |
| dual=False, | |
| fit_intercept=True, | |
| intercept_scaling=1, | |
| l1_ratio=None, max_iter=1000, | |
| multi_class=’auto’, n_jobs=None, | |
| penalty=’l2’, random_state=231, | |
| solver=’lbfgs’, tol=0.0001, | |
| verbose=0, | |
| warm_start=False))], | |
| verbose=False) |
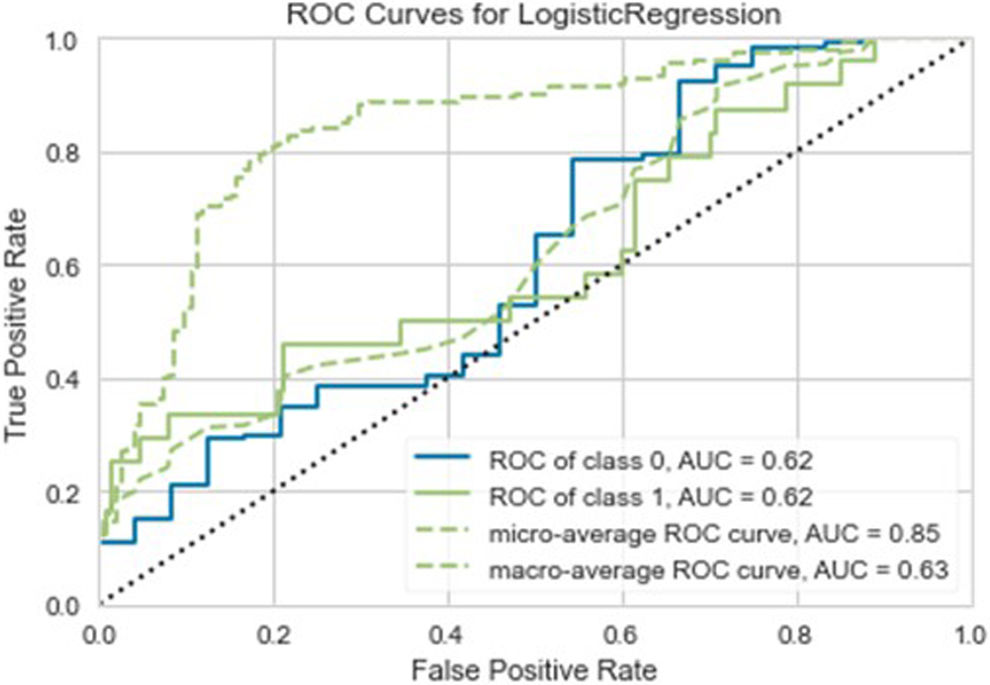
A pesar de que ha sido el modelo con mejor AUC, como se puede apreciar en la figura 3, la curva ROC del modelo seleccionado está cerca de la diagonal, lo que significa que la regresión lineal tiene un rendimiento muy limitado o apenas mejor que una predicción aleatoria. Esta observación se aprecia mejor en la matriz de confusión de los datos de test de la figura 4, en la que 30 casos son mal clasificados, y 16 de los 24 casos de pacientes fallecidos son clasificados como vivos. Se consigue una sensibilidad del 31%, una precisión del 34% y un AUC de 0,65.
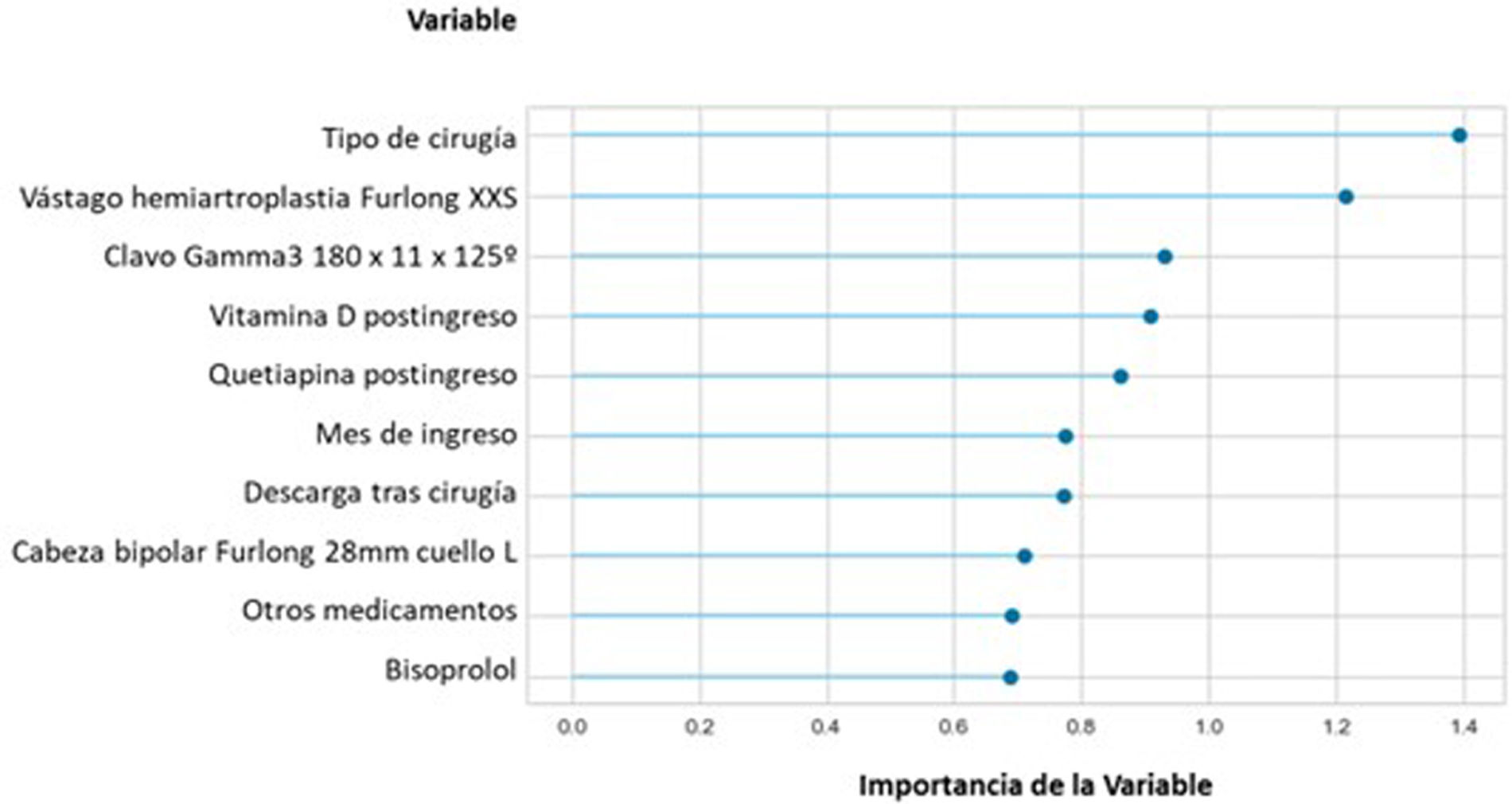
Finalmente, en cuanto al peso de las variables en el cálculo del modelo, en la figura 5 se observa que ninguna variable obtuvo una importancia predominante, siendo el tipo de cirugía la más influyente. También tuvieron importancia en el cálculo algorítmico algunos implantes específicos, como el vástago XXS de la hemiartroplastia cementada Furlong® (JRI), o el clavo intramedular corto de 125° Gamma3® (Stryker). Por otra parte, la necesidad de descarga tras la cirugía afectó a la supervivencia de estos pacientes. En cuanto a medicamentos, la vitaminaD, la quetiapina y el bisoprolol tuvieron influencia en la supervivencia (positiva para la primera, negativa para las dos últimas). Llama la atención que la variable «Otros medicamentos» aparezca entre las 10 variables con más peso. Esta variable se utiliza para indicar que se ha prescrito al paciente un inhibidor de la esclerostina (Evenity®, de Amgem). Sin embargo, en nuestra cohorte no se ha indicado este tratamiento antiosteoporótico. El hecho de que aparezca en el listado es debido a los casos de pacientes en los que este dato aparecía como «valor perdido» (0,6%), a los cuales se les imputó el código12, lo cual ha resultado de cierta importancia para el cálculo algorítmico de la regresión lineal, aunque se desconoce la razón de ello.
DiscusiónEn el presente trabajo hemos intentado encontrar un modelo predictor de fallecimiento a los 6meses tras fractura de cadera en pacientes mayores de 74años mediante la aplicación de técnicas de aprendizaje automático con datos de cinco diferentes fuentes de datos. En un estudio sobre el Registro Nacional Sueco de Fractura de Cadera (RIKSHOFT) se desarrolló un modelo de regresión lineal que predecía el riesgo de fallecimiento de los pacientes al año de una fractura de cadera con una sensibilidad del 75% (frente al 31% de nuestro trabajo), una precisión del 62% (frente al 34% de nuestro trabajo) y un AUC de 0,74 (frente el 0,65 de nuestro trabajo)7. Hay que tener en cuenta que el estudio sobre el RIKSHOFT incluía pacientes mayores de 18años (frente a 74años de nuestro trabajo), y que la variable con más peso en el algoritmo fue la de «carcinoma metastásico», seguida del riesgo ASA, sexo y edad. En nuestro caso varias de las variables con mayor peso en el algoritmo tienen una razón fisiopatológica clara, como lo puede ser la necesidad de descarga tras cirugía o la prescripción de ciertas medicaciones de las que se deducen importante deterioro cognitivo (quetiapina) o insuficiencia cardiaca congestiva (bisoprolol). Otras, como lo puede ser el mes de ingreso (enero, febrero… diciembre), o el uso de ciertos implantes específicos, no tienen explicación fisiopatológica clara.
Tanto el trabajo realizado con el RIKSHOFT como el nuestro han demostrado la superioridad de la regresión lineal sobre otros modelos que, a priori, podrían presuponerse más potentes a la hora de generar predicciones, como lo es el Gradient Boosting Classifier, tal y como han publicado algunos autores en otras disciplinas de cirugía ortopédica y traumatología8,9. Hay que tener en cuenta que cuanto más complejo es el modelo, mayor cantidad de datos es necesaria para su entrenamiento, lo cual puede explicar el bajo rendimiento de algunos de estos algoritmos, como el Gradient Boosting Classifier o el Ada Boost Classifier.
Por otra parte, nuestra cohorte ha presentado una mortalidad del 19,2% a los 6meses, similar a la publicada recientemente por otros autores españoles10.
Finalmente, aunque de manera generalizada, a la hora de trabajar con varias fuentes de datos se suelen utilizar bases de datos SQL, y la base de datos NoSQL MongoDB ha demostrado una gran flexibilidad y rendimiento, además de su fácil integración en código Python a través del módulo Pymongo. Su uso conjunto con las diferentes librerías de Python permite resolver el reto que plantean estas bases de datos a la hora de realizar informes y necesidades de análisis11.
ConclusionesA pesar del importante volumen de datos empleados en el entrenamiento de los modelos, no hemos conseguido generar uno que prediga con exactitud el estado vital de los pacientes de más de 74años a los 6meses del ingreso por fractura de cadera. Es posible que se necesiten un número mayor de pacientes y el uso de más variables, las cuales se podrían obtener de otras fuentes (por ejemplo, análisis clínicos, antecedentes personales o tratamientos previos al ingreso). Es por ello que creemos que el método utilizado tanto para la extracción, transformación y carga de los datos, como para el manejo de valores perdidos mediante el uso de una base de datos NoSQL MongoDB, y los módulos de Python Pandas, Pymongo y Pycaret, puede servir de referencia para futuros trabajos.
Nivel de evidenciaNivel de evidencia III.
FinanciaciónPropia.
Consideraciones éticasEsta investigación fue aprobada por el Comité Ético del Hospital Universitario Galdakao-Usansolo. Los autores han cumplido las normas éticas relevantes para la publicación. El consentimiento informado de los pacientes no se ha exigido por parte del CEIC.
Conflicto de interesesNinguno.