La longevidad viene determinada por la genética propia de cada especie y por factores externos, tales como nutricionales, ambientales, sociales, etc. Sin embargo, los individuos más longevos se caracterizan por presentar una mayor adaptación al entorno condicionada predominantemente por su propia genética. Dentro de una misma población con relativa homogeneidad genotípica, podemos encontrar cambios sutiles en la secuencia de ADN que afectan únicamente a un nucleótido. Estos cambios denominados polimorfismos de nucleótido simple (Single Nucleotide Polimorphisim [SNP]) se encuentran con una prevalencia mayor al 1-5% de la población. Por ello, nos planteamos estudiar en individuos centenarios si las posibles variaciones genéticas, analizando SNP, podrían tener alguna relevancia en la longevidad extrema que experimentan.
Material y métodosSe reclutó a 92 sujetos: 28 centenarios y 64 controles. Se les extrajo sangre, se aisló y amplificó ADN para el análisis de SNP mediante la tecnología Axiom™ Genotyping de Affymetrix. Los análisis estadísticos se realizaron mediante el programa Plink y varias bibliotecas de R para Windows (library SNPassoc, skatMeta).
ResultadosLos resultados del análisis muestran 12 SNP que presentan un valor de p inferior a 0,001, donde 5 de ellos (DACH1, LOC91948, BTB16, NFIL3 y HDAC4) tienen funciones reguladoras de la expresión de otros genes.
ConclusionesAsí pues, los resultados sugieren que las variaciones genéticas observadas entre centenarios y controles tienen lugar en 5 genes que están implicados en la regulación de la expresión génica, capacitándolos a adaptarse a diferentes condiciones ambientales con mejor éxito.
Longevity is determined by genetic and external factors, such as nutritional, environmental, social, etc. Nevertheless, when living conditions are optimal, longevity is determined by genetic variations between individuals. In a same population, with relative genotypic homogeneity, subtle changes in the DNA sequence affecting a single nucleotide can be observed. These changes, called single nucleotide polymorphisms (SNP) are present in 1-5% of the population.
Material and methodsA total of 92 subjects were recruited, including 28 centenarians and 64 controls, in order to find SNP that maybe implicated in the extreme longevity, as in the centenarians. Blood samples were collected to isolate and amplify the DNA in order to perform the analysis of SPN by Axiom™ Genotyping of Affymetrix technology. Statistical analyses were performed using the Plink program and libraries SNPassoc and skatMeta.
ResultsOur results show 12 mutations with a p<.001, where 5 of these (DACH1, LOC91948, BTB16, NFIL3 y HDAC4) have regulatory functions of the expressions of others genes.
ConclusionsTherefore, these results suggest that the genetic variation between centenarians and controls occurs in five genes that are involved in the regulation of gene expression to adapt to environmental changes better than controls.
La longevidad de los seres vivos viene determinada por la genética propia de cada especie y por factores externos a ellos, tales como los nutricionales, ambientales, sociales, etc. Sin embargo, dentro de una misma especie, se pueden observar diferencias de longevidad entre individuos. De hecho, cuando las condiciones de vida son óptimas, la supervivencia está únicamente condicionada por las variaciones genéticas entre individuos1. Además, dicha supervivencia se basa en el control homeostático que presentan los seres vivos, lo cual los capacita a adaptarse a diferentes condiciones ambientales, ya sean tanto en el medio interno como externo. Por tanto, el estudio de la secuencia genética, así como su control y regulación, constituyen unas herramientas clave para el entendimiento de la capacidad de adaptación al entorno de los seres vivos, de su longevidad y sobre todo en la longevidad extrema2.
Un ejemplo claro de este envejecimiento excepcional en el ser humano son los centenarios. Estos individuos, aproximadamente uno de cada 5.0003, han alcanzado una edad superior a la media de la población, debido a sus características genéticas. Por ello, en los últimos años, han sido considerados un modelo clave para el estudio del envejecimiento y longevidad en humanos.
Recientemente, un estudio, en el cual se ha analizado la expresión de micro-RNA (como control genético) en individuos de diferentes grupos de edad, ha mostrado cómo los centenarios poseen un patrón característico propio, muy similar al de los jóvenes y diferente al de los octogenarios. De forma similar, cuando se compara la expresión génica mediante el estudio de mRNA, los resultados muestran un patrón también característico propio en individuos centenarios, que es muy parecido a los jóvenes y no a octogenarios4.
Tanto la expresión génica (mRNA) como su regulación (micro-RNA) están codificadas en la secuencia de ADN. Esta secuencia no es exactamente igual entre razas ni entre individuos de una misma población, encontrándose cambios que afectan al fenotipo de las especies, como los que se describen entre razas, cambios por mutaciones, recombinaciones de genes, etc5. Dentro de una misma población con una relativa homogeneidad genotípica podemos encontrar, además, cambios sutiles en la secuencia de ADN que afectan únicamente a un nucleótido. Estos cambios denominados polimorfismos de nucleótido simple (del inglés Single Nucleotide Polimorphisim [SNP]), se encuentran con una prevalencia mayor del 1-5% de la población, según autores, y pueden o no traducirse en una alteración en la proteína codificada o en la regulación de la expresión génica6–8.
Por tanto, dada la existencia de patrones de expresión y regulación génica característicos de centenarios, nos planteamos estudiar si la presencia de variaciones en SNP, podría marcar un patrón de extrema longevidad en individuos centenarios en una población concreta. De esta manera, se podría ayudar a la identificación de aquellos factores genéticos que contribuyen al envejecimiento excepcional que presentan los individuos centenarios.
Material y métodosMuestraSe reclutaron un total de 92 sujetos: 28 centenarios (≥100 años) (22 mujeres y 6 hombres) y 64 controles (20-80 años) (41 mujeres y 23 hombres) pertenecientes al departamento de salud número 11 de la Comunidad Valenciana, España.
El estudio fue sometido a los principios de bioética recogidos en la declaración de Helsinki y a la legislación española pertinente. Además, todos los procedimientos experimentales fueron aprobados por el Comité de Ética en Investigación Clínica del Hospital Universitario de la Ribera (Alzira), desde donde se llevó a cabo el reclutamiento. Todos los pacientes y sus familiares fueron plenamente informados de los objetivos y el alcance de la investigación y firmaron un consentimiento informado.
Las muestras de sangre total fueron extraídas en ayunas por personal autorizado en el Hospital Universitario de la Ribera o en el propio domicilio del sujeto. Se emplearon tubos Vacutainer con EDTA como anticoagulante. Una vez alicuotadas en tubos tipo eppendorf, se congelaron a −80°C. Posteriormente, uno de estos crioviales se envió al Centro Nacional de Genotipado-Instituto Carlos III de Santiago de Compostela, para la extracción de ADN y genotipado de la muestra.
Genotipado de las muestrasEl ADN fue extraído mediante el Chemagic DNA blood kit, siguiendo las indicaciones del fabricante. Para el análisis de genotipado se utilizó la tecnología Axiom™ Genotyping de Affymetrix. Para ello, el ADN total se amplificó y fragmentó hasta 25-125 pares de bases. Estos fragmentos se purificaron y resuspendieron con la solución de hibridación que se transfirió posteriormente al GeneTitan Instrument de Affymetrix, para seguir su procesamiento completamente automatizado (hibridación en las placas de 96 pocillos, ligado, lavado, tinción, estabilización y escaneado). Las imágenes se procesaron automáticamente para obtener los genotipos con el algoritmo Axiom GT1, disponible a través del programa Genotyping Console (GTC) de Affymetrix.
Una vez obtenidas las imágenes de cada muestra en archivos, se descartaron aquellas que no sobrepasaron los siguientes controles de calidad: filtro por dishQC (se eliminaron las muestras con valor inferior a 0,82) y filtro por call rate (se eliminaron las muestras con valor inferior a 97%). Finalmente se genotiparon 295.988 marcadores.
Análisis estadísticoPrevio al análisis estadístico, los datos genéticos fueron sometidos a un procedimiento de control de calidad estándar de los estudios de asociación, en función de la desviación del equilibrio Hardy Weinberg y la tasa de genotipado. Los análisis estadísticos se realizaron mediante el programa Plink y varias bibliotecas de R para Windows (library SNPassoc, skatMeta).
En el análisis individual de los SNP, las diferencias en frecuencia de genotipos entre casos y controles para cada una de las variantes fueron evaluadas mediante regresión logística, empleando el programa Plink. La variable género fue incluida como covariable en el modelo de regresión. El análisis fue realizado asumiendo el modelo genético dominante. En dicho modelo, los genotipos son codificados como 0, 1 o 2 en función del número de copias del alelo menos frecuente.
Para controlar las múltiples comparaciones realizadas (por SNP), se aplicó la corrección False Discovery Rate (FDR-BH)9, de tal manera que una probabilidad ajustada por FDR (valor q) ≤ 0,05 se consideró significativa.
De los 295.988 marcadores genotipados, eliminando monomórficos, es decir, aquellos que muestran el mismo genotipo en todos los individuos analizados y, por tanto, no varían en la población de estudio, y aplicando los filtros pertinentes, quedaron un total de 37.457 SNP (definidos como aquellos marcadores con frecuencia superior al 5%) apropiados para este estudio.
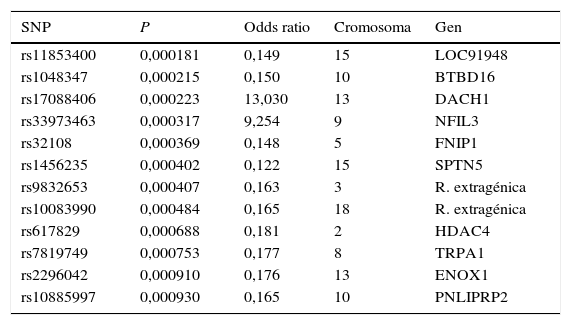
ResultadosLos resultados del análisis de SNP individuales muestran un grupo de 108 SNP con un p valor menor de 0,005 (material suplementario). Del total, se analizaron los 12 que presentaban un valor de p inferior a 0,001, aunque no se consideraron estadísticamente significativos tras aplicar la corrección de FDR, ya que son todas muy cercanas a 1, en concreto, el valor q para todos los SNP que se muestran en la tabla es constante, 0,9589, lo cual es frecuente en los estudios de asociación, donde se suele hablar de «SNP relacionados con la longevidad». En la tabla 1 se muestran dichos polimorfismos usando el sistema de nomenclatura «rs», su odd ratio que cuando es menor que uno determina menor prevalencia del polimorfismo en centenarios y cuando es mayor uno la mayor presencia. Así mismo, la tabla muestra el cromosoma y el gen donde se localizan.
polimorfismos con p valor menor a 0,001
| SNP | P | Odds ratio | Cromosoma | Gen |
|---|---|---|---|---|
| rs11853400 | 0,000181 | 0,149 | 15 | LOC91948 |
| rs1048347 | 0,000215 | 0,150 | 10 | BTBD16 |
| rs17088406 | 0,000223 | 13,030 | 13 | DACH1 |
| rs33973463 | 0,000317 | 9,254 | 9 | NFIL3 |
| rs32108 | 0,000369 | 0,148 | 5 | FNIP1 |
| rs1456235 | 0,000402 | 0,122 | 15 | SPTN5 |
| rs9832653 | 0,000407 | 0,163 | 3 | R. extragénica |
| rs10083990 | 0,000484 | 0,165 | 18 | R. extragénica |
| rs617829 | 0,000688 | 0,181 | 2 | HDAC4 |
| rs7819749 | 0,000753 | 0,177 | 8 | TRPA1 |
| rs2296042 | 0,000910 | 0,176 | 13 | ENOX1 |
| rs10885997 | 0,000930 | 0,165 | 10 | PNLIPRP2 |
Los 12 SNP analizados se localizan en diferentes regiones del genoma: 5 en los exones de los genes NFIL3, PNLIPRP2, SPBTN5, TRPA1 y BTBD16, otros 5 en la región intrónica de los genes DACH1, ENOX1, FNIP1, HDAC4 y LOC91948; y los otros 2 situados en el ADN extragénico. Los resultados muestran que excepto los SNP en DACH1 y NFIL3 que se encuentran con mayor prevalencia en centenarios, los demás son más prevalentes en la población control.
DiscusiónEn 2011 Rajpathak et al., después de entrevistar a 447 individuos de edades comprendidas entre 95-109 años, concluyeron que los estilos de vida de los individuos más longevos de los que habían entrevistado no diferían mucho de la población normal, lo cual parecía indicar que su incremento de longevidad no se debía al estilo de vida10. Asimismo, los descendientes de personas centenarias tienen mayor probabilidad de ser más longevos, y se estima que los hermanos de centenarios tienen entre 8 y 17 veces mayor probabilidad de llegar a serlo11. El componente genético, por lo tanto, juega un papel esencial en la longevidad extrema que presentan las personas que llegan a cumplir más de cien años.
Si bien, ya se han encontrado patrones de expresión de regulación característicos de centenarios4, estos deberían de estar codificados en la secuencia genética. Por ello, los estudios del Genome-wide association study cobran un papel importante en la identificación de las variantes genéticas que podrían ser en parte responsables de estas extremas longevidades.
El presente trabajo se basa en el estudio de variantes genéticas de un corte transversal de una población correspondiente a un área de salud de la Comunidad Valenciana. Mediante un análisis Genome-wide association study que realiza un cribado de 300.000 marcadores genéticos distribuidos en el genoma humano y después de las pertinentes correcciones, seleccionamos las 12 variantes más significativas y estudiamos el gen en el que se encuentra el polimorfismo. De los 12 SNP, encontramos 2 en regiones que no pertenecen a ningún gen, 5 que corresponden a proteínas con actividades concretas y 5 reguladores de la expresión génica.
De las proteínas cabe destacar FNIP1 que posee función de control energético celular siendo sensible a los niveles de ATP y con capacidad de interactuar con AMPK, el cual regula negativamente mTOR12. Esta vía, relacionada con la restricción calórica, se ha comprobado que está implicada en la longevidad de especies como levadura, gusanos e incluso en ratones13. En esta misma línea, ENOX1 constituye una proteína con capacidad de control de los niveles de NADH la cual también está implicada en el control energético celular14. El polimorfismo de estos dos genes está localizado en regiones potencialmente reguladoras y su presencia es menor en personas centenarias que en el resto de la población, lo cual indica que la forma menos frecuente del SNP es un factor de riesgo en relación a la longevidad.
Respecto el gen PNLIPRP2 corresponde a un enzima con capacidad galactolipasa, que participa en la digestión de grasas de la dieta. Alternativamente puede jugar un papel en el tráfico de gránulos de zimógenos, ya que se localiza en las membranas de dichos gránulos. Además, puede participar en la citotoxicidad mediada por células T, lo que siguiere que está implicada en la respuesta inmune15.
La regulación celular y el control de la homeostasia son factores claves para el mantenimiento de la supervivencia de los organismos. Este control se lleva a cabo mediante la continua síntesis de proteínas por parte de los tejidos que se van adaptando a las diferentes condiciones del medio interno. Entre las proteínas que se codifican en el genoma de las especies, unas tienen funciones específicas y muy concretas como estructurales, de transporte, transducción de señales, etc.; y otras se encargan de la regulación de la expresión génica, como los factores de transcripción y factores nucleares. Estas se encargan de controlar la expresión de numerosas proteínas constituyendo verdaderos maestros reguladores del genoma. Resulta interesante comprobar cómo de las 12 variantes más significativas encontradas en nuestro estudio, 5 de ellas corresponden a este tipo de proteínas reguladoras genéticas.
Encontramos BTBD16, este factor de transcripción resulta muy interesante porque parece tener la capacidad de interactuar con más de 200 micro-RNA, constituyendo una proteína con una alta capacidad de regulación génica. El polimorfismo se encuentra en la propia secuencia codificante genética de la proteína y tiene una menor prevalencia en personas centenarias, pudiendo considerarse como un factor de riesgo. NFIL3, otro factor de transcripción con capacidad de regular factores relacionados con la inflamación como las interleucinas, interviene en el control de la diferenciación de células inmunitarias como las T Helper o natural killer actuando por tanto en el control inmunitario del organismo16. Este SNP localizado en la zona codificante de la secuencia de la proteína se encuentra con mayor prevalencia en la población centenaria, al igual que DACH1 que constituye una proteína nuclear que regula la diferenciación y ciclo celular, y además, se ha comprobado que es capaz de reprimir ciertos tumores como el de mama y próstata, disminuyendo la expresión de receptores de crecimiento como EGFR. Así mismo, su disminución de la expresión se ha relacionado con tumores de orofaringe, esofaríngeo, renal, hepático, endometrial, etc.17; y otros como el de mama mediados por el receptor de IGF-118, del cual es evidente que cambios en su expresión pueden alargar la vida útil en los gusanos, moscas y ratones, lo que implica la conservación evolutiva de los mecanismos. Es importante destacar que estas mutaciones pueden mantener los animales sanos y durante más tiempo libre de enfermedad y pueden aliviar patologías específicas relacionadas con el envejecimiento, como es el cáncer19,20.
HDAC4, una enzima con actividad deacetilasa, posee la capacidad de regular la cromatina nuclear que envuelve al ADN, controlando la expresión de genes implicados en la regulación de la supervivencia, crecimiento y proliferación celular21. Su expresión aberrante está relacionada con la formación de numerosos tipos de cáncer. Regula la formación de condrocitos, desarrollo óseo, muscular y está implicada en patologías cardiovasculares. Además su sobreexpresión está relacionada con la disminución de la secreción de insulina y somatostatina22.
En conclusión, la longevidad extrema está condicionada por el componente genético, el cual se encuentra en la secuencia de nucleótidos del ADN. Esta secuencia contiene la información para el control homeostático de los individuos, el cual se pierde en las últimas etapas de la vida causando enfermedad y la muerte. Las personas centenarias tienen estos mecanismos homeostáticos mejor conservados que el resto de la población, y es en el ADN donde se encuentra la respuesta al porqué de la extrema longevidad. Por lo tanto, el conocimiento de esta secuencia nos llevará en el futuro a comprender la supervivencia de las especies.
FinanciaciónEste trabajo fue apoyado por becas SAF2010-19498 y SAF2013-44663-R, del Ministerio español de Educación y Ciencia (MEC); ISCIII2012-RED-43-029 de la «Red Temática de Investigación Cooperativa en Envejecimiento y Fragilidad» (RETICEF); PROMETEO2014/056 de la «Conselleria d’Educació, Cultura i Esport de la Generalitat Valenciana»; RS2012-609 Intramural Grant from INCLIVA y financiados por la UE CM1001 y FRAILOMIC-HEALTH.2012.2.1.1-2. El estudio ha sido cofinanciado por los Fondos de la Unión Europea.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.