Aun cuando es considerado un agente etiológico poco usual, Cyberlindnera (Candida) fabianii ha sido descrita como causante de septicemia en diversos reportes en los últimos años. Su identificación dudosa o incierta al utilizar métodos como CHROMagar Candida, API® Candida, API® ID32C o VITEK® MS, resulta en una subestimación de los casos producidos por esta levadura.
ObjetivosInformar por primera vez en Chile de un aislamiento de C. fabianii y su identificación.
MétodosSe utilizó la secuenciación de la región de los espaciadores internos transcritos para la identificación final de la levadura. Se evaluó su perfil de sensibilidad a los antifúngicos mediante la técnica de microdilución en caldo.
ResultadosLa identificación final solo fue posible mediante la secuenciación de las regiones ITS, lo que demuestra la limitada utilidad de las técnicas convencionales en la identificación de determinadas especies. La relación filogenética de la cepa aislada con otras especies de levadura queda reflejada en un dendrograma.
ConclusionesLas técnicas de secuenciación en la identificación de microorganismos fastidiosos o cuya identificación es dudosa con las metodologías clásicas, u otras más avanzadas como la espectrometría de masas, son imprescindibles.
Although considered an unusual etiological agent, Cyberlindnera(Candida)fabianii has been related to septicemia in several reports in recent years. Its doubtful or uncertain identification when using tests such as CHROMagar Candida, API® Candida, API® ID32C or VITEK® MS, leads to an underestimation of the cases produced by this yeast.
AimsTo report the first isolation of C. fabianii in Chile and its identification.
MethodsThe sequencing of the internal transcribed spacer region (ITS) was performed. Antifungal susceptibility profiles were obtained by means of the broth microdilution technique.
ResultsThe identification was only reached by sequencing the ITS regions, which shows the limited usefulness of the conventional techniques in the identification of some yeast species. A dendrogram shows the phylogenetic relationship of the isolated strain with some other yeast species.
ConclusionIn the identification of fastidious microorganisms or microorganisms whose identification is not completely reliable when using classical or even advanced methodologies, such as mass spectrometry, sequencing techniques are essential.
La enfermedad fúngica invasiva representa una amenaza constante para los pacientes hospitalizados e inmunocomprometidos. El aumento en el número de casos asociados a las infecciones fúngicas no solo contempla los hongos ya conocidos y más prevalentes, Candida albicans y Aspergillus fumigatus6,9, sino que incluye a agentes etiológicos emergentes. Una de las problemáticas asociadas a estos microorganismos es la difícil identificación en el laboratorio, aun cuando se utilicen equipos de última generación basados en espectrometría de masas. Asimismo, los clásicos esquemas de identificación, basados en las características fenotípicas, evidencian su limitada resolución, básicamente por no contemplar en sus bases de datos a estos raros agentes fúngicos. El hecho de no identificar, o hacerlo erróneamente, conlleva la subestimación de estos hongos poco usuales, lo que puede tener repercusión en el aspecto epidemiológico y, sobre todo, en el aspecto terapéutico. Entre estos raros agentes se encuentra Cyberlindnera fabianii, una levadura perteneciente al phylum Ascomycota, que ha sido aislada de suelo, cañas de azúcar, aceitunas y desde el ser humano4,14,17. El presente trabajo tiene como objetivo comunicar el hallazgo de C. fabianii en un hemocultivo por primera vez en Chile, así como su sensibilidad a los fármacos antifúngicos. Del mismo modo, demostramos una vez más la utilidad de las técnicas de biología molecular en estos casos en los que las metodologías clásicas, así como las avanzadas (no moleculares), presentan una resolución limitada.
MetodologíaDesde un laboratorio externo, y debido a la especialización de nuestro centro micológico, nos fue remitida la cepa de una levadura para su identificación. Las características morfológicas del aislamiento fueron determinadas mediante el uso de BBLTM agar dextrosa Sabouraud (Becton-Dickinson, EE. UU.) y BBLTM CHROMagar Candida (Becton Dickinson); los medios fueron incubados a 37°C durante 24h. Posteriormente, y una vez obtenidos y confirmados los cultivos puros, estos se sometieron a identificación mediante los métodos API®Candida (bioMérieux), API® ID32C (bioMérieux, Francia) y el sistema VITEK® MS (bioMérieux). Asimismo, y dadas las discrepancias, se evaluó el aislamiento mediante la técnica de producción de tubo germinal y la observación de la micromorfología según la técnica de Dalmau. En este último caso la cepa fue sembrada en BBLTM agar harina de maíz (Becton Dickinson) suplementado con Tween 80, e incubada a 30°C durante 24-48h.
Para la identificación molecular, el ADN fúngico se extrajo con el Mini-kit E.Z.N.A.® Fungal DNA (Omega Biotek Store, EE. UU.). La PCR para la amplificación de la región ITS1-5.8S-ITS2 se realizó en un volumen de 25μl siguiendo protocolos publicados anteriormente1. La secuencia de la cepa fue depositada en la base de datos GenBank bajo el número MT795190. Los análisis filogenéticos se realizaron utilizando el método de máxima verosimilitud en el software RAxML 8.0. La secuencia de Rhodotorula mucilaginosa CBS 329 fue utilizada como grupo externo.
La prueba de sensibilidad in vitro a los antifúngicos se realizó mediante el método de microdilución en caldo de acuerdo a los documentos CLSI M27 4 ed. y M607,8. Los valores de concentración mínima inhibitoria fueron obtenidos a las 24 y 48h. Polvos activos puros, de potencia conocida, de anfotericina B (Sigma-Aldrich), voriconazol (Sigma-Aldrich), itraconazol (Sigma-Aldrich), posaconazol (Sigma-Aldrich), fluconazol (Sigma-Aldrich), caspofungina (Sigma-Aldrich), micafungina (Astellas Pharma), y anidulafungina (Pfizer) fueron utilizados para la valoración. Todas las pruebas se realizaron por duplicado. El aislamiento de Candida parapsilosis ATCC 22019 fue incluido como control de calidad.
La cepa del estudio se encuentra depositada en la colección chilena de hongos (ChFC: Chilean Fungal Collection), Unidad de Micología, Universidad de Chile, bajo el código ChFC 481.
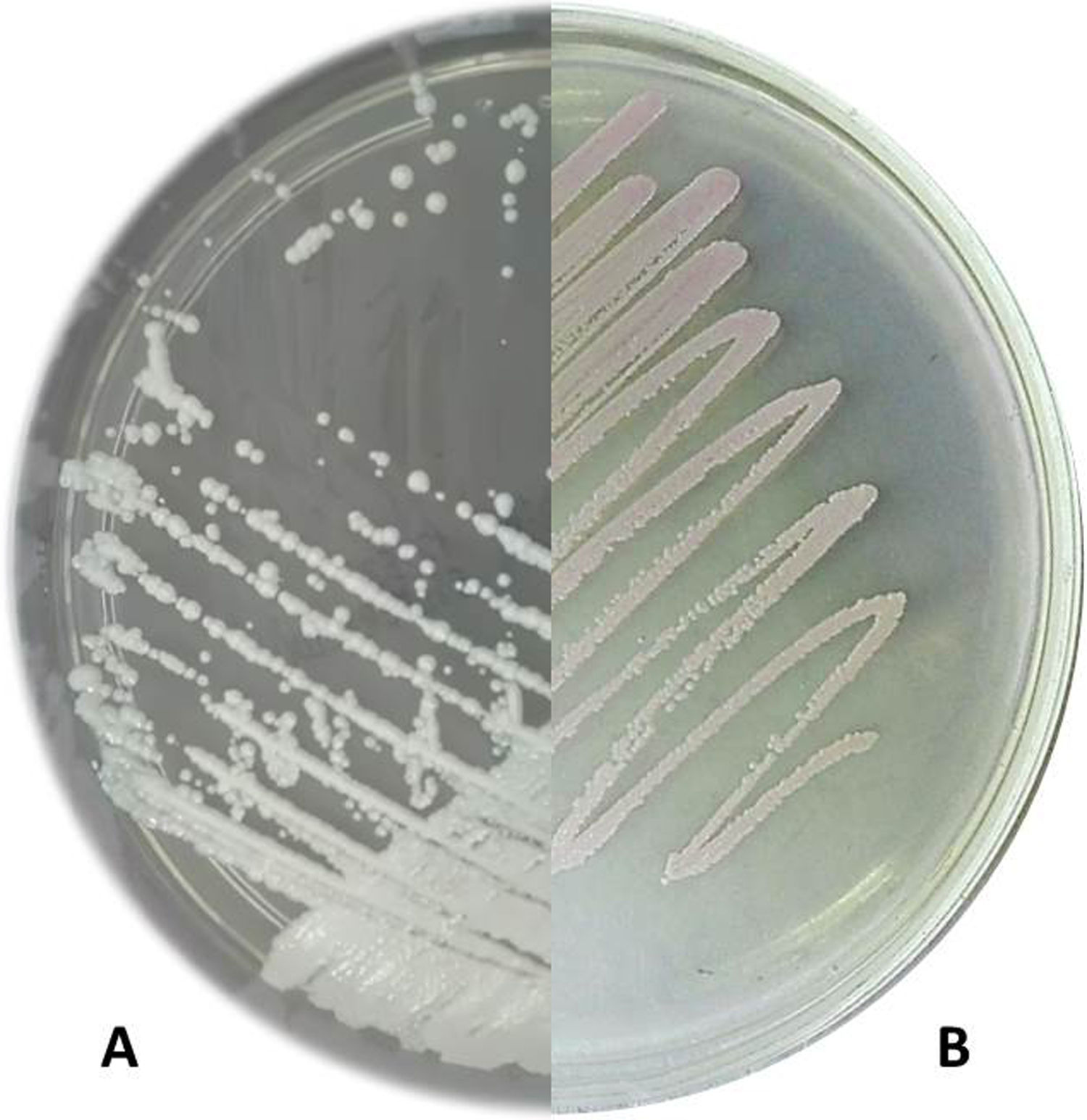
ResultadosTanto en el medio SDA como en CHROMagar Candida la cepa ChFC 481 creció, dando lugar a colonias levaduriformes en 24h. Las colonias exhibieron un color blanco, eran convexas y de bordes lisos en SDA, y de color rosado pálido, convexas y lisas en el agar cromogénico (fig. 1). La prueba de producción de tubo germinal resultó negativa. En el microcultivo solo fue posible observar células levaduriformes y blastoconidias.
Con las pruebas bioquímicas API® Candida y API® ID32C, y con el sistema Vitek® MS, las identificaciones fueron inconsistentes. El método API® Candida no arrojó porcentajes de identificación, y estos fueron del 57% para la especie Candidapelliculosa y 42,6% para Candidautilis con API® ID32C. En el caso de Vitek® MS el valor de identificación arrojado fue inferior a 1,7.
La identificación molecular permitió identificar con certeza la cepa ChFC 481 como C. fabianii. El análisis de secuencias mostró un grado de identidad del 100% con la secuencia de la cepa tipo de C. fabianii (CBS 5640). Las relaciones filogenéticas entre nuestra cepa y otros microorganismos relacionados se muestran en la figura 2.
Árbol filogenético obtenido con el análisis de las secuencias de la región ITS de la cepa ChFC 481 y de especies relacionadas mediante el método de máxima verosimilitud. En los nodos se indican los valores bootstrap≥70% en base al análisis de 1000 réplicas. En negrita se presenta la cepa de nuestro país.
A pesar de que tanto los puntos de corte clínicos como los puntos de corte epidemiológicos para esta especie no han sido establecidos, los perfiles de sensibilidad obtenidos mostraron que los antifúngicos más activos eran el voriconazol y la micafungina. Los valores de concentración mínima inhibitoria fueron los siguientes: anfotericina B 0,25μg/ml, itraconazol 0,25μg/ml, voriconazol≤0,03μg/ml, posaconazol 0,25μg/ml, fluconazol 1μg/ml, caspofungina 0,06μg/ml, micafungina≤0,03μg/ml y anidulafungina 0,06μg/ml.
DiscusiónEn el presente trabajo describimos el primer aislamiento de C. fabianii procedente del hemocultivo de un paciente pediátrico en nuestro país. A pesar de que este microorganismo es raramente aislado de muestras clínicas, su presencia ha ido cobrando relevancia en los últimos años, especialmente en hemocultivos provenientes de unidades de pediatría y neonatología12,19,20. Lo anterior representa un problema y un desafío para el diagnóstico de laboratorio dadas las cuestionables identificaciones que arrojan los sistemas basados en pruebas bioquímicas o espectrometría de masas11. En nuestro caso, tanto los perfiles de asimilación de hidratos de carbono como los perfiles proteicos obtenidos mediante los sistemas API® Candida, API® ID32C y VITEK® MS no nos permitieron identificar la levadura correctamente, obteniéndose identificaciones erróneas compatibles con C.utilis o C.pelliculosa. Lo anterior concuerda con gran parte de lo publicado, donde se habla de la errónea identificación inicial y de la necesidad de técnicas moleculares y de secuenciación3,5,12 para determinar correctamente la especie. En términos generales, la identificación fenotípica y la obtenida mediante VITEK® MS presentan un rendimiento cercano al 96,1%, si bien C. fabianii es una de las levaduras que se encuentran en el 3,9% de las especies no identificables o de las que se obtiene una identificación errónea18. La limitación a este respecto de los métodos utilizados se debe principalmente a la ausencia de los perfiles de C. fabianii en las bases de datos. Lo anterior es sumamente importante, y demuestra la necesidad de los laboratorios de contar con los sistemas, programas y bases de datos siempre actualizados. Asimismo, independientemente de la metodología empleada para la identificación, las características particulares de algunas cepas (mucosidad), los errores en la metodología de procesamiento y algunos factores inherentes a los equipos pueden ser determinantes en los valores/porcentajes bajos de identificación2,16.
Los valores de concentración mínima inhibitoria obtenidos con todos los antifúngicos fueron≤1μg/ml, y el fluconazol fue el menos activo. Estos resultados concuerdan con lo publicado previamente13,15. Sin embargo, un estudio de Hamal et al. mostró la capacidad de C. fabianii de formar biopelículas y, además, la capacidad de desarrollar resistencia secundaria a diferentes antifúngicos10. Asimismo, el uso profiláctico de fluconazol se ha asociado con fracaso en el tratamiento en un 50% de los casos15.
A pesar de las innumerables ventajas y del innegable avance que ha representado la espectrometría de masas en los diagnósticos, debe siempre tenerse en cuenta que, como toda técnica, tiene ciertas limitaciones. La correcta identificación de C. fabianii requiere de técnicas moleculares. Ante casos de identificación dudosa o errónea compatibles con C.utilis o C.pelliculosa recomendamos se utilicen las técnicas moleculares disponibles Asimismo, se debe prestar atención a los perfiles de sensibilidad, dado que terapias de larga duración podrían asociarse al desarrollo de resistencias.
FinanciaciónEstudio financiado con fondos propios.
Conflicto de interesesLos autores no tienen conflictos de interés que declarar.