Candida sake es una levadura que se aísla infrecuentemente en muestras clínicas y se asocia a procesos como candidemia, endocarditis o peritonitis, especialmente en ancianos y en pacientes con comorbilidad o tratamiento previo con antibióticos de amplio espectro o corticoides.
Empleando el sistema API 32C® (bioMérieux, Marcy-l’Étoile, Francia)2, durante 2011 se identificaron en nuestro servicio 3 aislamientos de C. sake en hemocultivos, en contraste con un único aislamiento en el período 2005-2010.
El objetivo de este estudio fue la caracterización de estos 4 aislamientos mediante 2 métodos distintos.
Se recuperaron de nuestro cepario las cepas de C. sake de hemocultivos, de pacientes distintos, entre los años 2005 y 2011, conservadas en crioviales a –80°C de temperatura en caldo cerebro-corazón con 20% de glicerol. En total fueron seleccionadas 4 cepas con una distribución de una en 2005 de un total de 33 candidemias en ese año y 3 de 45 candidemias en 2011 (tabla 1).
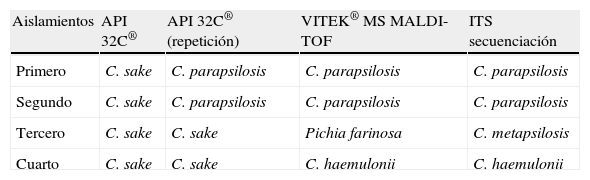
Tabla de pruebas identificativas para los 4 aislamientos de Candida
| Aislamientos | API 32C® | API 32C® (repetición) | VITEK® MS MALDI-TOF | ITS secuenciación |
| Primero | C. sake | C. parapsilosis | C. parapsilosis | C. parapsilosis |
| Segundo | C. sake | C. parapsilosis | C. parapsilosis | C. parapsilosis |
| Tercero | C. sake | C. sake | Pichia farinosa | C. metapsilosis |
| Cuarto | C. sake | C. sake | C. haemulonii | C. haemulonii |
Todos los hemocultivos positivos en que se observaron levaduras, tras la realización de tinción de Gram, se sembraron en agar Sabouraud con cloranfenicol y agar Sabouraud con cloranfenicol-actidiona (Bio-Rad, Marnes-la-Coquette, Francia). Además, todas las cepas fueron identificadas con medio CHROMagar Candida (Becton Dickinson, Heidelberg, Alemania) y con la galería API 32C® (bioMérieux).
La identificación de los aislamientos fue confirmada mediante amplificación y secuenciación de la región ITS (Internal Transcribed Spacer). El ADN fue extraído de colonias individuales mediante el método comercial InstaGene Matrix® (Bio-Rad, Hercules, CA, EE. UU.) y la región ITS se amplificó con los cebadores ITS86-F (5′-gTgAATCATCgAATCTTTgAAC-3′) e ITS4-R (5′-TCCTCCgCTTATTgATATgC-3′)6. El producto de PCR se purificó con el kit comercial High Pure PCR Product Purification Kit® (Roche, Mannheim, Alemania) y fue secuenciado con el aparato 377 de Applied Biosystems. Las secuencias de los aislamientos se compararon con aquellas depositadas en la base de datos del GenBank mediante BLAST.
Adicionalmente, se estudió la capacidad del sistema VITEK® MS™ -basado en MALDI-TOF®- (bioMérieux)7 para la identificación de los 4 aislamientos, siguiendo la metodología recomendada por el fabricante.
La identificación molecular demostró que los aislamientos correspondían a Candida parapsilosis (n=2), Candida metapsilosis (n=1) y Candida haemulonii (n=1). El sistema VITEK® MS identificó correctamente las 2 cepas de C. parapsilosis y la de C. haemulonii, pero identificó erróneamente C. metapsilosis como Pichia farinosa. Este sistema no incluye las especies del grupo «psilosis» en la base de datos de MS-ID v1 (bioMérieux).
Nuestros resultados muestran que la identificación de C. sake con API 32C® debería confirmarse con un método molecular. La literatura refiere hasta un 86% de eficacia para especies de Candida frecuentes y de un 85% en las infrecuentes4, pero el auténtico problema de esta prueba es la variabilidad entre observadores y la necesidad de una cierta experiencia a la hora de interpretar las galerías.
Es conveniente evaluar el sistema MALDI-TOF® VITEK® MS para comprobar su posible utilidad como método rápido alternativo, de la misma manera que sería necesario incrementar su base de datos. Algunos trabajos presentan excelentes resultados respecto a la identificación de levaduras por el sistema MALDI-TOF® con rangos de eficacia del 95 al 100%1,3,5.
FinanciaciónNinguna.
Conflicto de interesesNinguno.
Hospital Universitario Marqués de Valdecilla.