INTRODUCCIÓN
El gen HSPA2 codifica para la proteína de HSPA2 (Heat Shock Protein A2) y presenta un único exón de 1.920 pb1. Pertenece a la familia de proteínas de choque térmico (HSP) que son chaperonas moleculares que asisten a otras proteínas en su plegado y transporte. La proteína HSPA2 o HSPA70.2 es un tipo específico de HSP ubicado en la especie humana locus 14q24.1 y que se expresa especialmente en las células espermatogénicas2. Se han descrito diversas funciones en la espermatogénesis. Son esenciales para el proceso meiótico, se asocian al complejo sinaptonémico y se relacionan con la inhibición apoptótica3. También tienen un papel importante en la espermiogénesis durante la transición núcleo-histona núcleo-protamina en las espermátidas alongadas2. En general participan en la maduración del espermatozoide3,4. De relevancia se ha descrito que los ratones knock-out para HSPA2 presentan una parada madurativa en el estadio de espermatocitos primarios y son estériles5.
Adicionalmente, en estudios previos en nuestro grupo detectamos la proteína HSPA2 a través de técnicas proteómicas6. Subsiguientemente se detectó evidencia de un incremento de la HSPA2 en pacientes estériles con un incremento de lesión germinal determinada por TUNEL7. Más recientemente hemos podido demostrar la presencia de valores incrementados de HSPA2 en pacientes astenozoospérmicos8. También de relevancia, y utilizando el cerdo como modelo, se ha descrito una asociación del gen HSPA2 con la calidad espermática9,10. Todas estas razones sugerían la posibilidad de que variantes del gen HSPA2 en la especie humana pudiesen estar relacionadas con algunos casos de esterilidad. Por tanto iniciamos el presente trabajo con los objetivos de analizar en más detalle la relación entre incremento de HSPA2 y lesión germinal y de realizar un estudio mutacional del gen HSPA2 en pacientes varones estériles.
MÉTODOS
Sujetos y recogida de muestras
Las muestras de los pacientes estériles provienen del Laboratorio de Andrología y del Laboratorio de Genética Molecular del Hospital Clínic i Provincial de Barcelona. El comité de bioética del hospital aprobó este proyecto y se obtuvo consentimiento informado de los participantes. Las muestras de semen fueron recogidas por masturbación en envases estériles después de al menos 3 días de abstinencia sexual, pero no más de 7 días. Las muestras se mantuvieron entre 22 y 37 °C de temperatura y fueron evaluadas en el laboratorio dentro de 1 h desde su recogida. Posteriormente, la muestra sobrante que normalmente se desecha fue procesada para su análisis proteómico y de fragmentación del ADN, o para proceder al aislamiento de ADN.
Análisis proteómico de la HSPA2
Se incluyeron un total de 25 muestras de semen de 25 pacientes para el análisis proteómico de la proteína HSPA2. El plasma seminal se eliminó mediante gradiente de Percoll al 50%11. Los espermatozoides se resuspendieron en ham F10 1× y se lavaron 2 veces con el mismo medio. Se calculó un volumen de buffer de lisis para obtener una concentración de 230 millones de espermatozoides/ml en un volumen de 300 µl por cada tira de isoelectroenfoque (IEF). Las muestras se incubaron 1 h a temperatura ambiente y se centrifugaron a 3.000 g 5 min a 4 °C. Seguidamente se procedió a realizar el IEF y la electroforesis. Las proteínas solubilizadas se colocaron en la bandeja de rehidratación con tiras de 17 cm (pH: 5-8) lineales y rehidratadas durante 12 h. El IEF se realizó a 20 °C 15 min (0-250 V), 2 h (250-10.000 V), 45.000-60.000 V/h (10.000 V) y 10 h (50 V). Una vez terminado el IEF, las tiras se equilibraron en 6 M urea, 0,375 M tris-HCl, pH 8,8, 20% glicerol, SDS 2% y 2% DTT 10 min, seguido por el mismo tampón sin DTT y suplementado con 2,5% iodoacetamida durante 10 min. La segunda dimensión por electroforesis se realizó con una protean II a 300 V durante 3 h7,12. Finalmente se procedió a teñir los geles 2D con pink flamingo y se escanearon con typhoon SG-800. Las imágenes se procesaron con el software PDQuest 7.1.1., y la proteína HSPA2 se identificó por su movilidad relativa (basada en el punto isoeléctrico y peso molecular) en comparación con geles previos con las proteínas identificadas mediante espectrometría de masas6-8,12. La cuantificación de la intensidad de la proteína HSPA2 correspondiente a los 25 bidimensionales (1 de cada paciente) se realizó también con el software PDQuest 7.1.1.
Ensayo de TUNEL
Una alícuota de semen correspondiente a cada una de las 25 muestras procesadas para proteómica también fue procesada para su análisis mediante TUNEL (terminal deoxynucleotidyl transferase-mediated dUTP nick end-labeling) a través de microscopia óptica empleando el kit de detección de muerte celular in situ de Roche y un microscopio Olympus DX-50. Los datos fueron recogidos y procesados usando el software CELLQUEST y WinMDI v2.813.
Aislamiento del ADN
Para aislar el ADN correspondiente a los 7 pacientes teratozoospérmicos incluidos en el estudio, se partió de una alícuota de la muestra de semen y se utilizó el método de tiocinato de guanidina modificado14,15.
Brevemente se procesaron 5 millones de espermatozoides que se trataron con 1 ml de tampón de lisis (6 M tiocianato de guanidina; 30 mM citrato sódico a pH 7; 0,5% sarcosil; 0,20 mg/ml proteinasa K; 0,3 M β-mercaptoetanol) durante 4 h a 55 °C. A continuación se añadió el doble de volumen de alcohol isopropílico para precipitar el ADN. Subsiguientemente se realizaron 2 lavados con etanol 70% para eliminar las sales y, finalmente, disolviendo el ADN en TE (tris 10 mM pH 8,0, EDTA 0,1 mM) procediendo finalmente a la lectura de su concentración mediante el espectrofotómetro Nanodrop ND-100015. Para aislar el ADN correspondiente a los pacientes azoospérmicos, oligoastenoteratozoospérmicos y de los controles con fertilidad probada se partió de muestras de sangre periférica. El aislamiento de ADN se realizó mediante el kit QIAamp DNA Mini de Qiagen según su protocolo. Una vez obtenido el ADN se realizó su lectura mediante el espectrofotómetro Nanodrop ND-1000. Para proceder al estudio de mutaciones se utilizó el sobrante de ADN después de proceder a los estudios de microdeleciones del cromosoma Y practicados a los pacientes azoospérmicos o oligoastenoteratozoospérmicos (ninguno de estos pacientes presentaba una microdeleción del cromosoma Y).
Amplificación y secuenciación del gen HSPA2
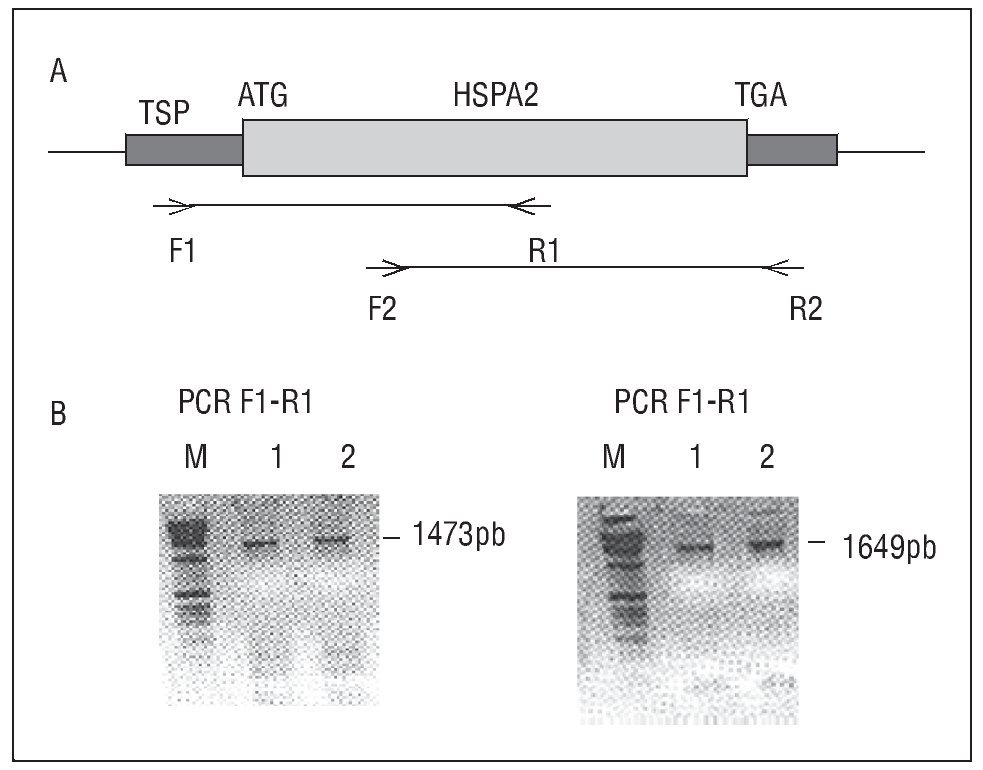
Previamente se diseñaron oligonucleótidos específicos con el programa Primer3 para conseguir amplificar y secuenciar de forma específica el gen HSPA2 (fig. 1), discriminándolo de seudogenes y genes homólogos pertenecientes a la familia de la HSP. La secuencia de los oligonucleótidos diseñados (fig. 1A) es F1 (5'-GTGGAGCTGGAAGAGTTTGTGAG-3'), R1 (5'-CC- ACTCATCAAGAGGAACACCAC-3'), F2 (5'-GAA- CGTGCTCATCTTTGACCTG-3'), R2 (5'-CACAC- CTGTTCTGTAGAAGCTTG-3'). Los reactivos incluidos en la reacción de amplificación fueron los siguientes: 1 µl de ADN; 1,25 µl de cada pareja de oligonucleótidos (20 pmol/µl); 0,5 µl de DMSO para evitar la formación de dímeros de oligonucleótidos; 1,5 µl de Cl2Mg, y 0,1 volumen de tampón de reacción 10× incluido en el kit de PCR (Biotherm DNA polimerasa) en un volumen final de 25 µl. Las condiciones de amplificación por PCR fueron las siguientes: 96 °C durante 5 min seguidos de 35 ciclos de 96 °C durante 1 min, temperatura de hibridación de los oligonucleótidos cebadores durante 30 s (F1-R1: 58 °C; F2-R2: 65 °C) y la extensión de bases a 72 °C (F1-R1: 4 min; F2-R2: 5 min). Una vez terminada la reacción de PCR se procedió a la comprobación del tamaño de los productos de amplificación obtenidos a través de su electroforesis en gel agarosa (fig. 2B).
Figura 1. Estructura del gen de la HSPA2 y oligonucleótidos utilizados para su amplificación por PCR. A) Se muestra la estructura del gen HSPA2 y se indica la combinación de los oligonucleótidos utilizados. B) Resultado de la amplificación del gen HSPA2. El producto de amplificación correspondiente a las 2 combinaciones de oligonucleótidos se ha separado mediante electroforesis en gel de agarosa. Se aprecian las bandas correspondientes a los productos de amplificación del gen HSPA2 esperados de 1.473 y 1.649 pares de bases, respectivamente.
Figura 2. Correlación entre los valores de HSPA2 y los resultados de TUNEL en pacientes estériles. A) Detección de la proteína HSPA2 de espermatozoides mediante electroforesis bidimensional identificada previamente por nuestro grupo a través de espectrometría de masas. B) Gráfico de dispersión entre los resultados de expresión de la proteína HSPA2 y la fragmentación del ADN determinada por TUNEL. Cada símbolo corresponde a un paciente independiente. Se detecta una correlación positiva entre la intensidad correspondiente a la HSPA2 y la fragmentación del ADN determinada por TUNEL en pacientes estériles (p = 0,044).
Para proceder a su secuenciación se realizó un tratamiento previo con 2 µl de exosap-IT por 5 µl de producto de PCR para eliminar los oligonucleótidos y los nucleótidos sobrantes de la PCR. Se incubaron a 37 °C durante 20 min y a 80 °C durante 10 min. La reacción de secuenciación se realizó con el kit de secuenciación cíclica BigDye Terminator v3.1 siguiendo las instrucciones del kit comercial. Para acabar se precipita el producto de secuenciación, para lo cual se añaden 5µl de EDTA 125 mM y 60 µl de etanol 100% a temperatura ambiente, se deja precipitar durante 20 min y se centrifuga a 12.000 rpm 15 min a 4 °C. Se descarta entonces el sobrenadante y se lava el sedimento con 50 µl de etanol al 70% en frío. Finalmente se centrifuga a 12.000 rpm, 2 min a 4 °C, se descarta el sobrenadante y se seca el pellet a temperatura ambiente. Una vez precipitado el ANA ya está preparado para cargar en el secuenciador automático DNA 3100. Una vez obtenida la secuencia se procedió al análisis de la región codificante mediante los programes CHROMAS y DNASTAR.
Análisis estadístico
Para el análisis de los resultados se utilizó el paquete estadístico SPSS 14.0. La búsqueda de correlaciones entre los valores de HSPA2 y los resultados de TUNEL se analizó mediante el test de Spearman. Las posibles asociaciones entre los polimorfismos identificados y los fenotipos se exploraron mediante el test de χ2. En todos los casos el valor aceptado de significación estadística fue de 0,05.
RESULTADOS
Correlación entre los niveles de HSPA2 y resultados del ensayo de TUNEL
Se ha detectado una correlación positiva entre la intensidad del punto proteico de las bidimensionales correspondiente a la HSPA2 (fig. 2A) y la fragmentación del ADN determinada por TUNEL en pacientes estériles (p = 0,044 -Mann-Whitney-). El detalle del diagrama de dispersión junto con la recta de regresión se muestran en la figura 2B7. A parte de la tendencia de correlación significativa indicada se aprecia también una elevada dispersión de los puntos, posiblemente debido a la existencia de diversos factores adicionales que afectan los valores de HSPA2 y los resultados de TUNEL.
Estudio mutacional del gen HSPA2
El análisis de las secuencias en el gen HSPA2 en las muestras estudiadas, no ha detectado ninguna mutación que resulte en un cambio del aminoácido o que provoque un truncamiento de la proteína o un cambio en la marca de lectura. Por tanto, no se ha encontrado ninguna mutación que pueda ser considerada patogénica. Se han detectado 3 polimorfismos (tipo SNP) que no comportan ningún cambio de aminoácido. Encontramos que 2 de los SNP ya se han descrito previamente (NCBI). Uno de estos SNP corresponde a la variante c.684 C>T (p.Asp228Asp) encontrada en heterocigosis en 10 pacientes azoospérmicos con parada madurativa, en 4 oligoastenoteratozoospérmicos, en 2 teratozoospérmicos y en 2 controles. Esta variante fue observada en homocigosis en 6 pacientes azoospérmicos con parada madurativa, en 1 paciente teratozoospérmico y en 1 paciente control (tabla 1). El otro SNP ya descrito previamente corresponde a la variante c.1653 C>T (p.Asn551Asn) detectada en heterocigosis en un paciente con azoospermia y parada madurativa y en un paciente con oligoastenoteratozoospermia severa (tabla 1). También hemos encontrado 1 SNP nuevo no descrito previamente. Este SNP corresponde a la variante c.1443 C>T (p.Phe481Phe) que se observa en heterocigosis en 1 paciente azoospérmico con parada madurativa y en un paciente oligoastenoteratozoospérmico (tabla 1). El análisis estadístico indica que los genotipos 684 TT y TC se detectan incrementados dentro del grupo de pacientes azoospérmicos en comparación con el resto de pacientes (16 pacientes azoospérmicos TT + TC y ninguno CC, en comparación con 7 pacientes oligosatenoteratozoospérmicos o teratozoospérmicos TT + TC y 6 pacientes CC; p < 0,005) (tabla 1). Adicionalmente, la frecuencia alélica del alelo 684 T resultó estar incrementada dentro del grupo de pacientes azoospérmicos (22 alelos T y 10 alelos C en comparación con 8 alelos T y 18 alelos C dentro del resto de pacientes; p < 0,005) (tabla 1).
DISCUSIÓN
En este trabajo hemos realizado un estudio mutacional del gen HSPA2 en distintos grupos de pacientes estériles y hemos detectado 3 polimorfismos, uno de los cuales no se había descrito previamente, y no se ha detectado ninguna mutación patogénica. El estudio mutacional del gen HSPA2 está justificado por los antecedentes en la bibliografía que indican una expresión del gen correspondiente durante la espermatogénesis2,3,9 y la existencia de parada madurativa e infertilidad en ratones knock-out para HSPA25, datos que sugerían que este gen era un buen candidato para explicar algunos de los casos de infertilidad. Este interés resulta apoyado por los resultados de estudios previos en nuestro laboratorio en los que detectamos la proteína HSPA2 mediante proteómica6 y un incremento de la HSPA2 en pacientes estériles con un incremento de lesión germinal determinada por TUNEL7 o un incremento de HSPA2 en pacientes astenozoospérmicos8. En el presente trabajo ampliamos la detección de la correlación mostrando el detalle de los diagramas de dispersión entre los valores de HSPA2 y los resultados de TUNEL (fig. 2). Los resultados confirman la existencia de una correlación positiva entre los valores de HSPA2 y los resultados de TUNEL, si bien también se evidencia un elevado grado de dispersión en los datos sugiriendo la concurrencia de otros factores que independientemente afecten a los resultados de TUNEL o de HSPA2 (fig. 2B).
La realización del estudio mutacional ha requerido el diseño de oligonucleótidos específicos para conseguir amplificar y secuenciar de forma específica el gen HSPA2 (fig. 1) discriminándolo de seudogenes y genes homólogos pertenecientes a la familia de la HSP1. La secuencia de estos oligonucleótidos y condiciones de PCR se describen por primera vez en este artículo y pueden servir como referencia para estudios posteriores. Los resultados del estudio mutacional excluyen la existencia de mutaciones patogénicas en el grupo de 29 pacientes en los que se ha secuenciado completamente la región codificante del gen HSPA2. Este resultado sugiere que la presencia de mutaciones patogénicas en el gen HSPA2, en caso de que existan, debe ser una causa relativamente infrecuente de bloqueo madurativo o de alteración de los parámetros seminales en pacientes con dificultades reproductivas. También es importante dar a conocer este resultado, aunque negativo, a la comunidad científica.
En el presente trabajo también hemos detectado la presencia de 3 polimorfismos tipo SNP que no comportan ningún cambio de aminoácido. Dos de los SNP (c.684 C>T, y c.1653 C>T) (tabla 1) ya habían sido comunicados previamente16, pero el tercer SNP encontrado corresponde a una variante nueva no descrita previamente (c.1443 C>T) (tabla 1) observada en heterocigosis en un paciente azoospérmico con parada madurativa y en un paciente oligoastenoteratozoospérmico (tabla 1). Es interesante notar que los genotipos 684 TT y TC se detectan incrementados dentro del grupo de pacientes azoospérmicos en comparación con el resto de pacientes (p < 0,005) (tabla 1). Este resultado sugiere que la presencia del alelo 684 T puede comportarse como un factor de riesgo de azoospermia. No obstante, este resultado no viene apoyado por los resultados recientemente comunicados en una población independiente16. Ninguno de los otros 2 genotipos detectados demostró estar presente con una frecuencia alélica significativamente incrementada en alguno de los subgrupos de pacientes. La detección de la relación detectada del polimorfismo c.684 C>T y esterilidad en nuestra población resulta coherente con los resultados de un estudio previo utilizando el cerdo como modelo, en donde se describió una asociación entre polimorfismos del gen HSPA2 y la calidad espermática4,10.
Como limitación del presente trabajo es preciso indicar que el número de muestras analizadas es relativamente bajo (tabla 1). Por tanto será interesante en un futuro ampliar el estudio para aumentar el tamaño muestral y, por tanto, incrementar la potencia estadística y confirmar la posible implicación de variaciones en el gen de la HSPA2 en la esterilidad masculina.
Financiación
Subvencionado con un proyecto del Ministerio de Ciencia y Tecnología BFU2009-07118.
Correspondencia: Dr. R. Oliva.
Grupo de Genética Humana. Unidad de Genética. Facultad de Medicina. Universidad de Barcelona. Casanova, 143. 08036 Barcelona. España.
Correo electrónico: roliva@ub.edu