Lilaea scilloides (Juncaginaceae) es una especie acuática de humedales temporales. Es una herbácea anual emergente que va desde Canadá hasta Argentina. Tiene reproducción sexual y asexual y es polinizada por viento. Su heteromorfismo floral es notorio con 5 diferentes tipos de flores. Se planteó la hipótesis de que, debido a su polinización, amplio intervalo de distribución y abundancia local, tendría alta diversidad genética, baja endogamia, baja diferenciación genética y alto flujo génico. Los objetivos de este estudio fueron determinar la diversidad y estructura genética de L. scilloides y compararlas con especies con características ecológicas y de historia de vida similares. Se analizaron 40 individuos en cada una de las 12 poblaciones con la técnica de electroforesis de isoenzimas en geles de almidón. Los resultados muestran baja diversidad genética dentro de las poblaciones (A= 1.77, Ae= 1.15, P= 56.6, Ho= 0.064 y He= 0.103), altos índices de endogamia (F15= 0.288), baja diferenciación genética entre las poblaciones (FST= 0.077) y alto flujo génico (Nm= 4.42). Se encontró evidencia de altos niveles de propagación clonal. Los resultados indican que la reproducción asexual es extremadamente importante, así como la posibilidad de que L. scilloides presente hidrofilia, un tipo de polinización no registrado para la especie.
Lilaea scilloides (Juncaginaceae) is an aquatic species inhabiting temporary wetlands. It is an annual herbaceous emergent plant distributed from Canada to Argentina. This species reproduces both sexually and asexually, and is wind-pollinated. Flowers are highly heteromorphic with 5 different types of flowers. We hypothesized that, because of its pollination syndrome, wide distribution and local abundance, the species would have high genetic diversity, low endogamy, low genetic differentiation, and high gene flow. The objectives of this study were to determine the genetic diversity and structure of L. scilloides and compare with species with similar ecological and life history traits. We analyzed 40 individuals from 12 populations using isozyme electrophoresis in starch gels. The results showed a low within population genetic diversity (A= 1.77, Ae= 1.15, P= 56.6, Ho= 0.064 and He= 0.103), a high endogamy index (FIS= 0.288), low genetic differentiation among populations (FST= 0.077), and high gene flow (Nm= 4.42). We also found evidence of a high degree of clonal propagation in the species. These results indicate that asexual reproduction is extremely important for the species, as well as the possibility that Lilaea scilloides has hydrophilous pollination, which has not been previously reported.
La diversidad y estructura genética están correlacionadas con las características ecológicas y de historia de vida de las plantas (e.g., Hamrick et al., 1979; Loveless y Hamrick, 1984; Hamrick y Godt, 1989; Hamrick y Godt, 1996). Sin embargo, las generalizaciones propuestas por estos autores se basan principalmente en estudios de plantas terrestres. Algunos estudios de plantas acuáticas muestran que en la categoría de especie generalmente se presenta mayor diversidad genética que en las plantas terrestres; por el contrario, a nivel de población presentan menor diversidad genética (Barrett et al., 1993; Laushman, 1993; Elam, 1998). La alta diversidad genética de las plantas acuáticas a nivel de especie se debe a los altos niveles de diferenciación genética entre poblaciones (Edwards y Sharitz, 2000; Chen et al., 2008; Wang et al., 2010), lo cual está relacionado con la distribución de sus hábitats, que generalmente se encuentran aislados dentro de paisajes terrestres (Elam, 1998). Esta distribución disminuye la probabilidad de flujo génico entre las poblaciones y aumenta la diferenciación a través de la deriva génica (Barrett et al., 1993; Elam, 1998; Chen et al., 2008). Otra característica de las plantas acuáticas es la diversidad clonal, que tiende a ser más alta en las poblaciones que presentan múltiples morfos florales (Shibayama y Kadono, 2007). Sin embargo, aún no se observan patrones claros para las especies de plantas acuáticas y es probable que su estructura y diversidad genética tenga mayor relación con su historia de vida particular, que con el hecho de vivir en ambientes acuáticos (Magallán et al., 2009; Honnay et al., 2010).
En el centro de México se ha descrito un tipo de humedales temporales con una alta diversidad florística (Martínez y García, 2001). Estos humedales, se distribuyen a manera de parches, lo cual es un aspecto de este tipo de ecosistemas que los hace atractivos para realizar estudios de genética de poblaciones y biología de la conservación (Elam, 1998). Estos humedales presentan una estacionalidad marcada, con un período de inundación de 6 a 8 meses y uno de sequía de 4 a 6 meses; se forman en depresiones poco profundas de suelos impermeables que se llenan únicamente por agua de lluvia, sin drenaje de ríos o arroyos, por lo que se pueden considerar como ecosistemas aislados. El período de inundación coincide con condiciones de temperatura ideal para el crecimiento de las plantas y el nivel de agua disminuye hasta secarse completamente entre diciembre y marzo dependiendo de la precipitación anual.
Lilaea scilloides (Poir.) Hauman (Juncaginaceae) es una monocotiledónea acuática que habita este tipo de humedales. Es una planta enraizada emergente anual con distribución desde Canadá hasta Argentina (Lot et al., 1999; Novelo y Lot, 2001). Esta especie ocupa una posición clave en la evolución floral de las monocotiledóneas (Posluszny et al., 1986). Presenta un notable heteromorfismo floral pues tiene 5 diferentes tipos de flores, 4 en la inflorescencia y una más en la base del escapo. La inflorescencia es una espiga con: i) las flores masculinas unisexuales en la punta de la espiga, ii) unas flores bisexuales con estilo corto en la parte media de la inflorescencia, iii) unas flores femeninas unisexuales con perianto y estilo corto en la base de la espiga y iv) unas flores femeninas unisexuales sin perianto con estilo corto. La quinta flor está en la base del escapo y es femenina con un estilo filamentoso muy largo (Posluszny et al., 1986). Lilaea scilloides es una especie con el síndrome de polinización anemófila debido a su morfología floral (Cook, 1988; Lot et al., 1999). Lilaea scilloides presenta 2 tipos de aquenios, los de la inflorescencia tienen 1 ala dorsal y 2 laterales, mientras que los de la base de la inflorescencia tienen estructuras parecidas a cuernos en su parte superior y no tienen alas (Lot et al., 1999; Novelo y Lot, 2001); la dispersión es hidrócora y ornitócora (Cook, 1988; Cook, 1996; Lot et al., 1999).
Los objetivos de este estudio fueron determinar la diversidad y estructura genética de las poblaciones de L. scilloides y compararlas con los patrones reportados para especies con características ecológicas y de historia de vida similares. Debido a su polinización anemófila, su amplia distribución y su abundancia en las poblaciones locales se hipotetizó que L. scilloides tendría alta diversidad genética, bajos índices de endogamia, baja diferenciación genética y alto flujo génico interpoblacional.
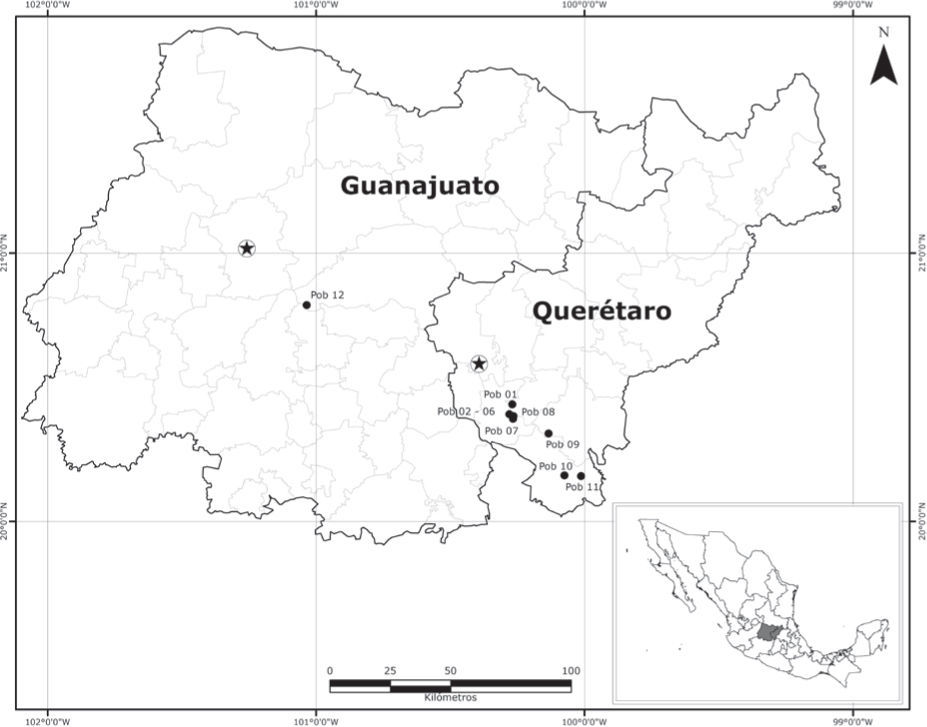
Materiales y métodosSe recolectaron 480 individuos de L. scilloides en 12 poblaciones; de éstas, 11 se distribuyen en Querétaro y 1 en Guanajuato, en el centro de México (Fig. 1). Las plantas se colectaron de forma aleatoria separadas entre sí al menos 1 m, se transportaron a 4° C y en el laboratorio se almacenaron en bolsas de plástico a -70° C.
Se usó la técnica de electroforesis horizontal de isoenzimas en geles de almidón (Cheliak y Pitel, 1984; Werth, 1985; Wendel y Weeden, 1989). Para la extracción de enzimas se usó el buffer "Microbuffer" (Werth, 1985), el procedimiento consistió en macerar un fragmento de hoja con 2 gotas del buffer de extracción y una pizca de arena esterilizada, con el extracto obtenido, se impregnaron los "wicks" hechos con papel de cromatografía Whatman 3MM. Los "wicks" se almacenaron a -70° C para ser utilizados al siguiente día. Los geles se prepararon a concentraciones del 12% de almidón (Wendel y Weeden, 1989). Se usaron 8 enzimas en 2 sistemas: C (APX, GOT, ME y PGI) y D (GDH, G-6PD, PGD y PGM). El sistema C se corrió a 65 mA durante 8 horas y el sistema D se corrió a 30 mA durante 9 horas. Los geles se tiñeron a 37° C. La nomenclatura de los loci y de los alelos se estableció de la siguiente manera: a los loci que estuvieran más cerca del ánodo se les asignó el número 1, los que seguían el número 2 y así sucesivamente conforme se acercaran al cátodo, se usó el mismo criterio para los alelos, asignando letras. Al momento de introducir los "wicks" en el gel se incluyó un "wick" testigo con genotipo conocido para asignar correctamente la nomenclatura de los loci y alelos. Se usaron "wicks" impregnados con amido black para determinar el frente de migración. La información de la estructura cuaternaria de las enzimas, que sirvió para llevar a cabo la adecuada interpretación de las bandas, se obtuvo de Wendel y Weeden (1989). Se llevó a cabo una primera lectura de los patrones de bandeo con los geles en fresco; éstos se fijaron en alcohol al 50% o en una solución 5:5:1 de metanol, agua y ácido acético.
Para los análisis estadísticos, se obtuvieron los genotipos individuales para cada población y se conformaron matrices de datos. Se utilizó el programa FSTAT (Goudet, 1995) para estimar en cada una de las poblaciones muestreadas el número promedio de alelos por locus (A), el número efectivo de alelos por locus (Ae), el porcentaje de loci polimórficos (P), la heterocigosidad observada (Ho), la heterocigosidad esperada (He) y el índice de fijación (F). La significancia de las desviaciones con respecto al equilibrio Hardy-Weinberg para cada locus en cada población se determinó mediante una prueba exacta, y la significancia de los valores globales mediante la prueba U, implementadas en el programa GENEPOP (Raymond y Rousset, 1995).
La base de datos se analizó para identificar individuos con genotipos multilocus idénticos (posibles clones), mediante el programa GENALEX 6.2 (Peakall y Smouse, 2006). Para cada genotipo multilocus repetido se calculó el valor de Psex, que es la probabilidad de observar n repeticiones de un genotipo dado en una muestra de tamaño N mediante reproducción sexual en una población con apareamientos aleatorios.
La diferenciación genética entre las poblaciones se estimó utilizando el método de Weir y Cockerham (1984) para calcular los estadísticos F de Wright usando el programa GENETIX 4 (Belkhir et al., 2004). El flujo génico entre poblaciones se calculó a partir del valor promedio de Fst mediante la fórmula Nm= (1 - Fst) / 4 Fst (Wright, 1951).
Adicionalmente, se realizó un análisis de varianza molecular (AMOVA) mediante el programa ARLEQUIN 3.11 (Excoffier et al., 2005), el cual subdivide la varianza molecular total en sus componentes inter-poblacional e intra-poblacional, mediante el cálculo de los estadísticos ?, que son análogos a los estadísticos F de Wright. La significancia de cada uno de los componentes de la varianza se estimó a partir de distribuciones generadas mediante 10 000 permutaciones al azar.
La estructura genética también se evaluó mediante el análisis de asignación Bayesiano implementado en el programa STRUCTURE ver. 2.3.1 (Falush et al., 2003). En este análisis los individuos son asignados probabilísticamente a uno de K grupos genéticos, donde K puede tomar sucesivos valores desde K= 1 hasta un número definido por el usuario. Al final del análisis se determina el valor de K que tiene mayor probabilidad dados los datos (Falush et al., 2003). En este caso, se probaron valores desde K= 1 hasta K= 12 y se realizaron 10 corridas para cada valor de K. Se utilizó el modelo que permite mezcla entre los grupos genéticos y frecuencias alélicas correlacionadas entre poblaciones. No se proporcionó al programa información previa sobre la población de origen de los individuos. Cada corrida tuvo un "burn-in" de 500 000 pasos, seguidos de 106 iteraciones. El número de grupos genéticos más probable se encontró determinando el valor máximo del estadístico AK, según el método de Evanno et al. (2005).
Finalmente, las relaciones de similitud entre las poblaciones se analizaron mediante la identidad genética (I) de Nei (1978) entre todos los pares posibles de poblaciones.
ResultadosSe registraron un total de 45 alelos en los 14 loci definidos. Todos los loci fueron polimórficos excepto PGD-1. El número promedio de alelos por locus (A), incluyendo los 14 loci, fue de 1.77 con un intervalo de 1.29 a 2.07, mientras que el promedio del número efectivo de alelos por locus (Ae) fue de 1.15 con un intervalo de 1.09 a 1.25. En todas las poblaciones Ae fue menor que A, lo cual indica presencia de alelos raros. El porcentaje de loci polimórficos (P) fue de 56.6% con un intervalo de 28.6 a 71.4%. El promedio de heterocigosidad observada (Ho) fue de 0.064 con un intervalo de 0.009 a 0.115 y el promedio de heterocigosidad esperada (He) fue de 0.103 con un intervalo de 0.057 a 0.156. Estos valores se muestran en el cuadro 1.
Parámetros de variación genética estimados para las doce poblaciones estudiadas de Lilaea scilloides. N= tamaño de la muestra, A= número de alelos por locus, Ae= número efectivo de alelos por locus, P= porcentaje de loci polimórficos, Ho= heterocigosidad observada, He= heterocigosidad esperada, F= índice de fijación, Nc= número de clones (individuos que tuvieron el mismo genotipo multilocus que al menos otro individuo), Ngc= Número de genotipos con clones (número de genotipos multilocus que se encontraron repetidos en la población). Los errores estándar asociados a los promedios están entre paréntesis
| Población | N | A | Ae | P | Ho | He | F | Nc | Ngc |
|---|---|---|---|---|---|---|---|---|---|
| P. Escobedo 1 | 39.1 (0.9) | 1.57 (0.2) | 1.08 (0.1) | 50.0 | 0.036 (0.01) | 0.057 (0.03) | 0.089 (0.09) | 33 | 6 |
| P. Escobedo 2 | 39.1 (0.5) | 1.79 (0.2) | 1.19 (0.08) | 57.1 | 0.061 (0.04) | 0.121 (0.04) | 0.433 (0.16) | 23 | 7 |
| Huimilpan 3 | 37.4 (1.4) | 1.29 (0.1) | 1.09 (0.06) | 28.6 | 0.017 (0.02) | 0.060 (0.04) | 0.574 (0.14) | 28 | 8 |
| Huimilpan 4 | 36.1 (1.9) | 1.93 (0.2) | 1.18 (0.06) | 64.3 | 0.082 (0.03) | 0.127 (0.04) | 0.272 (0.12) | 8 | 3 |
| Huimilpan 5 | 35.6 (1.4) | 1.86 (0.2) | 1.14 (0.06) | 64.3 | 0.009 (0.01) | 0.101 (0.03) | 0.641 (0.13) | 21 | 6 |
| Huimilpan 6 | 38.7 (0.4) | 2.07 (0.2) | 1.25 (0.09) | 71.4 | 0.111 (0.04) | 0.152 (0.05) | 0.177 (0.12) | 16 | 6 |
| Huimilpan 7 | 39.4 (0.3) | 1.71 (0.2) | 1.13 (0.06) | 57.1 | 0.081 (0.03) | 0.092 (0.04) | 0.184 (0.10) | 26 | 7 |
| Huimilpan 8 | 38.9 (0.7) | 1.79 (0.3) | 1.12 (0.06) | 50.0 | 0.051 (0.02) | 0.081 (0.03) | 0.171 (0.10) | 20 | 6 |
| San Juan 9 | 39.1 (0.7) | 1.86 (0.2) | 1.08 (0.03) | 71.4 | 0.064 (0.03) | 0.065 (0.02) | 0.119 (0.10) | 26 | 5 |
| Amealco 10 | 38.8 (0.9) | 1.64 (0.2) | 1.11 (0.05) | 42.9 | 0.076 (0.04) | 0.078 (0.03) | 0.033 (0.06) | 24 | 7 |
| Amealco 11 | 39.9 (0.1) | 1.79 (0.2) | 1.24 (0.10) | 57.1 | 0.115 (0.04) | 0.157 (0.05) | 0.169 (0.12) | 17 | 6 |
| J. Rosas 12 | 35.9 (1.8) | 2.00 (0.3) | 1.24 (0.1) | 64.3 | 0.069 (0.03) | 0.148 (0.05) | 0.424 (0.12) | 8 | 3 |
| Promedio | 38.1 (0.3) | 1.77 (0.06) | 1.15 (0.02) | 56.6 | 0.064 (0.01) | 0.103 (0.01) | 0.269 (0.04) |
Se obtuvieron 95 valores de índice de fijación o endogamia (F) en los 13 loci polimórficos de las 12 poblaciones estudiadas, de los cuales 30 fueron significativamente diferentes de cero y positivos, indicando exceso de individuos homocigotos, y 2 fueron significativamente diferentes de cero y negativos, indicando exceso de individuos heterocigotos. Las poblaciones Pedro Escobedo 1, Huimilpan 6, Huimilpan 7, San Juan 9, Amealco 10 y Amealco 11 no presentan valores significativamente diferentes de cero, indicando que estas poblaciones se encuentran en equilibrio de Hardy-Weinberg, mientras que las poblaciones Pedro Escobedo 2, Huimilpan 3, Huimilpan 4, Huimilpan 5, Huimilpan 8 y J. Rosas 12 presentan valores positivos y significativamente diferentes de cero, indicando endogamia en estas poblaciones. Los valores promedio de F por población se muestran en el cuadro 1.
El análisis de identificación de clones mostró un total de 182 genotipos multilocus únicos y 52 genotipos repetidos, cada uno entre 2 y 78 veces. Las población que mostró mayor presencia de clonalidad es Pedro Escobedo 1 (33 individuos tienen al menos un clon), mientras que las que tuvieron menor clonalidad fueron las poblaciones Humilpan 4 y J. Rosas 12 (con 8 individuos con algún clon) (Cuadro 1). Es interesante destacar que los genotipos multilocus con mayor número de repeticiones estuvieron presentes en varias poblaciones y no en una sola. Por ejemplo, el genotipo repetido más abundante se encontró en todas las poblaciones, excepto en la población 12. En todos los casos la probabilidad de repetición de los genotipos multilocus a través de reproducción sexual (Psex) fue baja (> 0.05).
Los estadísticos F de Wright: FIS FIT y FST calculados mediante el método de Weir y Cockerham (1984) se muestran en el cuadro 2. Las estimaciones de FIS fueron positivas y significativamente diferentes de cero en 8 loci, es decir, en 61.53% de los loci polimórficos existe exceso de homocigotos. El promedio de FIS fue de 0.288 y significativamente diferente de cero, indicando que existe endogamia al nivel de los individuos dentro de las poblaciones locales. Las estimaciones de FIT fueron positivas y significativamente diferentes de cero en 8 loci, indicando que 61.53% de los loci polimórficos presentan exceso de homocigotos. El promedio de FIT presentó un valor de 0.342 y fue significativamente diferente de cero. El promedio de FST tuvo un valor de 0.077 y fue significativamente diferente de cero, lo cual indica diferenciación genética moderada entre las poblaciones (Hartl y Clark, 1997).
Estadísticos F de Wright para Lilaea scilloides. FIS= coeficiente de endogamia entre individuos, FIT= coeficiente de endogamia entre individuos, FST= diferenciación genética entre subpoblaciones. Los valores significativamente diferentes de cero se denotan con asteriscos: *** p< 0.001; ** p< 0.01; * p< 0.05
| Locus | FIS | FIT | FST |
|---|---|---|---|
| APX-1 | 0.235*** | 0.285*** | 0.065*** |
| GOT-1 | 0.426*** | 0.482*** | 0.098*** |
| GOT-2 | 0.885*** | 0.895*** | 0.092*** |
| GOT-3 | 0.698*** | 0.721*** | 0.077*** |
| GDH-1 | 0.452*** | 0.468*** | 0.030*** |
| G6PD1 | -0.175*** | -0.017 | 0.134*** |
| G6PD2 | -0.013 | -0.001 | 0.011 |
| ME--1 | 0.222*** | 0.292*** | 0.091*** |
| ME--2 | 0.561*** | 0.616*** | 0.124*** |
| PGD-2 | 0.592*** | 0.616*** | 0.057*** |
| PGI-1 | -0.120** | -0.005 | 0.103*** |
| PGI-2 | -0.019 | 0.067 | 0.084*** |
| PGM-1 | -0.006 | 0.034 | 0.040*** |
| Promedio | 0.288*** | 0.342*** | 0.077*** |
El flujo génico entre poblaciones (Nm) presentó un valor de 4.421 (calculado a partir del promedio de FST). Como el valor de Nm es mayor a uno se considera que existe alto flujo génico entre las 12 poblaciones locales estudiadas de L. scilloides (Cuadro 3).
Flujo génico entre las doce poblaciones locales de Lilaea scilloides. FST= diferenciación genética entre subpoblaciones, Nm= flujo génico
| Locus | FST | Nm |
|---|---|---|
| APX-1 | 0.065 | 3.596 |
| GOT-1 | 0.098 | 2.301 |
| GOT-2 | 0.092 | 2.467 |
| GOT-3 | 0.077 | 2.997 |
| GDH-1 | 0.03 | 8.083 |
| G6PD-1 | 0.134 | 1.616 |
| G6PD-2 | 0.011 | 22.477 |
| ME-1 | 0.091 | 2.497 |
| ME-2 | 0.124 | 1.766 |
| PGD-2 | 0.057 | 4.136 |
| PGI-1 | 0.103 | 2.177 |
| PGI-2 | 0.084 | 2.726 |
| PGM-1 | 0.040 | 6.000 |
| Promedio | 0.077 | 4.421 |
El análisis de varianza molecular (AMOVA) indicó que el 15% de la varianza genética total se encuentra entre poblaciones (?st= 0.154; p= 0.01), mientras que el 85% de la variación genética se encuentra dentro de las poblaciones.
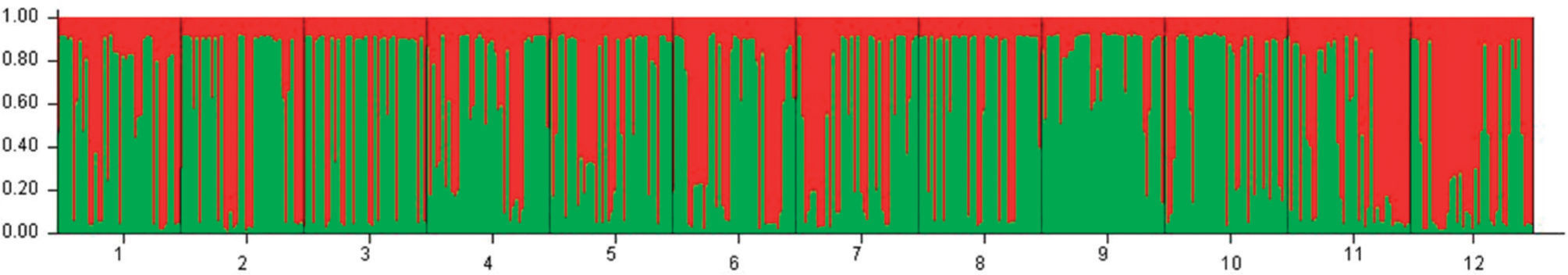
Los resultados de STRUCTURE, analizados mediante el estadístico ?K mostraron que K= 2 es el número de grupos genéticos más probable dados los datos (Figs. 2, 3). Ambos grupos genéticos se encuentran presentes en todas las poblaciones, aunque destaca que la población 12 (J. Rosas, Guanajuato) tiene la mayor proporción del grupo genético representado en rojo, mientras que la población 9 (San Juan, Querétaro) muestra una preponderancia del grupo genético representado en verde. La proporción de individuos que muestran evidencia de ancestría mezclada entre los 2 grupos genéticos es relativamente baja, lo cual puede deberse a una mayor frecuencia de reproducción clonal en comparación con la reproducción sexual, como indicó el análisis de los genotipos multilocus.
Resultados del análisis de asignación bayesiana mediante el programa STRUCTURE para 2 grupos genéticos (K= 2). Cada una de las barras horizontales delgadas representa a un individuo y la proporción de cada color es la proporción de ancestría derivada de cada uno de los 2 grupos genéticos. Las líneas negras separan a cada una de las 12 poblaciones.
Las identidades genéticas de Nei (1978) tienen un intervalo de 0.973 a 1.000, con un promedio de I= 0.990, indicando que las 12 poblaciones locales estudiadas son muy parecidas genéticamente (Cuadro 4).
Matriz de coeficientes de identidades genéticas de Nei (1978) para Lilaea scilloides. Bajo la diagonal: Identidades genéticas no sesgadas
| Población | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Pedro Escobedo 1 | **** | |||||||||||
| Pedro Escobedo 2 | 0.992 | **** | ||||||||||
| Huimilpan 3 | 0.995 | 0.991 | **** | |||||||||
| Huimilpan 4 | 0.990 | 0.988 | 0.990 | **** | ||||||||
| Huimilpan 5 | 0.997 | 0.991 | 0.996 | 0.988 | **** | |||||||
| Huimilpan 6 | 0.989 | 0.995 | 0.986 | 0.992 | 0.989 | **** | ||||||
| Huimilpan 7 | 0.995 | 0.987 | 1.000 | .992 | 0.995 | 0.983 | **** | |||||
| Huimilpan 8 | 0.998 | 0.995 | 0.999 | 0.994 | 0.997 | 0.992 | 0.998 | **** | ||||
| San Juan 9 | 0.995 | 0.990 | 0.988 | 0.993 | 0.990 | 0.990 | 0.987 | 0.993 | **** | |||
| Amealco 10 | 0.996 | 0.995 | 0.991 | 0.995 | 0.993 | 0.996 | 0.989 | 0.995 | 0.998 | **** | ||
| Amealco 11 | 0.988 | 0.992 | 0.987 | 0.984 | 0.987 | 0.993 | 0.983 | 0.991 | 0.982 | 0.989 | **** | |
| J. Rosas 12 | 0.988 | 0.991 | 0.988 | 0.973 | 0.988 | 0.981 | 0.985 | 0.988 | 0.976 | 0.983 | 0.985 | **** |
Al comparar la diversidad genética a nivel de población de L. scilloides (A= 1.77, P= 56.60, He= 0.103) con el promedio registrado por Hamrick y Godt (1989) para especies terrestres con características similares, tales como especies anuales (A= 1.48, P= 30.20, He= 0.105), con distribución amplia (A= 1.72, P= 43.00, He= 0.159) y con reproducción sexual y asexual (A= 1.47, P= 29.40, He= 0.103), se observa que L. scilloides presenta niveles similares de diversidad genética. En cambio, si se compara con especies que presentan polinización por viento (A= 1.79, P= 49.70, He= 0.148) se observa que sus niveles de diversidad genética son más bajos, lo que parece indicar que existen otros factores, además de la anemofilia, que están determinando la diversidad genética de la especie. La diversidad genética dentro de las poblaciones de L. scilloides (A= 1.77, P= 56.60, He= 0.103) fue comparada con los estudios en especies acuáticas anemófilas, como Carex basiantha (A= 1.46, P= 40.21, He= 0.138) y Carex willdenowii (A= 1.50, P= 40.00, He= 0.148) (Ford et al., 1998) y Typha minima (A= 1.50, He= 0.146) (Galeuchet et al., 2002), observándose que L. scilloides presenta valores muy similares en el número de alelos por locus y porcentaje de loci polimórficos y más bajos en la heterocigosidad esperada, que no corresponde con lo esperado para especies anemófilas.
Lilaea scilloides se considera anemófila debido a las características florales de las espigas (Cook, 1988), sin embargo no se ha considerado el gran heteromorfismo floral de la especie, que permite la posibilidad de autogamia, anemofilia e hidrofilia. Esta especie presenta un tipo de flores femeninas que crecen en la base de la inflorescencia y que nunca emergen del agua, en algunas de las cuales observamos la presencia de tubos polínicos y frutos maduros (Magallán, 2004), por lo que es posible suponer que L. scilloides presenta también hidrofilia. Las especies hidrófilas generalmente exhiben modificaciones estructurales en su polen, entre las que destacan el polen de forma globular que forma cadenas filiformes o granos muy alargados, probablemente para maximizar la eficiencia del transporte (Les, 1988). El polen de L. scilloides es elíptico, tripantocolpado, de 40 ?m en su eje ecuatorial y de 50 ?m en su eje polar y ornamentación reticulada (Magallán, 2004) el cual difiere del polen globular típico de las especies hidrófilas por lo que se requieren estudios ecológicos de la polinización más detallados en L. scilloides para explicar la discordancia entre el síndrome de polinización y el tipo de polen.
Al comparar la diversidad genética dentro de las poblaciones de L. scilloides con los registrados para especies acuáticas con hidrofilia tales como Ceratophyllum demersum (A= 1.22, P= 20.00, He= 0.064) (Les, 1991), Hydrilla verticillata (A= 1.00, P= 0.00, He= 0.000) (Hofstra et al., 2000), Najas marina (A= 1.05, P= 4.18, He= 0.015) (Triest, 1989), Vallisneria americana (A= 1.40, P= 38.50, He= 0.085) (Laushman, 1993) y Zostera marina (A= 1.30, P= 18.30, He= 0.063) (Laushman, 1993), se observa que L. scilloides presenta niveles de diversidad genética más altos.
Los resultados muestran que existe alta endogamia dentro de las poblaciones de L. scilloides (FIS= 0.288), lo cual es raro en especies anemófilas, por lo que es probable que exista autogamia en las flores hermafroditas de la espiga, dado que se observó que el androceo y el gineceo de dichas flores alcanzan su madurez al mismo tiempo. También es posible que el polen de las flores de las espigas llegue a través del agua a las flores femeninas sumergidas. En resumen, los diferentes modos de polinización que presenta L. scilloides junto con su modo de propagación vegetativa están influyendo de manera importante en la estructura y diversidad genética de la especie a nivel intra e interpoblacional.
El valor de FST (0.077) señala que existe poca diferenciación genética entre poblaciones lo cual contrasta con los patrones sugeridos para plantas acuáticas (Edwards y Sharitz, 2000; Chen et al., 2008; Wang et al., 2010). Asimismo, los valores de identidad genética entre las poblaciones locales de L. scilloides (I= 0.990) señalan que las poblaciones locales son muy parecidas genéticamente. La alta similitud genética puede deberse al alto flujo génico (Nm= 4.42) o a cuestiones históricas (polimorfismos ancestrales). Todos los humedales en los que L. scilloides fue colectada son ecosistemas aislados, algunos muy cercanos, como es el caso de la poblaciones 1 a 8, que como máxima distancia de separación tienen 6km. Por el contrario, otros humedales se ubican hasta 40km de distancia, como la población 12 que se ubica en el estado de Guanajuato (Fig. 1). La poca diferenciación genética entre poblaciones podría explicarse, por un lado, por el amplio alcance que tiene la dispersión de las diásporas, a través del agua y/o incluso por aves (Novelo y Lot, 2001); así mismo, la dispersión de los frutos que presentan estructuras aladas y están situados en las espigas, pueden ser llevados por el viento a largas distancias. Por otro lado, es posible que las poblaciones locales fueran fundadas por un pequeño número de individuos provenientes de una sola fuente, que después tuvieron una extensiva reproducción clonal y que históricamente no han sufrido procesos de colonización - extinción importantes. La reproducción clonal es muy común en plantas acuáticas llegándose a casos extremos de monoclonalidad en ciertas poblaciones estables en contraste con poblaciones policlonales que han estado sujetas a disturbios por actividades humanas (Lambertini et al., 2008).
Dado que L. scilloides habita ecosistemas aislados, se esperaba que la deriva génica fuera un proceso determinante en su estructura genética, sin embargo, esta fuerza evolutiva tiene como consecuencia alta diferenciación genética entre poblaciones (Hartl y Clark, 1997), que no se observa en las poblaciones estudiadas, por lo que es posible que no esté afectando la estructura poblacional de la especie. Es probable que la presencia de un banco de semillas esté eliminando los efectos de la deriva génica, debido a que los humedales temporales de estudio pasan por períodos de sequía - inundación, es necesario que las plantas tengan mecanismos que les permiten sobrevivir de un periodo a otro (banco de semillas). La persistencia del banco de semillas amortigua los "cuellos de botella" y permite que la diversidad genética alta se mantenga en las poblaciones (Edwards y Sharitz, 2000)
Los resultados obtenidos en este estudio no corroboran las hipótesis de alta diversidad y baja endogamia basadas en su distribución y polinización anemófila. Por el contrario, L. scilloides presenta baja diversidad genética y alta endogamia, debido a que no se habían tomado en cuenta variables como la polinización por autogamia, la posible hidrofilia y la extensa propagación clonal de la especie. Otro aspecto contrastante es que la mayoría de los estudios en plantas acuáticas muestran alta diferenciación poblacional (Barrett et al., 1993; Laushman, 1993; Edwards y Sharitz, 2000; Chen et al., 2008; Wang et al., 2010), mientras que L. scilloides presenta poblaciones muy parecidas genéticamente. Las características florales poco comunes de L. scilloides la hacen un objeto de estudio muy interesante desde el punto de vista evolutivo, por lo que es recomendable llevar a cabo estudios de su biología floral, mecanismos de dispersión y estudios de diversidad genética incluyendo más poblaciones, ya que esto contribuiría ampliamente a su entendimiento. Estudios en plantas acuáticas con heterostilia muestran que se puede mantener una alta diversidad y estructura genética en las poblaciones con distintas proporciones de morfos reproductivos, si se presentan mecanismos que garanticen la reproducción cruzada en poblaciones adyacentes (Shibayama y Kadono, 2007).
Finalmente, estudios con marcadores moleculares de ADN en plantas acuáticas pueden dilucidar las relaciones genealógicas e históricas entre los haplotipos distribuidos en poblaciones a diferentes escalas espaciales (Koga et al., 2008; Tan et al., 2008; Fehrmann et al., 2012), distinguir procesos de hibridación e introgresión (Ito et al., 2010; Yoshino et al., 2011) y robustecer los estudios taxonómicos y filogenéticos (Dkhar et al., 2010; Na et al., 2010; Telford et al., 2011). Son muy pocos los estudios de esta naturaleza realizados en plantas acuáticas mexicanas.
Los autores agradecen a D. Cabrera y a un revisor anónimo por sus valiosos comentarios y sugerencias; a P. Balderas por su ayuda en campo y a A. Beltrán por su ayuda en el laboratorio. A R. Luna-Reyes y N. Pérez-Naser por sus enseñanzas en cuanto a la técnica de electroforesis. C. Rojas elaboró el mapa de distribución. Esta investigación fue financiada por el Consejo Nacional de Ciencia y Tecnología (CONACYT 29104N) y por el Consejo de Ciencia y Tecnología de Querétaro (CONCYTEQ).