El hongo conocido popularmente como champiñón, Agaricus bisporus, es la especie comestible más cultivada comercialmente en el mundo. A pesar de su importancia y de que se ha incluido en listados de diferentes estados de México, su presencia de forma silvestre no se había corroborado. El objetivo de este trabajo fue determinar si esta especie crece de manera silvestre en México. Se realizaron recolectas en los estados de Tlaxcala y Veracruz, y se aislaron 7 cepas a partir de ejemplares silvestres. Se obtuvieron basidiomas de dichas cepas cultivadas en un sustrato a base de composta. Los ejemplares silvestres que se depositaron en el Herbario XAL, y los obtenidos en cultivo, se estudiaron macro y micromorfológicamente. La cepa IE 623 se utilizó para obtener la secuencia de la región ITS del ADN ribosomal. Los ejemplares revisados coinciden morfológicamente con A. bisporus y presentan en promedio 63.4% de basidios bispóricos. La secuencia de ADN corroboró la determinación taxonómica, y de acuerdo con los análisis de similitud y filogenéticos se concluyó que la cepa proviene de un ejemplar silvestre y no de una cepa comercial cultivada. Las cepas de A. bisporus silvestres en México podrían aportar germoplasma para mejorar su cultivo comercial.
The fungus commonly known as white button mushroom, Agaricus bisporus, is the most commercially cultivated species among edible ones worldwide. In spite of its importance, its presence in wild stage had not been confirmed even when it has been included in lists of species from different states of Mexico. The aim of this study was to determine if this species grows wild in Mexico. Collections were made in the states of Tlaxcala and Veracruz, where 7 strains were isolated from wild specimens. Basidiomes of these strains were grown in commercial compost-based substrate. Wild specimens, deposited at XAL Herbarium, and those obtained in culture were studied macro and micro morphologically. IE 623 strain was used to obtain the DNA ITS ribosomal sequence. The revised specimens match morphologically with A. bisporus and have an average of 63.4% of bisporic basidia. The DNA sequence confirmed the taxonomic identity and through similarity and phylogenetic analysis, it was concluded that the strain derived from a wild specimen and not from a commercially cultivated strain. Native strains of A. bisporus from Mexico could provide important germplasm to improve its commercial cultivation.
Las especies del género Agaricus L. son actualmente muy apreciadas, no solo por sus propiedades nutricionales, sino también por sus cualidades organolépticas y medicinales. El hongo conocido popularmente en México como champiñón, Agaricus bisporus (J.E. Lange) Imbach, es la especie más célebre del género y también la de mayor producción en el mundo entre las especies cultivadas (Arrillaga, 2004). Su producción mundial se estimó en más de 4 millones de toneladas en el año 2009 (Sonnenberg et al., 2011). Agaricus bisporus se diferencia de todas las especies del género por presentar 2 esterigmas en la mayoría de sus basidios (Parra, 2008), aunque con frecuencia se pueden observar basidios mono, tri e incluso tetraspóricos (Callac, Imbernon, Guinberteau, Desmerger y Theochari, 2003). A pesar de este carácter morfológico —2 esporas por basidio—, que hace inconfundible a la especie, existen 2 variedades silvestres, además de A. bisporus var. bisporus, que han mostrado ser principalmente tetraspóricas: A. bisporus var. burnettii Kerrigan y Callac con distribución en América del Norte y A. bisporus var. eurotetrasporus Callac y Guinb. presente en Europa (Callac, Billette, Imbernon y Kerrigan, 1993; Callac et al., 2003).
En México A. bisporus se ha citado en diversos trabajos como parte de inventarios en distintas entidades; sin embargo, en ninguno de ellos se ha proporcionado una descripción completa de la especie y en muy pocos se especificó el tipo de vegetación donde crecía. Se registró en el Distrito Federal, sin precisar el tipo de vegetación (Herrera y Guzmán, 1972) y de zonas urbanas, suburbanas y agrícolas (Zarco, 1986); en Jalisco (Guzmán-Dávalos, Nieves y Guzmán, 1983; Sánchez-Jácome y Guzmán-Dávalos, 2011); en Morelos en jardines y zonas perturbadas (López, Mora, Montiel y Guzmán, 1985). Agaricus bisporus var. albidus (J.E. Lange) Singer y A. bisporus var. bisporus se conocen en Michoacán (Conabio, 2005). Además, algunos trabajos han citado esta especie de cultivos comerciales en Baja California (Ayala y Guzmán, 1984), Veracruz (López-Ramírez, 2011). Guzmán (1977) citó A. bisporus var. albidus y A. bisporus var. bisporus cultivados comercialmente sin precisar la localidad. Por otra parte, Martínez-Carrera et al. (2001) citaron la especie en Tlaxcala, en el volcán La Malinche; sin embargo, dichos autores no mencionaron en qué herbario fue depositado el material estudiado y aislado, por lo que no se tiene un registro certero. Los objetivos del presente trabajo fueron: demostrar la presencia de esta especie de manera silvestre en México, corroborar la determinación de los materiales a través de la producción de basidiomas obtenidos a partir de las cepas aisladas y por medio de análisis filogenéticos, así como describir sus características morfológicas de valor taxonómico bajo condiciones experimentales de cultivo en compost utilizado para la producción comercial.
Materiales y métodosSe realizaron salidas de campo en los estados de Tlaxcala y Veracruz para recolectar ejemplares frescos. Se tomaron los datos de localidad, sustrato y hábitat de cada sitio de recolecta. Para el estudio macroscópico de los materiales se registraron los datos en fresco de píleo y estípite —forma, color y tamaño—, arreglo y color de las láminas, posición del anillo y el color del que se mancha el contexto. Además, se realizaron reacciones macroquímicas, Schäffer y KOH 5%, de acuerdo con Parra (2008). El estudio microscópico de los ejemplares se llevó a cabo bajo las técnicas rutinarias en micología (Largent, Johnson y Watling, 1977), con un microscopio estereoscópico (Carl Zeiss, Stemi DV4) y un microscopio compuesto (Carl Zeiss, Primo Star). Las fotografías se tomaron con una cámara digital (Sony Cyber-shot). Para la determinación de la especie se consultaron los trabajos de Arrillaga (2004); Cappelli (1984); Kerrigan (1986) y Parra (2008). De algunos ejemplares recolectados se aislaron cepas de manera vegetativa, de acuerdo con la metodología de Guzmán, Mata, Salmones, Soto-Velasco y Guzmán-Dávalos (2013) en medio de cultivo de glucosa y agar adicionado con extracto de compost o medio de cultivo de papa dextrosa agar (PDA, Bioxon) (Mata y Savoie, 2007).
Para la obtención de esporomas se preparó inóculo de 4 cepas, de acuerdo con la metodología de Mata y Savoie (2007), utilizando semillas de sorgo (Sorghum vulgare Pers.). Se prepararon muestras de 4kg de compost comercial, a las que se les agregó un 5% de inóculo, se mezcló uniformemente y se colocaron en bolsas de plástico. Después de 14 días de incubación a 25°C se añadió una capa de 5cm de tierra de cobertura para inducir la aparición de los esporomas, y las bolsas se colocaron en un cuarto a 18°C con humedad relativa de 90% y un fotoperiodo de 12/12h (Mata, Calderón-Fuentes y Savoie, 2012). Los ejemplares se cosecharon en diferentes estadios de desarrollo.
Todos los ejemplares estudiados, incluyendo los ejemplares provenientes de cultivo, están depositados en la colección micológica del Herbario XAL del Instituto de Ecología, A.C. En los ejemplares de las cepas cultivadas se determinó el porcentaje de basidios bispóricos en relación con los mono, tri y tetraspóricos, con un fragmento de una lámina madura colocado en un portaobjetos para su observación directa en el microscopio compuesto. Se determinó el número de esporas por basidio, en un total de 150 basidios por ejemplar, de acuerdo con la metodología de Imbernon, Callac, Gasqui, Kerrigan y Velcko (1996); Kerrigan y Ross (1987) y Callac et al. (2003) y el número promedio de esporas expresado como average spore number (ASN), de acuerdo con la fórmula establecida por Callac et al. (1996).
Análisis molecularLas secuencias de la región ITS —espaciador interno transcrito— del ADN ribosomal, que comprende el ITS1, 5.8S y el ITS2, se obtuvieron de la cepa IE 623 aislada del ejemplar Mata 641 (XAL). El ADN de la cepa se extrajo con el paquete XNAP (Sigma-Aldrich). Para esto se rasparon con una aguja de disección aproximadamente 1-2mm2 de micelio aéreo de la cepa cultivada en PDA. El micelio se colocó en un tubo Eppendorf® con 20μl de solución de extracción y se calentó por 10min a 65°C y 10min a 95°C en un termociclador (Bio-Rad, modelo T100), posteriormente se añadieron 20μl de solución de dilución y se incubó a temperatura ambiente por 30min (Garibay-Orijel, Morales-Marañón, Domínguez-Gutiérrez y Flores-García, 2013). La región ITS se amplificó con los cebadores ITS1F e ITS4 (Gardes y Bruns, 1993) bajo las condiciones descritas por Izzo, Agbowo y Bruns (2005). Los productos de PCR fueron revisados por medio de electroforesis en geles de agarosa al 1% en amortiguador TE. Los amplicones de buena calidad se limpiaron usando una mezcla de 1μl de ExoSAP-IT (USB-Affimetrix) con 1μl de agua por 3.5μl de producto de PCR. La reacción de secuenciación se realizó con Big Dye Terminator 3.1 (Applied Biosystems) según las instrucciones del fabricante. Se obtuvieron las secuencias de ambas direcciones en un secuenciador ABI 3100 (Applied Biosystems) en el Laboratorio de Secuenciación Genómica de la Biodiversidad y la Salud del Instituto de Biología, UNAM. Las secuencias se editaron y ensamblaron en Geneious Pro R7 (Biomatters).
Se construyó una matriz de secuencias con especies de Agaricus sección Bivelares utilizadas en estudios filogenéticos previos (Challen, Kerrigan y Callac, 2003; Geml, Geiser y Royse, 2004) y también se incluyeron aquellas secuencias de GenBank que resultaron similares a la secuencia de la región ITS de la cepa IE 623 mediante una búsqueda en Blast. Las secuencias fueron alineadas en Muscle (Edgar, 2004) y la matriz de datos se analizó mediante los métodos Neighbor-Joining, máxima verosimilitud e inferencia bayesiana en Geneious Pro R7 (Biomatters). Agaricus bitorquis (AF432899) se usó como grupo externo (Geml et al., 2004). Los árboles de Neighbor-Joining fueron construidos con el modelo de distancia genética Tamura-Nei remuestreando con bootstrap a través de 1,000 réplicas. Para los análisis de máxima verosimilitud se utilizó el programa PhyML (Guindon et al., 2010) buscando la topología de los árboles con SPRs y HKY85 como modelo de sustitución; se utilizó bootstrap con 1,000 réplicas para probar el soporte de las ramas. También se realizaron análisis Bayesianos en MrBayes (Huelsenbeck y Ronquist, 2001) usando el radio de variación Gamma, el modelo de sustitución GTR con 4 cadenas de Monte Carlo sobre 1,100,000 generaciones submuestreando cada 400 generaciones y un valor de temperatura de 0.2. Los mejores modelos de sustitución se eligieron con MrModelTest versión 2 (Nylander, 2004). Se almacenaron los valores de largo de las ramas y se calcularon las probabilidades posteriores. También se comparó la secuencia de la cepa IE 623 con 63 secuencias de la misma especie disponibles en GenBank. Las secuencias se alinearon y se determinaron las posiciones que presentaron heteroalelismos en la región ITS.
DescripciónAnálisis morfológicoSe revisaron 10 ejemplares silvestres, de los cuales se aislaron un total de 7 cepas que se depositaron en el Cepario de Hongos del Instituto de Ecología con las claves: IE 623, IE 673, IE 674, IE 708, IE 746, IE 790 e IE 802. Se revisaron también los ejemplares obtenidos de las muestras sembradas en compost de las cepas IE 623, IE 746 e IE 802. Todos los materiales revisados coinciden con la descripción morfológica de A. bisporus var. bisporus que se presenta a continuación.
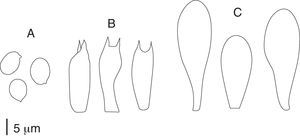
Agaricus bisporus var. bisporus (J.E. Lange) Imbach, Mitt. naturf. Ges. Luzern 15: 15, 1946 (figs. 1 y 2).
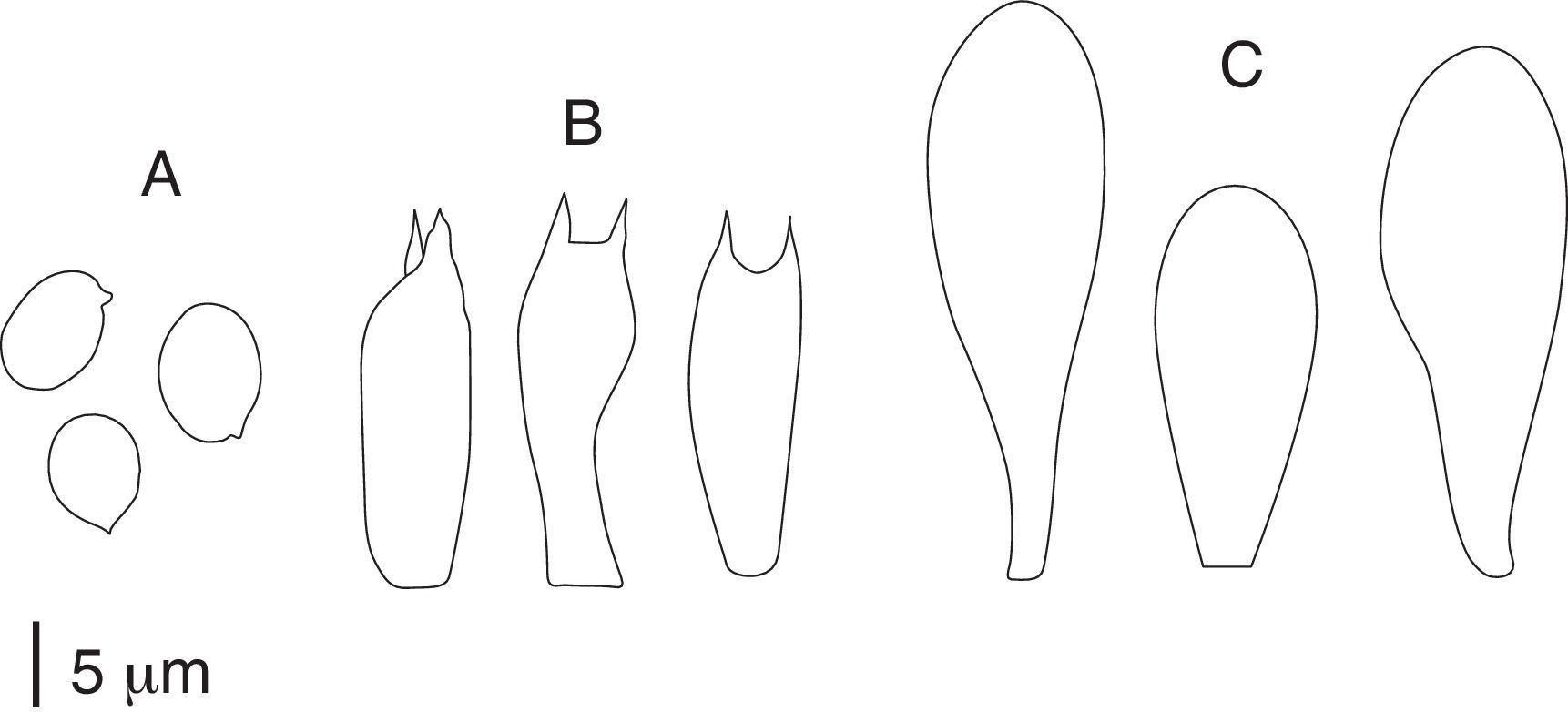
Píleo de 50-90mm de diámetro, globoso en etapas juveniles, convexo a plano-convexo y finalmente plano, blanquecino en etapas juveniles, beige, ocráceo a finalmente de color café claro, con el centro café oscuro, con fibrillas separadas hacia el margen de color café. Láminas libres, muy juntas, anchas, con lamélulas intercaladas, blanquecinas cuando son jóvenes, que cambian a color rosado y finalmente a café oscuro, casi negras, con tonos vináceos al madurar; arista blanquecina y ligeramente fimbriada. Contexto grueso, blanquecino que se mancha de color rojizo-vináceo al maltrato. Estípite de 50-55×12-17mm de cilíndrico a claviforme, a veces ligeramente bulboso en la base, hueco, liso, blanquecino, la parte interna se mancha de color rojizo-vináceo al maltrato, con frecuencia presenta cordones miceliales, los cuales pueden tener aspecto de rizomorfos cortos. Anillo ínfero o intermedio —en pocas ocasiones súpero—, membranoso, grueso, blanco. Basidiosporas de (6-) 7-8 (-9)×5.5-7 (-9)μm, ampliamente elípticas, de color café, con pared gruesa de 0.6-0.8μm de grosor. Basidios de 18-26 (-30)×6-8μm, clavados, la mayoría bispóricos —pocos trispóricos o tetraspóricos—, hialinos, de pared delgada. Queilocistidios de 15-35 (-45)×10-12 (-14)μm, clavados, hialinos, de pared delgada, abundantes. Reacciones macroquímicas: Schäffer y KOH negativas. Basidioma con olor débil, fúngico, agradable; sabor dulce típicamente fúngico, agradable.
Resumen taxonómicoHábitat. Terrícola, gregario, bajo árboles del género Cupressus en terrenos con cultivo de papa o en pastizales.
Material estudiado. Tlaxcala. Municipio de Cuapiaxtla, rancho El Tejocote a 2km de Cuapiaxtla, julio 19, 2001, Mata 641 (XAL), cepa aislada IE 623; octubre 3, 2002, Gándara 371-A, 376 (XAL); octubre 3, 2002, Ramírez-Guillén 174 (XAL), cepa aislada IE 673, 176 (XAL), cepa aislada IE 674, 178 (XAL); octubre 10, 2003, Mata 674 (XAL), cepa aislada IE 708; septiembre 18, 2006, Mata 719 (XAL), cepa aislada IE 746; agosto 29, 2007, Mata 755 (XAL), cepa aislada IE 790. Veracruz. Municipio de Coatepec, Unidad Deportiva Lic. Roberto Amorós Guiot, mayo 30, 2008, Mata 785 (XAL), cepa aislada IE 802. Ejemplares obtenidos en cultivo: Planta de cultivo de hongos del Instituto de Ecología, A.C., Xalapa, Veracruz, junio 28, 2002, Mata 647 (de la cepa IE 623, XAL); marzo 20, 2002, Mata 724 (de la cepa IE 746, XAL); junio 14, 2011, Mata 869 (de la cepa IE 802, XAL).
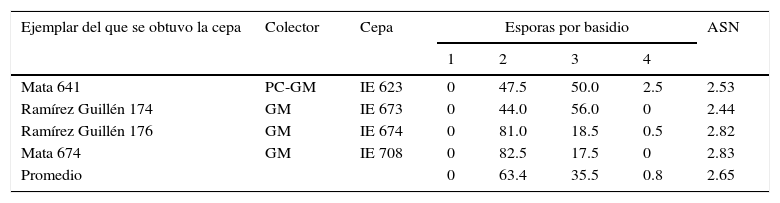
La tabla 1 muestra la proporción de basidios bispóricos respecto a los otros tipos de basidios, encontrada en los ejemplares analizados. Los esporomas de las cepas estudiadas mostraron variaciones entre 44% y 82.5% (promedio=64.3%) de basidios bispóricos típicos de la especie, de 18.5% a 56% (promedio=35.5%) de trispóricos y de 0.5-2.5% (promedio=0.8%) de tetraspóricos. Se obtuvo un ASN promedio de 2.65.
Variación en el número de esporas por basidio (%) y en el número promedio de esporas en diferentes cepas mexicanas silvestres de Agaricus bisporus.
| Ejemplar del que se obtuvo la cepa | Colector | Cepa | Esporas por basidio | ASN | |||
|---|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | ||||
| Mata 641 | PC-GM | IE 623 | 0 | 47.5 | 50.0 | 2.5 | 2.53 |
| Ramírez Guillén 174 | GM | IE 673 | 0 | 44.0 | 56.0 | 0 | 2.44 |
| Ramírez Guillén 176 | GM | IE 674 | 0 | 81.0 | 18.5 | 0.5 | 2.82 |
| Mata 674 | GM | IE 708 | 0 | 82.5 | 17.5 | 0 | 2.83 |
| Promedio | 0 | 63.4 | 35.5 | 0.8 | 2.65 | ||
ASN: número promedio de esporas por basidio (Average Spore Number); GM: Gerardo Mata; PC-GM: Philippe Callac-Gerardo Mata.
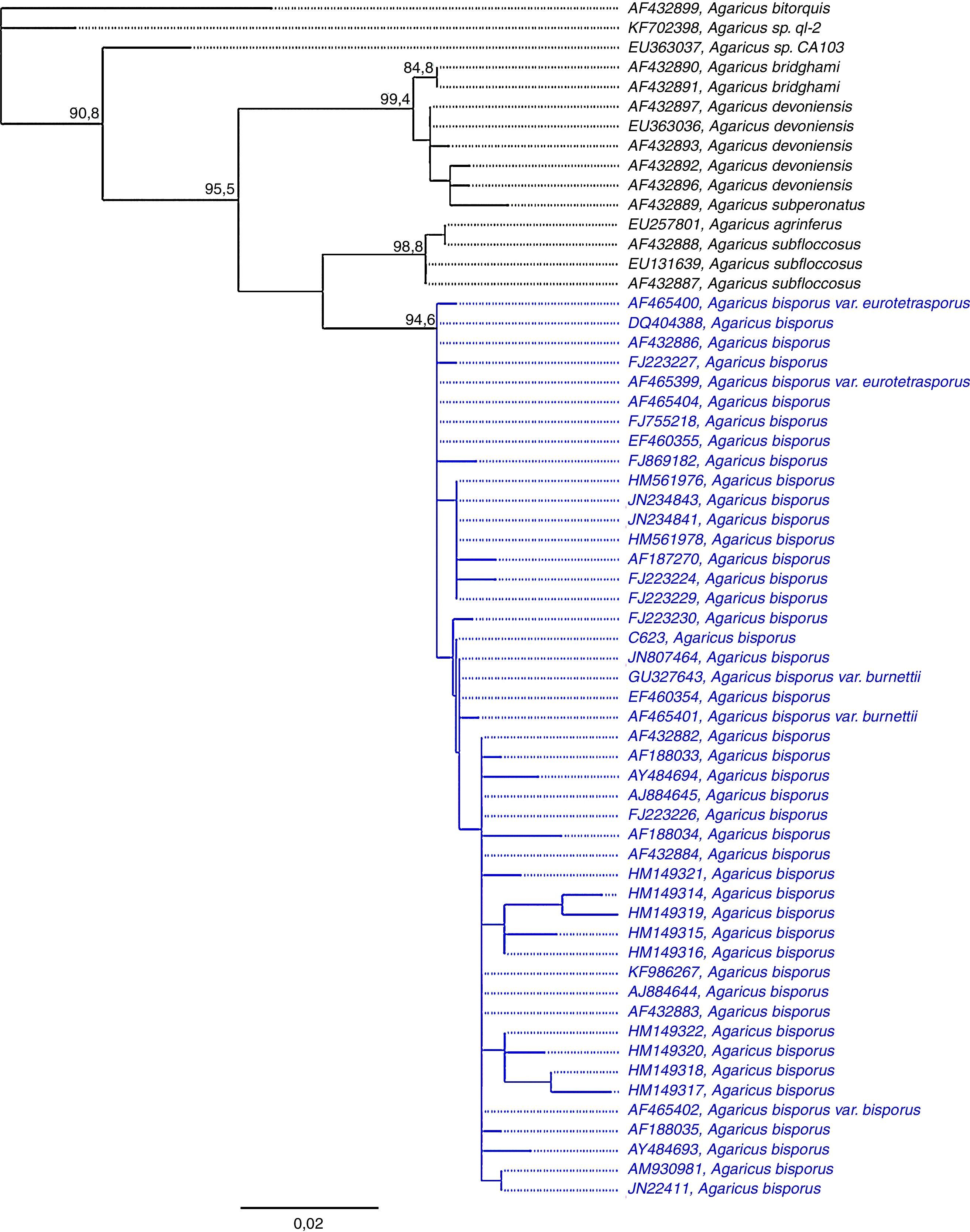
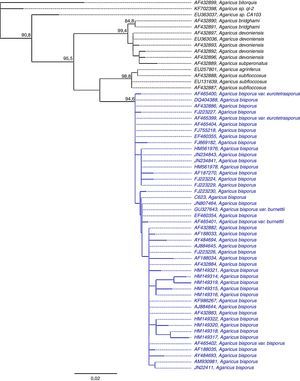
Se obtuvieron 4 secuencias de ADN en ambos sentidos de la cepa IE 623 de diferentes resiembras y medios de cultivo con entre 620 y 710 pares de bases. Entre ellas existió una similitud nucleotídica de 99.99% y su secuencia consenso de 712 pares de bases quedó registrada en GenBank bajo el número de acceso KM677957. Todos los análisis dieron resultados similares y se recuperó para Agaricus sección Bivelares, una estructura similar a la registrada por Challen et al. (2003). En todos los casos, el clado de A. bisporus tuvo un soporte alto —bootstrap de Neighbor Joining 99.4%, bootstrap de máxima verosimilitud 94.6%, probabilidades posteriores bayesianas 1—. En los 3 análisis la cepa IE 623 se ubicó dentro del clado de A. bisporus. La figura 3 muestra los resultados del análisis de máxima verosimilitud. Si bien dentro de A. bisporus hay poca estructura y los clados que se forman tienen poco soporte, la secuencia de la cepa IE 623 tiene una similitud genética mayor a cepas silvestres que a variedades cultivadas.
Árbol obtenido de un análisis de máxima verosimilitud de Agaricus sección Bivelares basado en secuencias de la región ITS del ADN ribosomal. Se muestran solo los valores de soporte de las ramas con un bootstrap mayor a 80.0%; en azul se indica el clado de A. bisporus y en azul y negritas la secuencia de la cepa IE 623.
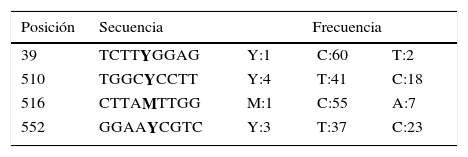
Se encontraron 4 posiciones —39, 510, 516, 552—, en las que se presentaron heteroalelismos en A. bisporus var. bisporus. La cepa IE 623 mostró C, Y, C y C en estas posiciones, respectivamente, y solo una de las secuencias de GenBank (JF223230) tiene la misma secuencia, pero esta última difiere, además, por una A en la posición 522. La cepa IE 623 tiene, por lo tanto, una secuencia única. En las 4 posiciones de importancia heteroalélica el genotipo «CYCC» de A. bisporus var. bisporus indica que uno de sus núcleos tiene un genotipo «CCCC» que aparece con mayor frecuencia (8/58; 13% de las secuencias), mientras que el otro núcleo que es «CTCC» es menos frecuente (2/58; 3%) (tabla 2).
Número de casos que comparten el orden en las 4 posiciones heteroalélicas (39, 510, 516, 552) de los genotipos encontrados en las 63 secuencias de Agaricus bisporus de las variedades bisporus, burnettii y eurotetrasporus, obtenidas de GenBank.
| Posición heteroalélica | Agaricus bisporus | ||
|---|---|---|---|
| 39, 510, 516, 552 | var. bisporus | var. burnettii | var. eurotetrasporus |
| CTCT | 35 | ||
| CTCC | 2 | 2 | |
| CCCC | 8 | 3 | |
| CCAC | 7 | ||
| TTCT | 2 | ||
| YYCY | 1 | ||
| CYMY | 1 | ||
| CYCY | 1 | ||
| CYCC | 1 | ||
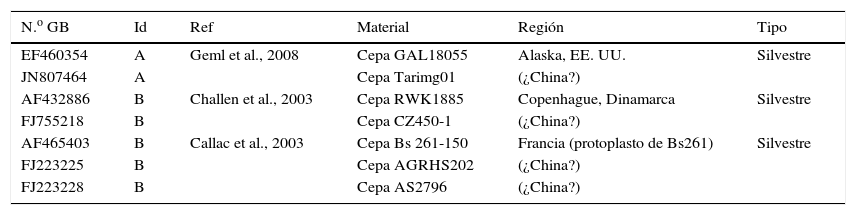
En la posición heteroalélica 510 de la región ITS las 63 secuencias de GenBank son C (18), T (41), o C/T=Y (4) (tabla 3). Sin embargo, estas 4 últimas secuencias difieren en otras posiciones de la cepa IE 623, lo que corrobora que no existe ninguna secuencia idéntica a la secuencia de dicha cepa. Por lo tanto, entre los 2 núcleos de la cepa heterocariótica IE 623 un núcleo que se podría denominar A contiene el nucleótido C, mientras que el otro núcleo llamado B contiene el nucleótido T. Se compararon las secuencias de estos 2 núcleos, A y B, con las 18 y 41 secuencias de GenBank, respectivamente conteniendo C o T en la posición 510. Entre ellas se encontró que 2 secuencias eran idénticas al núcleo A y 5 al núcleo B (tabla 4). Entre las 2 secuencias de tipo A se observó que una corresponde a una cepa silvestre proveniente de Alaska (EF460354; Geml, Laursen y Taylor, 2008); mientras que entre las 5 de tipo B, una es un homocarión aislado por el método de protoplastos a partir de una cepa silvestre procedente de Francia (AF465403; Callac et al., 2003) y otra es una cepa silvestre de Dinamarca (AF432886; Challen et al., 2003); de las 4 secuencias restantes de GenBank no existe información en la base o publicada sobre su procedencia.
Frecuencia heteroalélica en las 4 posiciones relevantes de los genotipos encontrados en las 63 secuencias de Agaricus bisporus obtenidas de GenBank.
| Posición | Secuencia | Frecuencia | ||
|---|---|---|---|---|
| 39 | TCTTYGGAG | Y:1 | C:60 | T:2 |
| 510 | TGGCYCCTT | Y:4 | T:41 | C:18 |
| 516 | CTTAMTTGG | M:1 | C:55 | A:7 |
| 552 | GGAAYCGTC | Y:3 | T:37 | C:23 |
Las letras en negrita indican los nucleótidos con variación heteroalélica.
Secuencias idénticas a los 2 tipos de núcleo de la cepa heterocariótica IE 623, encontrados en 63 secuencias depositadas en GenBank.
| N.o GB | Id | Ref | Material | Región | Tipo |
|---|---|---|---|---|---|
| EF460354 | A | Geml et al., 2008 | Cepa GAL18055 | Alaska, EE. UU. | Silvestre |
| JN807464 | A | Cepa Tarimg01 | (¿China?) | ||
| AF432886 | B | Challen et al., 2003 | Cepa RWK1885 | Copenhague, Dinamarca | Silvestre |
| FJ755218 | B | Cepa CZ450-1 | (¿China?) | ||
| AF465403 | B | Callac et al., 2003 | Cepa Bs 261-150 | Francia (protoplasto de Bs261) | Silvestre |
| FJ223225 | B | Cepa AGRHS202 | (¿China?) | ||
| FJ223228 | B | Cepa AS2796 | (¿China?) |
Id: identidad con el tipo de núcleo; N.o GB: número de acceso de GenBank.
El material estudiado coincide con Agaricus bisporus var. bisporus (Arrillaga, 2004; Cappelli, 1984; Parra, 2008), ya que la presencia de basidios bispóricos es un carácter distintivo, así como la medida de las basidiosporas mayores a 6μm de longitud y queilocistidios no mayores en promedio a 35μm de longitud (Callac et al., 1993; Cappelli, 1984; Parra, 2008). Kerrigan (1986) mencionó que las reacciones macroquímicas de Schäffer y KOH son negativas en A. bisporus, y que su característica principal es la presencia predominante de basidios bispóricos. Se ha clasificado en la sección Bitorques (Kuhn. y Rom. ex Hein.) Bon et Cappelli grupo Bisporus Cappelli (Arrillaga, 2004; Cappelli, 1984) y en la sección Bivelares (Kauffman) L.A. Parra subsección Hortenses (Kauffman) L.A. Parra (Kerrigan, Callac, & Parra, 2008; Parra, 2008). Arrillaga (2004) mencionó medidas más grandes del diámetro del píleo, hasta 100mm con escamas marrón pardusco, que no se observaron en los ejemplares estudiados. La proporción de basidios bispóricos y tetraspóricos se ha documentado en otros trabajos para A. bisporus var. bisporus; Martínez-Carrera et al. (2001) registraron 67.5% de basidios bispóricos y 27.5% de tetraspóricos, mientras que Callac et al. (2003) encontraron 80% y 1.27% de bi y tetraspóricos, respectivamente. Si bien la mayoría de las cepas han mostrado altos porcentajes de basidios bispóricos, en algunos casos este porcentaje ha variado de 17% a 55% en cepas silvestres (Callac et al., 1993, 2003). De acuerdo con los resultados obtenidos por Kerrigan, Billette, Callac y Velcko (1996) y Callac et al. (2003) tanto A. bisporus var. burnettii Kerrigan y Callac como la var. eurotetrasporus Callac y Ginb. poseen un porcentaje alto de basidios tetraspóricos, no así la var. bisporus, que presenta más bispóricos que tetraspóricos. En este trabajo se obtuvieron resultados similares a los registradas por Kerrigan et al. (1996) y Callac et al. (2003), aunque con menor porcentaje de basidios tetraspóricos. El ASN de los ejemplares obtenidos a partir de cepas cultivadas (promedio=2.65) se corresponde bien con los ejemplares considerados como bispóricos por Callac et al. (1996) (Callac, Theochari y Kerrigan, 2002), además, la cepa IE 623 presentó un ASN de 2.53 (tabla 1). El número de esporas por basidio es una característica determinada principalmente por un locus genético simple llamado BSN, el cual está ligado al locus de apareamiento MAT y a otros loci del cromosoma I (Imbernon et al., 1996). Agaricus y otros géneros cercanos de la tribu Agariceae Pat. son considerados ancestralmente tetraspóricos, es decir, que producen basidios predominantemente con 4 esterigmas (Kerrigan y Ross, 1987), lo que podría significar que el carácter bispórico de esta especie es resultado de un proceso de una adaptación evolutiva (Callac et al., 2003).
Las cepas comerciales provienen de un número limitado de genotipos tradicionales (Royse y May, 1982), y la mayoría son idénticas o muy similares a los primeros híbridos Horst U1® y Horst U3® obtenidos en la década de 1980 al cruzar las variedades white y off white (Fritsche, 1983). En contraste con la uniformidad encontrada entre las cepas cultivadas, las silvestres muestran una diversidad genética mayor (Foulogne-Oriol, Spataro y Savoie, 2009). La secuencia de la región ITS del ADN ribosomal de la cepa IE 623 apoyó la determinación taxonómica del ejemplar Mata 641 —del cual se obtuvo la cepa—. Los 3 análisis ubicaron esta secuencia dentro del clado de A. bisporus con alto nivel de soporte. El análisis de similitud nucleotídica (tabla 4) demuestra que la cepa IE 623 es genéticamente muy similar a cepas silvestres de diferentes países del Hemisferio Norte, como Estados Unidos de América, Dinamarca y Francia —de los ejemplares recolectados en China no se tiene certeza de que sean silvestres—. De hecho, las secuencias de las cepas comerciales más cercanas (HM149321, HM149322, FJ869182) son considerablemente diferentes (0.5%). Esto demuestra que al menos la cepa IE 623 es silvestre y se desarrolla de manera natural en el estado de Tlaxcala, y que no se trata de una cepa comercial cultivada que provenga de los cultivos de champiñones. La secuencia de la cepa IE 623 es típica de A. bisporus; sin embargo, representa una disposición única de los alelos conocidos en esta especie hasta ahora.
De acuerdo con Parra (2008)A. bisporus crece en Europa, principalmente en lugares donde se practica la ganadería, con frecuencia bajo árboles de la familia Cupressaceae, raras veces bajo otros árboles y en dunas; los materiales recolectados en Tlaxcala también se encontraron bajo árboles del género Cupressus, no así los materiales de Veracruz. Si bien el trabajo de Martínez-Carrera et al. (2001) citó esta especie de Tlaxcala, no mencionó en qué herbario se depositó el material estudiado, por lo que este trabajo corrobora la presencia de A. bisporus en Tlaxcala y lo registra por primera vez de Veracruz, en ambos casos creciendo de manera silvestre. Aunque el número de ejemplares recolectado fue bajo, los resultados sugieren que la especie podría tener una distribución amplia en México. A escala mundial A. bisporus se ha registrado de forma silvestre en América del Norte (Estados Unidos de América y Canadá), América del Sur (Argentina), Europa (Dinamarca, España, Francia, Grecia) y Asia (Israel, China) (Albertó, 1996; Callac et al., 2002; Challen et al., 2003; Geml et al., 2008; Niveiro y Albertó, 2013; Parra, 2008; Wang et al., 2008). El germoplasma encontrado en México puede ser una fuente importante de características genéticas a partir de las cuales se podría realizar un programa de trabajo encaminado a la obtención de cepas mejoradas capaces de desarrollarse bajo las condiciones ambientales locales. La capacidad de resistencia a plagas y enfermedades, el crecimiento adecuado en materiales regionales, así como la habilidad para fructificar a temperaturas superiores a 25°C son algunas de las cualidades deseables en las cepas de champiñón (Largeteau, Callac, Navarro-Rodríguez y Savoie, 2011; Navarro y Savoie, 2014; Salmones, Ballesteros-Hernández, Zulueta y Mata, 2012). El reto será lograr que el germoplasma mexicano se incorpore en corto tiempo a la producción comercial.
Este trabajo fue financiado en el marco de la cooperación bilateral México-Francia, a través del proyecto Agasub 115790 Conacyt-ANR. La secuenciación de ADN fue financiada por la red MEXBOL a través del proyecto Conacyt 1251085. Se agradece al técnico Ranulfo Castillo-del Moral, del INIFOR-UV, su asistencia en el trabajo de gabinete y elaboración de figuras. Al Dr. Gastón Guzmán y al técnico Juan Lara-Carmona, de la colección de hongos del Herbario XAL del INECOL, por su ayuda en la consulta y búsqueda de materiales. Al ingeniero Juan Carlos Sucarrats, de la empresa Riojal, la donación de compost para el cultivo de las cepas aisladas. Se agradece al M. en C. Rodolfo Ángeles-Argaiz, del Instituto de Biología de la UNAM, su apoyo en la obtención de las secuencias de ADN.
La revisión por pares es responsabilidad de la Universidad Nacional Autónoma de México.