Los métodos de laboratorio utilizados para el diagnóstico de blefaritis, conjuntivitis y queratitis incluyen desde exámenes en fresco para búsqueda de parásitos, tinciones hasta cultivos microbiológicos que pueden durar hasta 2 semanas para obtener el resultado.
ObjetivoImplementar una PCR para la detección de hongos y bacterias grampositivas y gramnegativas utilizando un método reportado previamente y adaptado a las condiciones del laboratorio de biología molecular del Centro de Investigación Biomédica de La Fundación Hospital Nuestra Señora de la Luz I.A.P. (CIB-HOL) utilizando el termociclador Axygen/Maxygene.
MétodosSe realizó una extracción de ADN de cultivos microbiológicos de bacterias grampositivas, gramnegativas y hongos. Se hicieron diluciones seriadas para determinar la sensibilidad del método. Se realizó PCR para hongos y bacterias. Se buscaron las condiciones en común para realizar la PCR en un solo paso.
ResultadosUtilizando las diluciones del ADN se obtuvo producto de amplificación utilizando una cantidad de 0.12ng de ADN bacteriano y de 0.15ng de ADN fúngico.
ConclusionesLa técnica de la PCR es una herramienta muy importante para la detección de ADN de bacterias y hongos causantes de infecciones oculares. Permite amplificar el material genético del agente etiológico y facilita su observación, obteniéndose los resultados en menor tiempo que con las técnicas convencionales.
Laboratory methods used for the diagnosis of blepharitis, conjunctivitis and keratitis ranging from fresh examinations to search for parasites, gram stain and microbiological cultures, can last up to two weeks to get the result.
ObjectiveTo implement a PCR for the detection of fungi and gram-positive and gram-negative bacteria using a previously reported method adapted to the molecular biology lab at Biomedical Research Center, Fundación Hospital Nuestra Señora de la Luz I.A.P. (CIB-HOL), using the thermocycler Axygen/Maxygene.
MethodsDNA from microbial cultures of Gram positive, Gram negative and fungi were obtained for the PCR. Serial dilutions were performed to determine the sensitivity of the method. PCR was performed for fungi and bacteria. We searched for common conditions for PCR in one step
ResultsWhen we used dilutions of DNA, the PCR was sensitive to 0.12ng to DNA bacterial and 0.15ng to DNA fungal.
ConclusionsThe PCR technique is a very important molecular biology tool to detect DNA from bacteria and fungi responsible of the ocular infections. This method allows amplifying the genetic material of the etiologic agent and the results are obtained in less time than using conventional techniques.
El ojo es el órgano responsable de la captación de la luz, que es el proceso inicial de la visión, y por su posición anatómica está constantemente expuesto a una variedad de patógenos. Sin embargo, cuenta con un sistema de producción de sustancias, entre ellas la lágrima que contiene una gran cantidad de compuestos químicos, como citocinas, enzimas, lípidos y electrólitos capaces de formar una barrera inmunológica para detener cualquier microorganismo que pudiera generar una infección1. El origen de una infección ocular puede ser producto de un trauma, cirugía, uso de lentes de contacto y otras enfermedades en las cuales el sistema inmunológico se encuentra comprometido, facilitando el desarrollo de los agentes causales. Las infecciones oculares son producidas por bacterias, hongos, virus o parásitos. Existen muchos tipos de infecciones oculares, y entre las más comunes podemos mencionar la conjuntivitis, la queratitis y la blefaritis o inflamación crónica de los párpados, mayoritariamente producida por estafilococos2–4.
Las infecciones oculares son una causa importante de morbilidad ocular en todo el mundo, con potencial riesgo de pérdida de la visión e incluso de la integridad ocular. La severidad depende tanto de las condiciones subyacentes del paciente como de la patogenicidad del agente infeccioso3,5. La conjuntivitis bacteriana, por ejemplo, se caracteriza por hiperemia conjuntival de inicio unilateral rápido acompañado de edema palpebral y secreción mucopurulenta. La infección a menudo se vuelve bilateral a los 2 días del inicio del padecimiento. Su incidencia es difícil de determinar debido a que la mayor parte de los pacientes no acuden a la ayuda profesional y se tratan empíricamente, lo que en ocasiones puede agravar la dolencia2,5. Otro padecimiento importante que pone en riesgo la salud visual es la queratitis asociada al uso de lentes de contacto. Existen reportes en los que se señala que la incidencia es de 30 a 60 por 10,000 ojos al año, o 1:200 a 1:500 usuarios por año5. Las bacterias que con mayor frecuencia producen queratitis asociada al uso de lentes de contacto son los bacilos gramnegativos, principalmente Pseudomonas aeruginosa (P. aeruginosa) y Serratia marcescens, aunque también puede estar involucrado Proteus vulgaris o Proteus mirabillis, así como bacterias grampositivas, entre ellas Staphylococcus aureus y Staphylococcus epidermidis (S. epidermidis), incluso levaduras del género Candida y amebas del género Acanthamoeba y en menor frecuencia los hongos filamentosos. La incidencia de queratitis en usuarios de lentes de contacto es muy baja, pero ocasionalmente los microorganismos pueden ser hallados en las soluciones y/o en los mismos lentes5–9.
Las cirugías oculares que involucran a la córnea tienen un cierto riesgo de infección por microorganismos que normalmente habitan en la superficie ocular. Se ha reportado que los microorganismos aislados con mayor frecuencia son bacterias grampositivas ocupando el primer lugar en frecuencia S. epidermidis, que proviene de la flora habitual de la superficie ocular5,10,11.
Existen otras enfermedades que están relacionadas con el riesgo de infección ocular. Entre las enfermedades que involucran la alteración del epitelio corneal podemos mencionar las enfermedades autoinmunes como la orbitopatía tiroidea, el síndrome de Sjögren y el penfigoide, entre otras. Las enfermedades sistémicas como la diabetes mellitus, las colagenopatías, el síndrome metabólico o los padecimientos que involucran un sistema de inmunosupresión también predisponen al desarrollo de queratitis infecciosa debido a la disminución de la defensa humoral y/o celular5,12–14.
En general, los microorganismos que han sido implicados como agentes causales de conjuntivitis, blefaritis y queratitis infecciosa son de origen bacteriano, predominando los grampositivos5. Los hongos representan alrededor del 5-30% de los casos, y con respecto a los parásitos, los porcentajes reportados son menores y se les atribuye un 1-15%. El parásito más común que provoca blefaritis es Demodex folliculorum mientras que la queratitis parasitaria está asociada con Acanthamoeba sp5,15,16.
Los métodos de laboratorio utilizados para el diagnóstico de blefaritis, conjuntivitis y queratitis incluyen desde exámenes en fresco para búsqueda de parásitos y tinciones hasta cultivos microbiológicos que pueden durar hasta 2 semanas para obtener el resultado17,18. La presencia de microorganismos puede estar en cantidades muy pequeñas y esto depende de la presentación clínica de la infección; por ejemplo, se pueden presentar desde abscesos corneales francos hasta formas más indolentes como la queratopatía cristalina en la que se puede encontrar un infiltrado intraestromal pero con una mínima respuesta inflamatoria y muestra insuficiente para realizar la microbiología clásica19–23. Debido a esto, se han desarrollado métodos de biología molecular para el diagnóstico microbiológico24–26. Se ha utilizado la reacción en cadena de la polimerasa (PCR) directa para virus, bacterias, hongos y parásitos; PCR anidada para identificación de bacterias; y PCR en tiempo real para todo tipo de microorganismos27–31.
En el año 2000 Jaegger et al. y Carrol et al. describieron un método por PCR, para la detección de hongos y bacterias respectivamente, en muestras de fluidos oculares32,33. Estos trabajos reportan una alta sensibilidad del método, algo muy importante en el diagnóstico de microbiología ocular debido a la escasa cantidad de material biológico que se puede obtener en una toma de muestra oftálmica, sin embargo, es necesario que cada laboratorio estandarice las condiciones de trabajo para la obtención de resultados satisfactorios. El objetivo del presente trabajo fue reproducir estas técnicas para la detección de hongos y bacterias obteniendo las condiciones adecuadas para hacerlo en un solo programa utilizando las mismas condiciones para la detección tanto de bacterias como de hongos; esto acortaría el tiempo de obtención de resultados que beneficiaría al paciente para obtener un resultado rápido y confiable. El trabajo se realizó en el laboratorio de biología molecular del Centro de Investigación Biomédica de La Fundación Hospital Nuestra Señora de la Luz I.A.P. (CIB-HOL).
Material y métodosSe obtuvieron muestras de cultivos microbiológicos de bacterias grampositivas (S. epidermidis), gramnegativas (P. aeruginosa) y hongos (Candida sp.) caracterizados previamente por observación microscópica, macroscópica y utilizando el sistema automatizado VITEK 2C (Laboratorio BioMerieux, Francia). Se realizó extracción de ADN utilizando un kit comercial (Puregene, Qiagen. Maryland, EE. UU.). La cuantificación del material genético se obtuvo por espectrofotometría utilizando el equipo NanoDrop 2000, Thermo Scientific. La PCR para hongos se llevó a cabo usando los oligonucleótidos 5′-CAGGGGAGGTAGTGACAATAA-3′ y 5′-ACAAATCACTCCACCAACTAAG-3′ utilizando una temperatura de alineamiento (Tm) de 59°C; para la PCR de bacterias se utilizaron los oligonucleótidos 5′-TTGGAGAGTTTGATCCTGGCTC-3′ y 5′-ACGTCATCCCCACCTTCCTC-3′ con una Tm de 56°C. Para la PCR anidada se emplearon los oligonucleótidos 5′-GGCGGCAKGCCTAAYACATGC-3′, 5′-GACGACAGCCATGCASCACCTG 3′ y 5′-GCGRCTCTCTGGTCTGA-3′ con Tm de 61.4°C. El programa de PCR en el termociclador Axygen/Maxygene para hongos y bacterias fue: 95°C/15min, después 35 ciclos con las temperaturas 94°C/1min Tm/1min, 72°C/1min; finalmente una temperatura de elongación de 72°C/10min. Se realizó una PCR en gradiente de temperatura para obtener una Tm en común, de tal forma que fuera posible obtener en un solo programa las condiciones necesarias para la detección de hongos y bacterias, optimizando el método y acortando el tiempo de obtención de los resultados. Se realizaron diluciones seriadas 1:10, 1:100, 1:1000 y 1:10000 para determinar la sensibilidad del método.
Para la PCR anidada se utilizaron los productos de amplificación obtenidos de la PCR para bacterias; las condiciones fueron: 95°C/10min, después 32 ciclos con las temperaturas 94°C/30s, 61.4/1min, 72°C/30s y finalmente una temperatura de elongación de 72°C/3min. Todos los productos de PCR se identificaron mediante electroforesis en gel de agarosa al 1.5% utilizando las condiciones de 90V durante 40min y Gelred 10X para visualizarlo en un transiluminador UVP BioLite, con luz UV a 302nm.
ResultadosDe la suspensión bacteriana, la concentración de ADN obtenido fue de 4ng/μl, mientras que para el ADN fúngico fue de 5ng/μl; para la PCR se utilizaron 3μl (12ng) de ADN bacteriano y 3μl (15ng) de ADN fúngico. Se obtuvieron resultados satisfactorios utilizando la Tm reportada para cada PCR. El producto de PCR para bacterias fue de aproximadamente 1,300pb, y el programa en el termociclador tuvo una duración de 3h y 20min (fig. 1). Para la PCR de hongos se obtuvo un producto de amplificación de aproximadamente 700pb y el tiempo del programa de corrida de la PCR en el termociclador fue de 3h y 20min (fig. 2). En la PCR en gradiente de temperatura, para realizar la PCR de hongos y bacterias en una sola ejecución, las mejores condiciones fueron: 95°C/10min, 35 ciclos con las temperaturas 94°C/1min 56°C/1min, 72°C/1min y finalmente una temperatura de elongación de 72°C/7min con una duración de ejecución de 3h y 10min. Con la Tm de 56°C se pudo amplificar tanto el ADN fúngico como el bacteriano.
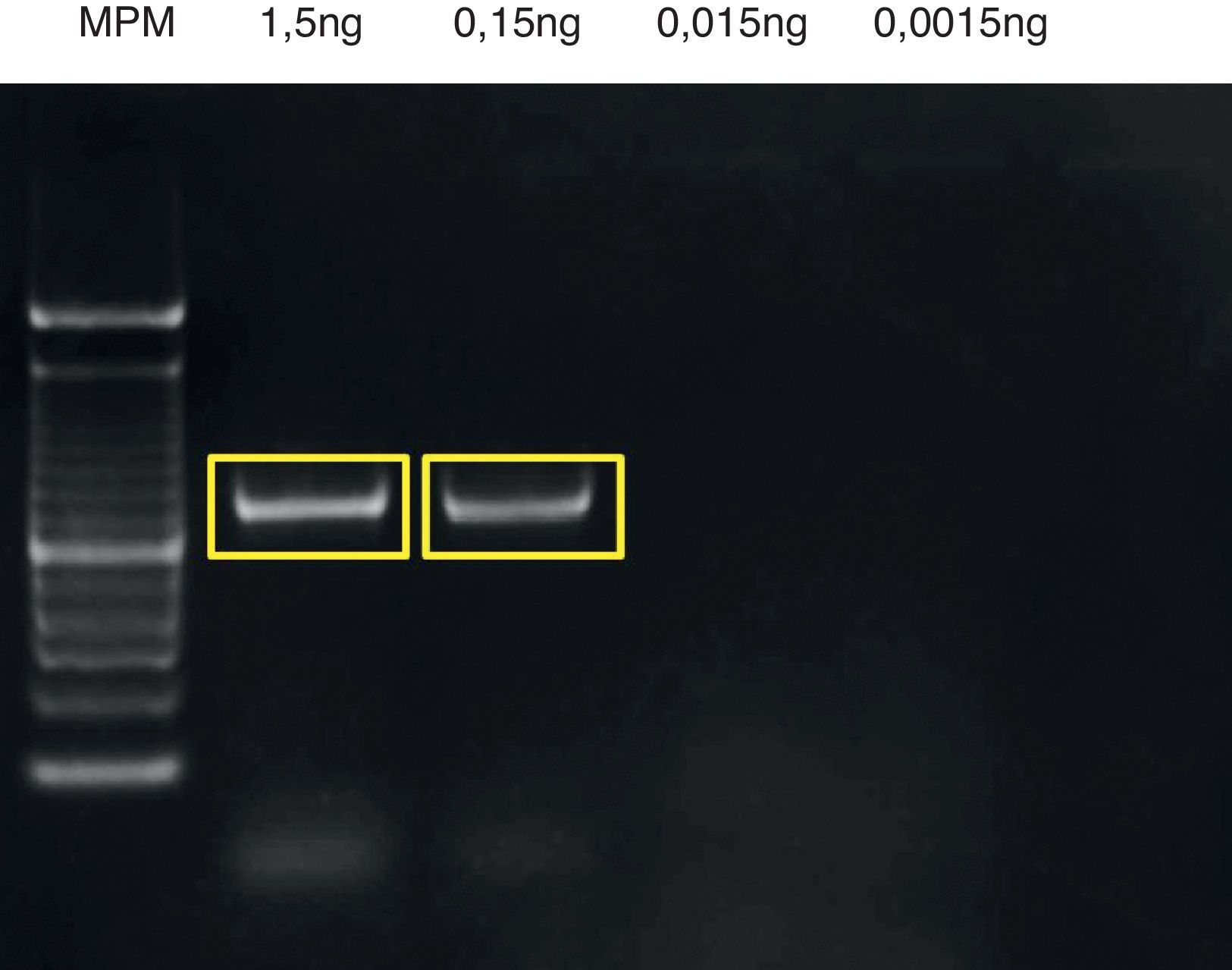
Utilizando este programa, se realizó PCR de bacterias y hongos usando diluciones del ADN respectivo y se obtuvo producto de amplificación utilizando una cantidad de hasta 0.12ng de ADN bacteriano (fig. 3) y 0.15ng de ADN fúngico (fig. 4).
Finalmente, en la PCR anidada se obtuvo una sola banda de aproximadamente 1,000pb para bacterias gramnegativas y 2 bandas para bacterias grampositivas, la primera de aproximadamente 1,050pb y la segunda de alrededor de 400pb (fig. 5).
Gel de agarosa al 1.5% de la PCR anidada. En el gel 1 (panel izquierdo) se observan 2 bandas de amplificación que corresponden a bacterias grampositivas (G+). En el gel 2 (panel derecho) se observa una sola banda de amplificación que corresponde a bacterias gramnegativas (G−). La PCR se ejecutó con un control negativo (C-).
La PCR es un método de biología molecular que permite amplificar el material genético de tal forma que una pequeña porción de ADN es suficiente para detectar la presencia de bacterias y hongos. En una muestra obtenida por raspado palpebral se obtiene una cantidad de 3 a 6ng/μl de ADN compuesto de una mezcla donde el mayor porcentaje proviene de las células del sujeto, mientras que en un raspado corneal la cantidad de ADN que se obtiene es menor, de 1 a 3ng/μl. En un paciente con infección ocular la cantidad de ADN obtenida sigue siendo la misma, pero el porcentaje de ADN proveniente de los microorganismos aumenta debido a la proliferación de los mismos en el sitio de la lesión, lo que hace posible su detección por PCR; utilizando los oligonucleótidos adecuados podemos amplificar e identificar si se trata de un hongo o una bacteria y, en caso de detectar un amplificado correspondiente a material genético bacteriano, es posible identificar si se trata de una bacteria grampositiva o gramnegativa usando la PCR anidada. Utilizando como base los trabajos de Jaegger et al. y Carrol et al.32,33 hemos logrado acortar el tiempo de duración de la PCR, obteniendo resultados en una sola ejecución utilizando una Tm en común para la detección de hongos y bacterias; esto es muy importante para el diagnóstico puesto que es un apoyo para la terapéutica oportuna y el restablecimiento de la salud del paciente.
ConclusionesLas pruebas de microbiología comúnmente utilizadas pueden tardar de una a 2 semanas para obtener el resultado de apoyo al diagnóstico oftalmológico, en cambio, utilizando técnicas de biología molecular podemos acortar ese tiempo lo cual es benéfico para el paciente que sufre de un evento clínico infeccioso pues para llevar a cabo un tratamiento eficaz y oportuno es necesario encontrar la causa. La técnica de PCR es una herramienta muy importante para la detección de ADN de bacterias grampositivas, gramnegativas y hongos causantes de infecciones oftalmológicas. Permite amplificar el material genético del agente etiológico y facilita su observación, obteniéndose los resultados en menor tiempo que utilizando las técnicas convencionales. Jaegger et al. y Carrol et al. fueron los pioneros en el desarrollo de una técnica de biología molecular para la detección de hongos y bacterias en fluidos oculares. Es muy importante que cada laboratorio establezca sus propias condiciones de trabajo y que se verifique tanto la reproducibilidad como la sensibilidad del método. En este trabajo pudimos establecer la PCR para la detección de bacterias y hongos utilizando una Tm en común que evita hacer 2 ejecuciones y acortar el tiempo en la obtención de resultados, optimizando la técnica ya reportada.
FinaciamientoLos autores no recibieron patrocinio para llevar a cabo este artículo.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Al Dr. Víctor Bautista de Lucio por la donación de material biológico.
A la Fundación Nacional Monte de Piedad.