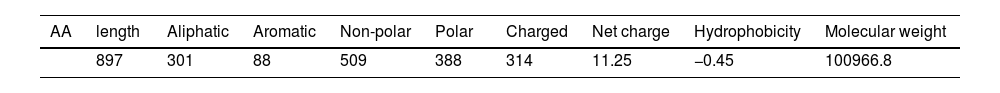Brucellosis infection could be eradicated by using an effective vaccine. Inf C gene expression generates one of the main Brucella pathogenesis proteins. The purpose of this study was to design a new vaccine against Brucella disease by in silico determination of epitopes of inf C protein.
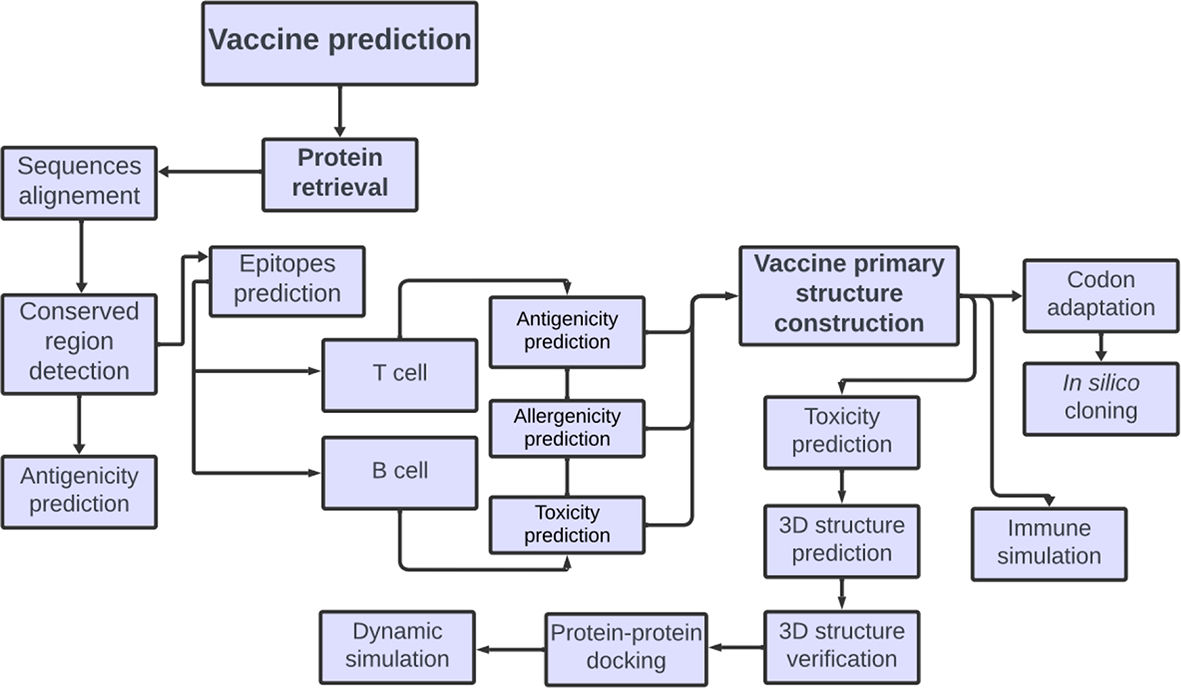
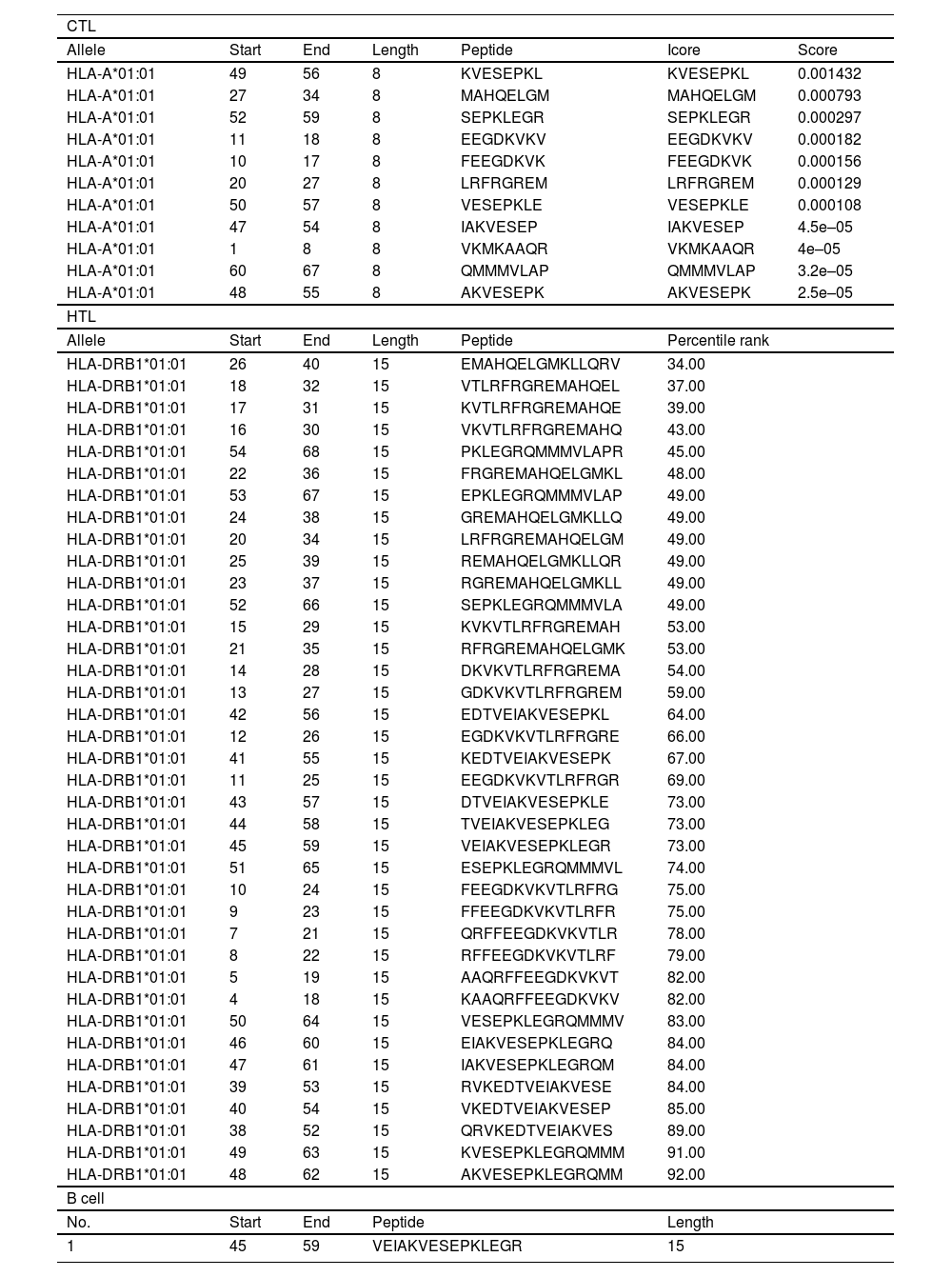
MethodsIn the first, Inf C amino acid sequences were extracted from the UniProt database and subjected to in silico analysis, including multiple sequence alignment, conserved region determination, allergenicity, antigenicity, and toxicity of the selected epitopes for TCL, HTL, and BCl. Vaccine-target (MHC alleles and TLRs) interactions, binding affinities, and dynamical stabilities were inspected through molecular docking and molecular dynamic simulation (MD) using Cluspro 2 server and GROMACS packages respectively. Further, the codon adaptation of the designed vaccine was determined by the JCat server and the obtained sequence was cloned in pET19b(+)vector by pDRAW32 software. Finally, the ability of the newer vaccine to stimulate the immune response was assessed using a computational immune simulation.
ResultsResults allowed us to select a peptide vaccine on basis of its good binding affinities with TLR-8 allele. The multi-peptide vaccine showed also to be highly antigenic, non-allergenic, non-toxic, and potential expression in E coli. Results showed also good stability of the vaccine–TLR8 complex and strong cellular and humoral immune response after three in silico injections of the vaccine construct.
ConclusionAll these theoretical results reveal that the conserved region of inf C protein could be used for designing of a new potent vaccine against Brucella.
la infección por brucelosis podría erradicarse mediante el uso de una vacuna eficaz. La expresión del Inf C gen genera una de las principales proteínas de patogénesis de Brucella. El propósito de este estudio fue diseñar una nueva vacuna contra la enfermedad de Brucella mediante la determinación in silico de los epítopos de la inf C proteína.
MétodosEn primer lugar, las secuencias de Inf C aminoácidos se extrajeron de la base de datos UniProt y se sometieron a análisis in silico, incluida la alineación múltiples de secuencias, la determinación de la región conservada, la alergenicidad, la antigenicidad y la toxicidad de los epítopos seleccionados para TCL, HTL y BCl. Las interacciones vacuna-objetivo (alelos MHC y TLR), afinidades de unión y estabilidades dinámicas se inspeccionaron mediante el acoplamiento molecular y molecular dinámica simulación (MD) utilizando el servidor Cluspro 2 y los paquetes GROMACS, respectivamente. Además, el servidor JCat determinó la adaptación de codones de la diseñada vacuna y luego la secuencia obtenida se clonó en el vector pET19b(+) mediante el software pDRAW32. Finalmente, se evaluó la capacidad de la nueva vacuna para estimular la respuesta inmunitaria mediante una computacional inmunitaria simulación.
Resultadoslos resultados nos permitieron de seleccionar una peptídica vacuna sobre la base de sus buenas afinidades de unión con el alelo TLR-8. La multipéptido vacuna demostró también ser altamente antigénica, no alergénica, no tóxica y con potencial expresión en E coli. Los resultados también mostraron una buena estabilidad del constructo complejo TLR8-vacuna y unas fuerte humoral y celular inmune respuestas después tres inyecciones in silico del constructa vacuna.
ConclusiónTodos estos teóricos resultados revelan que la región conservada de la inf C proteína podría usarse para diseñar una nueva potente vacuna contra Brucella.
Artículo
Comprando el artículo el PDF del mismo podrá ser descargado
Precio 19,34 €
Comprar ahora