La medicina personalizada entrega un nuevo enfoque a la prevención, diagnóstico y tratamiento de cada paciente, basándose en la genética y el estilo de vida de cada persona. Para este objetivo se utilizan biomarcadores que permiten identificar y clasificar a los pacientes según sus características individuales. La anatomía patológica no ha estado exenta de esta nueva estrategia terapéutica, y gracias a los estudios moleculares diseñados en los últimos años se han identificado firmas genéticas en numerosas neoplasias. Estas marcas clave se pueden estudiar mediante diferentes enfoques técnicos, y permiten al médico patólogo integrar la información histológica y molecular, con el fin de abordar estas enfermedades de una manera más segura y efectiva. En esta revisión se detallan los principales biomarcadores utilizados para el estudio de tumores en anatomía patológica, que constituyen una herramienta vital para determinar el tratamiento personalizado adecuado de nuestros pacientes
Personalized medicine provides a new approach to the prevention, diagnosis and treatment of each patient, based on the genetics and lifestyle of each person. For this purpose, biomarkers are used to identify and classify patients according to their individual characteristics. Pathology has not been exempt from this new therapeutic strategy, and thanks to molecular studies designed in recent years, genetic signatures have been identified in numerous neoplasms. These key marks can be studied using different technical approaches, and allow the pathologist to integrate histological and molecular information, in order to address these diseases in a safer and more effective way. This review details the main biomarkers used for the study of tumors in Pathology, which constitute a vital tool to determine the appropriate personalized treatment of our patients.
Desde hace más de una década, la medicina personalizada (medicina de precisión) entró en el mundo científico, mostrando un singular crecimiento desde entonces. Se define como un nuevo enfoque en las modalidades de prevención y tratamiento de pacientes, basado en la genética del individuo y la variabilidad de su estilo de vida. Notables hitos como la publicación de la secuencia del genoma humano y los numerosos avances tecnológicos de los últimos años han permitido enfocar el tratamiento mediante la medicina de precisión, teniendo como objetivo personalizar la atención médica con decisiones terapéuticas adaptadas a cada persona, considerando las diferencias individuales en la clínica, ambiente, estilos de vida e información de biomarcadores (DNA, RNA, proteínas y metabolitos)1.
La medicina de precisión ha rediseñado todas las áreas de la salud, incluida la anatomía patológica, en donde el patólogo emerge como una figura clave integrando la histología e información molecular y genética, especialmente en el estudio de tumores2. Gracias al estudio molecular realizado en neoplasias, se han encontrado varios tipos de mutaciones (cambios de base, amplificaciones, deleciones, traslocaciones) en todos los tipos de cáncer. Esta firma genética del tumor constituye una gran oportunidad en el manejo de los pacientes oncológicos, ya que permite al equipo médico realizar el mejor diagnóstico, así como evaluar el pronóstico y las implicaciones terapéuticas en cada caso3.
Las muestras de tumor fijadas en formalina e incluidas en parafina permiten el estudio de diferentes biomarcadores a través de diferentes aproximaciones técnicas. Entre ellas destacan la inmunohistoquímica (IHQ), la reacción en cadena de la polimerasa (PCR), hibridación in situ (ISH) y la secuenciación de nueva generación (NGS)4.
La siguiente revisión tiene como objetivo resumir los principales biomarcadores determinados en el estudio histopatológico de tumores, con enfoque en la práctica clínica y en el uso de terapias específicas.
2Consideraciones inicialesPara el estudio de biomarcadores en muestras de pacientes oncológicos, los equipos de salud deben trabajar con enfoque multidisciplinario, en donde médicos oncólogos, cirujanos, radiólogos y patólogos definan que test diagnósticos entregan información clave para el manejo correcto del paciente. Frente a este escenario el rol de anatomía patológica será esencial para garantizar que los pacientes reciban informes de calidad.
Para cumplir con este objetivo, se deben considerar procesos con metodologías que aseguren todos los estándares nacionales e internacionales de calidad, desde la obtención de la muestra hasta el informe final. Dentro de los puntos críticos que se deben monitorear están: tamaño de la muestra de biopsia, tiempos cortos de isquemia, y procesos óptimos de fijación con soluciones estandarizadas para el análisis posterior de biomarcadores (ej: formalina neutra tamponada). Una vez que la muestra es procesada en el laboratorio, el médico patólogo debe seleccionar el tejido que será evaluado para cada biomarcador. Para ello debe considerar tanto la naturaleza de la muestra, además de cumplir con los requisitos de tamaño, celularidad, interferentes, entre otros. Finalmente, una vez realizado el test del biomarcador particular, el patólogo debe analizar e integrar el resultado al reporte anátomo-patológico final del paciente5.
3Cáncer de pulmónEl cáncer de origen pulmonar es una de las neoplasias con mayores tasas de mortalidad en hombres y mujeres en Chile y el mundo6,7. El carcinoma pulmonar es una enfermedad heterogénea, cuyo diagnóstico se realiza con un enfoque multidisciplinario integrando la clínica, imágenes y el estudio histológico. Se reconocen dos grupos: de célula pequeña (SCLC) y de célula no pequeña (NSCLC), siendo el adenocarcinoma el subtipo más frecuente en este último grupo8. Estudios genéticos realizados en los últimos años, han determinado varios biomarcadores clave en la evaluación de pacientes con adenocarcinomas (Tabla 1), entre ellos EGFR, ALK, ROS1, RET, BRAF, MET, NTKR y KRAS, con un enfoque en terapia personalizada. En la actualidad estos marcadores poseen tratamientos dirigidos aprobados por la FDA, y se describen a continuación9–11:
Biomarcadores en cáncer de pulmón
| Biomarcador | Mutación buscada | Método de testeo |
|---|---|---|
| EGFR | Mutaciones puntuales, deleciones e inserciones | PCR-NGS-FISH |
| ALK | Rearreglos | IHQ-FISH-NGS-PCR |
| ROS1 | Rearreglos | IHQ-FISH-NGS-PCR |
| RET | Rearreglos | FISH |
| BRAF | Mutación puntual | PCR-NGS-FISH |
| MET | Mutación puntual y deleciones | NGS-PCR |
| NTRK | Rearreglos | FISH-NGS-PCR-IHQ |
| KRAS | Cambio de base nucleotídica | PCR-NGS |
Abreviaturas: PCR: reacción en cadena de la polimerasa; NGS: secuenciación de próxima generación; FISH: hibridación in situ fluorescente; IHQ: inmunohistoquímica.
-EGFR (receptor del factor de crecimiento epidérmico): pacientes con cáncer de pulmón etapa IV que poseen mutaciones en el gen EGFR pueden ser tratados con inhibidores tirosina quinasa (TKI), como son: afatinib, dacomitinib, erlotinib, gefitinib, osimertinib, exceptuando aquellos que presenten inserciones en el exón 20, en donde a los pacientes se les recomienda tratarse con quimioterapia e inmunoterapia.
-ALK (receptor tirosina quinasa ALK): pacientes con cáncer pulmonar etapa IV que presenten rearreglos del gen ALK pueden ser tratados con TKI, incluidos: crizotinib, ceritinib, alectinib, brigatinib, lorlatinib.
-ROS1 (receptor tirosina quinasa ROS proto-oncogen 1): pacientes con cáncer de pulmón etapa IV que presenten rearreglos del gen ROS1 pueden ser tratados con TKI, entre ellos crizotinib y entrectinib.
-RET (RET proto-oncogen): pacientes con cáncer pulmonar etapa IV que presenten rearreglos del gen RET pueden ser tratados con TKI como son selpercatinib y pralsetinib.
-BRAF (serina-treonina quinasa B-Raf proto-oncogen): pacientes con cáncer de pulmón que presenten la mutación de BRAF V600E pueden ser tratados con terapia combinada de dabrafenib y trametinob o con inmunoterapia.
-MET (receptor tirosina quinasa, MET proto-oncogen): pacientes con cáncer de pulmón que presenten la alteración MET conocida como exón 14 skipping (salto del exón 14) pueden ser tratados con capmatinib o tepotinib. Otras alteraciones en este gen, como es su amplificación, también se ha detectado en pacientes con NSCLC. Sin embargo, en estos casos se recomienda uso de inmunoterapia.
-NTRK (receptor tirosina quinasa neurotrófico): este biomarcador corresponde a tres genes: NTRK1, NTRK2 y NTRK3, los cuales, si bien presentan baja frecuencia de alteraciones en cáncer de pulmón, poseen tratamiento personalizado disponible. En el escenario de fusión de uno de los tres genes, el tratamiento de elección sería con drogas como entrectinib y larotrectinib.
-KRAS (GTPasa KRAS proto-oncogen): gen frecuentemente mutado en pacientes con NSCLC, para el cual no se encontraban drogas que pudieran contrarrestar sus alteraciones. Recientemente la FDA ha aprobado un inhibidor contra la mutación de KRAS G12C, llamado sotorasib.
Es importante destacar que, en este tipo de neoplasias, en donde el material para estudio histológico puede ser limitado y el número de marcadores moleculares a estudiar es amplio, se debe preferir emplear técnicas que permitan maximizar el DNA que se obtiene de la muestra. Por ello, el uso de plataformas que permiten identificar múltiples genes y/o alteraciones a la vez es lo más recomendado (por ejemplo: secuenciación masiva (NSG) y PCR en tiempo real como la plataforma Idylla™)12.
4Cáncer de mamaPara carcinoma de mama invasor, en tumores recurrentes y/o metastásicos, el Colegio Americano de Patólogos (CAP) y la Sociedad Americana de Oncología Clínica (ASCO) recomiendan realizar inicialmente la determinación de tres biomarcadores (Tabla 2): receptor de estrógeno (RE), receptor de progesterona (RP) y el receptor 2 del factor de crecimiento epidérmico humano (HER2). Estos marcadores poseen valores predictivos y de pronóstico13–15. A nivel diagnóstico, el cáncer de mama se clasifica en cinco subtipos, en función de la expresión de estas moléculas. A nivel de tratamiento, determinar el estatus de los receptores hormonales (RE y RP) permitirá identificar pacientes que podrían beneficiarse de la terapia hormonal16. El tercer marcador evaluado, receptor HER2, ha mostrado estar sobre-expresado en 15-30% de los pacientes con cáncer de mama, asociándose con tumores más agresivos. Sin embargo, este criterio permite seleccionar pacientes que se beneficiarán con terapias anti-HER2, entre ellas trantuzumab, pertuzumab y lapatinib, lo que permite mejorar el pronóstico de pacientes que reciben estos medicamentos14,17. Además, en pacientes con cáncer de mama metastásico positivo para RE y RP y negativo para HER2, se debe determinar el estatus mutacional de PIK3CA (fosfatidilinositol-4-fostato 3 quinasa subunidad catalítica tipo 2 alfa), debido a que desde el año 2019 se encuentra aprobado el tratamiento de estos pacientes con inhibidores para PI3K, como es el alpelisib18. Recientemente, la FDA ha aprobado el uso de abemaciclib junto a la terapia endocrina, en pacientes con alto riesgo de recurrencia con receptores hormonales positivos, HER2 negativo, nódulos positivos y con valores de Ki-67 igual o superior al 20%19. En cáncer de mama triple negativo (RE, RP, y HER2 no expresados) se recomienda determinar el estatus PD-L1, ya que estas pacientes pueden ser tratadas con inmunoterapia20.
Biomarcadores en cáncer de mama
| Biomarcador | Método de testeo | Objetivo |
|---|---|---|
| RE | IHQ | Determinar porcentaje de expresión por células tumorales |
| RP | IHQ | Determinar porcentaje de expresión por células tumorales |
| PI3K | PCR/NGS | Búsqueda de mutaciones activantes |
| Ki-67 | IHQ | Determinar porcentaje de expresión por células tumorales |
| HER2 | IHQ/FISH | Búsqueda de sobreexpresión de la proteína/amplificación del gen |
Abreviaturas: PCR: reacción en cadena de la polimerasa; NGS: secuenciación de próxima generación; FISH: hibridación in situ fluorescente: IHQ: inmunohistoquímica.
El cáncer colorrectal (CCR), al igual que el cáncer de pulmón, es una de las neoplasias malignas de mayor mortalidad a nivel mundial21. En el CCR existen una amplia gama de biomarcadores que se utilizan en su manejo, permitiendo determinar predisposición y detección de la enfermedad en etapas tempranas, así como monitorear el éxito del tratamiento y recurrencia de la enfermedad22. Dentro de los biomarcadores diagnósticos podemos encontrar una vasta gama de moléculas que se pueden determinar en muestras de tejido (citoqueratinas, CDX2, etc.), en sangre (inestabilidad cromosómica (CIN), DNA libre circulante (cfDNA), etc.), en heces (sangre oculta, DNA, RNA)23. Respecto a biomarcadores pronósticos y predictivos en muestras de tejido tumoral, el CAP y la ASCO recomiendan realizar determinación de los siguientes biomarcadores: inestabilidad microsatelital (MSI) y/o proteínas de reparación del DNA (MMR), RAS (KRAS y NRAS), HER2, NTRK y BRAF24–26, los que se detallan a continuación:
-BRAF: se recomienda evaluar el estado mutacional de BRAF en todos los pacientes con CCR metastásico en etapa IV antes de iniciar terapias con inhibidores anti-EGFR, ya que mutaciones activantes en esta proteína predicen respuesta a estos medicamentos. Inhibidores anti-BRAF aún no están disponibles para este tipo de tumores.
-KRAS y NRAS: se recomienda evaluar el estado mutacional de KRAS y NRAS en todos los pacientes con CCR metastásico en etapa IV antes de iniciar terapias con inhibidores anti-EGFR. Esta determinación se sugiere realizar en el tumor primario, así como en las metástasis. Pacientes que porten mutaciones en RAS no deben tratarse con inhibidores anti-EGFR.
-HER2: se recomienda evaluar esta mutación en todos los pacientes con CCR metastásico en etapa IV. Pacientes con amplificación de HER2 (con RAS y BRAF sin mutaciones) pueden tratarse con trastuzumab, pertuzumab o lapatinib.
-Fusiones NTRK: pacientes con fusiones en los genes
NTRK 1, 2 o 3 pueden ser tratados con larotrectinib o entrectinib.
-MMR: en este caso se determina que el tumor tenga deficiencias en los sistemas de reparación del DNA, para ello se evalúan al menos 4 proteínas: MLH1, MSH2, MSH6 y PMS2. Pacientes en donde el proceso de reparación está defectuoso se correlacionan como alta inestabilidad microsatelital (MSI-H). Por ello se recomienda evaluar en tumores CCR metastásicos en etapa IV, los cuáles podrían ser tratados con inmunoterapia (Tabla 3).
Biomarcadores en cáncer colorrectal
| Biomarcador | Mutación buscada | Método de testeo |
|---|---|---|
| BRAF | Cambio de base nucleotídica | PCR/NGS/IHQ |
| KRAS | Cambio de base nucleotídica | PCR/NGS |
| NRAS | Cambio de base nucleotídica | PCR/NGS |
| HER2 | Amplificación | IHQ/FISH |
| NTRK | Rearreglos | NGS/PCR |
| MMR/MSI | Expresión disminuida/Fenotipo alta MSI | IHQ/PCR |
Abreviaturas: PCR: reacción en cadena de la polimerasa; NGS: secuenciación de próxima generación; FISH: hibridación in situ fluorescente; IHQ: inmunohistoquímica.
Tumores con MSI-H puede presentarse como mutaciones no hereditarias. Sin embargo, en aproximadamente un 3-5% de los casos pueden ser causados por el síndrome de Lynch, en donde la mutación es germinal y puede trasmitirse de generación a generación. En estos casos el consejo genético cobra gran relevancia.
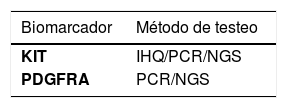
6Tumor estromal gastrointestinal (GIST)Los GIST son los tumores mesenquimáticos más comunes del tracto gastrointestinal. Histológicamente los GIST pueden ser similares a otras neoplasias mesenquimáticas. Sin embargo, es necesario diferenciarlo de otras entidades, ya que existen terapias dirigidas disponibles para GIST avanzados o metastásicos, en donde se utilizan inhibidores de tirosina quinasa27. Por ello, los biomarcadores moleculares clave (Tabla 4) que deben analizarse son28:
-KIT (receptor tirosina quinasa, proto-oncogen KIT): esta alteración es la más frecuente en GIST. Pacientes que portan esta mutación pueden beneficiarse del tratamiento con imatinib, sunitunib, regorafenib.
-PDGFRA (receptor alfa del factor de crecimiento derivado de plaquetas): pacientes con GIST metastásico que poseen alteraciones en este gen pueden ser candidatos al tratamiento con avapritinib.
7Adenocarcinoma de estómago y unión gastroesofágicaLa mayoría de los pacientes diagnosticados con cáncer gástrico presentan una enfermedad avanzada o metastásica29. El CAP recomienda determinar el estatus del gen y la expresión de la proteína HER2 en todos los pacientes con adenocarcinoma no operable localmente avanzado, recurrente o metastásico, ya que el tratamiento con terapias anti-HER2, como el trastuzumab, ha mostrado ser efectivo en aumentar la sobrevida de los pacientes cuando se compara sólo con quimioterapia30. Otros marcadores útiles de determinar en estos pacientes son MSI y PD-L1, para los cuales también existen disponibles terapias dirigidas31.
8MelanomaDesde el año 2015 el CAP recomienda realizar análisis de BRAF. Mutaciones en este gen ocurren en más del 50% de los melanomas. Pacientes con mutaciones en BRAF pueden beneficiarse con drogas como vemurafenib, dabrafenib o encorafenib, las que han demostrado aumentar en seis meses la sobrevida32. En el caso de pacientes con melanoma metastásico se debe determinar si los pacientes son candidatos a inmunoterapia, ya que el uso de nivolumab y pembrolizumab está autorizado33.
9Tumores del sistema nervioso centralLos tumores cerebrales comprenden un amplio y heterogéneo grupo de enfermedades que comprometen al cerebro. Se dividen en dos grandes grupos: primarios (los originados en componentes del sistema nervioso central [SNC]), y secundarios (metástasis de tumores que se originan fuera del SNC)34. Según el reporte estadístico CBTRUS 2013-2017 la tasa de incidencia anual de tumores cerebrales primarios en Estados Unidos es de 23,79 por cada 100.000 habitantes, siendo el meningioma el principal tumor benigno y el glioblastoma el tumor maligno más frecuente35. En Chile no existe estadística sobre tumores cerebrales primarios, aunque estudios iniciales basados en datos de egresos hospitalarios muestran que la mortalidad por tumores del SNC en el año 2013 en nuestro país fue de 2,6 por 100.000 habitantes34. La decisión del tratamiento de los pacientes con estos tumores dependerá de varios factores como son el tamaño, la ubicación y el tipo histológico.
Recientemente, la Organización Mundial de la Salud (OMS) introdujo un nuevo sistema de clasificación que incluyo la histología del tumor y parámetros moleculares36,37. Este diagnóstico integrado de los tumores ha permitido mayor precisión diagnóstica, determinaciones más precisas del pronóstico y selección adecuada de tratamiento38,39. Dentro de los biomarcadores relevantes a determinar (Tabla 5) en estos tumores están: IDH, codeleción 1p/19q, MGMT, Histona H3.3, TERT, CDKN2A, entre otros40–43], los cuales se detallan a continuación:
Biomarcadores en tumores del sistema nervioso central
| Biomarcador | Alteración estudiada | Método de testeo |
|---|---|---|
| IDH | Cambio de base nucleotídica | IHQ/PCR/NGS |
| Codeleción 1p/19q | Deleción | FISH |
| MGMT | Metilación | PCR |
| Histona H3.3 | Cambio de base nucleotídica | PCR/NGS/IHQ |
| TERT | Cambio de base nucleotídica | PCR/NGS |
| CDKN2A | Deleción | FISH |
Abreviaturas: PCR: reacción en cadena de la polimerasa; NGS: secuenciación de próxima generación; FISH: hibridación in situ fluorescente: IHQ: inmunohistoquímica.
IDH (Isocitrato Deshidrogenasa): este marcador corresponde a dos genes, IDH1 e IDH2. En IDH1, se ha observado una mutación puntual recurrente que genera un cambio en el residuo aminoacídico 132 de la secuencia de la proteína. Por otro lado, la mutación en gen IDH2 produce cambios en el residuo aminoacídico R172. Se debe determinar el estatus IDH en astrocitomas difusos grados 2 y 3, oligodendrogliomas grados 2 y 3, y glioblastomas. Mutaciones en uno de los dos genes se ha asociado con mejor pronóstico y respuesta a la terapia en pacientes con gliomas.
Codeleción 1p/19q (deleción de brazo corto del cromosoma 1 y deleción del brazo largo del cromosoma 19): El estatus 1p/19q es un marcador importante en el diagnóstico de tumores con componente oligodendroglioglial. Su uso en la clínica ha permitido definir el diagnóstico de este tipo de tumores, especialmente en el caso de neoplasias de morfología compleja.
Además, desde estudios iniciales se observó que los pacientes que presentan esta alteración poseen un mejor pronóstico y respuesta a la terapia con fármacos quimioterapéuticos como procarbazin, lomustina, vincristina y temozolomida.
MGMT (O6-metilguanina DNA Metiltrasferasa): esta proteína es responsable de reparar errores en el DNA. Por ello, al encontrarse silenciada por metilación en células del tumor, los pacientes responden mejor a la quimioterapia con drogas como la temozolomida. Se recomienda determinar este biomarcador en todos los gliomas de alto grado.
Histona H3.3: la mutación puntual en el gen de la histona H3.3 produce cambio a nivel de la secuencia de proteína en el aminoácido número 27. Este biomarcador permite predecir respuesta a tratamiento en gliomas de línea media difusos pediátricos.
TERT (Telomerasa trascriptasa inversa): en este biomarcador se evalúan mutaciones que ocurren en la secuencia promotora del gen TERT. Si las células tumorales presentan dichos cambios se asocia con tumores más agresivos y que no responden bien a los tratamientos (quimioterapia y radioterapia).
CDKN2A (Inhibidor de quinasa dependiente de ciclina 2A): Se debe determinar la deleción homocigota de CDKN2A en pacientes con gliomas IDH mutados, ya que se ha observado que pacientes con esta alteración poseen mal pronóstico y con supervivencia limitada.
BIOMARCADORES PARA INMUNOTERAPIA EN CÁNCER
Este tipo de biomarcador permite determinar la relación del sistema inmunológico del paciente con su tumor. En la práctica clínica se han incorporado para reconocer a pacientes que se beneficiaran del uso de este tipo de terapia44–46. Entre los más utilizados actualmente se encuentran:
-PD-1/PD-L1: la expresión de este biomarcador se debe estudiar para seleccionar pacientes que se beneficiaran de inmunoterapia. Tumores con altos niveles de expresión, estudiados por IHQ, tiene mejor respuesta que aquellos que no presentan expresión. Actualmente existen varios tipos de tumores con varias terapias disponibles, ejemplo de ello es el uso de pembrolizumab para tratar tumores NSCLC, CCR, gástrico, melanoma, o nivolumab para tratar tumores NSCLC, melanoma, CCR.
-Carga mutacional del tumor (TMB): este biomarcador hace referencia a las mutaciones que acumulan los tumores. Su estudio debe realizarse por medio de secuenciación de próxima generación (NGS). Dichas mutaciones generan producción de proteínas mutadas en las células del tumor, las cuáles el sistema inmunológico puede reconocer como neoantígenos. Recientemente se ha documentado que pacientes con alto TMB responden a terapias con inhibidores de punto de control inmunitario. Actualmente, este biomarcador se recomienda evaluar en pacientes con NSCLC.
10Biopsia líquidaLa biopsia líquida comprende una serie de procedimientos mínimamente invasivos que permiten obtener a través de muestras de sangre y otros fluidos orgánicos, componentes clave de la biología del tumor, tales como células tumorales circulantes, material genético y proteínas. Por ello, ha surgido como un complemento al estudio histopatológico clásico de tumores en el manejo del cáncer. Es especialmente valiosa cuando los procedimientos quirúrgicos son invasivos, con potenciales complicaciones, o cuando no se puede acceder adecuadamente al tumor. A partir de los componentes que se obtienen de este tipo de muestra se pueden estudiar biomarcadores especialmente útiles para el monitoreo de los pacientes, así como detectar respuestas a tratamiento y resistencia a los fármacos47,48. En el año 2016, la FDA aprobó el primer test diagnóstico que utilizaba como muestra inicial biopsia líquida, en donde a partir de muestras de plasma se pudo detectar mediante PCR mutaciones en el gen EGFR en pacientes con NSCLC, pudiendo ser tratados con erlotinib49. Hoy en día, plataformas como FoundationOne Liquid CDx utilizando NGS, permiten detectar más de 300 alteraciones, especialmente útiles en guiar la selección de terapias de pacientes con tumores sólidos, como cáncer de próstata y de pulmón50.
10.1Perspectivas a futuroBasado en los últimos aconteceres científicos tanto en Chile como en el mundo entero, se espera que el cáncer llegue a convertirse en la primera causa de muerte. En este contexto la medicina personalizada será una herramienta fundamental en el tratamiento de los pacientes oncológicos. El uso de la droga precisa para cada enfermedad, basado en la evidencia genética individual, parece vislumbrarse en el futuro próximo. El uso de compuestos terapéuticos que respondan a las características moleculares de cada tumor, con menos efectos indeseados, permitirán controlar la enfermedad de manera eficiente y a la vez mejorar la calidad de vida de los pacientes.
Dentro de los desafíos que presenta la medicina de presión en tumores sólidos están la heterogeneidad tumoral y la resistencia de los tumores a las terapias dirigidas. Por ello los pacientes deberán estar en constante seguimiento durante su tratamiento y la figura de la biopsia líquida, en conjunto a los estudios clásicos, emerge como una gran alternativa que permitirá monitorizar enfermedad mínima residual, cambios en estados mutacionales y detectar resistencias a los fármacos en forma temprana. Bajo esta mirada, se espera que el rol del especialista en anatomía patológica sea clave en la integración y aplicación que nos ofrecerá la medicina de precisión del futuro.
Finalmente, es de vital importancia que las entidades de salud implementen de manera rutinaria el uso de herramientas moleculares en la práctica médica, las cuales, junto al diagnóstico histológico, permitirán tomar las mejores decisiones terapéuticas en beneficio del paciente.
Declaración de conflicto de interésLos autores declaran no tener conflictos de interés, en relación a este artículo.