En España, al igual que en otros países donde el sarampión endémico ha sido eliminado, es necesario utilizar de forma rutinaria las herramientas diagnósticas que confirmen los casos para su prevención y control de la diseminación. Se describen los diferentes ensayos microbiológicos utilizados para su diagnóstico durante un brote de sarampión en 2019 en la provincia de Guadalajara (España).
MétodosLas pruebas serológicas y moleculares se realizaron en el laboratorio de Microbiología del Hospital Universitario de Guadalajara y en el Centro Nacional de Microbiología del Instituto de Salud Carlos III (Majadahonda, España). Los datos de los pacientes se obtuvieron del sistema epidemiológico de vigilancia.
ResultadosSe diagnosticaron de sarampión un total de 43 pacientes por métodos microbiológicos: 29 casos por PCR (exudado faríngeo u orina) junto con IgM específica positiva, 11 pacientes solamente por PCR, y 3 pacientes exclusivamente por presencia de IgM. El genotipo D8 fue identificado en 35 pacientes y el genotipo A en 2 casos descartados como postvacunal. La PCR en suero fue positiva en 11 de 14 pacientes con ausencia de IgM en su primera muestra recogida de suero. Once casos confirmados habían recibido una o 2 dosis de la vacuna. Doce adultos fueron ingresados, todos diagnosticados de hepatitis.
ConclusionesLa combinación de pruebas moleculares y la presencia de IgG e IgM específicas son necesarias para un diagnóstico correcto y la clasificación de los pacientes como fallo vacunal (primario o secundario). El genotipado es una herramienta fundamental para la correcta clasificación de los pacientes en el contexto de un programa de eliminación del sarampión.
In Spain, like in other countries where endemic measles has been eliminated, there is a need for available diagnostic tools for confirming any cases in order to prevent and control its transmission. We describe the different microbiological tests used for the diagnosis of measles during an outbreak that occurred in 2019 in the province of Guadalajara (Spain).
MethodsSerological and molecular tests were performed at the Microbiology laboratory of the Guadalajara University Hospital and at the National Center for Microbiology of the Carlos III Health Institute (Majadahonda, Spain). Patient data were obtained from the surveillance system.
ResultsA total of 43 patients had a laboratory diagnosis of measles: 29 cases by PCR (pharyngeal exudate or urine) and positive specific IgM, 11 cases by PCR, and 3 cases only by a positive IgM. Genotype D8 was identified in 35 confirmed cases and genotype A in 2 that were discarded as post-vaccination cases. PCR was positive in the acute sera of 11 out of 14 patients with a negative IgM. Eleven confirmed cases had recieved one or 2 vaccine doses. Twelve adult patients were hospitalizated, all of them with a diagnostic of hepatitis.
ConclusionsThe combination of molecular tests and the presence of specific IgG and IgM are necessary for a correct diagnosis of measles and also to classify patients with a breakthrough infection or vaccine failures (primary or secondary). Genotyping is essential for the correct classification of the patients in the context of a measles elimination program.
El sarampión es una enfermedad altamente contagiosa por vía respiratoria que afecta únicamente a humanos. La existencia de una vacuna específica de elevada eficacia ha significado un descenso importante en la incidencia de la enfermedad. Esto ha llevado a la Organización Mundial de la Salud (OMS)1 a fijar como objetivo la eliminación de esta enfermedad. En el año 2017 la OMS declaró que España había alcanzado la eliminación del sarampión al no registrar transmisión endémica los 3 años anteriores, en presencia de un sistema de vigilancia sensible y específico. Así, los casos de sarampión que se dan en nuestro país son importados o relacionados con la importación y los brotes que se producen son pequeños y limitados en el tiempo, afectando principalmente a adultos no vacunados o con vacunación incompleta2. Los casos en personas correctamente vacunadas en España se habían estimado en un 3% hasta el año 20143, sin embargo, en los últimos años han ido en aumento, como está descrito en otros países en situación de eliminación y con elevadas coberturas vacunales. Para el correcto control y prevención de casos secundarios e identificación de cadenas de transmisión, es necesaria la investigación de laboratorio de los casos en el menor tiempo posible, incluyendo la caracterización molecular de las cepas. Clásicamente, el diagnóstico se basaba en la detección de anticuerpos específicos de la clase IgM contra el virus. El retraso en la detección de anticuerpos de fase aguda específicos (IgM) o en la seroconversión del título de anticuerpos IgG ha favorecido el uso cada vez más extenso de la detección de genomas víricos para el diagnóstico debido a su positividad más precoz.
Además, la existencia de casos de sarampión en personas correctamente vacunadas plantea grandes retos diagnósticos tanto desde el punto de vista clínico, puesto que pueden presentar una forma atípica de la enfermedad, como desde el punto de vista del diagnóstico de laboratorio, puesto que no se detecta IgM específica o aparece de forma más tardía. Esto disminuye el valor predictivo negativo de esta técnica, cobrando una mayor relevancia el diagnóstico molecular para la adecuada clasificación de los casos. Así mismo, en países con baja circulación del virus, una IgM específica en un caso sin una vinculación epidemiológica clara o fuerte sospecha clínica no permite confirmar el caso. De este modo, la OMS ha establecido algoritmos de diagnóstico específicos para países en situación de eliminación del sarampión1.
En el año 2019 se notificaron en España 606 casos sospechosos de sarampión. La tasa de incidencia nacional fue de 0,61 casos/100.000 habitantes. La mayoría de los casos se registraron en las comunidades autónomas de Cataluña, Madrid, Comunidad Valenciana y Castilla-La Mancha. La tasa más elevada por comunidades autónomas se registró en esta última, con 2,85 casos/100.000 habitantes2.
En el presente trabajo se describen las características epidemiológicas, microbiológicas y clínicas de los casos diagnosticados en la provincia de Guadalajara (España) pertenecientes a un brote que también afectó a las comunidades autónomas de Madrid y Aragón, y las dificultades para el diagnóstico de los casos.
MétodosEl estudio se realizó con las muestras recibidas entre el 8 de abril y el 25 de agosto de 2019 en el Hospital Universitario de Guadalajara, que atiende a una población aproximada de 250.000 habitantes. Las muestras procedían de pacientes que acudían al Servicio de Urgencias del hospital o bien de distintos centros de atención primaria de toda la provincia. Los datos clínico-epidemiológicos de todos los pacientes se recogieron a partir de la información proporcionada por la Sección de Epidemiología de la Delegación de Sanidad y Asuntos Sociales de Guadalajara. Estos datos incluían: edad, sexo, fecha de inicio de loa síntomas, fecha de inicio del exantema, signos y síntomas clínicos, contactos conocidos, antecedente de viajes, vacunación previa, complicaciones y necesidad de hospitalización.
Definición de casoLos casos se clasificaron siguiendo los criterios descritos en el protocolo de vigilancia de la Red Nacional de Vigilancia Epidemiológica4:
Caso sospechoso (caso clínicamente compatible): persona que cumple los criterios clínicos en el que no ha sido posible recoger muestras para su confirmación serológica y que no ha estado en contacto con un caso confirmado por laboratorio.
Caso probable (caso confirmado por vínculo epidemiológico): persona que cumple los criterios clínicos y que tiene vínculo epidemiológico con un caso confirmado por laboratorio.
Caso confirmado (caso confirmado por laboratorio): persona no vacunada recientemente que satisface los criterios clínicos y de laboratorio (presencia de IgM específica en suero o por detección de ARN del virus en muestra de exudado faríngeo y/o orina). Persona recientemente vacunada en la que se detecta el genotipo salvaje del virus.
Caso descartado: un caso que cumple los criterios clínicos de sarampión y que tiene resultados de laboratorio negativos o que está vinculado epidemiológicamente con un caso confirmado por laboratorio de otra enfermedad exantemática.
Estudio microbiológicoLa detección de IgG/IgM específicas se llevó a cabo en muestras de suero mediante inmunoanálisis quimioluminiscente (Liaison® Measles IgG/IgM, DiaSorin) en el analizador Liaison® (DiaSorin, Saluggia, Italia).
La extracción de ácidos nucleicos se llevó a cabo con el extractor automático MagCore® (RBC Bioscience, New Taipei City, Taipei) en el laboratorio de Microbiología del Hospital Universitario de Guadalajara. La amplificación del genoma del virus sarampión se realizó mediante una PCR en tiempo real, específica del gen de la nucleoproteína (RealCycler®, Progenie Molecular).
Para la confirmación de los 17 primeros casos y el genotipado de los virus, las muestras se enviaron al Centro Nacional de Microbiología del Instituto de Salud Carlos III (Majadahonda, España). La extracción de ácidos nucleicos totales se llevó a cabo utilizando el kit comercial QIAsymphony® Virus/Bacteria Midi Kit (96) (Qiagen) mediante un extractor automático (QIAsymphony, Qiagen, Hilden Germany). La detección del genoma de sarampión se realizó utilizando una PCR multiplex en tiempo real para virus exantemáticos (rubéola, sarampión y parvovirus B19), incluida en el Programa de Vigilancia Microbiológica de sarampión y rubéola5. Así mismo, la determinación del genotipo y el análisis de variantes se realizó por secuenciación de un fragmento de 450 nucleótidos del gen de la nucleoproteína (N450), recomendado por la OMS, tras su amplificación mediante una PCR en tiempo real descrita previamente6. La determinación de IgM e IgG en el Centro Nacional de Microbiología se llevó a cabo mediante ELISA (Enzygnost® Anti-Measles Virus [IgM/IgG], Siemens).
ResultadosDurante el periodo entre el 8 de abril y el 25 de agosto de 2019, se notificaron 88 casos sospechosos de sarampión en la provincia de Guadalajara (España). De ellos, 48 se clasificaron como confirmados por cumplir los criterios clínicos y de laboratorio para sarampión. Cuarenta y tres fueron diagnosticados en el Hospital Universitario de Guadalajara y los otros 5 en las comunidades autónomas de Madrid o de Aragón. Ocho casos se clasificaron como sospechosos y 3 como probables. Se descartaron 29 casos. Entre ellos, 2 casos al comprobar por genotipado (genotipo A, cepa vacunal) que se trataba de exantemas postvacunales y otro caso con IgG e IgM positivas por no presentar clínica y ser negativas por PCR 2 muestras de orofaringe y una de orina. En otros casos descartados se detectó otro agente etiológico, parvovirus B19 en 3 casos y virus de la rubéola en un caso.
Se recibieron 53 muestras de suero de los casos (entre 0 y 2 por paciente), 41 exudados faríngeos y 15 muestras de orina.
Los diagnósticos microbiológicos de los 43 casos se realizaron en 29 casos por presencia de IgM específica y PCR positiva (65,9%); en 11 casos por PCR positiva y en 3 por presencia de IgM (tabla 1). En 5 de los casos diagnosticados por PCR e IgM, esta se detectó en una segunda muestra de suero.
Método diagnóstico microbiológico de los 43 casos de sarampión y su relación temporal con la aparición del exantema
| Días en relación con la aparición del exantema | ||||||
|---|---|---|---|---|---|---|
| Caso | IgM | IgG | PCR (faringe/orina) | −2 a 0 | 1 a 4 | >4 |
| 1 | Pos | Neg | Pos | x | ||
| Pos | Neg | x | ||||
| 2 | Neg | Neg | NR | x | ||
| Pos | Pos | Pos | x | |||
| 3 | Pos | Neg | Pos | x | ||
| 4 | Pos | Neg | Pos | x | ||
| 5 | Posbajo | Neg | Pos | x | ||
| Pos | Pos | x | ||||
| 6 | Neg | Pos | Pos | x | ||
| 7 | Pos | Neg | Pos | x | ||
| 8 | Posbajo | Neg | Pos | x | ||
| 9 | Posbajo | Pos | Pos | x | ||
| 10 | Neg | Pos | Pos | x | ||
| 11 | Posbajo | Neg | Pos | x | ||
| 12 | Pos | Neg | Pos | x | ||
| Pos | Pos | x | ||||
| 13 | Neg | Pos | Pos | x | ||
| 14 | Pos | Pos | Pos | x | ||
| 15 | Pos | Pos | Pos | x | ||
| 16 | Pos | Neg | Pos | x | ||
| 17 | Pos | Pos | Pos | x | ||
| 18 | Neg | Neg | Pos | x | ||
| 19 | Neg | Neg | Pos | x | ||
| Pos | Pos | x | ||||
| 20 | Neg | Neg | Pos | x | ||
| 21 | Neg | Neg | Pos | x | ||
| 22 | NR | NR | Pos | x | ||
| 23 | Pos | Neg | Pos | x | ||
| 24 | NR | NR | Pos | x | ||
| 25 | Pos | Neg | Pos | x | ||
| 26 | Pos | Pos | Pos | x | ||
| 27 | Pos | Pos | Pos | x | ||
| 28 | Pos | Pos | Pos | x | ||
| 29 | Pos | Neg | Pos | x | ||
| 30 | Neg | Neg | x | |||
| Pos | Neg | Pos | x | |||
| 31 | Neg | Pos | Pos | x | ||
| Neg | Pos | x | ||||
| 32 | Neg | Neg | Pos | x | ||
| Pos | Pos | x | ||||
| 33 | Pos | Neg | NR | x | ||
| 34 | Neg | Neg | Pos | x | ||
| Pos | Pos | x | ||||
| 35 | Pos | Neg | Pos | x | ||
| 36 | Pos | Pos | NR | x | ||
| 37 | Posbajo | Neg | Pos | x | ||
| 38 | Neg | Pos | Pos | x | ||
| 39 | Pos | Neg | Pos | x | ||
| 40 | Pos | Neg | Pos | x | ||
| 41 | Neg | Pos | NR | x | ||
| Pos | Pos | x | ||||
| 42 | Pos | Neg | Pos | x | ||
| 43 | NR | NR | Pos | x | ||
Neg: negativo; NR: no realizado; PCR: reacción en cadena de la polimerasa; Pos: positivo; Posbajo: positivo débil (1-1,4 index).
De los 11 casos diagnosticados exclusivamente por PCR solo se dispuso de suero en 8 de ellos, en los que la toma de la muestra se realizó de forma temprana, antes de 4 días tras la aparición del exantema (suero basal para estudio de seroconversión).
En los 3 casos en los que el diagnóstico microbiológico se realizó exclusivamente mediante presencia de IgM, no se dispuso de muestra para PCR.
En la primera muestra de suero de 14 pacientes había presencia de anticuerpos IgG específicos. En 6 pacientes se evidenció seroconversión de IgG con una segunda muestra de suero.
Se determinó el genotipo en 37 casos; en 35 se identificó la variante MVs/GirSomnath.IND/42.16/ del genotipo D8, y en los otros 2, el genotipo A (vacunal), por lo que fueron descartados.
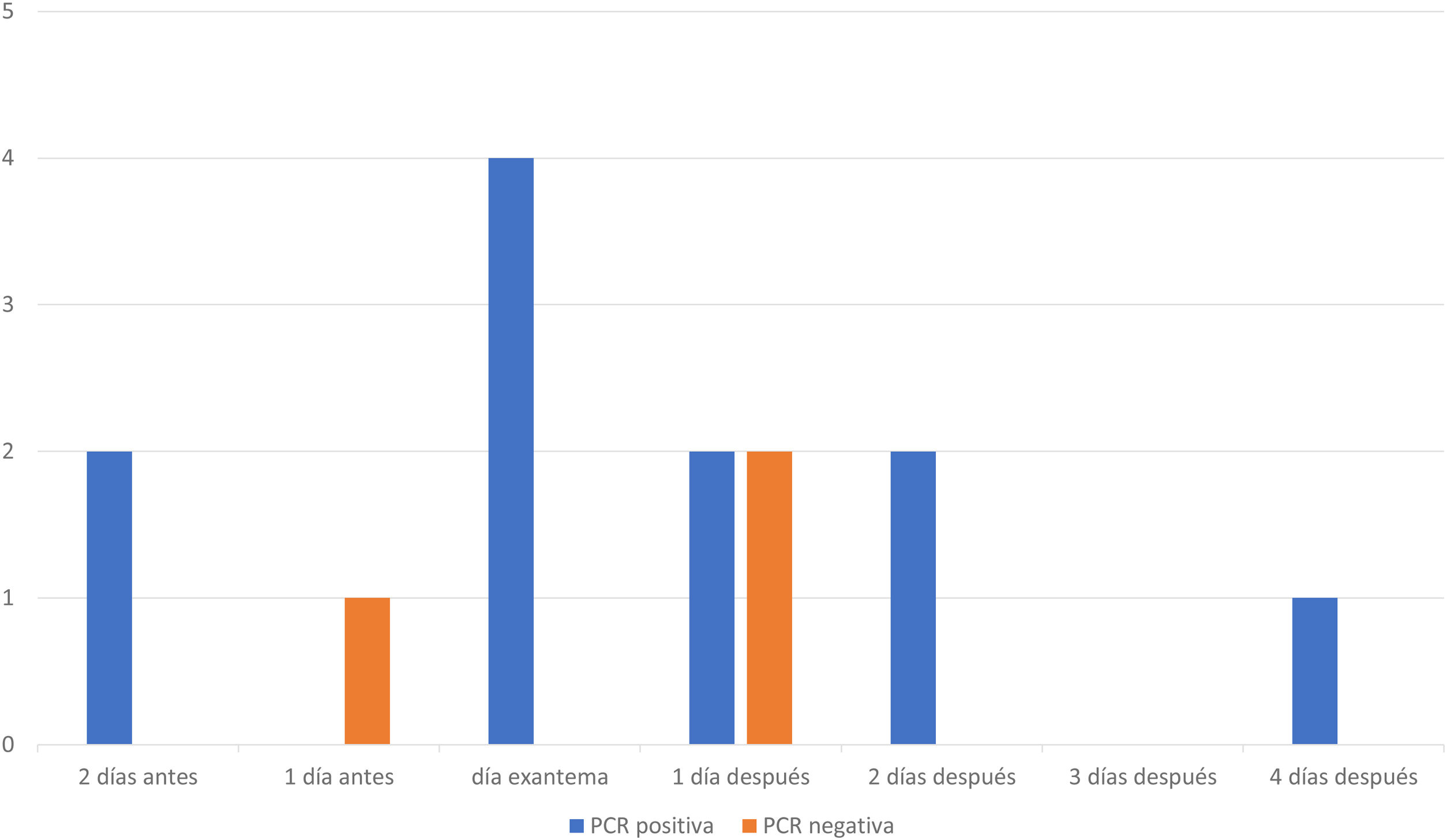
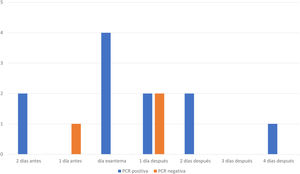
En 14 muestras de suero de 14 pacientes con IgM negativa en su muestra inicial y en las que posteriormente se confirmó el diagnóstico de sarampión se realizó PCR de suero. En 11 casos fue positivo. Estas muestras se recogieron entre 2 días preexantema y 4 días postexantema. Las otras 3 PCR de suero fueron negativas (fig. 1). De las 3 muestras negativas, 2 pertenecían a pacientes vacunados. En la tercera, aunque se desconocía el estado vacunal del paciente, presentaba IgG positiva e IgM negativa en el inicio del cuadro clínico.
El caso índice del brote en Guadalajara fue una mujer que acudió al hospital para visitar a un familiar, ocasionando casos secundarios en el centro sanitario. A partir de este caso, se consiguió establecer hasta 10 cadenas de transmisión, 4 de las cuales estaban relacionadas con centros sanitarios2.
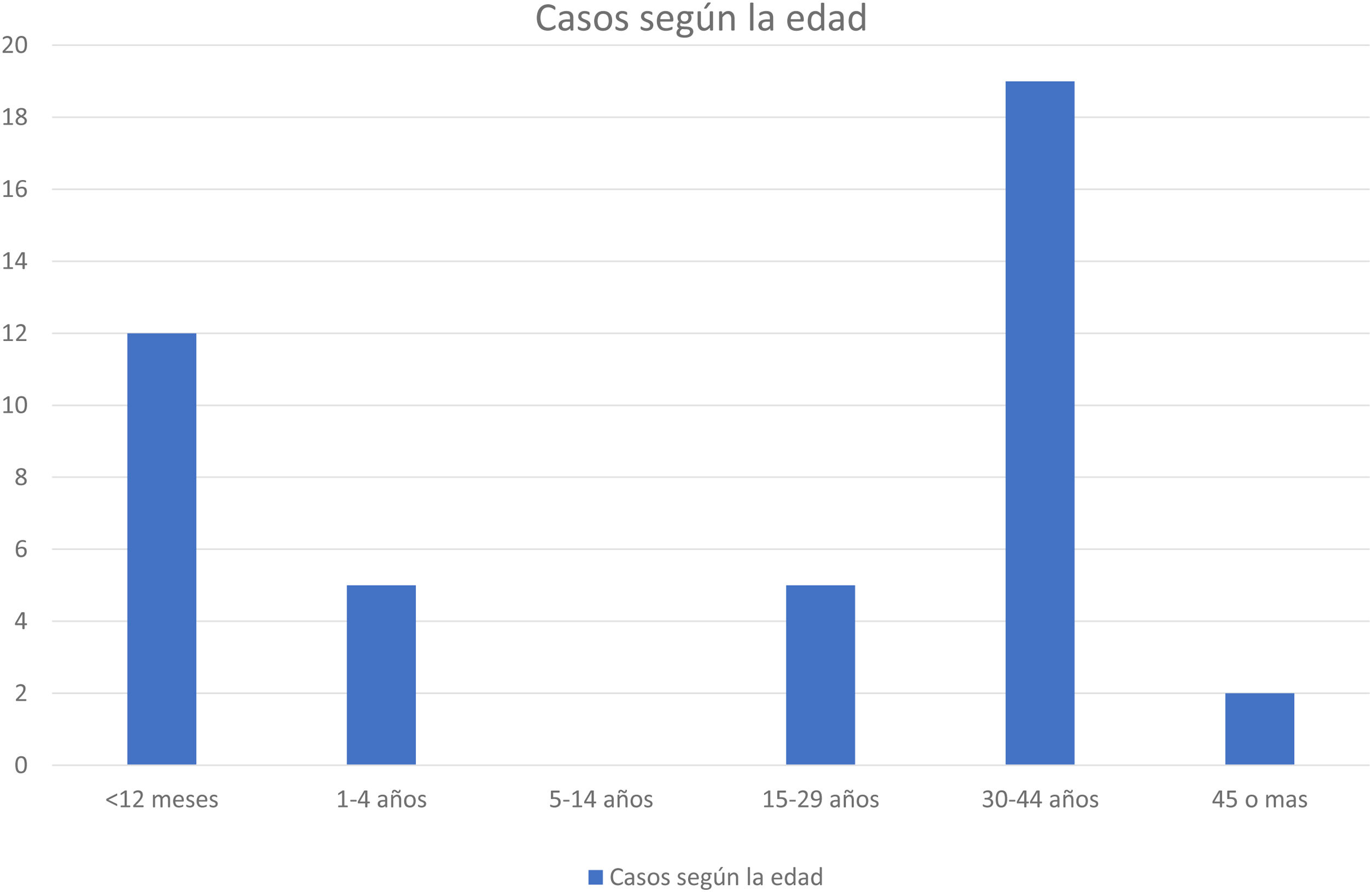
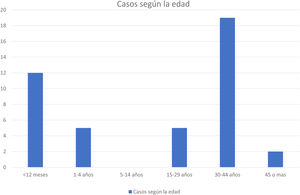
De los casos diagnosticados, 22 fueron mujeres y 21 hombres. En cuanto a la distribución por edad, se observaron 2 grupos: 26 adultos con edades comprendidas entre los 25 y los 53 años y 17 niños entre 4 meses y 3 años (fig. 2). Destaca el diagnóstico de 2 niños de 4 meses de edad, hermanos gemelos con IgG negativa al inicio del cuadro en ambos casos, y cuya madre también fue diagnosticada de sarampión. En 11 pacientes hay constancia de haber recibido alguna dosis de la vacuna, en 9 de haber recibido una dosis y en 2, de 2 dosis. En 3 pacientes vacunados el perfil serológico de la primera muestra recibida fue IgG positiva e IgM negativa (tabla 2). El diagnóstico se realizó en todos ellos mediante PCR de exudado de orofaringe.
Casos de pacientes vacunados diagnosticados de sarampión
| Caso | Edad | N.o de dosis | IgM | IgG inicio cuadro | Incremento IgGa | PCR faríngea | Genotipo |
|---|---|---|---|---|---|---|---|
| 1 | 34 a | 1 | Pos | Neg | NR | Pos | D8 |
| 2 | 34 a | 1 | Neg | Pos | NR | Pos | D8 |
| 3 | 25 a | 1 | Pos | Pos | Sí | Pos | D8 |
| 4 | 28 a | 2 | Neg | Pos | NR | Pos | D8 |
| 5 | 3 a | 1 | Pos | Pos | NR | Pos | D8 |
| 6 | 3 a | 1 | Pos | Pos | NR | Pos | D8 |
| 7 | 13 m | 1 | Neg | Neg | NR | Pos | D8 |
| 8 | 7 m | 1 | Neg | Neg | NR | Pos | D8 |
| 9 | 13 m | 1 | NR | NR | NR | Pos | D8 |
| 10 | 24 a | 2 | Neg | Pos | Sí | Pos | D8 |
| 11 | 36 a | 1 | Neg | Pos | NR | Pos | D8 |
a: años; m: meses; Neg: negativo; NR: no realizado; PCR: reacción en cadena de la polimerasa; Pos: positivo.
Se diagnosticaron las siguientes complicaciones: 13 casos de hepatitis aguda, 5 de diarrea, 2 de otitis y uno de queratitis. Los 13 pacientes con hepatitis presentaron un aumento importante de la enzima ALT (mayor del doble del nivel superior considerado normal). Esto afectó fundamentalmente a sujetos jóvenes no relacionados entre sí, 12 adultos (25-47 años, media: 36 años) y un niño. La afectación hepática fue el motivo de ingreso de todos los pacientes adultos.
Los síntomas más frecuentes de este grupo de hospitalizados fueron: el rash cutáneo (presente en el 100%), la fiebre (presente también en el 100%), las artromialgias (que aparecieron en el 77%) y las manchas de Koplik (objetivadas en un 54%).
En cuanto a las alteraciones analíticas, todos los pacientes adultos presentaron al ingreso, junto con el aumento de ALT, incremento también de las enzimas AST, GGT y LDH; en el caso del paciente pediátrico, no se objetivaron estas alteraciones durante el ingreso, sino en la analítica de control un mes después del alta hospitalaria.
Cinco de los pacientes con afectación hepática presentaron además elevación de la bilirrubina total. El 46%, conjuntamente, presentó ictericia con cifras de bilirrubina>1,2mg/dL. En todos estos pacientes se solicitó serología de virus hepatotropos (VHA, VHB, VHC, CMV y VEB), resultando negativa en todos los casos. Se descartó mediante anamnesis el diagnóstico de hepatitis tóxica. Con motivo de la alteración analítica del perfil hepático, se solicitó ecografía abdominal para completar el estudio, sin encontrar hallazgos relevantes en ninguna de las mismas.
Tras un periodo breve de estancia hospitalaria, en ningún caso superior a los 7 días, los pacientes evolucionaron de forma favorable mejorando tanto clínica como analíticamente, recibiendo tratamiento sintomático con antitérmicos y sueroterapia. Ningún paciente presentó complicación alguna que conllevase prolongar el ingreso o requiriese medidas intensivas, ni realizar ingreso en UCI.
DiscusiónEl sarampión es actualmente la causa más frecuente de muerte infantil por una infección que sin embargo es prevenible mediante vacunación. La mayoría de las muertes se producen en países en vías de desarrollo y como consecuencia de las complicaciones de la enfermedad1.
Esta situación ha llevado a la OMS a la realización de programas para conseguir su eliminación. Estos programas se basan en la vacunación correcta de la población, con unas coberturas de vacunación≥95%, así como la implantación de sistemas de vigilancia sensibles y específicos basados en la detección del caso. Dada su elevada contagiosidad, con una tasa de ataque secundario superior al 90%, el diagnóstico precoz es esencial.
El diagnóstico se basaba tradicionalmente en la clínica del paciente con el exantema característico, junto a fiebre elevada y conjuntivitis, coriza y/o tos y la aparición de IgM específica en suero. Sin embargo, toda sospecha clínica debe confirmarse con pruebas de laboratorio. La IgM suele tardar varios días en ser detectable (≥4 tras el exantema), por lo que conlleva un retraso en el diagnóstico7. Por otro lado, en países en situación de posteliminación con baja circulación del virus, el valor predictivo negativo de la IgM disminuye, puesto que los casos en vacunados aumentan y en estos puede no aparecer4,8, así como el valor predictivo positivo disminuye, aumentando los casos de falsos positivos de IgM9. En nuestro estudio, en 14 casos no se detectó IgM en la primera muestra de suero obtenida. Así mismo, una sanitaria vacunada y asintomática con contacto con un caso e IgG e IgM positivas fue descartada. Se ha descrito previamente que en algunas personas con contacto previo con el virus puede ocurrir una estimulación inmunológica antigénica tras un nuevo contacto, provocando la producción de IgM10.
El uso de técnicas de detección genómica como la PCR de muestras de orofaringe u orina proporciona un diagnóstico de laboratorio anterior a la aparición de IgM. En nuestro estudio, 15 pacientes acudieron a un centro sanitario entre 48h antes y el mismo día de aparición del exantema. Esto se debió tanto a la presencia de síntomas inespecíficos como a la existencia de contactos diagnosticados en el contexto de un brote epidémico. En el caso del suero, si bien no es una muestra óptima para la detección de ARN vírico, se ha descrito que se puede detectar en los primeros días de la enfermedad, siendo habitualmente negativo cuando aparecen las IgG11. Su utilidad fundamental sería en pacientes de corta evolución en los que, excepcionalmente, no dispongamos de otras muestras (exudado de orofaringe u orina) para PCR y que no hayan recibido la vacuna. En un estudio, la mayor rentabilidad se encontró en las primeras 72h tras la aparición del exantema12. Sin embargo, la obtención de exudado faríngeo, que puede completarse con orina, es imprescindible en todos los casos sospechosos de sarampión para asegurar un adecuado rendimiento diagnóstico13,14.
En el presente estudio, así como en otros anteriores3,15,16, se describen casos de sarampión en pacientes vacunados. Esto puede deberse tanto a un fallo vacunal primario como secundario. Para diferenciar ambas situaciones sería necesario un estudio serológico postvacunal. Sin embargo, la presencia de IgG con ausencia de IgM en el momento del diagnóstico sugiere un fallo vacunal secundario17. Se considera que los pacientes vacunados son menos contagiosos, pero pueden ocasionar brotes18. En estos pacientes, el diagnóstico es más complejo debido a que la clínica es más leve y por la dificultad en la interpretación serológica con IgM ausente junto con una menor excreción viral17. Esto refuerza la necesidad de tomar un grupo de muestras completo en todos los casos sospechosos de sarampión. Además, en la vigilancia de los brotes debemos tener en cuenta la existencia de estos casos en pacientes vacunados, y no descartar casos por la presencia de IgG en el inicio del cuadro clínico junto con ausencia de IgM en muestras recogidas de manera precoz. En estos casos es fundamental el diagnóstico molecular.
El estudio de avidez de IgG en estos pacientes es una herramienta que puede complementar al resto de las herramientas diagnósticas y permite caracterizar el fallo vacunal19.
El análisis de los genotipos detectados en un brote aporta información local, permitiendo descartar casos que son reacciones vacunales4,20, como los 2 niños de 12 meses que recibieron la primera dosis en nuestro estudio. Por otra parte, la vigilancia molecular posibilita relacionar los casos con otros que puedan concurrir temporalmente en otras localizaciones y proporciona información de los genotipos predominantes a nivel global4,7,21–23. En nuestro caso, la variante identificada del genotipo D8 (MVs/GirSomnath.IND/42.16/-variant) circulaba de forma predominante en Europa. En España, a lo largo del 2018 y el 2019 se han identificado diversos casos esporádicos de esta variante, originaria de la India e importada al resto de Europa principalmente desde Ucrania. En 2019 se ha detectado esta variante en los brotes de Valencia, que comenzó a finales de 2018, Barcelona, Tenerife y Madrid.
El brote descrito en este estudio presenta las características de los brotes que tienen lugar en un país en fase de posteliminación, afectando a una franja etaria de adultos jóvenes al inicio del brote y posteriormente a niños no vacunados o con una dosis de vacuna. Dentro del grupo de adultos hay 2 subgrupos: uno mayoritario, de personas no vacunadas o con vacunación incompleta, ya que desde la introducción de la vacuna triple vírica en 1981 se tardaron algunos años en conseguir coberturas altas, y como la circulación del virus se redujo notablemente, tampoco adquirieron la infección de forma natural; y otro grupo de vacunados correctamente que presentan fallo vacunal, probablemente por un fenómeno de evanescencia con una disminución con el tiempo de la protección debida a la vacuna24. Este dato ha sido ya comunicado en Holanda en personas nacidas entre los años 1975 y 198525. La baja incidencia de la enfermedad hace que no exista un efecto booster en los vacunados, incrementando la población susceptible en el tiempo.
Además de los síntomas característicos de sarampión, en ocasiones se observan cuadros de neumonía o hepatitis, esta última fundamentalmente en adultos26,27. En el brote ocurrido en el área sanitaria de Guadalajara llama la atención la afectación hepática como síntoma de la enfermedad. En 13 pacientes se observó un aumento de ALT. La hepatitis asociada a sarampión suele detectarse como un aumento transitorio de transaminasas durante la fase aguda de la enfermedad. La afectación hepática asociada al sarampión puede presentar 2 patrones: por un lado, daño hepatocelular caracterizado por un aumento de los niveles de transaminasas que es un reflejo de efecto citopático por acción directa del virus28; y por otro, un segundo patrón colestásico tardío como consecuencia de la acción del sistema inmunológico del paciente29.
Ante cualquier sospecha de sarampión es necesaria la notificación urgente a las autoridades sanitarias de estos casos y la posterior confirmación de los mismos4,7. Para ello, deben recogerse muestras de orofaringe, orina y suero para estudio molecular y serológico y conocer la relación temporal de la obtención de la muestra con el cuadro clínico. La recogida de las muestras para el estudio molecular debe ser lo más temprana posible, mientras que para el estudio serológico puede ser necesaria una segunda muestra de suero30. Esto es aún más importante en los casos de personas vacunadas y adultas.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.