Introducción
Aspergillus fumigatus es un hongo oportunista que causa infecciones invasoras graves en personas inmunodeprimidas. El diagnóstico precoz de la aspergilosis invasora es fundamental, ya que permite adelantar el inicio del tratamiento antifúngico, del que suele depender la supervivencia del enfermo 1. No obstante, actualmente existen dudas sobre cuáles son las técnicas diagnósticas y las terapéuticas más adecuadas para controlar esta micosis. Los ensayos clínicos necesarios para determinar estos aspectos son de difícil diseño, financiación y realización, por lo que deben buscarse métodos alternativos.
Una de estas alternativas es el desarrollo de modelos animales que permiten reproducir infecciones humanas como la aspergilosis invasora. Los modelos experimentales de infección se han utilizado y se utilizan hoy en día para desarrollar nuevas técnicas diagnósticas y para evaluar tratamientos novedosos, como las nuevas moléculas antifúngicas y la terapia combinada 2. Los resultados obtenidos en los modelos experimentales tienen sus limitaciones, pero permiten llegar a conclusiones que pueden considerarse más cercanas a la práctica clínica que aquellas obtenidas en experimentos in vitro 3.
Se han desarrollado varios modelos animales de aspergilosis invasora. En la mayoría de ellos se han empleado roedores inmunodeprimidos a los que se les administra conidias del hongo por vía intranasal, en un intento de reproducir la cadena de infección de la aspergilosis, es decir, inhalación, infección pulmonar y posterior diseminación. Mediante estos modelos de infección se han evaluado técnicas diagnósticas y diversos tratamientos 4-6. En todos ellos se ha necesitado un método de cuantificación de la infección por Aspergillus, que permitiera evaluar la respuesta a la terapia o la eficacia de una técnica diagnóstica.
El método de cuantificación más empleado es el recuento de colonias (unidades formadoras de colonias, UFC) por gramo de tejido cultivado. Sin embargo, el recuento de colonias puede verse alterado por la presencia de hifas en los tejidos que al fragmentarse, pueden aumentar los recuentos o, en otras ocasiones, rendir un número escaso de colonias. Además, el recuento también puede alterarse por la supervivencia en el tracto respiratorio de conidias no germinadas, pertenecientes al inóculo administrado al animal 2. Por eso, se han desarrollado otras técnicas de cuantificación basadas en tinciones histológicas y en la detección de la biomasa fúngica, como la cuantificación de quitina, ergosterol o antígenos fúngicos 7-10. Estas técnicas son laboriosas y, en muchas ocasiones, poco reproducibles.
En los últimos años, se han aplicado algunos métodos de cuantificación basados en la detección de ácidos nucleicos, demostrando una sensibilidad y una especificidad variables 8,11,12. Uno de estos métodos es la reacción en cadena de la polimerasa (PCR) en tiempo real, que permite cuantificar los ácidos nucleicos amplificados y que, por tanto, puede emplearse en modelos animales de infección para evaluar la carga fúngica en los tejidos infectados, constituyendo una alternativa a las técnicas de cuantificación mencionadas anteriormente.
El objetivo de este trabajo es la valoración de la utilidad de una técnica basada en la PCR en tiempo real para la detección y cuantificación de ADN fúngico, en un modelo animal murino de infección pulmonar por A. fumigatus.
Métodos
Modelo murino deaspergilosisinvasora
Se realizó un modelo estandarizado siguiendo el descrito por Smith et al 13. Se utilizaron ratones machos de 6-7 semanas, ICR, libres de patógenos, con un peso medio de 30 g (Criffa, Barcelona, España). Se emplearon 30 ratones que se distribuyeron en grupos de cinco ratones por jaula. El cálculo del tamaño muestral se efectuó con la aplicación STATCAL del programa EPI INFO 2002 (Centres for Diseases Control and Prevention, Atlanta, EE.UU.). Con un intervalo de confianza del 95% y con una potencia estadística 1-beta del 80%, se necesitaban 30 ratones, para que los resultados tuvieran peso estadístico. Cada animal se identificó individualmente para facilitar el seguimiento del experimento. Para la estabulación de los animales se utilizaron jaulas en estanterías especiales de aluminio, conectadas con filtros HEPA y un sistema de ventilación positiva (Tecniplast, Barcelona, España). Se utilizó para las jaulas un lecho de viruta irradiada (Lignocel, Harlan Interfauna, Barcelona, España) y los animales recibieron pienso estéril de mantenimiento (TRT, Harlan Interfauna) y agua estéril complementada con 1 mg/ml de tetraciclina (Sigma-Aldrich, Madrid, España), para evitar la contaminación bacteriana. Se cumplió estrictamente la normativa recogida en el real Decreto 223/1988 sobre protección de los animales de experimentación.
La tabla 1 incluye un resumen de los grupos de animales incluidos en el estudio. Se emplearon un grupo control que no recibió inóculo ni tratamiento inmunosupresor, y otro grupo control de la inmunosupresión que recibió terapia inmunosupresora y que no fue inoculado con conidias. Los cuatro grupos restantes fueron inoculados con conidias de una cepa clínica de A. fumigatus (CNM-CM-237, Colección de Hongos Miceliales del Centro Nacional de Microbiología). La cepa se subcultivó en tubos de agar patata glucosa (APD, Oxoid, Madrid, España), a 37 °C durante 3-5 días. Las conidias se recogieron con solución salina (0,85%) estéril complementada con Tween (0,1% v/v, Sigma-Aldrich), lavadas y filtradas (filtros de 11 μ, Millipore, Madrid, España) para evitar la formación de cúmulos. Tras esto, se prepararon las suspensiones de inoculación mediante recuento en cámara hematocitométrica de Neubauer (Brand, Merck, Madrid, España). Se realizaron recuentos de colonias en agar Sabouraud (Oxoid) para evaluar la fiabilidad del procedimiento de preparación del inóculo. Cada grupo experimental fue inoculado con un número de conidias diferente, 10 3, 10 4, 10 5 y 10 6/ml.
La inmunodepresión de los animales se llevó a cabo con ciclofosfamida (Genoxal. Prasfarma, Barcelona, España) (200 mg/kg) y acetato de cortisona (Sigma-Aldrich) (112,5 mg/kg). Los ratones recibieron cinco tandas de inmunodepresión, 3 y 1 día antes de la inoculación y 3, 6 y 9 días después de la inhalación de las conidias (días 3, 1, +3, +6 y +9).
El inóculo se administró intranasalmente. Los grupos experimentales fueron etiquetados como G-10 6, G-10 5, G-10 4, G-10 3, según el tamaño del inóculo recibido (tabla 1). Los animales fueron anestesiados mediante inyección intramuscular de 100 ml de una mezcla de ketamina 12,5 mg/ml (Ketolar, Parke-Davis, Madrid, España) y xylazina 2,3 mg/ml (Rompun, Bayer, Madrid, España). En este estado, los ratones fueron inoculados por vía intranasal con 30 ml de la solución de esporas previamente preparada.
Tras la inoculación, los animales fueron observados diariamente, en busca de los síntomas propios de una aspergilosis pulmonar: inmovilidad, letargo y disnea. Cuando se observó alguno de estos signos, los animales se sacrificaron mediante dislocación cervical. Los ratones sin signos de infección fueron sacrificados al décimo día de observación, considerado como el punto final de los experimentos. La disección de los animales se realizó en condiciones asépticas. Se extrajeron los pulmones, se pesaron y resuspendieron en 0,5 ml de suero salino estéril. Los pulmones fueron triturados y homogeneizados manualmente, tras lo que se separó una alícuota de 50 μl, que se sembró en agar Sabouraud-cloranfenicol (Oxoid). Las placas se incubaron a 35 °C durante 24-48 h y se contó el número de colonias cuando éstas fueron visibles. Los resultados obtenidos se expresaron en UFC por gramo de tejido analizado. Las colonias recuperadas en placa se analizaron mediante Southern-Blot, siguiendo los protocolos habituales 14 para verificar que se trataba de la cepa inoculada y no de una contaminación. El resto del homogeneizado se reservó para realizar un posterior ensayo de detección de ADN fúngico mediante PCR en tiempo real.
Detección de ADN fúngico mediante PCR en tiempo real
Para la realización de las técnicas moleculares se utilizaron 20 mg de los homogeneizados pulmonares. Para el proceso de extracción se empleó el sistema Wizard SV Genomic DNA Purification System (Promega, Innogenetics, Madrid, España), siguiendo las instrucciones del fabricante. Las reacciones de PCR en tiempo real se realizaron en el termociclador LightCycler 2.0 System (Roche Aplied Science, Madrid, España), siguiendo el método descrito por Loeffler et al 15. Este método se basa en la utilización de dos sondas FRET (Fluorescence Resonance Energy Transfer) específicas marcadas con fluoresceína y RED640, y dos iniciadores para la amplificación de una región que codifica el fragmento 18S del ADN ribosómico de Aspergillus spp.16. Esta sonda es específica para A. fumigatus, aunque se han descrito algunas reacciones cruzadas con otras especies de Aspergillus15. Las reacciones de PCR en tiempo real se realizaron empleando el sistema LightCycler-FastStart DNA hibridization probes (Roche Aplied Science), en un volumen final de 20 μl y empleando 3 mM de Cl2Mg, 1 μM de cada iniciador, 0,2 μM de cada sonda FRET, y 10 μl del ADN obtenido. En la tabla 2 se muestran las secuencias de los iniciadores y de las sondas FRET utilizadas en los experimentos.
La cuantificación del ADN amplificado se realizó mediante la construcción de una recta patrón, en las que se emplearon cantidades conocidas de ADN de la cepa inoculada, en cantidades decrecientes (100 ng hasta 10 fg). A estas muestras se les realizó una PCR en tiempo real, obteniéndose la amplificación de las mismas en distintos ciclos de la reacción (crossing point). Tras ello se realizó una regresión lineal en la que se utilizaron los logaritmos de las concentraciones de ADN utilizadas y el crossing point. La cantidad de ADN amplificado en las muestras procedentes de los animales se calculó mediante una regresión lineal. Una vez realizadas las reacciones de PCR se comprobó la presencia del amplicón que se quería detectar, así como el tamaño de éste, mediante electroforesis en gel de agarosa al 0,8% (Sigma-Aldrich) con TAE (Tris acetate-EDTA) siguiendo los protocolos habituales 14. Asimismo se procedió a secuenciar el fragmento amplificado para verificar la presencia de A. fumigatus. Para las reacciones de secuenciación se utilizaron 4 μl de un sistema de secuenciación (BigDye Terminator Cycle Sequencing Ready Reaction, Applied Biosystem, Madrid, España), 1 μM de los iniciadores utilizados en la reacción de PCR y 5 μl del producto final de la amplificación. Las secuencias fueron ensambladas y comparadas empleando los programas SeqMan II y EditsEq (DNAstar, Inc. Lasergene, Madison, EE.UU.).
Análisis estadístico
El análisis de los resultados se realizó con la ayuda del programa informático SPSS 12.0 (SPSS SL, Madrid, España). Se efectuó un análisis descriptivo de los días de supervivencia de cada grupo experimental, así como de los recuentos de colonias por gramo de tejido pulmonar y de los resultados de la PCR en tiempo real.
Los días de supervivencia fueron tomados como la variable principal para realizar las comparaciones y los análisis de regresión. En primer lugar, se analizaron las diferencias en el tiempo de supervivencia por grupo experimental, mediante tablas de supervivencia y el test de Kaplan-Meier, previa comprobación de la normalidad de los resultados. Tras ello, se realizó un estudio de correlación entre la supervivencia, los recuentos de colonias y los datos de la PCR en tiempo real mediante coeficientes de correlación de Pearson (r), previa conversión logarítmica de las UFC por gramo de tejido obtenidas. Las diferencias entre grupos en la cuantificación del ADN fúngico se calcularon mediante análisis de la varianza con el test Post hoc de Tukey. El nivel de significación estadística que se aceptó fue p < 0,01.
Resultados
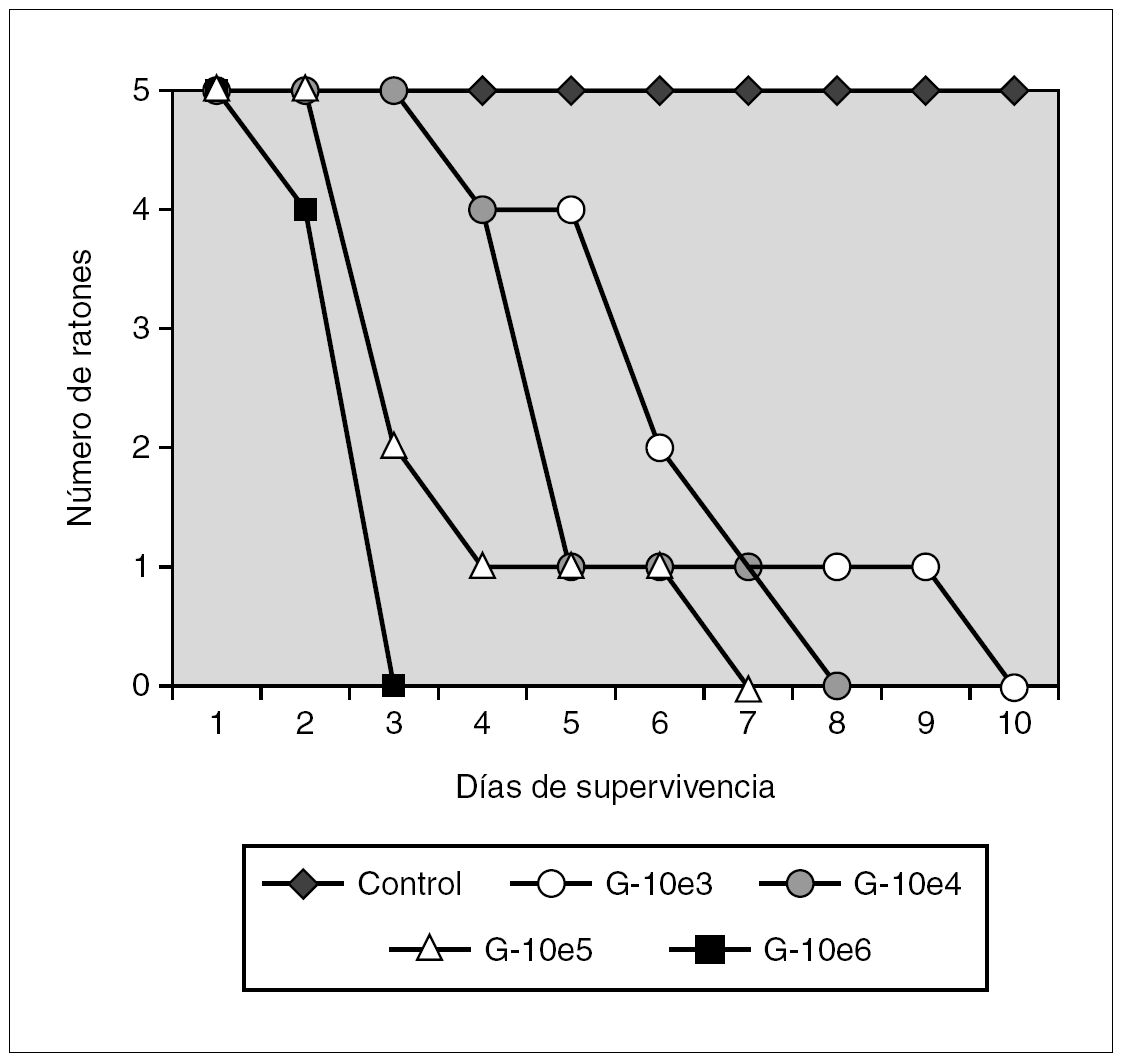
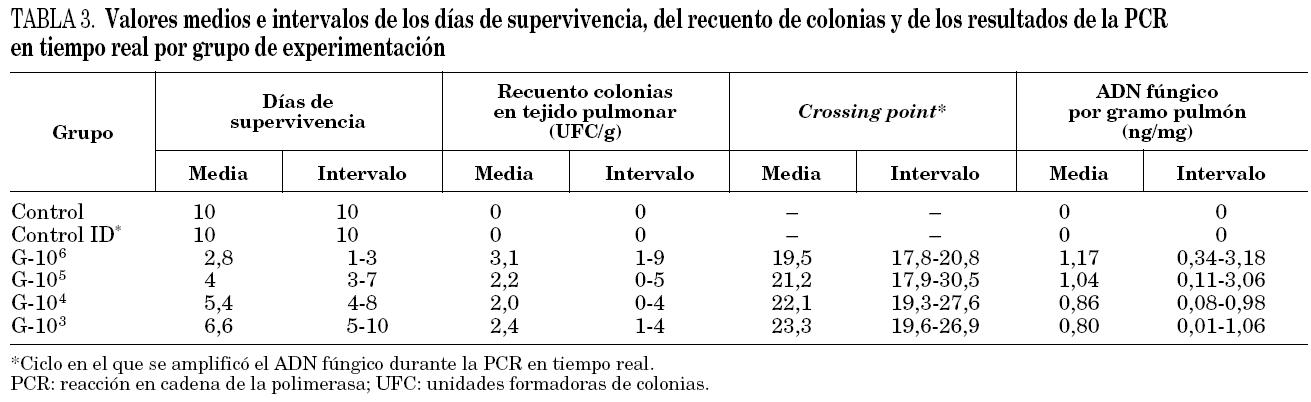
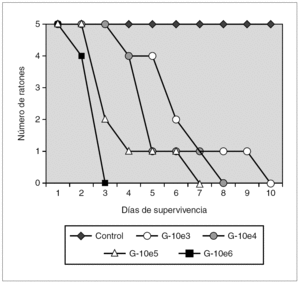
En la figura 1 se representan las curvas de supervivencia de los grupos experimentales incluidos en el modelo murino de aspergilosis pulmonar. En la figura se representa el número acumulado de animales sacrificados, por día de experimentación. En los grupos que recibieron los inóculos más elevados, 10 6 y 10 5 conidias/ml, la mayoría de los ratones presentaba signos de enfermedad al tercer o cuarto día de experimentación. Mientras que con los otros dos inóculos, los animales empezaron a ser sacrificados el cuarto día. Estas diferencias resultaron estadísticamente significativas (p < 0,01) cuando se analizaron en tablas de supervivencia y con el test de Kaplan-Meier. Las medias de los días de supervivencia por grupo de experimentación se resumen en la tabla 3, destacando la diferencia entre la supervivencia media de los animales inoculados con 10 6 conidias/ml (2,8 días) y la de los animales inoculados con 10 3 conidias/ml (6,6 días). Por lo tanto, el tiempo medio de supervivencia tuvo una relación inversa y estadísticamente significativa con el tamaño del inóculo empleado.
Figura 1. Curvas de supervivencia por grupo de experimentación. G-10e6: grupo de ratones inoculados con 10 6 conidias/ml; G-10e5: inoculados con 10 5 conidias/ml; G-10e4: inoculados con 10 4 conidias/ml; G-10e3: inoculados con 10 3 conidias/ml; Control: grupo control sin inmunodepresión.
Una vez halladas estas diferencias significativas en la supervivencia, se calcularon las CFU por gramo de tejido pulmonar y se realizaron las pruebas con la técnica de PCR en tiempo real, con la intención de correlacionar los resultados de la supervivencia con ambas técnicas de cuantificación de la infección por Aspergillus. En primer lugar debe indicarse que el recuento de colonias fue positivo en el 90% de los animales inoculados. En dos ratones, uno del grupo G-10 5 y otro del G-10 4, no se detectó la infección mediante el cultivo del tejido pulmonar. Además, la correlación entre los días de supervivencia y el recuento de colonias por gramo de tejido pulmonar no fue estadísticamente significativa (r = 0,123; p > 0,01). En la tabla 3 se muestran los valores de los recuentos por gramo de tejido pulmonar cultivado, clasificado por grupo experimental.
Los resultados obtenidos con la PCR en tiempo real también se resumen en la tabla 3. La técnica de PCR fue positiva en el 100% de los ratones incluidos en el modelo. Además, los valores de crossing point y la cantidad amplificada de ADN fúngico (ng/mg de tejido pulmonar) se correlacionaron significativamente con los días de supervivencia de los animales. El coeficiente de correlación entre el crossing point y los días de supervivencia fue de 0,66 (p < 0,01), y entre la cantidad de ADN fúngico y el tiempo de supervivencia de 0,81 (p < 0,01), indicando una relación inversa y significativa entre ambas variables. Además, las diferencias halladas en los valores del crossing point y del ADN fúngico fueron estadísticamente significativa (p < 0,01) entre el grupo G-10 6 y el resto de grupos con animales inoculados. Las diferencias entre los otros tres grupos (G-10 5, G-10 4 y G-10 3) no fueron significativas mediante el test de Tukey.
Discusión
Una de las principales limitaciones que siempre han mostrado los modelos animales es la dificultad para evaluar o cuantificar la infección fúngica por hongos filamentosos. Estos microorganismos tienen la capacidad de generar hifas en los tejidos que invaden, por lo que la cuantificación de la infección es compleja. La técnica de referencia, que es el recuento de colonias por gramo de tejido cultivado 17, no puede utilizarse con fiabilidad, ya que los filamentos pueden fragmentarse, lo que hace que se sobreestime el número de colonias, con el consiguiente error en estudios de valoración de antifúngicos o de virulencia de las cepas. Por lo tanto, la mayoría de los estudios con modelos animales han empleado la supervivencia como variable principal 2. Sin embargo, las actuales recomendaciones sobre la disminución del sufrimiento de los animales de laboratorio y el desarrollo de los modelos discriminatorios, que evalúan varias variables en un mismo modelo e intentan reproducir la infección como ocurre en el ser humano, han hecho que se necesiten otras formas de analizar la infección fúngica en los animales 7-10.
Uno de estos métodos puede ser la PCR en tiempo real 15. Esta técnica es un tipo de PCR cuantitativa, basada en la medición de la fluorescencia emitida por el producto de la reacción de amplificación. Pueden utilizarse diferentes formas de marcaje fluorescente y la cuantificación se realiza determinando en qué ciclo se empieza a detectar la fluorescencia. En este estudio se realizó un modelo animal de aspergilosis pulmonar, en el que se administraron diferentes inóculos, con la intención de evaluar diferencias en el tiempo de supervivencia, en el recuento de colonias por gramo de tejido pulmonar cultivado y en la cantidad de ADN fúngico amplificado por PCR en tiempo real.
Nuestros resultados demuestran que el tiempo de supervivencia era menor cuanto mayor era el inóculo. Esta tendencia tuvo significación estadística, por lo que los días de supervivencia fueron tomados como variable de referencia para comparar los recuentos y los resultados de la PCR. La PCR en tiempo real resultó ser más sensible que los recuentos de colonias (100 frente a 90%), pero además, tanto los valores de crossing point como la cantidad de ADN amplificado por mg de tejido pulmonar, tuvieron una relación con los días de supervivencia de los animales. Estos resultados significan que a mayor inóculo, los animales vivían menos días de media, y que se detectaba una mayor cantidad de ADN fúngico. Esta relación no se obtuvo con el recuento de colonias, ya que los valores de esta variable no fueron dependientes del tamaño de inóculo empleado, lo que la invalida como método de cuantificación de la infección. En nuestro estudio pudo observarse además que la técnica es sensible y específica, ya que se detectó ADN fúngico en todos los ratones inoculados con la cepa clínica de Aspergillus y la amplificación fue negativa, en todos los controles sin inocular.
La PCR en tiempo real constituye un método fiable para cuantificar la infección pulmonar por A. fumigatus en este modelo murino. Es un método sencillo, rápido y reproducible por lo que puede emplearse en modelos que evalúen la eficacia de antifúngicos, en estudios que analicen la virulencia de determinadas especies o cepas y en trabajos que determinen la cadena de infección de las micosis, la posibilidad de realizar un diagnóstico precoz o la eficacia de la respuesta inmunitaria del huésped. Las técnicas de amplificación de ácidos nucleicos no tienen las limitaciones de los estudios de supervivencia y pueden sustituir a métodos complejos como la cuantificación de quitina, ergosterol u otros componentes fúngicos. La difusión de las técnicas de cuantificación de ácidos nucleicos, como la PCR en tiempo real, puede ayudar a la implantación del diagnóstico molecular en la práctica microbiológica asistencial, de la que se encuentra alejada en la actualidad. Estas técnicas están automatizadas, parcial o totalmente, y aportan sistemas comerciales de extracción, purificación, amplificación y detección de ácidos nucleicos, por lo que existe la posibilidad de elaborar protocolos estandarizados, aplicables a los laboratorios clínicos, con fiabilidad metodológica y, quizá, rentabilidad diagnóstica.
Agradecimientos
Agradecemos la excelente asistencia técnica de Victoria Ruiz Gutiérrez y el apoyo en la realización de este trabajo de la red temática de investigación RESITRA (Red de Estudio de la Infección en el Trasplante) financiada por el Fondo de investigaciones Sanitarias del Instituto de Salud Carlos III. Alicia Gómez-López disfruta de un contrato de investigación de la red RESITRA asociado al proyecto FIS G03/075. Emilia Mellado tiene un contrato del programa Ramón y Cajal del Ministerio de Educación y Ciencia.