La carga viral es un marcador muy útil para realizar el seguimiento de los pacientes infectados por VHB y VHC. Este trabajo compara ensayos basados en amplificación mediada por transcripción y en PCR a tiempo real con el objetivo de comprobar si pueden ser intercambiables.
Material y métodosEstudio bicéntrico en el que se analizó la carga viral de 147 muestras de plasma de pacientes infectados por VHB y 229 por VHC, mediante ensayos basados en amplificación mediada por transcripción (Aptima® HBV Quant y Aptima® HCV Quant Dx, que utilizan el sistema Panther (Hologic®)) y PCR a tiempo real (COBAS® AmpliPrep / COBAS® TaqMan® y COBAS® 6800), calculando el grado de concordancia entre ellos.
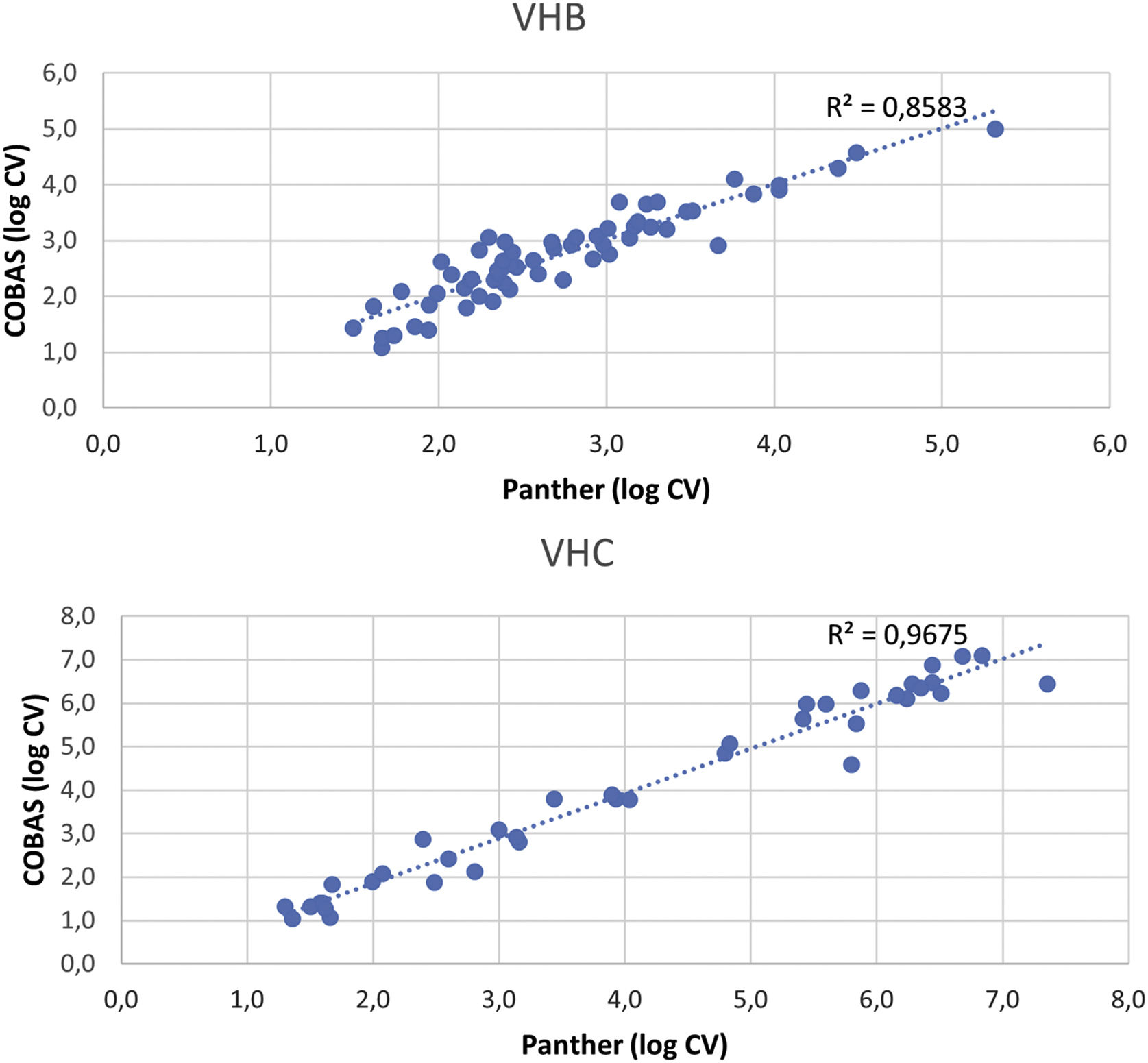
ResultadosSe detectó carga viral en ambos equipos en 60 (40,82%) muestras de VHB (mediana del log de la carga viral: COBAS®: 2,51UI/mL (RIC 2,20-3,17), Panther: 2,71UI/mL (RIC 2,21-3,22)) y en 39 (16,96%) muestras de VHC (mediana del log de la carga viral: COBAS®: 3,93UI/mL (RIC 2,24-6,01), Panther: 3,80UI/mL (RIC 1,99-6,14)). La concordancia entre ambos equipos fue de κ=0,943 para VHB y κ=0,925 para VHC. La comparación de las muestras con carga viral detectada mediante los 2 ensayos mostró una correlación alta tanto para VHB (R2=0,86) como para VHC (R2=0,97).
ConclusionesLos ensayos basados tanto en amplificación mediada por transcripción como en PCR a tiempo real pueden ser intercambiables para el manejo de pacientes infectados con VHB y VHC.
Viral load is a very useful marker for monitoring patients infected with HBV and HCV. This work compares assays based on transcription-mediated amplification and on real-time PCR to verify whether they can be interchangeable.
Material and methodsa bicentric study, in which 147 plasma samples from patients infected with HBV and 229 with HCV were analyzed, was carried out. Transcription-mediated amplification-based assays (Aptima® HBV Quant and Aptima® HCV Quant Dx, employing Panther system (Hologic®)) and on real-time PCR (COBAS® AmpliPrep / COBAS® TaqMan® and COBAS® 6800) were used and the degree of concordance between them was calculated.
ResultsViral load was detected in both systems in 60 (40.82%) HBV samples (median log viral load: COBAS®: 2.51IU/mL (IQR 2.20-3.17), Panther: 2.71IU/mL (IQR 2.21-3.22)) and in 39 (16.96%) HCV samples (median log viral load: COBAS®: 3.93IU/mL (IQR 2.24-6.01), Panther: 3.80IU/mL (IQR 1.99-6.14)). The agreement between both systems was κ=0.943 for HBV and κ=0.925 for HCV. Comparison of viral load samples detected by both assays showed a hight correlation for HBV (R2=0.86) and for HCV (R2=0.97).
ConclusionsBoth transcription-mediated amplification and on real-time PCR based assays may be interchangeable for the management of patients infected with HBV and HCV.
Las infecciones por los virus de la hepatitis B (VHB) y de la hepatitis C (VHC) suponen una preocupación sanitaria importante en todo el mundo. Por lo tanto, resulta de gran relevancia la identificación de las personas infectadas por estos virus, así como conocer la evolución de su enfermedad.
Según la Organización Mundial de la Salud, la prevalencia del VHB representa el 3,5% de la población mundial, variando en función de la región1. La Organización Mundial de la Salud también afirma que 820.000 defunciones fueron debidas a la enfermedad por este virus en 20192.
Cerca de 300.000 personas fallecieron en 2019 debido a la enfermedad causada por el VHC3. La prevalencia global de esta infección es de aproximadamente un 3%, si bien en nuestro medio se estima en torno a un 2-2,5%4. La hepatitis crónica por VHC es la principal causa de cirrosis hepática y de trasplante hepático4.
La carga viral es un indicador directo de la replicación viral y su cuantificación es primordial para el manejo de los pacientes infectados por estos virus. Este marcador es útil en el diagnóstico de la infección, en la progresión de la enfermedad, en la monitorización del tratamiento, en la toma de decisiones cuando este necesita modificarse, así como en la evaluación de la infectividad de los pacientes infectados5–8. Además, con respecto a los pacientes infectados por VHC, las guías de práctica clínica recomiendan conocer la carga viral al inicio del tratamiento y a las 12 semanas tras finalizarlo para poder confirmar la respuesta viral sostenida9. Por todo ello, se necesitan pruebas sensibles, específicas, seguras, precisas, reproducibles, automatizables y con un amplio rango dinámico de cuantificación que permita la interpretación fiable de los resultados de carga viral5–8.
En la actualidad existen diferentes ensayos moleculares a través de sistemas comerciales automatizados disponibles para cuantificar el material genético de estos virus en plasma o suero. Todos ellos son sistemas que detectan y cuantifican ADN de VHB y ARN de VHC, permitiendo monitorizar y diagnosticar la infección.
Los ensayos Aptima® HBV Quant y Aptima® HCV Quant Dx utilizan el sistema Panther (Hologic®) basado en amplificación mediada por transcripción (TMA). Por otro lado, los equipos de Roche COBAS® AmpliPrep / COBAS® TaqMan® y COBAS® 6800 (Roche Molecular Systems, Inc.) emplean una amplificación basada en PCR a tiempo real.
La sensibilidad analítica (LoD≥95%) de los ensayos de Aptima® es mayor comparada con los de COBAS® para la determinación de VHB y VHC: para VHB estos valores son de 5,58UI/mL para los ensayos Aptima® y 20,0UI/mL y 7,6UI/ml para los de Roche. En el caso de VHC las sensibilidades analíticas son 3,9UI/mL y 15,0UI/ mL y 12,0UI/mL, respectivamente.
En este trabajo se comparó el rendimiento de 2 ensayos moleculares con distinto diseño (TMA vs. PCR a tiempo real) empleando los sistemas Aptima® Quant y Roche COBAS® AmpliPrep / COBAS® TaqMan® o Roche COBAS® 6800 tanto para la determinación de ADN de VHB como para ARN de VHC. Publicaciones previas muestran resultados de estudios comparativos entre ensayos cuantitativos de VHB y VHC individualmente10–14. El objetivo de este estudio es cuestionar la creencia de que estas determinaciones han de realizarse siempre con la misma técnica y el mismo equipo, y demostrar que los ensayos estudiados son, efectivamente, intercambiables.
Material y métodosMuestras clínicasEn este estudio se incluyeron muestras de plasma obtenidas de pacientes infectados por VHB y VHC recibidas en los Servicios de Microbiología y Parasitología de 2 hospitales españoles: el Complejo Hospitalario Universitario de Santiago de Compostela, A Coruña, y el Hospital Universitario San Cecilio de Granada.
Se analizaron un total de 147 muestras para determinación de ADN de VHB y 229 muestras para determinación de ARN de VHC. Las técnicas moleculares comparadas son TMA vs. PCR a tiempo real empleando los sistemas Aptima® Quant y Roche COBAS® AmpliPrep / COBAS® TaqMan® o Roche COBAS® 6800. Cada muestra se probó paralelamente en los ensayos de Aptima y Roche (en el Complejo Hospitalario Universitario de Santiago de Compostela el sistema evaluado fue Roche COBAS® AmpliPrep / COBAS® TaqMan® mientras que el Hospital Universitario San Cecilio empleó el Roche COBAS® 6800) siguiendo las instrucciones del fabricante, sin otro criterio de selección que el volumen de muestra disponible. El Complejo Hospitalario de Santiago de Compostela analizó 105 (71,43%) muestras de VHB y 161 (70,31%) de VHC, y el Hospital Universitario de San Cecilio 42 (28,57%) de VHB y 68 (29,69%) de VHC.
Método estadístico empleadoLa concordancia entre los resultados cualitativos (negativos, detectados pero por debajo del límite de cuantificación, cuantificados y por encima del límite superior de cuantificación) se evaluó calculando el grado de concordancia kappa (κ). Para las muestras clínicas cuantificadas por ambos ensayos, se determinó la mediana de la carga viral, el rango intercuartílico (RIC) y el cuadrado del coeficiente de correlación de Pearson (R2).
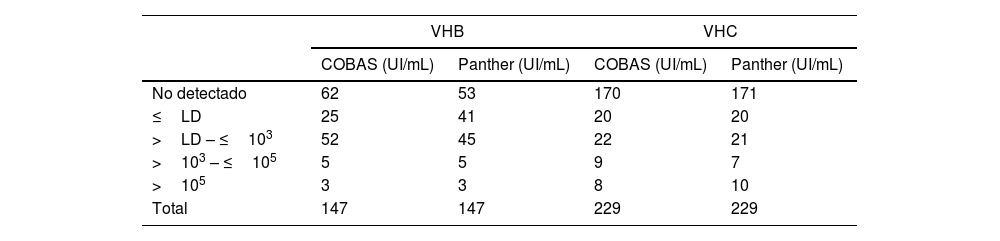
ResultadosEl número de muestras estratificadas por rango de UI/mL según su resultado puede observarse en la tabla 1.
De las 147 muestras de VHB analizadas, 87 fueron negativas o por debajo del límite detección de COBAS® y 60 (40,82%) detectaron carga viral de VHB en ambos equipos, siendo la mediana del log de la carga viral mediante COBAS® de 2,51UI/mL (RIC 2,20-3,17), y mediante Panther de 2,71UI/mL (RIC 2,21-3,22), no encontrándose diferencias significativas entre ambos valores (p=0,9343). La concordancia entre ambos equipos para el VHB medida mediante el índice de kappa fue de 0,943.
Con respecto a las determinaciones de VHC, de las 229 muestras analizadas, 190 fueron negativas o por debajo del límite de detección de COBAS y 39 (16,96%) tuvieron carga viral positiva en ambos equipos, siendo la mediana del log de la carga viral mediante COBAS® de 3,93UI/mL (RIC 2,24-6,01), y mediante Panther de 3,80UI/mL (RIC 1,99-6,14), no encontrándose diferencias significativas entre ambos valores (p=0,9764). La concordancia entre ambos equipos para el VHC medida mediante el índice de kappa fue de 0,925.
La comparación de las muestras con carga viral mediante los 2 ensayos (fig. 1) mostró una alta correlación tanto para VHB (R2=0.86) como para VHC (R2=0.97).
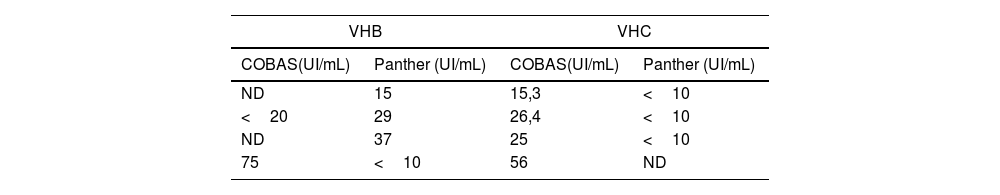
Con respecto a las muestras discordantes entre ambos equipos, se detectaron 4 (2,7%) en las correspondientes a la determinación de ADN de VHB, de las cuales 3 presentaron carga viral positiva mediante Panther y por debajo del límite de cuantificación o no detectable (ND) mediante COBAS®, mientras que una muestra presentó carga viral positiva mediante COBAS® e inferior al límite de cuantificación mediante Panther (tabla 2).
En el caso de las muestras para determinación de ARN de VHC, se detectaron 4 (1,7%) muestras discordantes con carga viral positiva en el sistema COBAS® y por debajo del límite de cuantificación o carga viral no detectable mediante el sistema Panther (tabla 2). No se detectaron muestras con carga viral positivas en Panther y negativas en COBAS® para VHC.
DiscusiónDada la gran importancia de detectar las infecciones por VHB y VHC, así como de conocer la carga viral de los pacientes infectados, son precisas técnicas que ofrezcan resultados fiables. En este trabajo se comparan 2 ensayos moleculares a través del rendimiento de métodos automatizados para la determinación de la carga viral de VHB y de VHC. Los ensayos comparados son TMA a través del sistema Aptima® Quant (Hologic®) y PCR a tiempo real, a través de los sistemas Roche COBAS® AmpliPrep / COBAS® TaqMan® y Roche COBAS® 6800. Las 2 técnicas son adecuadas para la detección cualitativa y la cuantificación de ADN de VHB y ARN de VHC en muestras clínicas de pacientes infectados. Los equipos empleados son altamente sensibles, detectan con precisión todos los genotipos de VHB y los de VHC y son completamente automatizados, con la ventaja que eso supone en el flujo de trabajo de un laboratorio. Además, el sistema Aptima® permite la posibilidad de carga continua de muestras, así como a su vez dar prioridad a las muestras urgentes. Otra ventaja de este sistema es la posibilidad de realizar más de una determinación al mismo tiempo a partir de la misma muestra, agilizando los resultados.
Los resultados obtenidos en este estudio muestran un alto nivel de correlación entre los sistemas cuando se determina tanto VHC como VHB, no mostrando diferencias significativas en situaciones de relevancia clínica. En consecuencia, se puede afirmar que estos ensayos son intercambiables, resultando apropiados en situaciones tales como la caracterización de la hepatitis o la infección crónica, así como en la monitorización del tratamiento en pacientes infectados por VHB o en el diagnóstico del VHC y la detección de la respuesta viral sostenida en pacientes infectados por VHC.
Estudios previos mostraron resultados similares a los obtenidos en este trabajo. Así, en el caso de muestras de pacientes infectados por VHB, Schønning et al. y Schalasta et al. demostraron que los ensayos Aptima® HBV Quant Assay y COBAS® Ampliprep / COBAS® TaqMan® HBV test v2.0 están altamente correlacionados y son comparables para la cuantificación de la mayoría de estas muestras empleando muestras de plasma en el primer caso y tanto plasma como suero en el segundo10,11. Con respecto a las muestras de pacientes infectados por VHC, los autores mencionados obtuvieron resultados comparables al cotejar las citadas técnicas, empleando igualmente muestras de plasma Schønning et al. y suero y plasma Schalasta et al.12,13. Worlock et al. lograron asimismo buenas correlaciones entre los ensayos previamente descritos y Abbott RealTime HCV Assay14.
La principal limitación de este estudio es el bajo número de muestras positivas analizadas por ambos métodos para estudiar cada carga viral. Aunque la correlación encontrada es buena, sería recomendable realizar un estudio con una cantidad superior de muestras para aumentar la potencia estadística del estudio.
Por lo tanto, en nuestra experiencia, los ensayos moleculares basados en TMA de Aptima® Quant (Hologic®) y en PCR a tiempo real de Roche COBAS® AmpliPrep / COBAS® TaqMan® y Roche COBAS® 6800 son métodos adecuados para el estudio molecular ADN del VHB y ARN del VHC en muestras clínicas de pacientes infectados, pudiendo ser intercambiables para el manejo de pacientes infectados con VHB y VHC.
FinanciaciónLa presente investigación no ha recibido ayudas específicas provenientes de agencias del sector público, sector comercial o entidades sin ánimo de lucro.
Conflicto de interesesNinguno.