A partir del 2008 se detectaron varios aislados de Staphylococcus hominis (S. hominis) multirresistentes, incluyendo resistencia al linezolid y a la teicoplanina, en pacientes ingresados en dos hospitales de Mallorca. Por ello, se inició un estudio para determinar la epidemiología molecular y el mecanismo de resistencia al linezolid.
MétodosEl estudio de epidemiología molecular se realizó mediante electroforesis en campo pulsado (ECP), tras digestión con ApaI. Se efectuó amplificación de un fragmento de los genes ARNr 23S (con secuenciación posterior) y cfr.
ResultadosDesde marzo de 2008 hasta febrero de 2009 se detectaron 15 aislados de S. hominis resistentes al linezolid y a la teicoplanina, procedentes de 14 pacientes. Todos ellos excepto uno habían ingresado en las Unidades de Cuidados Intensivos de alguno de los dos hospitales. La mayoría de los aislados (9) se obtuvieron en hemocultivos. Gran parte de los pacientes infectados (12 de los 15 episodios infecciosos, el 80,0%) recibieron pautas de linezolid antes de la detección del aislado resistente. La ECP reveló la presencia de un único clon entre los aislados de S. hominis resistentes al linezolid. Se detectó la mutación G2576T en todas las cepas resistentes, mientras que la PCR del gen cfr fue negativa en las mismas. Todos los aislados fueron también resistentes a la penicilina, oxacilina, trimetoprim-sulfametoxazol, ciprofloxacino, levofloxacino y tobramicina; y sensibles a la eritromicina, tetraciclina, gentamicina y daptomicina. La CMI a la vancomicina fue de 4μg/ml en todos ellos.
ConclusionesLa detección de cepas de estafilococos resistentes al linezolid resalta la necesidad de racionalizar el uso del linezolid y mantener un control activo de dicha resistencia con objeto de preservar la utilidad clínica de este antimicrobiano.
Since March 2008, several linezolid and teicoplanin-resistant Staphylococcus hominis (S. hominis) isolates have been recovered from patients admitted to the two major hospitals on the island of Majorca, Spain. For this reason, a study was conducted to determine the molecular epidemiology of these isolates and the mechanism of linezolid resistance.
MethodsThe molecular epidemiology study was performed by pulsed-field gel electrophoresis (PFGE) analysis, after digestion with ApaI. Linezolid resistance mechanisms were evaluated by PCR amplification of a fragment of the domain V of the 23S rRNA gene (followed by sequencing) and cfr gene.
ResultsFrom March 2008 to February 2009, 15 linezolid and teicoplanin-resistant S. hominis isolates were recovered from 14 patients. All of them, except one, were hospitalised in the intensive care units of either of the two institutions. Isolates were obtained mainly from blood cultures (9). The majority of infected patients (12 of 15 infectious episodes, 80.0%) had received courses of linezolid prior to detection of the resistant isolate. PFGE analysis revealed the presence of a unique clone among linezolid resistant S. hominis isolates. The G2576T mutation was detected in all the linezolid resistant strains. None of the resistant isolates showed a positive PCR for the cfr gene. All of the isolates were also resistant to penicillin, oxacillin, trimethoprim-sulfamethoxazole, ciprofloxacin, levofloxacin, and tobramicin; whereas all of them were susceptible to erythromycin, tetracycline, gentamicin, and daptomycin. The MIC of vancomycin was 4μg/ml for all the strains.
ConclusionsThe detection of linezolid resistant Staphylococci highlights the need to rationalise the use of linezolid, and maintain an active surveillance of its resistance to preserve the clinical usefulness of this antimicrobial.
Los estafilococos coagulasa negativa (SCN) se encuentran frecuentemente como residentes ubicuos en la piel y membranas mucosas de personas sanas. Sin embargo, constituyen también la principal causa de bacteriemia y de infecciones relacionadas con catéteres, especialmente en los pacientes ingresados en las Unidades de Cuidados Intensivos (UCI)1,2. En España, en dos estudios multicéntricos, entre los aislados clínicamente significativos de SCN, Staphylococcus hominis (S. hominis) ocupó el segundo lugar en frecuencia tras Staphylococcus epidermidis3,4.
La mayoría de aislados de SCN son habitualmente resistentes a múltiples antibióticos, incluyendo las penicilinas resistentes a penicilinasa5. Por ello, se recomienda el tratamiento empírico con glucopéptidos (vancomicina o teicoplanina) para cubrir las infecciones producidas por estos microorganismos. No obstante, la aparición de cepas de SCN con sensibilidad disminuida a los glucopéptidos (incluyendo aislados resistentes a glucopéptidos), los parámetros farmacocinéticos subóptimos para este grupo antibiótico y la toxicidad limitan su utilidad6. Por todo ello se ha considerado la administración de otras familias de antimicrobianos, entre ellas las oxazolidinonas. El mecanismo de acción de las oxazolidinonas se basa en la unión a la subunidad 50S del ribosoma bacteriano (concretamente al dominio V del ARNr 23S), inhibiendo la síntesis proteica7. El linezolid es, hasta el momento, la única oxazolidinona aprobada para su uso.
La resistencia al linezolid en SCN es extremadamente rara, aunque incrementándose en los últimos años. En un informe que recoge la resistencia al linezolid en cepas de grampositivos detectadas en el año 2008 (Zyvox®Annual Appraisal of Potency and Spectrum [ZAAPS]), en el que participaron 24 países del mundo, entre 748 aislados de SCN, solamente 3 de ellos (0,4%) fueron resistentes al linezolid5. El mecanismo de resistencia al linezolid más frecuentemente detectado en estafilococos se produce por mutaciones en el dominio V del gen ARNr 23S, mayoritariamente la mutación G2576T (según la numeración para Escherichia coli [E. coli])5,7–9. La resistencia al linezolid también puede producirse por mutaciones en las proteínas ribosomales L3 o L4, y por la presencia del gen cfr, que forma parte de un plásmido que puede ser horizontalmente transferido a otros estafilococos10.
Desde marzo del 2008, se detectaron los primeros aislados de S. hominis resistentes al linezolid y a la teicoplanina, en dos hospitales terciarios de Mallorca, principalmente a partir de hemocultivos de pacientes hospitalizados en UCI. Este trabajo se llevó a cabo con el fin de investigar el mecanismo de resistencia y la epidemiología molecular de los aislados de S. hominis resistentes al linezolid detectados en los dos hospitales desde marzo de 2008 hasta febrero de 2009.
Material y métodosHospitales participantesEste estudio fue llevado entre marzo de 2008 y febrero de 2009 en los dos principales hospitales públicos de la isla de Mallorca, España. El Hospital Son Llàtzer (HSLL) es un hospital terciario con 377 camas. La UCI del HSLL es una unidad médico-quirúrgica con 18 camas. El Hospital Universitario Son Dureta (HUSD) es el hospital terciario de referencia para las Islas Baleares con 740 camas. La UCI del HUSD tiene 30 camas divididas en 5 unidades según enfermedad Coronaria, Médica, Quirúrgico-Traumatológica, Neurocrítica y de Cirugía Cardíaca.
Identificación bacterianaLos aislados se identificaron mediante el sistema Vitek® 2 (bioMérieux, Francia) en HSLL o API® ID 32 STAPH (bioMérieux, Francia) en HUSD. Para confirmar la identificación de S. hominis, se realizó en una cepa resistente al linezolid una amplificación del gen ARNr 16S usando los cebadores 27F (5’- AGA GTT TGA TCM TGG CTC AG -3’) y 907R (5’- CCG TCA ATT CMT TTR AGT TT -3’)11. Posteriormente se realizó secuenciación de dicho gen con los cebadores 27F y 519R (5’- GWA TTA CCG CGG CKG CTG -3’)11 y se consultaron las bases de datos del Genbank y del Ribosomal Database Project.
Pruebas de sensibilidad antimicrobianasEl estudio de sensibilidad inicial se realizó con la tarjeta Vitek® 559 (HSLL) o por disco-difusión (HUSD), según las recomendaciones del Clinical and Laboratory Standards Institute (CLSI)12. Posteriormente, se determinó la concentración mínima inhibitoria (CMI) de los aislados de S. hominis resistentes al linezolid mediante tiras de E-test® (bioMérieux, Francia), para los siguientes antimicrobianos: linezolid, oxacilina, vancomicina, teicoplanina, ciprofloxacino, eritromicina, clindamicina, trimetoprim-sulfametoxazol (cotrimoxazol), y daptomicina. Se consideraron los puntos de corte definidos por el CLSI12.
Detección de mutaciones en ARNr 23SPara identificar las posibles mutaciones, amplificamos el dominio V del gen ARNr 23S en tres aislados de S. hominis sensibles al linezolid (todos fueron detectados en hemocultivos, uno del HSLL y dos del HUSD), y en cinco aislados de S. hominis resistentes al linezolid (tres del HSLL y dos del HUSD). Se usaron los cebadores 5’-TGG GCA CTG TCT CAA CGA-3’ (correspondientes a las bases 1984-2001 del ARN 23S de E. coli) y 5’-GGA TAG GGA CCG AAC TGT CTC-3’ (correspondientes a las bases 2597-2617 del ARN 23S de E. coli) para amplificar un fragmento de 634 pb8. Los productos de PCR fueron secuenciados y alineados con las correspondientes secuencias de oligonucleótidos de la cepa de referencia de Staphylococcus aureus (número de acceso en GenBank X68425).
Detección del gen cfrSe realizó una PCR para detectar la presencia del gen cfr, según las condiciones descritas por Kehrenberg13. Como control positivo para esta PCR, se usó una cepa de S. aureus resistente al linezolid y a la meticilina procedente del Hospital Clínico San Carlos, Madrid14.
Estudios de epidemiología molecularLa relación clonal de los aislados de S. hominis resistentes al linezolid se determinó mediante electroforesis en campo pulsado (ECP). En cada ECP, se incluyeron algunos aislados de S. hominis sensibles al linezolid. Al principio del estudio, se usó SmaI como enzima de restricción, pero sólo se visualizaron 3-5 bandas para cada cepa. Luego, en una segunda fase, se empleó la enzima de restricción ApaI, según el protocolo descrito por Sorlozano y Vindel15. Los fragmentos de ADN cromosómicos se separaron usando el sistema Chef-DR III® (Bio-Rad, Richmond, EE.UU.), con las siguientes condiciones: pulso inicial 0,1s, pulso final 30s, tiempo 24h a 6V/cm. Los patrones de bandas de la ECP se interpretaron según los criterios de Tenover16.
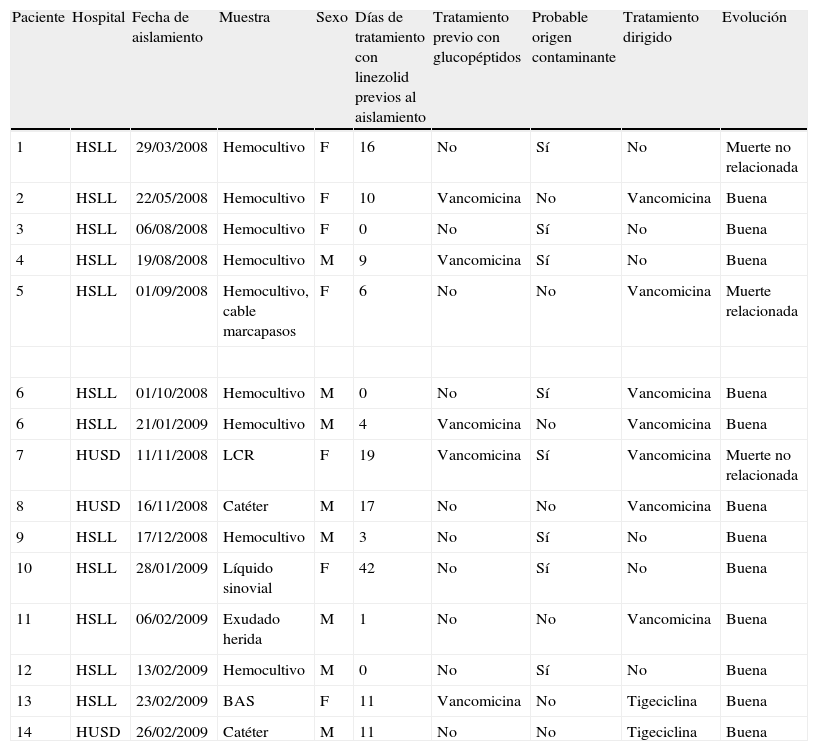
ResultadosDurante el periodo del estudio (marzo 2008-febrero 2009), se identificaron en los dos hospitales un total de 15 aislados clínicos de S. hominis subsp. hominis resistentes al linezolid en 14 pacientes distintos (tabla 1). Un paciente tuvo dos hemocultivos positivos a este microorganismo (con el mismo antibiograma) en dos ingresos diferentes separados por 100 días. De los 15 aislados, 12 se detectaron en pacientes ingresados en el HSLL (11 de ellos en la UCI) y los otros 3 en el HUSD (todos ellos en la Subunidad Quirúrgica-Traumatológica de la UCI). La evolución en los 15 episodios infecciosos fue buena en 12 de ellos, otros 2 pacientes fallecieron por causas no relacionadas, mientras que el paciente restante desarrolló una bacteriemia con infección del cable de marcapasos por este microorganismo, falleciendo a los pocos días (tabla 1).
Características de los pacientes con Staphylococcus hominis resistente al linezolid
| Paciente | Hospital | Fecha de aislamiento | Muestra | Sexo | Días de tratamiento con linezolid previos al aislamiento | Tratamiento previo con glucopéptidos | Probable origen contaminante | Tratamiento dirigido | Evolución |
| 1 | HSLL | 29/03/2008 | Hemocultivo | F | 16 | No | Sí | No | Muerte no relacionada |
| 2 | HSLL | 22/05/2008 | Hemocultivo | F | 10 | Vancomicina | No | Vancomicina | Buena |
| 3 | HSLL | 06/08/2008 | Hemocultivo | F | 0 | No | Sí | No | Buena |
| 4 | HSLL | 19/08/2008 | Hemocultivo | M | 9 | Vancomicina | Sí | No | Buena |
| 5 | HSLL | 01/09/2008 | Hemocultivo, cable marcapasos | F | 6 | No | No | Vancomicina | Muerte relacionada |
| 6 | HSLL | 01/10/2008 | Hemocultivo | M | 0 | No | Sí | Vancomicina | Buena |
| 6 | HSLL | 21/01/2009 | Hemocultivo | M | 4 | Vancomicina | No | Vancomicina | Buena |
| 7 | HUSD | 11/11/2008 | LCR | F | 19 | Vancomicina | Sí | Vancomicina | Muerte no relacionada |
| 8 | HUSD | 16/11/2008 | Catéter | M | 17 | No | No | Vancomicina | Buena |
| 9 | HSLL | 17/12/2008 | Hemocultivo | M | 3 | No | Sí | No | Buena |
| 10 | HSLL | 28/01/2009 | Líquido sinovial | F | 42 | No | Sí | No | Buena |
| 11 | HSLL | 06/02/2009 | Exudado herida | M | 1 | No | No | Vancomicina | Buena |
| 12 | HSLL | 13/02/2009 | Hemocultivo | M | 0 | No | Sí | No | Buena |
| 13 | HSLL | 23/02/2009 | BAS | F | 11 | Vancomicina | No | Tigeciclina | Buena |
| 14 | HUSD | 26/02/2009 | Catéter | M | 11 | No | No | Tigeciclina | Buena |
HSLL: Hospital Son Llàtzer; HUSD: Hospital Universitario Son Dureta; LCR: líquido cefalorraquídeo; BAS: aspirado bronquial.
Los aislados se obtuvieron principalmente de hemocultivos (9), seguido de catéteres (2), y líquidos estériles (2). Además, se llevó a cabo un estudio de colonización (nasal, axilar, e inguinal) en los últimos tres pacientes ingresados en HSLL. Todos ellos tuvieron colonización axilar e inguinal por S. hominis multirresistente, con el mismo antibiotipo que su cepa clínica correspondiente.
La secuenciación del gen ARNr 16S confirmó la identificación de S. hominis subsp. hominis. Todos estos aislados fueron resistentes a la penicilina, oxacilina (CMI > 256μg/ml), teicoplanina (CMI > 256μg/ml), trimetoprim-sulfametoxazol (CMI > 32μg/ml), ciprofloxacino (CMI > 32μg/ml), levofloxacino, tobramicina y linezolid (CMI ≥ 96μg/ml). Todos estos aislados fueron sensibles a la eritromicina, tetraciclina, gentamicina y daptomicina. La CMI de la daptomicina fue de 0,25μg/ml en todos ellos. Respecto a la vancomicina, todas las cepas presentaron una CMI de 4μg/ml, todavía sensibles según las recomendaciones del CLSI12 (S ≤ 4μg/ml, I 8-16μg/ml, R >32μg/ml) pero resistentes según los criterios del European Committee on Antimicrobial Susceptibility Testing (EUCAST) de enero de 201117 (S ≤ 2μg/ml, R >2μg/ml).
En relación con la clindamicina, casi todos los aislados tuvieron una sensibilidad intermedia a este antibiótico (CMI = 0,75μg/ml), excepto en dos aislados. Uno de ellos fue sensible (CMI = 0,5μg/ml) y el otro resistente a la clindamicina (CMI = 4μg/ml). Todas las cepas fueron sensibles a la rifampicina, excepto una que fue intermedia.
Los cinco aislados de S. hominis resistentes al linezolid secuenciados tuvieron la mutación G2576T (según numeración de E. coli) en el dominio V del gen ARNr 23S, mientras que ninguno de los tres aislados de S. hominis sensibles al linezolid secuenciados presentaron dicha mutación. En comparación con la secuencia del dominio V del gen ARNr 23S de la cepa control de S. aureus (número acceso GenBank X68425), nosotros también encontramos en todos los ocho aislados de S. hominis (tanto sensibles como resistentes al linezolid) la sustitución C2163T. Por ello, consideramos que esta sustitución es un polimorfismo de especie. No se detectó el gen cfr en ninguno de los aislados de S. hominis.
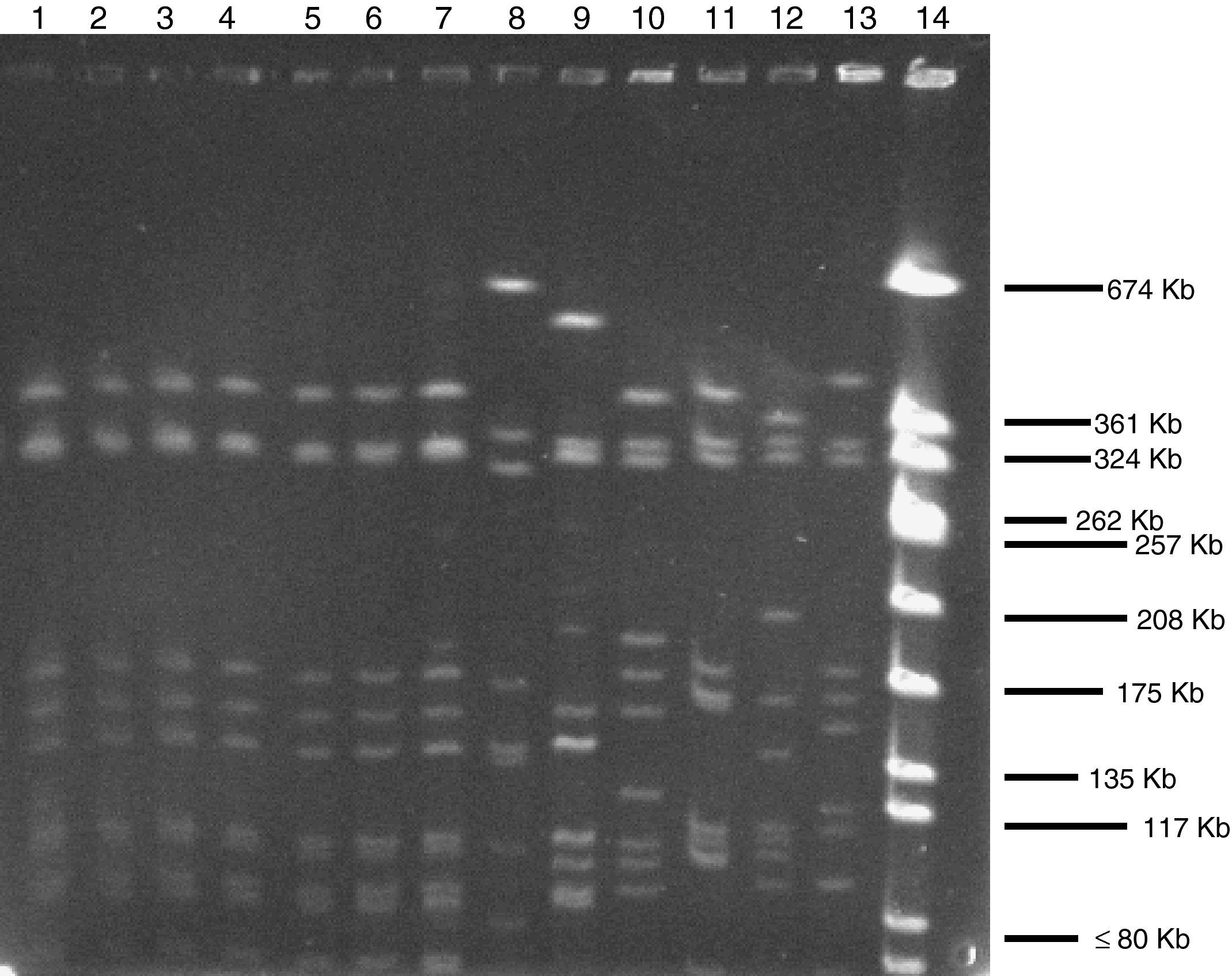
La ECP, tras digestión con SmaI, mostró que todos los aislados de S. hominis resistentes al linezolid tenían el mismo patrón electroforético, aunque mostraron sólo tres bandas, mientras que los aislados sensibles tuvieron 3-4 bandas en diferentes posiciones. La ECP usando ApaI como enzima de restricción mostró que todos los aislados resistentes al linezolid pertenecían al mismo clon, mientras que todos los aislados de S. hominis sensibles pertenecían a diferentes clones (fig. 1).
Electroforesis en campo pulsado de trece aislados de Staphylococcus hominis utilizando ApaI como enzima de restricción. Líneas 1-6: aislados de S. hominis resistentes al linezolid del Hospital Son Llàtzer (HSLL). Línea 7: aislado de S. hominis resistente al linezolid del Hospital Universitario Son Dureta (HUSD). Línea 8: aislado de S. hominis sensible al linezolid del HUSD. Líneas 9-13: aislados de S. hominis sensibles al linezolid del HSLL. Línea 14: cepa de referencia de Staphylococcus aureus NCTC 8325.
El linezolid fue aprobado para uso clínico en Estados Unidos en el año 2000 y en Europa en el 2001, con una excelente actividad contra la mayoría de los cocos grampositivos7. No se encontraron aislados de cocos grampositivos resistentes al linezolid antes de su aprobación y además, la resistencia por mutación es difícil de seleccionar in vitro18. No obstante, en el año 2001, un año después de su comercialización, se detectó en Boston, EE.UU., el primer aislado clínico de estafilococo resistente al linezolid. Este primer caso estaba producido por una cepa de S. aureus resistente a la meticilina (SARM) con la mutación G2576T en el gen ARNr 23S, que se había detectado en un paciente que había recibido diálisis peritoneal en tratamiento previo con linezolid8. El primer SCN resistente al linezolid publicado fue una cepa de S. epidermidis detectada en EE.UU. en el 2002, también con la misma mutación19. Los primeros dos brotes descritos de estafilococos resistentes al linezolid ocurrieron en el 2005, uno de ellos en Pittsburgh, EE.UU.20, y el otro en Dublín, Irlanda21; ambos producidos por dos cepas de S. epidermidis. El mecanismo de resistencia al linezolid en los aislados de S. epidermidis del hospital americano no es conocido, pero todas las cepas resistentes del hospital irlandés contenían la mutación G2576T. En España, se han descrito varios brotes de SCN resistentes al linezolid con la mutación G2576T en aislados de S. epidermidis22, Staphylococcus haemolyticus23 y S. hominis15. Por lo que respecta al gen cfr, la primera cepa de S. aureus detectada en humanos con dicho gen ocurrió en 2005 en Colombia24, mientras que el primer SCN con el gen cfr se aisló en 2007 en EE.UU. en una cepa de S. epidermidis25. Asimismo, en 2008 se detectó el primer brote de SARM con el gen cfr en el Hospital Clínico San Carlos, Madrid14.
En marzo de 2008, se detectó la primera cepa de S. hominis resistente al linezolid en Mallorca, en una paciente ingresada en la UCI del HSLL, que comenzó tratamiento con linezolid 16 días antes del aislamiento de la cepa resistente. El segundo aislado se observó en mayo 2008, en otra paciente de la misma UCI también tratada con linezolid. Pero, en agosto del 2008, a pesar de las medidas de control implantadas (aislamiento de los pacientes infectados y administración de un tratamiento antibiótico efectivo) se estableció en la UCI del HSLL una situación endémica, que persiste actualmente. Además, en enero de 2009, se detectó en el HSLL el primer paciente infectado por esta cepa hospitalizado fuera de la UCI. En el HUSD, los aislados de S. hominis resistentes al linezolid se detectaron por primera vez en dos pacientes ingresados en la UCI de este hospital en noviembre del 2008. Posteriormente, se aisló de nuevo en febrero del 2009 también en una paciente ingresada en la UCI.
De modo similar al primer brote descrito de S. hominis resistente al linezolid en Granada, España15, en la mayoría de nuestros pacientes, este microorganismo se aisló solamente en un vial de hemocultivo de un total de cuatro, por lo que podría considerarse como un contaminante de los hemocultivos. Pero, en algunos casos, detectamos S. hominis resistente al linezolid en líquidos corporales, aspirados bronquiales, o simultáneamente en catéter y hemocultivos. Llama la atención que algunos de los pacientes infectados tenían además una colonización axilar e inguinal por la misma cepa, pero no colonización nasal. Esto está en concordancia con el trabajo de Center et al en el que encontraron, en neonatos, una mayor detección de aislados de Staphylococcus warneri con sensibilidad disminuida a la vancomicina en muestras cutáneas y de heces (32 aislados en 39 muestras) que en nasofaringe (7 de 39)26.
Nosotros encontramos la mutación G2576T en el gen ARNr 23S en todos los cinco aislados de S. hominis resistentes al linezolid que secuenciamos. La aparición de la resistencia al linezolid se desarrolla en un proceso de dos pasos: inicialmente tiene lugar una mutación en la posición 2576 de una de las copias del gen ARNr 23S (los estafilococos poseen cinco o seis copias de este gen), seguida de una recombinación intracromosómica (conversión de genes), en la que se distribuye dicha mutación a las otras copias de los genes, confiriendo resistencia al linezolid9. Los aislados de estafilococos con un mayor número de copias mutadas del gen ARNr 23S suelen tener unos valores de la CMI del linezolid más altos27.
Los mecanismos de resistencia al linezolid en S. hominis se caracterizaron por primera vez en los aislados del brote de Granada15. Ese trabajo describió la presencia de dos «nuevas mutaciones» simultáneamente en aislados de S. hominis resistentes al linezolid: C2190T y G2603T. Sin embargo, ellos no usaron la numeración de E. coli para las mutaciones, sino la numeración de la cepa de referencia de S. aureus X68425. De hecho, su mutación G2603T corresponde realmente a la clásica G2576T, como encontramos en nuestro estudio. Además, en el estudio de Granada no secuenciaron aislados de S. hominis sensibles al linezolid, con lo que la mutación C2190T que encontraron en los aislados resistentes (C2163T con la numeración de E. coli) es de hecho un polimorfismo de la especie S. hominis, también presente uniformemente en los aislados sensibles al linezolid de esta especie. No se ha estudiado específicamente si este polimorfismo C2163T confiere alguna ventaja a S. hominis en relación a otras especies de SCN, si bien, al detectarse la resistencia al linezolid en diferentes especies de estafilococos sin este polimorfismo, es probable que éste no juegue ningún papel en la resistencia al linezolid. Recientemente, se ha publicado la detección de la mutación G2576T en cinco aislados de S. hominis resistentes al linezolid en pacientes ingresados en la UCI de dos hospitales de Sicilia28.
Dos aspectos adicionales no quedan claros en nuestro brote. Uno de ellos es la diseminación de la cepa desde la UCI del HSLL a la UCI del HUSD. En 2008, no se produjo ninguna transferencia de pacientes entre las UCI de ambos hospitales y tampoco hubo ningún traslado de instrumental entre ellas. No obstante, dos enfermeras estaban trabajando en las dos UCI al mismo tiempo, aunque no se realizó estudio de colonización del personal sanitario. De modo similar, se desconocen los factores responsables de la persistencia de la cepa en la UCI del HSLL, produciendo 1-2 infecciones cada mes. La cepa podría transmitirse de paciente a paciente a través de las manos del personal sanitario colonizado29, o bien, podría haber un reservorio en el ambiente de la UCI. En el brote irlandés de S. epidermidis resistente al linezolid, los autores encontraron la misma cepa resistente en las cercanías de los pacientes colonizados y en un ordenador usado por el personal del la UCI21.
Una larga duración del tratamiento con linezolid, una dosificación insuficiente, y la administración repetida del linezolid son factores de riesgo para desarrollar resistencia a este antimicrobiano30. La resistencia al linezolid no sólo se ha observado en pacientes con un tratamiento prolongado a dicho antimicrobiano, sino también en casos sin exposición obvia20–23. En nuestra serie, la mayoría de pacientes infectados (12 de 15 episodios infecciosos, el 80,0%) habían recibido ciclos de linezolid antes de la detección del aislado resistente. Con todo, 3 pacientes (20,0%) no recibieron linezolid, lo que sugiere la adquisición por transmisión cruzada21,23.
Todos los aislados de S. hominis resistentes al linezolid fueron también resistentes a la teicoplanina y tuvieron por E-test CMI a la vancomicina de 4,0μg/ml. En el momento actual, 4,0μg/ml es el límite superior del rango de sensibilidad según el CLSI12, y resistente según el EUCAST17. Este perfil de resistencia a glucopéptidos está también presente en aislados resistentes al linezolid de S. epidermidis20, S. haemolyticus23 y S. hominis15,28, todos ellas con la mutación G2576T. La relación entre esta mutación en el gen ARNr 23S y la resistencia a los glucopéptidos es desconocida. La resistencia a los glucopéptidos en Staphylococcus spp. es multifactorial31, siendo el engrosamiento de la pared celular una característica común32.
La incidencia creciente de infecciones causadas por aislados de estafilococos multirresistentes, incluyendo resistencia a la teicoplanina y al linezolid, es preocupante. Es crucial hacer un uso racional del linezolid y mantener un control activo de dicha resistencia para preservar la utilidad clínica de este antimicrobiano.
FinanciaciónEste trabajo ha sido parcialmente sufragado por el Ministerio de Ciencia e Innovación de España, Instituto de Salud Carlos III, a través de la Red Española de Investigación en Patología Infecciosa (REIPI C03/14 y RD06/0008).
Conflicto de interesesLos autores declaran no tener conflicto de intereses.
Agradecemos a la Dra. Carmen Vidal de la Unidad de Secuenciación del HUSD (ahora Son Espases) su ayuda con los experimentos de las diferentes secuenciaciones, y a la Dra. Carmen Betriu y la Dra. Gracia Morales del Hospital Clínico San Carlos, Madrid, el envío de una cepa de S. aureus positiva para el gen cfr.