Uno de los principales problemas en el diagnóstico de la gripe es el tipo de muestra y la edad del paciente. En general, la mayoría de las técnicas diagnósticas se han mostrado muy efectivas en los pacientes pediátricos y en los aspirados nasofaríngeos. Sin embargo, su eficacia se ha mostrado mucho menor en la población adulta y los frotis faríngeos.
ObjetivoSe ha realizado un estudio prospectivo comparativo sobre la eficacia de una técnica de RT-PCRtr (reverse transcription polymerase chain reaction in real time ‘reacción en cadena de la polimerasa en transcripción reversa en tiempo real’) comercial, un enzimoinmunoanálisis (EIA) y el cultivo celular shell-vial (SV) en la detección de virus gripales A y B en 125 frotis faríngeos de pacientes adultos con sospecha clínica de gripe durante la temporada 2007–2008.
Material y métodosA los frotis faríngeos se les realizó la detección antigénica gripal mediante un EIA dot-blot comercial. Para la RT-PCRtr se extrajo el ácido ribonucleico de 200μl de la muestra mediante el sistema de extracción automatizado EZ1 virus Mini Kit v2.0. La amplificación genómica se realizó mediante una RT-PCRtr utilizando el sistema comercial automatizado OneStep RT-PCR FluA + FluB y el SmartCycler como sistema de amplificación. Las muestras se inocularon en 2 viales de la línea celular Madin-Darby de riñón canino. El tiempo de respuesta se calculó como el transcurrido entre la llegada de la muestra hasta la obtención del resultado definitivo.
ResultadosEl sistema EIA detectó 27 muestras positivas (21,6%), la RT-PCRtr 62 muestras positivas (49,6%) y el cultivo SV 56 muestras positivas (44,8%). De las 62 muestras positivas, el EIA detectó 27 muestras (43,5%), la RT-PCRtr 62 muestras (100%) y el SV 56 muestras (90,3%). El empleo de la RT-PCRtr permitió que en el 38,4% de los adultos el diagnóstico se obtuviera el mismo día de recepción de la muestra. Así, el 67,2% de los resultados se obtuvieron en las primeras 24h con un tiempo de respuesta medio de 1,1 días.
ConclusiónLa técnica RT-PCRtr estudiada ha mostrado una elevada sensibilidad y especificidad, en comparación con las otras técnicas, en la detección de los virus gripales en los frotis faríngeos de la población adulta. Mediante esta técnica se puede procesar con facilidad un gran número de muestras, obtieniéndose resultados en el mismo día de la recepción de la muestra.
The age of the patients and the type of sample are major problems in the diagnosis of influenza. Most available diagnostic techniques are highly effective in pediatric patients and in nasopharyngeal aspirates. However, in the adult population and using throat swabs, these techniques are much less reliable.
AimWe performed a prospective study comparing the efficacy of a commercial real-time reverse transcription PCR assay (RT-PCR) with that of an enzyme immunoassay (EIA) or shell vial culture (SV) in the detection of influenza A and B viruses in 125 throat swabs from adults with clinically suspected influenza during the 2007–2008 flu season.
Material and methodsThroat swabs were subjected to rapid antigen detection for influenza viruses by means of a commercial dot-blot EIA. For the RT-PCR technique, RNA was extracted from 200μL of each sample by the automated extraction system, EZ1 virus minikit (version 2.0). Genomic amplification of the extracted viral RNA was carried out using the OneStep RT-PCR FluA+FluB automated system with the SmartCycler amplification system. Each sample was inoculated into 2 SV of the MDCK cell line. Turnaround times were calculated from the time specimens were received in the laboratory to the time the result was reported to clinicians.
ResultsThe EIA system detected 27 (21.6%) positive samples, RT-PCR 62 (49.6%) positive samples, and SV 56 (44.8%) positive samples. Among the 62 positive samples, EIA detected 27 (43.5%), RT-PCR 62 (100%) and SV 56 (90.3%). With the use of RT-PCR, 38.4% of the adults studied were diagnosed on the same day samples were received. Among the total, 67.2% of diagnostic results were obtained within the first 24 hours; turnaround time was 1.1 days.
ConclusionThe real-time RT-PCR method studied displayed high sensitivity and specificity in the detection of influenza virus in adult patients, when compared with the conventional techniques. With real-time RT-PCR, large numbers of samples can be rapidly tested and results provided the same day samples are received.
La gripe es una enfermedad infecciosa causada por virus gripales A y B que se presenta como epidemias anuales en los meses invernales. Los subtipos H3N2 y H1N1 de virus gripal A y de virus gripal B1 causan estas epidemias en la actualidad.
Desde el punto de vista epidemiológico, es muy importante realizar un diagnóstico etiológico al inicio de cada nueva temporada epidémica, así como durante y al final de ésta, para establecer la prevalencia y las características antigénicas de las cepas gripales circulantes en ésta. Asimismo, la existencia de los inhibidores de la exo-a-sialidasa para el tratamiento de esta entidad aconseja realizar el diagnóstico etiológico en un período no superior a las 48h tras el inicio de los síntomas1,2. Además de esto, el diagnóstico rápido y específico de la gripe se ha mostrado coste-efectivo en la gripe pediátrica3 así como en el control de las epidemias gripales que se producen en las residencias de ancianos4.
El aislamiento vírico se ha considerado como el método de referencia para el diagnóstico de gripe. Éste puede realizarse mediante inoculación en huevos embrionados o mediante cultivo celular (método convencional o en shell-vial [SV])5,6. Sin embargo, estos métodos presentan el inconveniente de ser lentos y laboriosos, ya que requieren de 2 a 7 días para obtener los resultados5,6. Debido a esto, se han desarrollado métodos de diagnóstico rápidos basados en la detección antigénica y, preferentemente, en la detección genómica5.
Para la detección antigénica, los métodos de enzimoinmunoanálisis (EIA) son los que han mostrado una mayor sensibilidad y especificidad. La mayoría se realizan sobre membranas de nitrocelulosa y permiten la obtención de resultados luego de 10 a 30min de incubación5–7.
Diferentes estudios han demostrado que los métodos moleculares (reacción en cadena de la polimerasa en transcripción reversa) presentan mayor sensibilidad que los métodos antigénicos y que el propio cultivo celular en el diagnóstico de las infecciones gripales5,8,9. La técnica de RT-PCRtr (reverse transcription polymerase chain reaction in real time ‘reacción en cadena de la polimerasa en transcripción reversa en tiempo real’) permite la obtención de resultados en menos de 4h y reduce el número de falsos positivos que puedan deberse a las contaminaciones cruzadas entre diferentes muestras8,9.
Uno de los problemas en el diagnóstico de la gripe es el tipo de muestra utilizada y la edad del paciente. En general, la mayoría de las técnicas diagnósticas son muy eficaces en la población pediátrica y en los aspirados nasofaríngeos10. Sin embargo, el rendimiento de estas técnicas en la población adulta y en los frotis faríngeos es mucho más bajo11,12. Por esto, se ha realizado un estudio sobre la eficacia de una RT-PCRtr frente a un método de detección antigénica y del cultivo SV en la detección de virus gripales A y B en una población adulta con sospecha de gripe.
Material y métodosSe ha realizado un estudio comparativo entre 3 técnicas diagnósticas en la detección de virus gripales A y B, en frotis faríngeos de personas adultas (mayores de 18 años) con sospecha clínica de gripe durante la temporada 2007–2008, procedentes de la red de vigilancia de la gripe (pacientes comunitarios) y de pacientes hospitalizados.
Los frotis faríngeos se enviaron lo antes posible al laboratorio en un medio de transporte para virus (VTM, Vircell, Granada, España) sin refrigerar. A cada uno de éstos se les realizó la detección antigénica rápida frente a virus gripales A y B mediante un EIA comercial (Directigen FluA+B, Becton & Dickinson) de acuerdo con las instrucciones del fabricante.
Para la técnica de la RT-PCRtr se realizó la extracción del ácido ribonucleico (ARN) a partir de 200μl de cada muestra. Para este proceso se utilizó el sistema automatizado de extracción EZ1 virus Mini Kit v2.0 (BioRobot EZ1, Qiagen, Alemania) de acuerdo con las instrucciones del fabricante. El ARN se eluyó en 60μl de tampón y se conservó a 4°C entre uno y 3 días. El proceso de amplificación genómica del ARN vírico extraído se realizó mediante una RT-PCRtr utilizando el sistema comercial automatizado OneStep RT-PCR FluA+FluB (Qiagen, Alemania) con el sistema de amplificación SmartCycler (Cepheid, The Netherlands). El proceso se basa en la utilización de 5μl de extracto de ARN obtenido previamente de la muestra y 20μl de la mezcla de reacción y amplificación con los cebadores específicos de virus gripales (Influenza virus A/B primer and probe set analyse specific reagent, Cepheid, The Netherlands). La amplificación se realiza mediante cebadores, sentido y sinsentido, dirigidos contra la proteína de matriz (M1) de cada uno de virus gripales A y B. Tras el proceso de amplificación, los amplicones se detectan mediante la utilización de una sonda específica para virus gripal A marcada con el fluorocromo FAM y para el virus gripal B con el fluorocromo Alexa-532. El proceso se realizó con el aparato SmartCycler de acuerdo con las instrucciones del fabricante.
A su vez, las muestras se inocularon en 2 cultivos SV de la línea celular Madin-Darby de riñón canino (Vircell, Granada, España), centrifugadas a 3.500r.p.m. por 15min e incubadas durante 72h, y posteriormente se tiñeron las monocapas con anticuerpos monoclonales frente a virus gripal A (clon IA52/9) y frente a virus gripal B (clon IB82/2) (Monofluokit Influenza, Sanofi Diagnostics Pasteur, Marnes la Coquette, Francia), mediante una técnica de inmunofluorescencia indirecta. Se consideró una muestra como positiva si técnica una RT-PCRtr y/o un cultivo SV positivo para alguno de los virus gripales.
Se ha definido el tiempo de respuesta como aquel que transcurría entre la recepción de la muestra en el laboratorio y la obtención del resultado definitivo. Asimismo, se ha comparado el tiempo de respuesta entre las muestras estudiadas en la temporada gripal 2006–2007 (sin el empleo de la RT-PCRtr) y las de la temporada gripal 2007–2008 (con el empleo de la RT-PCRtr).
Los cálculos estadísticos comparativos entre las diferentes técnicas diagnósticas se realizaron mediante el empleo del programa informático SPSS versión 11.0.
ResultadosEn este estudio se han analizado 125 frotis faríngeos de pacientes adultos. Se detectó la presencia de algún virus gripal en 62 muestras (49,6%). El virus gripal A se detectó en 34 muestras (54,8%) y el virus gripal B en 28 muestras (45,2%).
Globalmente, el método EIA detectó 27 muestras positivas (21,6%), la RT-PCRtr 62 muestras positivas (49,6%) y el cultivo SV 56 muestras positivas (44,8%). De las 62 muestras positivas, el método EIA detectó 27 muestras (43,5%), la RT-PCRtr detectó 62 muestras (100%) y el cultivo SV detectó 56 muestras (90,3%).
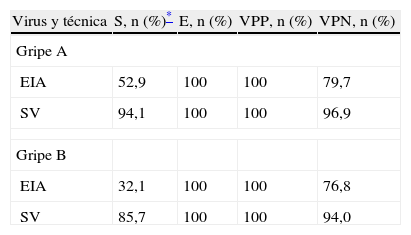
De las 34 muestras positivas al virus gripal A, el método EIA detectó 18 (52,9%), la RT-PCRtr detectó 34 (100%) y el cultivo SV detectó 32 (94,1%) (tabla 1). De las 28 muestras positivas a virus gripal B, el método EIA detectó 9 (32,1%), la RT-PCRtr detectó 28 (100%) y el cultivo SV detectó 24 (85,7%) (tabla 2).
Resultados comparativos obtenidos en la detección de virus gripal A
| EIA, n (%) | RT-PCRtr, n (%) | SV, n (%) | No, n (%)* |
| + | + | + | 18 (52,9) |
| − | + | + | 14 (41,1) |
| − | + | − | 2 (5,8) |
| 18 (52,9) | 34 (100) | 32 (94,1) | 34 |
EIA: enzimoinmunoanálisis; RT-PCRtr: reverse transcription polymerase chain reaction in real time ‘reacción en cadena de la polimerasa en transcripción reversa en tiempo real’; SV: cultivo shell-vial.
Resultados comparativos obtenidos en la detección de virus gripal B
| EIA, n (%) | RT-PCRtr, n (%) | SV, n (%) | No, n (%)* |
| + | + | + | 9 (32,1) |
| − | + | + | 15 (53,5) |
| − | + | − | 4 (14,2) |
| 9 (32,1) | 28 (100) | 24 (85,7) | 28 |
EIA: enzimoinmunoanálisis; RT-PCRtr: reverse transcription polymerase chain reaction in real time ‘reacción en cadena de la polimerasa en transcripción reversa en tiempo real’; SV: cultivo shell-vial.
La tabla 3 presenta los resultados obtenidos al comparar la RT-PCRtr con las otras técnicas diagnósticas. Asimismo, no se han obtenido resultados falsos positivos con el método EIA y el cultivo SV y ambas técnicas presentan una especificidad y un valor predictivo positivo del 100%.
Resultados obtenidos en la comparación entre la reacción en cadena de la polimerasa en transcripción reversa en tiempo real y las otras técnicas diagnósticas
| Virus y técnica | S, n (%)* | E, n (%) | VPP, n (%) | VPN, n (%) |
| Gripe A | ||||
| EIA | 52,9 | 100 | 100 | 79,7 |
| SV | 94,1 | 100 | 100 | 96,9 |
| Gripe B | ||||
| EIA | 32,1 | 100 | 100 | 76,8 |
| SV | 85,7 | 100 | 100 | 94,0 |
E: especifidad; EIA: enzimoinmunoanálisis; S: sensibilidad; SV: cultivo shell-vial; VPN: valor predictivo negativo; VPP: valor predictivo positivo.
La incorporación de la RT-PCRtr ha permitido que en el 38,4% de los pacientes se obtuviera el resultado en el mismo día de la recepción de la muestra. Con esta nueva técnica el 67,2% de los resultados diagnósticos se obtuvo en las primeras 24h, con un tiempo de respuesta medio de 1,1 días (rango de 0 a 3 días) frente a los 3,6 días (rango de 2 a 6 días) obtenidos en las 93 muestras estudiadas en la temporada 2006–2007.
DiscusiónEl diagnostico rápido y específico de la gripe es actualmente un elemento esencial en el tratamiento de los pacientes con infecciones respiratorias agudas del tracto respiratorio inferior.
Las técnicas de detección antigénica frente a virus gripales A y B han mostrado un valor de sensibilidad y un valor predictivo negativo muy variables. De este modo, aunque pueden utilizarse como técnicas iniciales de selección, obligan a realizar técnicas alternativas de mayor eficacia diagnóstica. Los principales motivos de esta variabilidad son el tipo de muestra analizada y la población estudiada11,12.
Diferentes estudios han demostrado que los aspirados nasofaríngeos muestran un mayor rendimiento diagnóstico que los frotis faríngeos o nasales, probablemente debido a la mayor carga vírica de estas muestras5,10. Por otro lado, se ha demostrado que la población infantil muestra una mayor carga y un mayor tiempo de excreción vírica que la población adulta10,13. Además, en general, en los pacientes adultos es mas difícil obtener un aspirado nasofaríngeo, especialmente si son ambulatorios y debe recurrirse a la toma de un simple frotis faríngeo. Así, pues, la población adulta representa un reto en el diagnóstico de la infección gripal debido a la baja carga vírica de su tracto respiratorio y a la utilización de una muestra respiratoria no adecuada4,5,10.
En este estudio, realizado durante la temporada gripal 2007–2008, se ha detectado la presencia de virus gripales en el 49,6% de la población adulta analizada. En el estudio de D′Heilly et al11 realizado en 84 adultos, se detectó un virus gripal en el 27% de los casos. La prevalencia que comunicó este autor está por debajo de otros estudios, con valores de prevalencia del 66 al 77%, en los que se establecían criterios mínimos de sospecha clínica para estudiarse14. Los estudios realizados con pacientes ambulatorios sin aplicación de criterios mínimos demuestran una positividad del 21 al 32%15. Un estudio que realizó Reina et al12 sobre 67 pacientes adultos obtuvo una positividad del 37,8% frente a los virus gripales. Debe tenerse en cuenta que los valores de positividad pueden variar ampliamente de acuerdo con la temporada y las características antigénicas de los virus circulantes.
Hay pocos estudios sobre la eficacia de las técnicas de detección antigénica rápida sólo en la población adulta con infección gripal. Así, D’Heilly et al11 comunicaron que la técnica antigénica que utilizaron detectó esta infección en tan sólo el 18% de los pacientes. El porcentaje global de positividad en la técnica antigénica utilizada en este estudio fue del 43,5%, aunque ha mostrado diferencias entre virus gripal A (52,9%) y virus gripal B (32,1%). Los valores diferentes entre los 2 tipos víricos ya se han descrito en otros estudios y han demostrado la mayor dificultad de estas técnicas antigénicas en la detección del virus gripal B en muestras respiratorias, tanto en niños como en adultos11,12,16–18.
En este estudio, el cultivo SV ha permitido detectar el 90,3% de las muestras positivas por RT-PCRtr, presentando un mayor rendimiento para virus gripal A (94,1%) que para virus gripal B (85,7%). Ruest et al16 ya habían comunicado que tanto las técnicas antigénicas como la PCR (polymerase chain reaction 'reacción en cadena de la polimerasa') presentaban una sensibilidad global superior del 10 al 15% en la detección del virus gripal A. Herrmann et al18 han comunicado que la PCR multiplex permite detectar un 27% más de positividades frente ambos virus; ésta fue la técnica que detectó la totalidad de las muestras positivas por otros métodos.
Diferentes estudios2,16–18 han demostrado que las técnicas de amplificación genómica (PCR) detectan un mayor número de muestras positivas, presentando una mayor sensibilidad que las técnicas antigénicas y el cultivo celular. Este incremento oscila entre el 3 y el 46%, especialmente en la poblacion adulta11,16,17. Ruest et al16 obtuvieron en su estudio un 7% más de positividad con la PCR. En este estudio el 100% de las muestras positivas se detectó mediante la RT-PCRtr y, además, el 9,6% se detectó sólo mediante esta técnica.
En este estudio las muestras sólo positivas en la RT-PCRtr tardaron más de 4 días en llegar al laboratorio, lo que puede determinar el deterioro de ésta y la incapacidad de los virus presentes en éstas para crecer en el cultivo celular17,18. La PCR permitiría en estos casos procesar muestras tomadas en diferentes tiempos y lugares sin que se produjera una pérdida de la capacidad de detección genómica18. Por otro lado, algunos estudios han comprobado que las muestras positivas en el cultivo y negativas en la PCR se pueden deber a la inactivación de los virus durante el transporte o la conservación de la muestra o por procesos técnicos de inhibición de la amplificación. La posibilidad de una inhibición en la prueba de la PCR se calcula en cerca del 2% de las muestras del tracto respiratorio, aunque si se repite la técnica con otra alícuota se disminuye este porcentaje19.
Una de las principales ventajas de la RT-PCRtr es la rapidez (tiempo de respuesta) en el diagnóstico, no sólo en comparación con las técnicas clásicas sino también con la PCR convencional20–22. La duración de la técnica que se estudió fue de unas 4h, lo que permite realizar varias veces la misma técnica en una sola jornada de trabajo. En el estudio de Zitterkopf et al9 se ha comunicado un tiempo de respuesta de 14,8h para virus gripal A frente a las 49,3h del cultivo SV. En este estudio los tiempos de respuesta fueron de 26,4h para la RT-PCRtr y de 86,4h para el cultivo SV. La utilización habitual de la RT-PCRtr ha permitido que en el 38,4% de los adultos estudiados se obtuviera el resultado en el mismo día de la recepción de la muestra. De este modo, el 67,2% de los resultados diagnósticos se obtuvo en las primeras 24h, con un tiempo de respuesta de 1,1 días frente a los 3,6 días obtenidos en las 93 muestras estudiadas en la temporada 2006–2007.
En resumen, la RT-PCRtr estudiada ha mostrado una elevada sensibilidad y especificidad en la detección de los virus gripales en comparación con las técnicas convencionales. Como consecuencia de esto, se cree que la técnica de la RT-PCRtr se debe considerar como la técnica de referencia para el diagnóstico de gripe en los pacientes adultos y en las muestras con menor carga vírica, como los frotis faríngeos. Esta técnica reduce el tiempo de respuesta en los resultados y el número de falsos positivos de las muestras. Además, permite estudiar un gran número de muestras y obtener resultados en el mismo día de su procesamiento.