En España el aislamiento de cepas de Pseudomonas aeruginosa productoras de metalobetalactamasas (MBL) es poco frecuente. En este artículo se describe la caracterización de 9 aislados clínicos de P. aeruginosa multirresistentes clonalmente relacionados, aislados en Cantabria (España) que poseen el gen blaVIM−2.
MétodosLos aislados de P. aeruginosa (uno por paciente) se han obtenido en el Servicio de Microbiología del Hospital Universitario Marqués de Valdecilla entre enero de 2004 y diciembre de 2006. La identificación y los estudios preliminares de sensibilidad a los antimicrobianos se determinaron con el sistema MicroScan Walkaway (Dade Behring, Sacramento, California) y se confirmaron mediante microdilución de referencia.
ResultadosLas concentraciones inhibitorias mínimas de imipenem (IMP) fueron de 32 a 128 μg/ml y de 16 a 64μg/ml para meropenem. Los 9 aislados presentaron un único patrón de Rep-PCR y tenían sensibilidad intermedia o resistencia a ceftacidima, cefepima, gentamicina, tobramicina, amikacina y ciprofloxacina. Ocho de los 9 aislados fueron sensibles a aztreonam. La tasa de hidrólisis de IMP por extractos crudos de los aislados estudiados fue de 162 ± 18 a 235 ± 28pmol/min/μg proteína y se inhibió en presencia de 5mM de ácido etilendiaminotetraacético. Todos los aislados poseían un integrón con los genes aac (6′)32, blaVIM−2 y un gen que codifica para una probable transposasa, flanqueados por las regiones conservadas 5′CS y 3′CS.
ConclusiónLa presencia de la MBL VIM-2 en estas cepas es suficiente para justificar su resistencia a carbapenémicos.
Pseudomonas aeruginosa strains producing metallo-β-lactamases (MβL) are uncommon in Spain. This study describes the characterization of 9 new clonally related multiresistant P. aeruginosa isolates possessing the blaVIM−2 gene in Cantabria (Northern Spain).
MethodsP. aeruginosa clinical strains (1 per patient) were isolated in the Microbiology Service of Marqués de Valdecilla University Hospital between January 2004 and December 2006. Identification and preliminary susceptibility studies were performed with the MicroScan WalkAway system (Dade Behring, Sacramento, CA) and results were verified by a microdilution reference method.
ResultsMICs of imipenem and meropenem for the 9 isolates ranged from 32 to 128 and 16 to 64μg/mL, respectively. Nine isolates had a single Rep-PCR pattern and were intermediate or resistant to ceftazidime, cefepime, gentamicin, tobramycin, amikacin and ciprofloxacin. Eight of the 9 isolates were susceptible to aztreonam. Hydrolysis activity of imipenem in MβL-positive isolates ranged from 162±18 to 235±28pmol/min/μg protein and was abolished in the presence of 5 mM EDTA. All isolates possessed an integron with genes aac(6′)32, blaVIM−2 and a putative transposase-encoding gene, flanked by the conserved 5’CS and 3’CS regions.
ConclusionIn the clinical isolates studied, the presence of MβL VIM-2 sufficed to explain their resistance to carbapenems.
Los carbapenémicos son antimicrobianos ampliamente empleados en el tratamiento de infecciones producidas por Pseudomonas aeruginosa. La resistencia a estos compuestos se ha asociado clásicamente al efecto aditivo de 3 mecanismos moleculares: la pérdida de la porina OprD junto con la hiperproducción de la betalactamasa cromosómica AmpC y con el incremento en la expresión de la bomba de expulsión activa MexAB-OprM (que afecta a la expulsión del meropenem [MPM])1. La resistencia a carbapenémicos se ha incrementado recientemente debido a la diseminación de metalobetalactamasas (MBL) pertenecientes a la clase B de Ambler, capaces de hidrolizar estos compuestos además de las penicilinas y cefalosporinas, pero no el aztreonam. Estas enzimas requieren zinc para su actividad catalítica y su actividad es inhibida por agentes quelantes como el ácido etilendiaminotetraacético (EDTA, ethylenediaminetetraacetic acid).
Según el estudio multicéntrico llevado a cabo en hospitales españoles por Gutiérrez et al2, la frecuencia de aislados clínicos de P. aeruginosa resistentes a imipenem (IMP) o a MPM es del 18,9% y la presencia de MBL en estos aislados se da con una frecuencia del 0,4%.
Las MBL descritas hasta la fecha en Pseudomonas spp. se agrupan en 4 grupos moleculares: IMP, VIM, GIM y SPM3. Un quinto grupo corresponde a SIM4, que se ha descrito únicamente en Acinetobacter baumannii. Los genes causantes de la producción de MBL se encuentran formando parte de un integrón5, que puede estar localizado en un plásmido movilizable o insertado en el cromosoma.
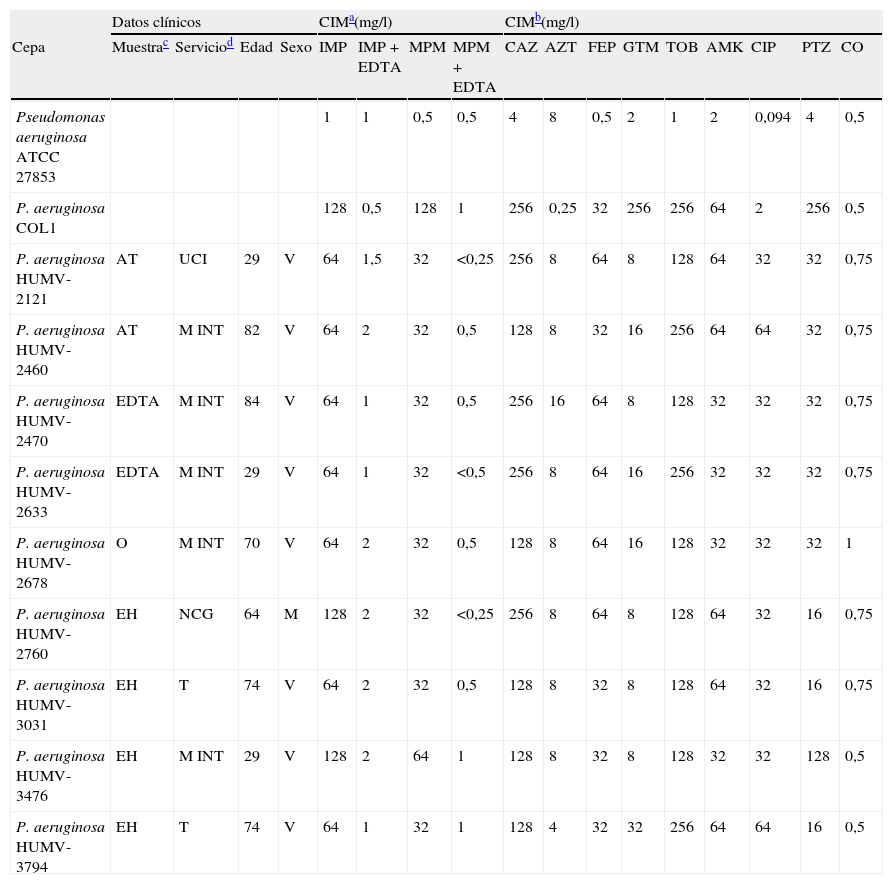
El objetivo de este estudio ha sido la caracterización fenotípica y genotípica de 9 aislados clínicos de P. aeruginosa que codifican la MBL VIM-2, detectados en el Servicio de Microbiología del Hospital Universitario Marqués de Valdecilla durante los años 2004 a 2006 (tabla 1).
Susceptibilidad a antimicrobianos en aislados clínicos productores del gen blaVIM-2
| Datos clínicos | CIMa(mg/l) | CIMb(mg/l) | |||||||||||||||
| Cepa | Muestrac | Serviciod | Edad | Sexo | IMP | IMP + EDTA | MPM | MPM + EDTA | CAZ | AZT | FEP | GTM | TOB | AMK | CIP | PTZ | CO |
| Pseudomonas aeruginosa ATCC 27853 | 1 | 1 | 0,5 | 0,5 | 4 | 8 | 0,5 | 2 | 1 | 2 | 0,094 | 4 | 0,5 | ||||
| P. aeruginosa COL1 | 128 | 0,5 | 128 | 1 | 256 | 0,25 | 32 | 256 | 256 | 64 | 2 | 256 | 0,5 | ||||
| P. aeruginosa HUMV-2121 | AT | UCI | 29 | V | 64 | 1,5 | 32 | <0,25 | 256 | 8 | 64 | 8 | 128 | 64 | 32 | 32 | 0,75 |
| P. aeruginosa HUMV-2460 | AT | M INT | 82 | V | 64 | 2 | 32 | 0,5 | 128 | 8 | 32 | 16 | 256 | 64 | 64 | 32 | 0,75 |
| P. aeruginosa HUMV-2470 | EDTA | M INT | 84 | V | 64 | 1 | 32 | 0,5 | 256 | 16 | 64 | 8 | 128 | 32 | 32 | 32 | 0,75 |
| P. aeruginosa HUMV-2633 | EDTA | M INT | 29 | V | 64 | 1 | 32 | <0,5 | 256 | 8 | 64 | 16 | 256 | 32 | 32 | 32 | 0,75 |
| P. aeruginosa HUMV-2678 | O | M INT | 70 | V | 64 | 2 | 32 | 0,5 | 128 | 8 | 64 | 16 | 128 | 32 | 32 | 32 | 1 |
| P. aeruginosa HUMV-2760 | EH | NCG | 64 | M | 128 | 2 | 32 | <0,25 | 256 | 8 | 64 | 8 | 128 | 64 | 32 | 16 | 0,75 |
| P. aeruginosa HUMV-3031 | EH | T | 74 | V | 64 | 2 | 32 | 0,5 | 128 | 8 | 32 | 8 | 128 | 64 | 32 | 16 | 0,75 |
| P. aeruginosa HUMV-3476 | EH | M INT | 29 | V | 128 | 2 | 64 | 1 | 128 | 8 | 32 | 8 | 128 | 32 | 32 | 128 | 0,5 |
| P. aeruginosa HUMV-3794 | EH | T | 74 | V | 64 | 1 | 32 | 1 | 128 | 4 | 32 | 32 | 256 | 64 | 64 | 16 | 0,5 |
AMK: amikacina; AT: aspirado traqueal; AZT: aztreonam; CAZ: ceftacidima; CIM: concentración inhibitoria mínima; CIP: ciprofloxacina; CO: colistina; E: esputo; EDTA: ácido etilendiaminotetraacético (ethylenediaminetetraacetic acid); EH: exudado herida; FEP: cefepima; GTM: gentamicina; IMP: imipenem; M: mujer; M INT: medicina interna; MPM: meropenem; NCG: neurocirugía; O: orina directa; PTZ: piperacilina con tazobactam; T: traumatología; TOB: tobramicina; UCI: unidad de cuidados intensivos; V: varón.
CIM (mg/l) determinadas mediante microdilución de IMP y de MPM en presencia y ausencia de 0,4mM de EDTA y de 0,04mM 1,10-fenantrolina.
Los aislados de P. aeruginosa (uno por paciente) se han obtenido de muestras clínicas enviadas al Servicio de Microbiología del Hospital Universitario Marqués de Valdecilla entre enero de 2004 y diciembre de 2006. Como posibles productores de MBL se seleccionaron los aislados con una concentración inhibitoria mínima (CIM) de IMP o de MPM superior o igual a 8mg/l (tabla 1). La identificación y los estudios preliminares de sensibilidad a los antimicrobianos se determinaron con el sistema MicroScan Walkaway (Dade Behring, Sacramento, California, EE. UU.) y con tiras de Etest (AB Biodisk, Solna, Suecia).
Las CIM de IMP y de MPM se confirmaron mediante microdilución de referencia de acuerdo con las normas del Clinical and Laboratory Standards Institute6. También se ha empleado la microdilución para la detección fenotípica de MBL: se consideró indicativa de la presencia de MBL una relación superior o igual a 8 del cociente entre la CIM de IMP o de MPM solos y el mismo agente combinado con 0,4mM de EDTA (Sigma, Madrid, España) y 0,04mM 1,10-fenantrolina (Sigma, Madrid, España). La obtención del ácido desoxirribonucleico (ADN) y las reacciones para llevar a cabo la detección simultánea de los genes blaIMP, blaVIM, blaGIM, blaSPM y blaSIM mediante Multiplex PCR (polymerase chain reaction 'reacción en cadena de la polimerasa') se han realizado usando los oligonucleótidos y las condiciones previamente descritas3.
Se han utilizado como controles en todas las reacciones de amplificación las cepas portadoras de los diferentes tipos de MBL: P. aeruginosa (blaVIM−2), P. aeruginosa 07-406 (blaVIM−7), P. aeruginosa 86-6348 (blaIMP−2), A. baumannii 48-695 (blaIMP−1), P. aeruginosa 86-16734 (blaIMP−13) y P. aeruginosa 73-5671 (blaGIM−1), cedidas por el Dr. Toleman (Universidad de Cardiff, Reino Unido); A. baumannii T104 YMC 03/9/T104 (blaSIM−1), enviada por el Dr. Rossolini (Universidad de Siena, Italia) y P. aeruginosa P1088 (blaSMP−1), donada por el Dr. Mendes (Universidad Federal de São Paulo, Brasil).
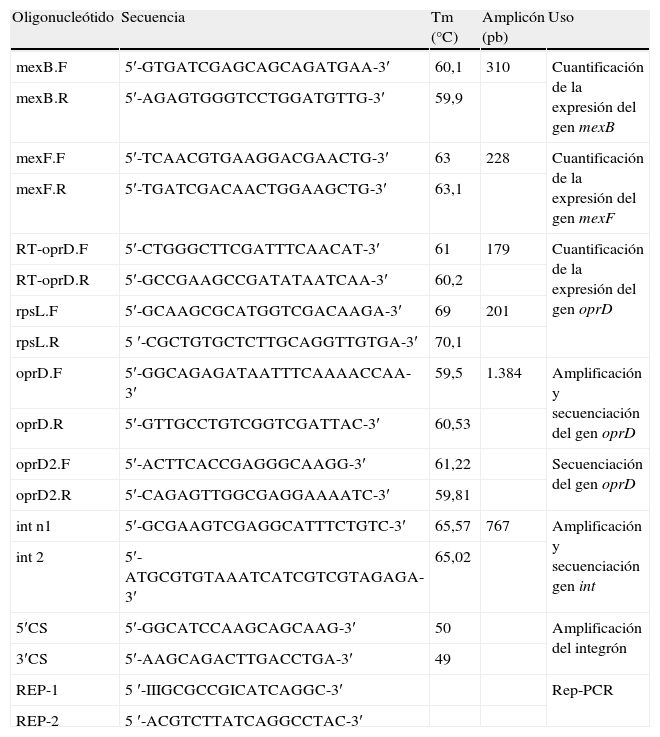
Las reacciones de amplificación y secuenciación de los integrones que codifican el blaVIM se realizaron con los oligonucleótidos 5′CS y 3′CS (tabla 2) que hibridan con regiones conservadas de los extremos del integrón. También se ha diseñado otro grupo de oligonucleótidos (no mostrados) para secuenciar la región interna del integrón.
Oligonucleótidos usados en este trabajo
| Oligonucleótido | Secuencia | Tm (°C) | Amplicón (pb) | Uso |
| mexB.F | 5′-GTGATCGAGCAGCAGATGAA-3′ | 60,1 | 310 | Cuantificación de la expresión del gen mexB |
| mexB.R | 5′-AGAGTGGGTCCTGGATGTTG-3′ | 59,9 | ||
| mexF.F | 5′-TCAACGTGAAGGACGAACTG-3′ | 63 | 228 | Cuantificación de la expresión del gen mexF |
| mexF.R | 5′-TGATCGACAACTGGAAGCTG-3′ | 63,1 | ||
| RT-oprD.F | 5′-CTGGGCTTCGATTTCAACAT-3′ | 61 | 179 | Cuantificación de la expresión del gen oprD |
| RT-oprD.R | 5′-GCCGAAGCCGATATAATCAA-3′ | 60,2 | ||
| rpsL.F | 5′-GCAAGCGCATGGTCGACAAGA-3′ | 69 | 201 | |
| rpsL.R | 5 ′-CGCTGTGCTCTTGCAGGTTGTGA-3′ | 70,1 | ||
| oprD.F | 5′-GGCAGAGATAATTTCAAAACCAA-3′ | 59,5 | 1.384 | Amplificación y secuenciación del gen oprD |
| oprD.R | 5′-GTTGCCTGTCGGTCGATTAC-3′ | 60,53 | ||
| oprD2.F | 5′-ACTTCACCGAGGGCAAGG-3′ | 61,22 | Secuenciación del gen oprD | |
| oprD2.R | 5′-CAGAGTTGGCGAGGAAAATC-3′ | 59,81 | ||
| int n1 | 5′-GCGAAGTCGAGGCATTTCTGTC-3′ | 65,57 | 767 | Amplificación y secuenciación gen int |
| int 2 | 5′-ATGCGTGTAAATCATCGTCGTAGAGA-3′ | 65,02 | ||
| 5′CS | 5′-GGCATCCAAGCAGCAAG-3′ | 50 | Amplificación del integrón | |
| 3′CS | 5′-AAGCAGACTTGACCTGA-3′ | 49 | ||
| REP-1 | 5 ′-IIIGCGCCGICATCAGGC-3′ | Rep-PCR | ||
| REP-2 | 5 ′-ACGTCTTATCAGGCCTAC-3′ |
Rep-PCR: reacción en cadena de la polimerasa repetida; Tm: temperatura.
La actividad hidrolítica de los carbapenémicos se determinó mediante ensayos espectrofotométricos de acuerdo con lo descrito anteriormente5. De forma breve, las reacciones se llevaron a cabo en tampón de 50mM hepes (Sigma, Madrid, España), pH 7,0 suplementado con 5μM de sulfato de cinc (Sigma, Madrid, España) a 25°C en un volumen de 1ml. La hidrólisis del IMP se midió a 297nm usando una concentración inicial de 150mM. En todas las reacciones se emplearon como controles positivo y negativo P. aeruginosa COL-1 productora de VIM-27 y P. aeruginosa ATCC 27853, respectivamente.
El gen oprD que codifica una porina de membrana externa se amplificó y se secuenció con los oligonucleótidos oprD.F y oprD.R (tabla 2).
La expresión de los genes mexB, mexF y oprD se cuantificó mediante RT-PCR (real time polymerase chain reaction 'reacción en cadena de la polimerasa en tiempo real'). Se partió del ácido ribonucleico (ARN) total obtenido con el equipo RNeasy (Qiagen, Barcelona, España); para eliminar el posible ADN contaminante se trataron las muestras con desoxirribonucleasa de acuerdo con las instrucciones del fabricante. La concentración y la calidad de las muestras se determinaron con el sistema Experion Automated Electrophoresis System (Bio-Rad, California, EE. UU.). Un μg de ARN total de cada muestra se transcribió con el equipo i-script® Select cDNA Synthesis (Bio-Rad, California, EE. UU.); a continuación, los ADN complementarios de los genes mexB, mexF y oprD se cuantificaron usando el sistema ABI 7000 Real Time PCR (Applied Biosystems, California, EE. UU.). Para la reacción de amplificación se usó el equipo iTaq® SYBR Green with Rox (Bio-Rad, California, EE. UU.). Los oligonucleótidos empleados para la amplificación de estos genes aparecen indicados en la tabla 2. Para corregir los posibles errores en la cuantificación debidos a diferencias en la cantidad inicial de ARN total empleado y así comparar los resultados de las diferentes muestras se eligió como control interno de la reacción el gen ribosómico rpsL8. Los resultados se presentan como la relación de la expresión entre los genes estudiados (mexB, mexF o oprD) y el gen de referencia (rpsL)9. La expresión relativa de cada gen en cada uno de los aislados se compara con respecto a la expresión del mismo gen en la cepa control P. aeruginosa ATCC 27853. Se considera que los genes mexB y mexF se encuentran sobreexpresados en un aislado clínico con respecto a la cepa parental cuando hay un incremento en la expresión de mexB superior o igual a 2 órdenes de magnitud y de mexF superior o igual a 10 órdenes de magnitud. Estos valores se encuentran dentro del rango de otros estudios1.
La P. aeruginosa JFL30 y la P. aeruginosa JFL0510 son cepas de referencia que sobreexpresan los genes que codifican las bombas de expulsión MexAB-OprM y MexEF-OprN, respectivamente y se han empleado como controles positivos.
La relación clonal de los aislados productores de MBL se analizó mediante detección de regiones extragénicas palindrómicas repetidas con PCR (Rep-PCR)11 con los oligonucleótidos REP-1 y REP-2. Los aislados se han considerado no relacionados genéticamente cuando se han detectado 2 o más bandas de diferencia entre éstos.
Resultados y discusiónDurante los 2 años de estudio, se aislaron en el laboratorio 163 aislados clínicos no sensibles a IMP o a MPM (CIM para IMP o para MPM superior o igual a 8mg/l). En 9 de los aislados con CIM de IMP de 32 a 128μg/ml y de MPM de 16 a 64μg/ml, (tabla 1) se ha detectado la presencia de una MBL mediante el método fenotípico de microdilución en caldo. Este resultado se ha corroborado mediante Multiplex PCR, obteniéndose en los 9 aislados un amplicón de 380pb. La secuenciación de este fragmento ha hecho posible la identificación del gen blaVIM−2 en todos los amplificados.
Los aislados fueron también resistentes a cefepima y a ceftacidima, y todos éstos menos uno (HUMV-2470) son sensibles a aztreonam. Salvo este último, los aislados presentan, pues, el patrón característico de producción de MBL. Como en otros casos fueron también resistentes a los aminoglucósidos evaluados (gentamicina, tobramicina y amikacina) y mostraron un alto grado de resistencia a ciprofloxacina (CIM > 32μg/ml). Todos fueron sensibles a colistina (tabla 1).
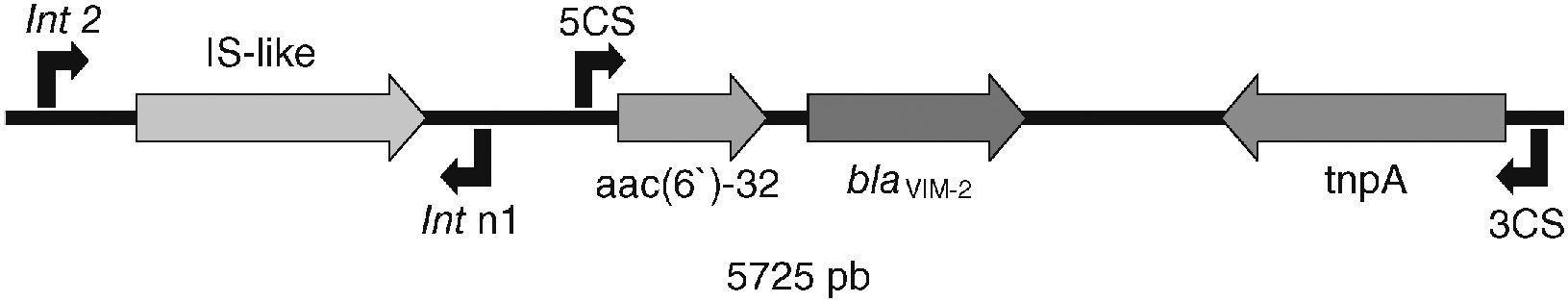
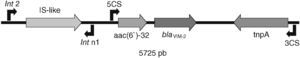
La amplificación mediante PCR del gen de la integrasa intI1 con los oligonucleótidos int n1 e int2 (tabla 2) permitió obtener un fragmento de un tamaño mayor al esperado en los 9 aislados objeto de estudio. La secuenciación del fragmento ha hecho posible identificar la presencia de una secuencia de inserción de 1,1 kb (IS-like element) que interrumpe el gen de la integrasa (fig. 1). Esto se ha descrito recientemente en cepas de P. aeruginosa portadoras de MBL aisladas en España por Gutiérrez et al2.
Estructura del integrón que contiene los aislados HUMV-2121 y HUMV-2633. Aparecen señalados con flechas los sitios en los que alinean los oligonucleótidos 5′CS y 3′CS empleados para la amplificación de los casetes de resistencia y la transposasa, así como los oligonucleótidos int n1 e int 2 usados para la amplificación del gen de la integrasa.
El análisis clonal que se realizó mediante Rep-PCR englobó en un único grupo a los 9 aislados de P. aeruginosa. Se han seleccionado 2 de éstos (HUMV-2121 y HUMV-2633) como representantes para estudios posteriores.
Para la secuenciación completa del integrón en los aislados HUMV-2121 y HUMV-2633 se ha amplificado en primer lugar la región que contiene los casetes de resistencia con oligonucleótidos (tabla 2) que hibridan con las regiones conservadas de los extremos 5′CS y 3′CS del integrón (fig. 1). El amplicón obtenido tiene un tamaño de 3.552pb; el gen blaVIM−2 se encuentra precedido por otro gen que codifica la acetiltransferasa aac (6′) 32 recientemente descrita2, que confiere resistencia a gentamicina, tobramicina y amikacina. Este resultado explica la resistencia a aminoglucósidos que se ha señalado previamente. En el extremo 3′CS, el integrón contiene un gen que codifica una transposasa. Todos los datos indican que se trata del mismo integrón descrito por primera vez por Gutiérrez et al2. Los ensayos de conjugación llevados a cabo utilizando como donadores los aislados clínicos HUMV-2121 y HUMV-2633 y como recipiente la cepa Escherichia coli DH5α no han permitido en ningún caso obtener transconjugantes resistentes a IMP. El plásmido obtenido a partir de las cepas HUMV-2121 y HUMV-2633 se introdujo mediante transformación química en E. coli DH5α y se observó que la CIM de IMP en E. coli DH5α pasa de 0,19μg/ml a 1μg/ml en la misma cepa con el plásmido. Este resultado indica la localización plasmática del gen blaVIM−2 y, por tanto, del integrón que lo contiene.
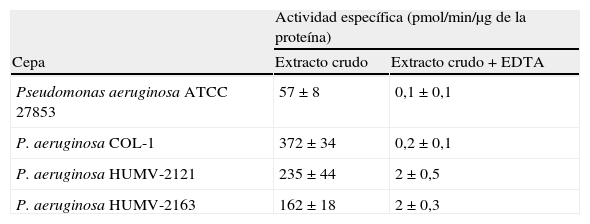
La actividad de hidrólisis del IMP observada en extractos crudos de los aislados HUMV-2121 y HUMV-2633 fue de 235 ± 44pmol/min/μg y de 162 ± 18pmol/min/μg, respectivamente; esta capacidad de hidrólisis es totalmente inhibida tras incubar las muestras con 5mM de EDTA (tabla 3). Estos resultados indican que VIM-2 se expresa en las cepas estudiadas.
Actividad de hidrólisis del imipenem en extractos crudos
| Actividad específica (pmol/min/μg de la proteína) | ||
| Cepa | Extracto crudo | Extracto crudo + EDTA |
| Pseudomonas aeruginosa ATCC 27853 | 57 ± 8 | 0,1 ± 0,1 |
| P. aeruginosa COL-1 | 372 ± 34 | 0,2 ± 0,1 |
| P. aeruginosa HUMV-2121 | 235 ± 44 | 2 ± 0,5 |
| P. aeruginosa HUMV-2163 | 162 ± 18 | 2 ± 0,3 |
Actividad de hidrólisis del imipenem en extractos crudos de Pseudomonas aeruginosa ATCC 27853, P. aeruginosa COL-1 (control positivo), de los aislados clínicos HUMV-2121 y HUMV-2633. en ausencia y presencia de EDTA.
EDTA: ethylene diamine tetra-acetic 'ácido edético'.
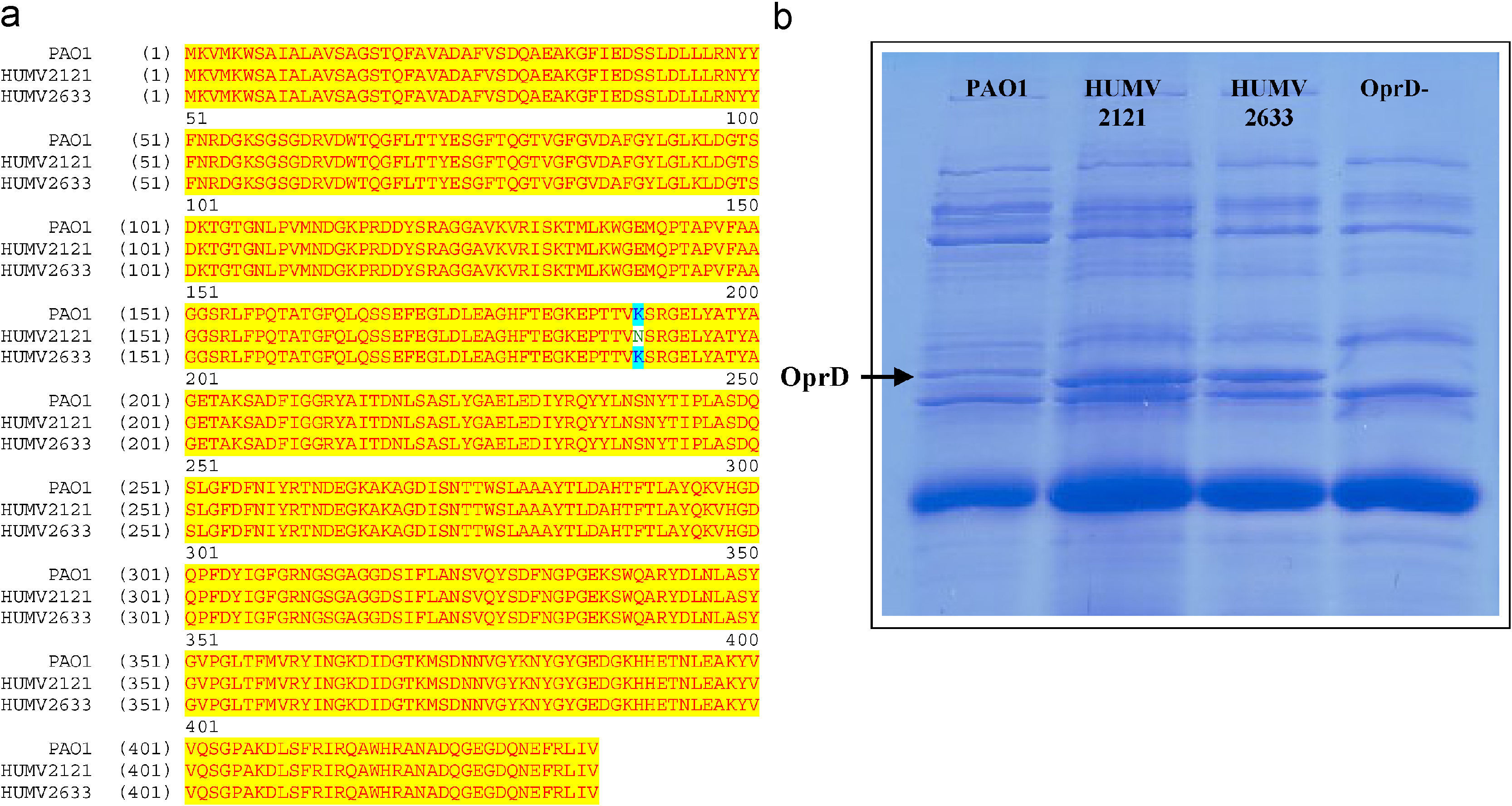
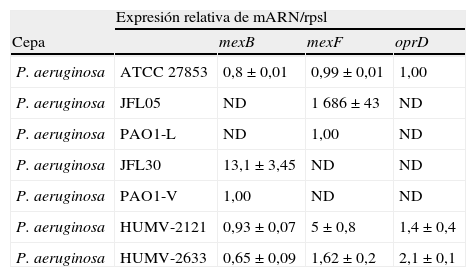
La porina OprD es el canal de entrada de los carbapenémicos de uso clínico en P. aeruginosa. Las mutaciones del gen oprD son bastante frecuentes y se relacionan de forma directa con la resistencia a carbapenémicos en esta especie12. Aunque algunas cepas de P. aeruginosa que producen MBL poseen también mutaciones de oprD, no se ha observado esta asociación en estas cepas (fig. 2). En los 2 aislados estudiados, HUMV-2121 y HUMV-2633, la secuencia y la expresión del gen oprD no presenta diferencias con respecto a la cepa de referencia P. aeruginosa ATCC 27853. Además, el estudio de las proteínas de membrana externa de ambos aislados (separadas mediante electroforesis en geles de poliacrilamida con SDS) permitió comprobar la presencia de una proteína de 46 kD, compatible con la expresión de OprD (datos no mostrados), y los valores de expresión del gen oprD medidos por RT-PCR en los aislados HUMV-2121 y HUMV-2633 no muestran diferencias estadísticamente significativas con respecto a los de la cepa de referencia (tabla 4). Todos estos datos indican que OprD no está implicada en la resistencia a carbapenémicos en los aislados clínicos de este estudio.
Expresión relativa de los genes mexB , mexF y oprD
| Expresión relativa de mARN/rpsl | ||||
| Cepa | mexB | mexF | oprD | |
| P. aeruginosa | ATCC 27853 | 0,8 ± 0,01 | 0,99 ± 0,01 | 1,00 |
| P. aeruginosa | JFL05 | ND | 1 686 ± 43 | ND |
| P. aeruginosa | PAO1-L | ND | 1,00 | ND |
| P. aeruginosa | JFL30 | 13,1 ± 3,45 | ND | ND |
| P. aeruginosa | PAO1-V | 1,00 | ND | ND |
| P. aeruginosa | HUMV-2121 | 0,93 ± 0,07 | 5 ± 0,8 | 1,4 ± 0,4 |
| P. aeruginosa | HUMV-2633 | 0,65 ± 0,09 | 1,62 ± 0,2 | 2,1 ± 0,1 |
Incremento relativo en la expresión de mARN del gen mexB (MexAB-oprM) respecto a P. aeruginosa PAO1-V, mexF (MexEF-OprN) respecto a P. aeruginosa PAO1-L y oprD (OprD) respecto a P. aeruginosa ATCC 27853.
mARN: messenger ribonucleic acid 'ácido ribonucleico mensajero'; ND: No definido.
La expresión de la bomba de expulsión MexAB-OprM se relaciona con la expulsión del MPM y su sobreexpresión en cepas clínicas se traduce en un aumento de la CIM de este antimicrobiano13. Por otra parte, la sobreexpresión de MexEF-OprN suele asociarse a la represión de la expresión de oprD, siendo este último hecho (y no la actividad de la bomba propiamente dicha) la causa de un aumento de la CIM de IMP14. En los 2 aislados, HUMV-2121 y HUMV-2633, los valores de expresión de ARN mensajero de mexB y mexF determinados mediante RT-PCR no se observaron diferencias significativas con respecto a P. aeruginosa ATCC 27853 (tabla 4). En conjunto, la presencia de la MBL VIM-2 en estas cepas es suficiente para justificar su resistencia a carbapenémicos. Ninguno de los otros mecanismos de resistencia estudiados contribuye al fenotipo de resistencia a estos antimicrobianos.
Aunque la prevalencia de clones epidémicos multirresistentes de P. aeruginosa productores de MBL es todavía baja en el hospital, se sigue llevando a cabo una vigilancia continua que permita su detección, con el objetivo de prevenir su diseminación. Hay que tener en cuenta, además, que la existencia de integrones que contienen casetes de resistencia puede contribuir a agravar el problema en otras especies. Durante el período de estudio, además de los 9 aislados de P. aeruginosa que se describen aquí, también se han podido identificar otros 3 aislados clínicos de Pseudomonas putida procedentes de hemocultivos que contienen un integrón de clase 1 con los genes blaVIM−2 y aacA4 (manuscrito en preparación).
Los aislados clínicos de P. aeruginosa y P. putida productores de VIM-2 suponen un grave problema terapéutico que es necesario controlar, tanto para reducir su expansión a nuevos pacientes como para prevenir la diseminación de la resistencia a otros microorganismos capaces de causar infecciones nosocomiales.
El Ministerio de Sanidad y Consumo, el Instituto de Salud Carlos III (proyecto PI050690), la Red Española de Investigación de Enfermedades Infecciosas (REIPI RD06/0008) y el Instituto de Formación e Investigación Marqués de Valdecilla (IFIMAV) financiaron este estudio.