Se estudiaron los genotipos circulantes de Chlamydia trachomatis que causan infección en nuestra área geográfica con el fin de detectar posibles peculiaridades epidemiológicas.
MétodosSe genotiparon 177 cepas obtenidas entre 2006 y 2008 empleando una reacción en cadena de la polimerasa con cebadores del gen ompA y posterior secuenciación.
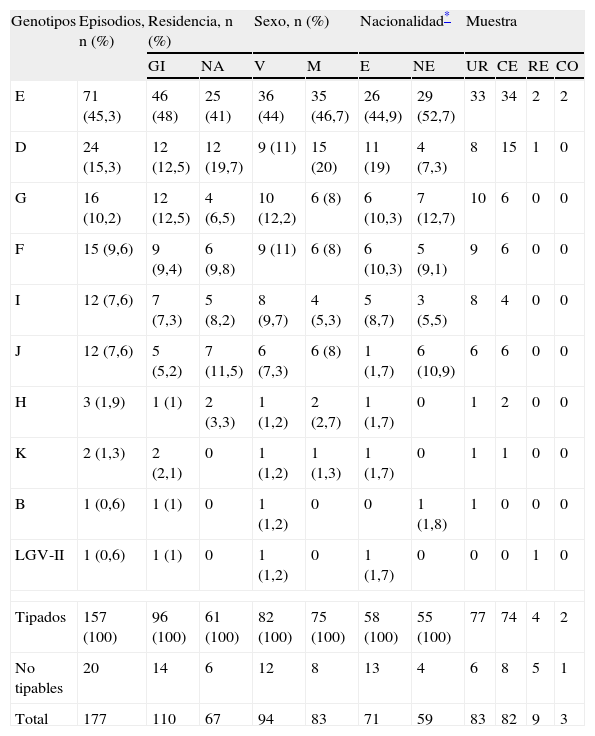
ResultadosLos genotipos más frecuentes fueron el E (45,3%), el D (15,3%), el G (10,2%) y el F (9,6%). Otros genotipos encontrados fueron B, H, I, J, K y LGV II.
ConclusiónLa técnica empleada tuvo una alta rentabilidad (89%).
Circulating Chlamydia trachomatis genotypes that cause infection in our geographic area were studied with the aim of detecting possible epidemiological peculiarities.
MethodsA total of 177 strains obtained between 2006 and 2008 were genotyped using a PCR with primers targeting the ompA gene, and later sequenced.
ResultsThe most frequent genotypes were: E (45.3%), D (15.3%), G (10.2%) and F (9.6%). Other genotypes found were: B, H, I, J, K and LGV II.
ConclusionThe molecular assay used had a high yield (89%).
El interés epidemiológico de las infecciones por Chlamydia trachomatis ha aumentado notablemente en los últimos años. La C. trachomatis es ya la principal causa de infecciones de transmisión sexual (ITS), que puede pasar inadvertida y ocasionar graves complicaciones1. Según la técnica empleada se clasifica en serotipos y genotipos. El serotipado se realiza con anticuerpos monoclonales epítopo específicos frente a la MOMP (major outter membrane protein ‘proteína principal de la membrana externa’), pero está en desuso debido a la complejidad y baja rentabilidad del cultivo de C. trachomatis. Actualmente hay varios métodos de genotipado, entre los que destacan la PCR (polymerase chain reaction 'reacción en cadena de la polimerasa') convencional o anidada seguida de secuenciación, PCR en tiempo real empleando sondas específicas y el análisis del polimorfismo de longitud de los fragmentos de restricción (RFLP, restriction fragment length polymorphism)2,3.
Debido a la gran diversidad existente de genotipos, el genotipado de C. trachomatis puede resultar de interés por varios motivos, entre los que cabe destacar: conocer su distribución geográfica, facilitar el seguimiento terapéutico y de contactos (trazabilidad), dilucidar si algunos genotipos se asocian más a ciertas manifestaciones clínicas y aportar información útil para el desarrollo de vacunas. El objetivo de este trabajo fue estudiar los genotipos circulantes de C. trachomatis en nuestra área geográfica, en una época en que la población inmigrante todavía era inferior al 10%, con el fin de detectar posibles peculiaridades epidemiológicas que originen futuras líneas de investigación.
Material y métodosEntre enero de 2006 y agosto de 2008 se analizaron 1.500 muestras (878 de cérvix, 495 uretrales, 99 rectales, 11 conjuntivales y 17 de otras localizaciones) de 1.299 episodios de 1.176 pacientes (451 varones y 725 mujeres) con una edad media de 33 años (mediana de 31 años, rango de 0,4 a 83 años). Se consideró que las muestras de un mismo paciente recibidas en 6 meses correspondieron a un mismo episodio, salvo que en ese período tuviera una primera muestra negativa y otra segunda positiva. Las muestras procedieron principalmente de pacientes sintomáticos en los que se sospechaba ITS, tanto de urgencias hospitalarias como de consultas hospitalarias o ambulatorias (mayoritariamente de una consulta de ITS), y de pacientes asintomáticos (aproximadamente uno de cada 3) con exposiciones de riesgo que acudieron para realizarse controles. También se incluyeron en el estudio 67 muestras de diferentes episodios de pacientes ya diagnosticados en Navarra. Se recogieron datos clinicoepidemiológicos como sexo, edad, nacionalidad, residencia, conductas de riesgo, localización de la infección y tipo de muestra, aunque por razones de confidencialidad, en algunos pacientes los datos epidemiológicos fueron incompletos.
La obtención y transporte de las muestras se realizó con el medio ViralPack (Biomedics). Se emplearon 2 métodos de obtención de ácidos nucleicos a partir de las muestras. Desde enero de 2006 hasta diciembre de 2007 y en todas las muestras procedentes de Navarra se realizó una extracción manual de las muestras con el equipo Amplicor CT/NG Specimen Preparation (Roche). Desde diciembre de 2007 se utilizó un método de extracción automatizada con el equipo NucliSens en el robot EasyMAG (BioMérieux). Para la detección se empleó el COBAS TaqMan CT Test (Roche), cuya diana es el plásmido críptico de C. trachomatis. En el momento de la realización de este trabajo este método no detectaba la variante con una deleción de 377pb en el plásmido críptico, recientemente descrita en Suecia, si bien su hallazgo en otros países europeos ha sido anecdótico4. Para el genotipado de las muestras positivas se realizó una PCR anidada de un fragmento del gen ompA empleando los cebadores descritos por Lysén et al2 a partir del ácido desoxirribonucleico extraído en el robot easyMAG (BioMérieux). Los amplicones se secuenciaron mediante la utilización del cebador MOMP87 en un ABIPRISM 3130 (Applied Biosystems). Se obtuvieron secuencias de unos 800 nucleótidos que se analizaron en el Basic Local Alignment and Search Tool (http://www.ncbi.nlm.nih.gov/blast/Blast.cgi).
ResultadosSe estudiaron los genotipos de C. trachomatis detectados en 177 episodios (tabla 1). Las muestras procedieron de 173 pacientes (91 varones y 82 mujeres) con una edad media de 30 años. El 55% eran sujetos autóctonos y el 45% eran inmigrantes. Se identificó el genotipo de 157 de los 177 episodios (89%). En 115 episodios se pudo secuenciar el amplicón obtenido en la primera PCR y en 42 episodios fue necesario realizar la PCR anidada. Globalmente, el genotipo más frecuente fue el E (en 71 de 157 [45,3%]) seguido de los genotipos D (15,3%), G (10,2%), F (9,6%), I (7,6%), J (7,6%), H (1,9%), K (1,3%), B (0,6%) y LGV-II (0,6%). No hubo diferencias estadísticamente significativas en los porcentajes obtenidos en Gipuzkoa y Navarra. En Gipuzkoa el genotipo G fue el segundo más frecuente después del E y 9 de 12 episodios (75%) correspondieron a varones. También en el grupo de varones y en inmigrantes el genotipo más frecuente después del E fue el G (el 12,2 y el 12,7% respectivamente).
Número y porcentaje de episodios genotipados
| Genotipos | Episodios, n (%) | Residencia, n (%) | Sexo, n (%) | Nacionalidad* | Muestra | ||||||
| GI | NA | V | M | E | NE | UR | CE | RE | CO | ||
| E | 71 (45,3) | 46 (48) | 25 (41) | 36 (44) | 35 (46,7) | 26 (44,9) | 29 (52,7) | 33 | 34 | 2 | 2 |
| D | 24 (15,3) | 12 (12,5) | 12 (19,7) | 9 (11) | 15 (20) | 11 (19) | 4 (7,3) | 8 | 15 | 1 | 0 |
| G | 16 (10,2) | 12 (12,5) | 4 (6,5) | 10 (12,2) | 6 (8) | 6 (10,3) | 7 (12,7) | 10 | 6 | 0 | 0 |
| F | 15 (9,6) | 9 (9,4) | 6 (9,8) | 9 (11) | 6 (8) | 6 (10,3) | 5 (9,1) | 9 | 6 | 0 | 0 |
| I | 12 (7,6) | 7 (7,3) | 5 (8,2) | 8 (9,7) | 4 (5,3) | 5 (8,7) | 3 (5,5) | 8 | 4 | 0 | 0 |
| J | 12 (7,6) | 5 (5,2) | 7 (11,5) | 6 (7,3) | 6 (8) | 1 (1,7) | 6 (10,9) | 6 | 6 | 0 | 0 |
| H | 3 (1,9) | 1 (1) | 2 (3,3) | 1 (1,2) | 2 (2,7) | 1 (1,7) | 0 | 1 | 2 | 0 | 0 |
| K | 2 (1,3) | 2 (2,1) | 0 | 1 (1,2) | 1 (1,3) | 1 (1,7) | 0 | 1 | 1 | 0 | 0 |
| B | 1 (0,6) | 1 (1) | 0 | 1 (1,2) | 0 | 0 | 1 (1,8) | 1 | 0 | 0 | 0 |
| LGV-II | 1 (0,6) | 1 (1) | 0 | 1 (1,2) | 0 | 1 (1,7) | 0 | 0 | 0 | 1 | 0 |
| Tipados | 157 (100) | 96 (100) | 61 (100) | 82 (100) | 75 (100) | 58 (100) | 55 (100) | 77 | 74 | 4 | 2 |
| No tipables | 20 | 14 | 6 | 12 | 8 | 13 | 4 | 6 | 8 | 5 | 1 |
| Total | 177 | 110 | 67 | 94 | 83 | 71 | 59 | 83 | 82 | 9 | 3 |
CE: cérvix; CO: conjuntival; E: española; GI: Gipuzkoa; M: mujeres; NA: Navarra; NE: no española; RE: rectal; UR: uretral; V: varones.
Las infecciones por C. trachomatis son un importante problema de salud pública mundial y en Europa su tasa de incidencia presenta una tendencia ascendente, con cifras de 100 casos por cada 100.000 habitantes (510 por cada 100.000 en el grupo de edad entre 15 y 24 años)1.
La distribución de los grupos genotipados en este trabajo fue similar a la descrita en otras partes del mundo, quizás con una mayor frecuencia relativa del genotipo G (especialmente en las cepas de Gipuzkoa)2,3. Los genotipos D-K son los más frecuentes en Occidente y, de ellos, el genotipo E es el más prevalente, lo que también se observó en la serie del presente estudio (45%). Estos genotipos causan generalmente enfermedades genitales (uretritis, epididimitis, cervicitis, salpingitis, etc.) y sus complicaciones (enfermedad inflamatoria pélvica, embarazo ectópico, infertilidad, etc.), pero también pueden estar relacionados con otras enfermedades, como el síndrome de Reiter, neumonía y conjuntivitis (2 casos por el genotipo E)1,5. En el presente trabajo los síntomas más referidos fueron uretritis en los varones y dolor abdominal o sospecha de enfermedad inflamatoria pélvica en las mujeres. Tres episodios sucedieron en gestantes con ITS previas. Se detectaron 12 episodios en mujeres asintomáticas al realizarse controles por tener factores de riesgo de contraer ITS. Los genotipos LGV-I, LGV-II y LGV-III se asocian al linfogranuloma venéreo. En este estudio se detectó el genotipo LGV-IIb en una muestra de un varón homosexual de 46 años con rectitis, proveniente de Vitoria, que refirió como factor de riesgo una pareja de origen sudamericano. El linfogranuloma venéreo es una infección más frecuente en países tropicales y subtropicales (África, Asia, América del Sur y Caribe), aunque en los últimos años se han descrito numerosos casos en Europa, en varones homosexuales que presentaban típicamente proctitis, asociados al genotipo LGV-IIb6,7. Los genotipos A, B, Ba y C se relacionan con el tracoma, una de las principales causas de ceguera en el mundo y que se distribuye fundamentalmente en África, Asia, América Central y del Sur y Australia. En un episodio se detectó el genotipo B en una muestra uretral de un varón de 26 años de Europa del Este con uretritis y coinfección por Haemophilus parainfluenzae. Su detección, aunque en una localización inusual, así como la del LGV-IIb, pone de manifiesto la capacidad de exportación geográfica de cepas de C. trachomatis y la validez del método de genotipado empleado para detectar la amplia variedad de genotipos, incluso los poco frecuentes en esta zona. En este estudio, la representación de la población inmigrante (45%) fue mayor que la que le correspondería por su presencia en la población general (inferior al 10%), ya que a una parte importante de los pacientes se los atendió en una consulta de ITS en la que uno de los motivos habituales de consulta fue el ejercicio de la prostitución.
Se han descrito varios métodos para el genotipado de C. trachomatis. El más utilizado se basa en la secuenciación del gen ompA y en el análisis de las diferencias en sus 4 dominios variables2,3. La técnica utilizada en este trabajo emplea una PCR anidada convencional2. Jalal et al emplearon una PCR anidada en tiempo real que permitiría la detección de hasta un 10% de infecciones mixtas, pero con un costo superior debido a la utilización de sondas. El RFLP es otro método capaz de detectar infecciones mixtas, pero su poder discriminatorio y su reproducibilidad entre distintos laboratorios es menor. La estrategia empleada en este trabajo con la técnica de genotipado tuvo una rentabilidad alta (89%), aunque entre sus limitaciones se debe considerar que no discrimina infecciones mixtas. De las 20 cepas que no se pudieron genotipar, 15 correspondieron a muestras con detecciones en ciclos de amplificación altos y, por tanto, con baja carga bacteriana. Nueve de ellas no se detectaron empleando la extracción manual de ácidos nucleicos y sí con la extracción automatizada que permitió, desde diciembre de 2007, aumentar la sensibilidad de detección de C. trachomatis8.
Este es el primer estudio sobre genotipado de C. trachomatis realizado en España. El genotipado basado en el gen ompA permitió conocer la distribución de los genotipos circulantes en un área del norte de España, confirmando el predominio del genotipo E. Sin embargo, para poder aportar una información epidemiológica más detallada (diferentes variantes genotípicas, trazabilidad de contactos) es necesario aplicar nuevas técnicas moleculares descritas recientemente que permitan una mayor resolución y discriminación genotípica9,10.