Estudiar la importancia de la correcta identificación a nivel de especie así como la interpretación de las pruebas de sensibilidad en aislados de Aeromonas spp. productoras de bacteriemia mediante los métodos convencionales rutinarios y los nuevos métodos moleculares.
Material y métodosEl estudio incluyó a 22 pacientes con bacteriemia por Aeromonas hydrophila grupo, identificadas mediante el sistema MicroScan. La identificación posterior a nivel de especie se realizó por espectrometría de masas y se confirmó mediante la secuenciación del gen rpoB.
La actividad de imipenem, cefotaxima, piperacilina/tazobactam, ciprofloxacino y cotrimoxazol se estudió por microdilución comercial y tiras de gradiente de antibiótico con bajo y alto inóculo. La detección de carbapenemasas se realizó mediante el test de Hodge modificado y su confirmación mediante la detección por PCR del gen cphA.
ResultadosSe identificaron 9 (40,9%) aislamientos como Aeromonas hydrophila, 8 (36,4%) como Aeromonas veronii y los 5 (22,7%) restantes como Aeromonas caviae.
La resistencia a los antibióticos betalactámicos mediante microdilución comercial y tiras de gradiente de CMI fue, respectivamente, del 36-50% para imipenem; del 4-56% para cefotaxima; y de 27-56% para piperacilina/tazobactam.
La concordancia entre el sistema automatizado y el sistema de difusión con tira de gradiente antibiótico fue, globalmente para las 3 especies, del 68% para imipenem, del 50% para cefotaxima y del 46% para piperacilina/tazobactam.
No se detectó resistencia a cotrimoxazol y ciprofloxacino por ambos métodos, aunque el 22,7% de las cepas fueron resistentes a ácido nalidíxico.
ConclusionesEs fundamental la identificación a nivel de especie de los aislamientos de Aeromonas spp. ya que la resistencia a betalactámicos es especie y método dependiente. Los altos porcentajes de resistencia antibiótica encontrados no aconsejan el uso de antibióticos betalactámicos y quinolonas como tratamiento empírico de la infección invasiva por Aeromonas ssp.
To assess the relevance of correct identification and interpretation of susceptibility testing of Aeromonas spp. bacteremia isolates using newly developed molecular methods in comparison to previous conventional methods.
Material and methodsThe study included 22 patients with bacteremia due to Aeromonas hydrophila group, microbiologically characterized using the MicroScan system. Further identification to species level was performed by mass spectrometry, and confirmed by sequencing the rpoB gene.
The MIC of imipenem, cefotaxime, piperacillin-tazobactam, ciprofloxacin and cotrimoxazole was studied using a commercial broth microdilution and antibiotic gradient strips with low and high inocula. Detection of carbapenemase production was performed using the modified Hodge test, and was confirmed by amplifying the cphA gene by PCR.
ResultsA total of 9 (40.9%) isolates were identified as Aeromonas hydrophila, 8 (36.4%) as Aeromonas veronii, and the remaining 5 (22.7%) isolates as Aeromonas caviae.
Resistance to beta-lactams according to both the commercial microdilution and MIC gradient strips methods was: 36%-50% to imipenem; 4%-56% to cefotaxime, and 27%-56% to piperacillin/tazobactam.
The agreement between results generated by the automated system and the diffusion antibiotic gradient strip was, for all 3 species, 68% for imipenem, 50% to cefotaxime, and 46% to piperacillin/tazobactam.
No resistance to cotrimoxazole and ciprofloxacin was found by either of the two methods, although 22.7% of the strains were resistant to nalidixic acid.
ConclusionsIt is essential to identify the isolates of Aeromonas spp. at the species level, due to the fact that beta-lactam resistance is species- and method-dependent. The high rate of resistance to beta-lactam and quinolones reduce their application as empiric treatments for invasive infection by Aeromonas ssp.
Aeromonas spp. son bacilos gramnegativos anaerobios facultativos ampliamente distribuidos en el medio ambiente, pudiéndose encontrar en numerosos ecosistemas acuáticos de agua dulce o salobre, así como en aguas residuales1.
A pesar de que el tracto gastrointestinal es la localización más frecuente de las infecciones producidas por Aeromonas spp., también se han comunicado infecciones de localización extraintestinal, principalmente infecciones del tracto biliar, peritonitis, infecciones de piel y tejidos blandos y bacteriemia1,2.
La bacteriemia y la septicemia son entidades relativamente frecuentes en el contexto de la infección por Aeromonas y generalmente afectan a pacientes con neoplasias hematológicas, enfermedad hepatobiliar u otras situaciones de inmunodepresión3–5. Aeromonas veronii (A. veronii), Aeromonas hydrophila (A. hydrophila) y Aeromonas caviae (A. caviae) son las especies implicadas en la bacteriemia6. No siempre es posible identificar la fuente primaria de la bacteriemia, pero parece razonable asumir que sea el tracto gastrointestinal. En los últimos años se ha observado un aumento de los episodios de bacteriemia asociados a infección nosocomial y/o a los cuidados sanitarios4,7, con una mortalidad muy alta en los pacientes inmunodeprimidos oscilando entre el 24 y el 68%4,7-9.
El tratamiento de la infección bacteriémica por Aeromonas spp. no está claramente definido. Fluoroquinolonas, cefalosporinas de tercera generación o cotrimoxazol son opciones de tratamiento, según UptoDate con un grado de evidencia 2C10, mientras que otras guías de terapéutica antimicrobiana incluyen también carbapenemas dentro de estas opciones11–13.
Estas recomendaciones se basan en que los aislamientos clínicos de Aeromonas se consideran sensibles in vitro a cefalosporinas de tercera generación y carbapenemas14–21. Sin embargo, la presencia en el género de diversas betalactamasas cromosómicas inducibles, como metalobetalactamasas, cefalosporinasas de clase C y penicilinasas de clase D, podrían conducir a una falta de eficacia terapéutica de estos antibióticos3,20. Por otro lado, la distribución de las betalactamasas es especie-dependiente21 y los métodos convencionales de identificación son poco precisos en la diferenciación de las especies; por ello la aplicación de nuevas técnicas moleculares como la espectrometría de masas podría ser de gran ayuda en la caracterización precisa del género a nivel de especie22–25.
Además, no existe acuerdo entre las distintas guías en los criterios de interpretación de la sensibilidad del género Aeromonas a los antibióticos. Así, la guía americana CLSI26 presenta puntos de corte y la guía europea EUCAST no los ofrece, al ser un tema sujeto a discusión.
El objetivo del presente trabajo ha sido llegar a una correcta identificación a nivel de especie haciendo uso de la tecnología MALDI-TOF27 basándonos en la secuenciación parcial del gen rpoB como método de referencia; así como la interpretación adecuada de las pruebas de sensibilidad en aislados de Aeromonas spp. productoras de bacteriemia comparando los métodos convencionales que se utilizan en la actualidad con los nuevos métodos moleculares.
Material y métodosAislamientos clínicosSe estudiaron 22 aislamientos de Aeromonas spp. procedentes de hemocultivos de pacientes ingresados en el hospital Virgen del Rocío en el periodo comprendido entre 2003 y 2013, que el sistema convencional de identificación (MicroScan, Siemens AG, Alemania) caracterizaba como A. hydrophila grupo.
IdentificaciónLa identificación definitiva de especie se realizó mediante secuenciación parcial del gen rpoB28,29 utilizando los cebadores Pasrpob-L (5′-GTGAAAGARTTCTTTGGTTC-3′) y Rpob-R (5′-TTGCATGTTNGNACCCAT-3′).
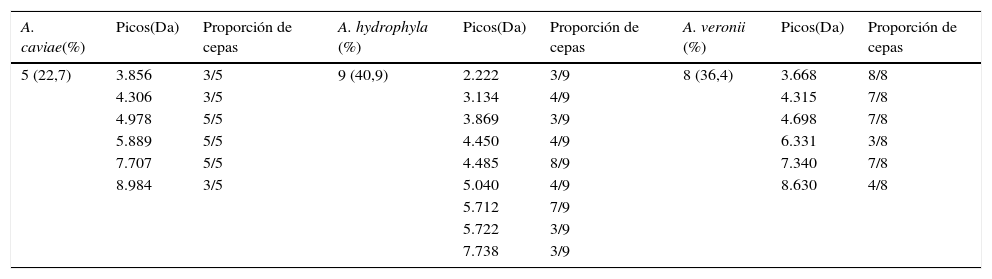
Asimismo, se procedió a una identificación a nivel de especie mediante un espectrómetro de masas Microflex II (Bruker Daltonik, Alemania) equipado con un láser de 60Hz directamente de la colonia y por extracción con protocolo estándar27, considerando una puntuación superior a 2,2 indicativa de identificación a nivel de especie. Adicionalmente se procedió a un análisis manual de los espectros obtenidos con el objetivo de identificar aquellos picos característicos de especie.
Sensibilidad a antimicrobianosLa CMI de imipenem, cefotaxima y piperacilina/tazobactam, así como de ciprofloxacino; cotrimoxazol y aminoglucósidos como amikacina y gentamicina se determinó mediante el método de microdilución en caldo en los paneles combo NC53 (MicroScan, Siemens AG, Alemania). Además, se estudió mediante difusión en disco en agar Mueller Hinton la sensibilidad a ácido nalidíxico. Los puntos de corte utilizados para establecer la categoría clínica se basaron en recomendaciones CLSI26.
Adicionalmente, se realizó la determinación de la CMI de imipenem, cefotaxima y piperacilina/tazobactam mediante difusión en agar con tiras de gradiente de antibiótico (Liofilchem, Italia) con 2 tamaños de inóculo (0,5 y 1,5 McFarland), según el método descrito por Wu y el CLSI26,28. Las cepas que presentaban colonias en el interior de la elipse de inhibición fueron consideradas resistentes con valores de CMI superiores a la concentración máxima de la tira utilizada.
El estudio fenotípico de producción de carbapenemasas se realizó mediante el test de Hodge modificado utilizando una cepa Escherichia coli ATCC 25922 y un disco de ertapenem de 10μg, según el protocolo descrito por Wu et al.28.
Detección del gen cphASe realizó mediante amplificación por PCR, siguiendo el protocolo establecido por Wu et al.28, utilizando los siguientes cebadores: cphA- F: 5′-GCT TAG AGC TCC TAA GGA GCA AGA TGA AAG GTT GG-3′ y cphA-R: 5′-GCA TAG GTA CCT TAT GAC TGG GGT GCG GCC TTG–3′. Este método, amplifica un fragmento de 720pb aproximadamente, que detecta mediante electroforesis en gel de agarosa al 1,2%.
ResultadosIdentificación de los aislamientos clínicosLos resultados de la secuenciación parcial del gen rpoB fueron los siguientes: 9 (40,9%) cepas se identificaron como A. hydrophila, 8 (36,4%) como A. veronii y las 5 (22,7%) restantes como A. caviae.
La espectrometría de masas permitió la identificación exacta a nivel de especie (puntuación>2,2) en 13 (59%) aislados empleando el protocolo de extracción, mientras que a partir de la colonia directa se identificaron 16 (73%) aislados.
El 100% de las cepas de A. veronii se identificaron de forma exacta por ambos métodos. Mediante el análisis manual de los espectros se obtuvieron una serie de picos característicos de especie que permitían la identificación de la especie (tabla 1). Nueve (40,9%) cepas fueron identificadas como A. hydrophila, 8 (36,4%) como A. veronii y las 5 (22,7%) restantes como A. caviae, obteniéndose los mismos resultados que mediante el método de referencia.
Identificación de las especies de Aeromonas obtenida mediante MALDI-TOF y análisis de los espectros
| A. caviae(%) | Picos(Da) | Proporción de cepas | A. hydrophyla (%) | Picos(Da) | Proporción de cepas | A. veronii (%) | Picos(Da) | Proporción de cepas |
|---|---|---|---|---|---|---|---|---|
| 5 (22,7) | 3.856 | 3/5 | 9 (40,9) | 2.222 | 3/9 | 8 (36,4) | 3.668 | 8/8 |
| 4.306 | 3/5 | 3.134 | 4/9 | 4.315 | 7/8 | |||
| 4.978 | 5/5 | 3.869 | 3/9 | 4.698 | 7/8 | |||
| 5.889 | 5/5 | 4.450 | 4/9 | 6.331 | 3/8 | |||
| 7.707 | 5/5 | 4.485 | 8/9 | 7.340 | 7/8 | |||
| 8.984 | 3/5 | 5.040 | 4/9 | 8.630 | 4/8 | |||
| 5.712 | 7/9 | |||||||
| 5.722 | 3/9 | |||||||
| 7.738 | 3/9 |
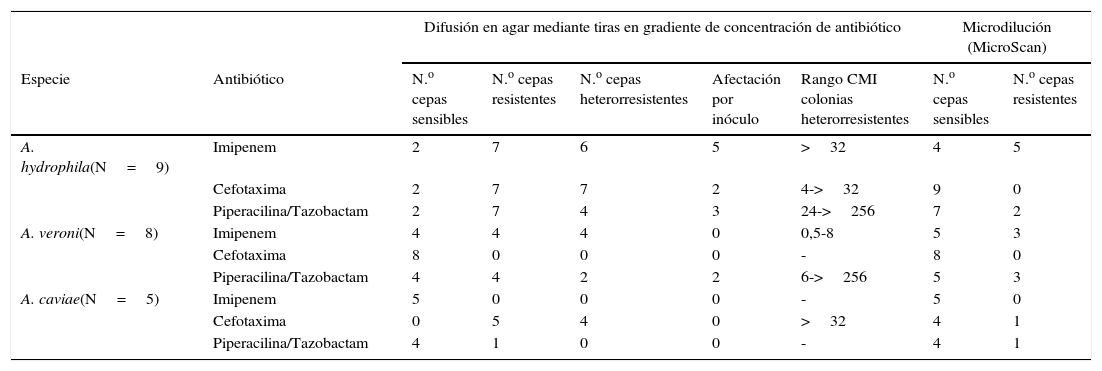
La aparición de colonias en el interior de la elipse de inhibición se consideró como criterio de resistencia con un valor de CMI mayor de la máxima concentración de la tira de gradiente antibiótico. De esta forma podemos ver cómo el fenómeno de heterorresistencia está implicado en la práctica totalidad de los casos de resistencia a imipenem y cefotaxima (10/11 y 11/12 respectivamente) y también en una proporción importante (6/12) de los casos de resistencia a piperacilina/tazobactam, obteniendo unos altos porcentajes de resistencia a los 3 antibióticos betalactámicos probados (tabla 2).
Actividad de imipenem, cefotaxima y piperacilina/tazobactam sobre las distintas especies de Aeromonas mediante gradiente de concentración de antibiótico y microdilución (MicroScan)
| Difusión en agar mediante tiras en gradiente de concentración de antibiótico | Microdilución (MicroScan) | |||||||
|---|---|---|---|---|---|---|---|---|
| Especie | Antibiótico | N.o cepas sensibles | N.o cepas resistentes | N.o cepas heterorresistentes | Afectación por inóculo | Rango CMI colonias heterorresistentes | N.o cepas sensibles | N.o cepas resistentes |
| A. hydrophila(N=9) | Imipenem | 2 | 7 | 6 | 5 | >32 | 4 | 5 |
| Cefotaxima | 2 | 7 | 7 | 2 | 4->32 | 9 | 0 | |
| Piperacilina/Tazobactam | 2 | 7 | 4 | 3 | 24->256 | 7 | 2 | |
| A. veroni(N=8) | Imipenem | 4 | 4 | 4 | 0 | 0,5-8 | 5 | 3 |
| Cefotaxima | 8 | 0 | 0 | 0 | - | 8 | 0 | |
| Piperacilina/Tazobactam | 4 | 4 | 2 | 2 | 6->256 | 5 | 3 | |
| A. caviae(N=5) | Imipenem | 5 | 0 | 0 | 0 | - | 5 | 0 |
| Cefotaxima | 0 | 5 | 4 | 0 | >32 | 4 | 1 | |
| Piperacilina/Tazobactam | 4 | 1 | 0 | 0 | - | 4 | 1 | |
Los valores de CMI encontrados en las colonias del interior de las elipses de inhibición fueron muy variables, oscilando desde la sensibilidad hasta la resistencia de alto nivel. En el caso de imipenem, con valores desde 0,5mg/L a >32mg/L, en el caso de cefotaxima desde 4mg/L hasta >32mg/L y desde 6mg/L hasta >256mg/L en el caso de piperacilina/tazobactam.
Asimismo, el efecto inóculo ha sido observado en una proporción variable de casos afectando tanto a cepas sensibles como resistentes. Este efecto se observa con mayor frecuencia en imipenem y piperacilina/tazobactam que con cefotaxima.
Existen diferencias notables entre las 3 especies (tabla 2). El mayor grado de resistencia lo encontramos con A. hydrophila siendo resistentes 7 de las 9 cepas estudiadas a los 3 betalactámicos con distinto grado de heterorresistencia y afectación del inóculo según el antibiótico considerado. Sin embargo, en A. veronii destaca la total sensibilidad a cefotaxima frente a un alto nivel de resistencia a imipenem y piperacilina/tazobactam, siendo el imipenem el antibiótico donde aparecen más casos de heterorresistencia y afectación por inóculo. Por último, imipenem y piperacilina/tazobactam muestran una buena actividad frente a A. caviae mientras que todas las cepas son resistentes a cefotaxima, desempeñando un papel muy importante en ello la heterorresistencia.
En la detección de carbapenemasas mediante el test de Hodge modificado, encontramos que fue positivo en 16 (72,7%) de las 22 cepas estudiadas: en 8 (89%) de las 9 cepas de A. hydrophila, en todas las cepas de A. veronii y en ninguna de las A. caviae.
La amplificación del gen cphA fue positiva en 14 de las 22 (63,6%) cepas estudiadas. Diferenciándose por especies, en ninguna de las A. caviae se detectó el gen, mientras que el resultado fue positivo en 7 de 9 (77,7%) A. hydrophila y en 7 de 8 (87,5%) A. veronii. La detección del gen cphA coincidió en su totalidad con el método de tiras de gradiente antibiótico en el caso de A. caviae y A. hydrophila; en la cual 6 de las 7 cepas resistentes a imipenem se detectaron por el fenómeno de la heterorresistencia. No obstante, en el caso de A. veronii, solo se detectaron mediante tiras de gradiente antibiótico 4 cepas con resistencia a imipenem, siendo 7 de ellas positivas para el gen cphA.
Los resultados de sensibilidad antimicrobiana obtenidos por los diferentes métodos se recogen en la tabla 2.
La concordancia entre el sistema automatizado y el sistema de difusión con tira de gradiente antibiótico fue, globalmente para las 3 especies, del 68% para imipenem, del 50% para cefotaxima y del 46% para piperacilina/tazobactam.
Veintiuna (95,4%) de las cepas fueron sensibles a cotrimoxazol y todas las cepas fueron sensibles a ciprofloxacino; aunque el 23% de las cepas fueron resistentes a ácido nalidíxico. En cuanto a los aminoglucósidos, se encontró un 100% de cepas sensibles a amikacina y un 88% de sensibilidad a gentamicina.
DiscusiónEste trabajo constata que la sensibilidad antibiótica en el género Aeromonas es especie y método dependiente3,21, por lo que la identificación precisa a nivel de especie tiene relevancia clínica y la espectrometría de masas podría ser una alternativa superior a los métodos fenotípicos convencionales.
Recientemente, se ha publicado un artículo25 en el que los picos característicos para las 3 especies estudiadas y los propuestos en nuestro trabajo coinciden parcialmente en el caso de A. hydrophila, y en el 100% de A. veronii y A. caviae, En el caso de A. hydrophila, de los 4 picos característicos que señalan en el trabajo, coincidimos en 2 y añadimos 7 picos más. No obstante, estas coincidencias se dan en menor grado22 o no se aprecian con otros trabajos publicados23.
A pesar de ello, la precisión en la identificación de nuestras cepas mediante MALDI-TOF queda constatada por la concordancia total con la secuenciación del gen rpoB.
A diferencia de los métodos basados en la amplificación de ácidos nucleicos y secuenciación, es un método sencillo, rápido y relativamente poco costoso, aunque son necesarias mejoras en las bibliotecas de los sistemas MALDI-TOF para la identificación automática de estos microorganismos y evitar la necesidad de un análisis manual.
El cotrimoxazol y las fluoroquinolonas suelen ser los fármacos de primera línea en el tratamiento de las infecciones graves por Aeromonas spp., pudiendo también usarse aminoglucósidos, cefalosporinas de tercera generación, piperacilina/tazobactam y carbapenemas, especialmente imipenem10–13. No obstante, existen discrepancias entre los diversos autores con respecto a la actividad y eficacia de los mismos4,6,18,20,21,30–32.
Los métodos automatizados convencionales no son capaces de detectar todos los posibles casos de resistencia en los antibióticos betalactámicos en Aeromonas spp., clasificando como sensibles a microorganismos resistentes.
Aeromonas spp. presenta distinto grado de heterorresistencia y afectación por el inóculo en función del antimicrobiano estudiado; así, es más frecuente en cefotaxima e imipenem que en piperacilina/tazobactam. Este hecho determina la falta de concordancia entre el método de microdilución automatizado y el de difusión con tira de gradiente de antibiótico. Esta variabilidad se debe a los diferentes tipos betalactamasas cromosómicas inducibles: metalobetalactamasa, cefalosporinasa de clase C y penicinilasa de clase D que posee Aeromonas spp., distribuidas según especie33. Estas enzimas se inducen mediante un sistema único de expresión coordinada, el sistema blrAB TCR34 y esta inducción en ocasiones es dependiente de inóculo28. Al aumentar el inóculo, se incrementa la CMI e incluso cambiaría la categoría clínica de sensible a resistente como podría ocurrir en las infecciones con una alta carga bacteriana.
En el caso de la cefotaxima, la heterorresistencia aparece en el 80% de las cepas de A. caviae y en el 77,7% de las cepas de A. hydrophila debido a la presencia de la betalactamasa cromosómica e inducible de clase C (AmpC), la cual no aparece en A. veronii. Sin embargo, los métodos automatizados habituales no detectan la heterorresistencia, obteniéndose un 96% de cepas sensibles, cifras muy similares a las descritas en estudios previos18,21,30,31–33. Sin embargo, ninguno de ellos utilizó la metodología necesaria para la detección de heterorresistencia. Por todo ello, el método de difusión con tira de gradiente de antibiótico es el que mejor puede detectar la posible aparición de estas cepas resistentes inducidas durante el tratamiento con cefalosporinas de tercera generación.
Con respecto a piperacilina/tazobactam, la literatura arroja resultados similares a cefotaxima; con altos niveles de sensibilidad encontrados en la mayoría de los estudios18,21,30,32. En nuestro trabajo los porcentajes de resistencia son iguales que para cefotaxima si tenemos en cuenta el global de las especies; no obstante, es el antibiótico donde encontramos menos casos de heterorresistencia, estando este hecho tal vez relacionado con la menor potencia como inductor de la piperacilina/tazobactam de las enzimas betalactamasas35.
Wu et al.28 evaluaron diversos métodos fenotípicos para la detección de carbapenemasas en este género, concluyendo que el test de Hodge modificado es el más sensible, al detectar el 97% de los aislamientos que poseían el gen cphA. Nuestros resultados coinciden en la especie A. caviae; en todas ellas fue negativo como era esperable ya que esta especie carece de carbapenemasa cromosómica. Sin embargo, en algunas cepas de A. hydrophila y A. veronni hemos detectado falsos positivos.
En las infecciones invasivas por A. caviae, imipenem puede ser un tratamiento efectivo y recomendable. En los casos de infección por A. veronii y A. hydrophila existe un porcentaje de cepas que se categorizan por los métodos habituales como sensibles, haciéndose evidente la importancia de la correcta identificación a nivel de especie.
La concordancia entre la amplificación del gen cphA y la detección del fenómeno de la heterorresistencia, en el caso de las resistencias inducibles a imipenem, ha demostrado la fiabilidad del método de tiras de gradiente antibiótico en las especies de A. hydrophila y A. caviae, pero no en A. veronii para la detección de carbapenemasas, lo que nos permite suponer que sería también adecuado para el resto de los antibióticos probados así como en todos aquellos que puedan afectarse por la inducción de las enzimas cromosómicas que albergan estas especies.
Finalmente, aunque el 100% de nuestras cepas es sensible a ciprofloxacino, el 23% de ellas presenta resistencia a ácido nalidíxico, lo que implica una mutación de primer paso en el gen gyrA que podría conducir una segunda mutación que invalide el tratamiento con ciprofloxacino4,6,18,21. Asimismo, cotrimoxazol y aminoglucósidos como amikacina y gentamicina pueden ser un tratamiento eficaz, como se recoge en distintas guías6,10,12,13, por presentar unas altas tasas de sensibilidad.
Para concluir, queremos resaltar la importancia de la correcta identificación de las especies de Aeromonas spp. más frecuentemente encontradas en clínica así como la interpretación adecuada de las pruebas de sensibilidad.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.