La espectrometría de masas MALDI-TOF es ya una herramienta de trabajo rutinaria en Microbiología Clínica, por su rapidez y fiabilidad en la identificación de microorganismos. Sus resultados están perfectamente contrastados en la identificación de bacterias y levaduras. La identificación de micobacterias y hongos filamentosos presenta mayor complejidad, por la mayor heterogeneidad de espectros dentro de cada especie. La metodología es algo más compleja, y la ampliación del número de especies de referencia, y del número de espectros de cada especie, serán cruciales para alcanzar mayor eficacia. La identificación directa a partir de hemocultivos se ha implantado dada su aportación al manejo de pacientes graves, pero su aplicación a otras muestras es más compleja.
Los medios de cultivos cromogénicos han supuesto también una aportación al diagnóstico rápido tanto en bacterias como en levaduras, ya que aceleran el diagnóstico, facilitan la detección de cultivos mixtos y permiten un diagnóstico rápido de especies resistentes.
MALDI-TOF mass spectrometry is now a routine resource in Clinical Microbiology, because of its speed and reliability in the identification of microorganisms. Its performance in the identification of bacteria and yeasts is perfectly contrasted. The identification of mycobacteria and moulds is more complex, due to the heterogeneity of spectra within each species. The methodology is somewhat more complex, and expanding the size of species libraries, and the number of spectra of each species, will be crucial to achieve greater efficiency. Direct identification from blood cultures has been implemented, since its contribution to the management of severe patients is evident, but its application to other samples is more complex.
Chromogenic media have also contributed to the rapid diagnosis in both bacteria and yeast, since they accelerate the diagnosis, facilitate the detection of mixed cultures and allow rapid diagnosis of resistant species.
La introducción de la espectrometría de masas (EM) matrix-assisted laser desorption/ionization time of flight (MALDI-TOF) ha sido, con toda probabilidad, el cambio tecnológico de mayor calado acaecido en la Microbiología Clínica en la última década. En pocos años ha pasado de ser una prometedora novedad, a ser una tecnología totalmente integrada en la actividad clínica diaria y disponible, en nuestro país, en los Servicios de Microbiología de numerosos centros hospitalarios1. Para ello ha sido necesario que los espectrómetros de masas sufrieran una importante evolución tecnológica. A este respecto, hubo dos avances que resultaron cruciales. En primer lugar, el diseño del sistema Time of Flight (TOF) por W.E. Stephens, en 1946, que permitió la separación de masas diferentes. Al acelerarse los iones en un campo eléctrico y adquirir todos la misma energía cinética, la velocidad que adquieran, y por tanto el tiempo empleado en recorrer el tubo de vacío, depende de la masa de la molécula ionizada, que podrá ser inferida a partir del tiempo empleado en dicho recorrido.
El segundo hito fue el desarrollo de métodos que permitían ionizar proteínas intactas que, por su tamaño, hasta entonces no habían sido susceptibles de ionización, ya que la alta energía requerida para la misma acababa alterando o destruyendo la propia proteína. En 1987, Köichi Tanaka presenta un nuevo método de análisis (Soft Laser Desorption) que permitía transferir a las moléculas la energía necesaria para ionizarlas sin romper los frágiles enlaces químicos. Este descubrimiento le valió el Premio Nobel de Química en 2002, junto con John B. Fenn, por «el desarrollo de métodos de identificación y de análisis estructural de macromoléculas biológicas». No tardaron en desarrollarse espectrómetros de masas basados en este tipo de análisis, aunque fueron necesarios algunos cambios en el método para que finalmente apareciese la EM MALDI-TOF, en la cual la desorción de los iones es favorecida por una matriz que absorbe la energía del láser y la transfiere parcialmente a las moléculas objeto de estudio. A pesar de que estas modificaciones mejoraron la técnica, haciéndola más sencilla y sensible, los responsables de estas mejoras, Michael Karas y Franz Hillenkamp, no fueron incluidos en el premio Nobel, lo cual generó una importante controversia. A partir de las aportaciones de estos autores empezó a ser posible el estudio de macromoléculas y biopolímeros mediante EM, lo que abrió nuevos campos de aplicación para esta tecnología.
En sus casi 30 años de vida, la EM MALDI-TOF se ha utilizado para el análisis cuantitativo y cualitativo de proteínas de orígenes diversos. Inicialmente se aplicó a proteínas aisladas previamente o a pequeños conjuntos de proteínas, pero gracias a los avances técnicos tanto en la instrumentación como en las herramientas para el análisis informático de los datos, actualmente se puede abordar el estudio de grandes grupos de proteínas.
La posibilidad de empezar a estudiar proteínas complejas, y no solo pequeños péptidos, amplió mucho los campos potenciales de aplicación de la EM. Uno de ellos fue la identificación de microorganismos que, a medida que se simplificó el procedimiento y se mejoró el software necesario para explotar los datos crudos proporcionados por el espectrómetro, derivó rápidamente en su aplicación clínica.
Espectrometría de masas MALDI-TOF en Microbiología ClínicaHasta la introducción de la EM MALDI-TOF, la identificación bacteriana, aun con avances significativos como la creación de galerías de identificación miniaturizadas y la automatización de su inoculación y lectura, seguía nutriéndose de los métodos desarrollados por la bacteriología clásica. La práctica totalidad de los sistemas de identificación seguían basándose en la fermentación de azúcares y su detección a través del cambio de pH generado, la metabolización de otros sustratos y la producción de diferentes metabolitos y actividades enzimáticas detectables por métodos químicos. Todos estos métodos adolecían de varias limitaciones:
- •
Requerían crecimiento bacteriano, lo que suponía, en la mayor parte de los casos, un periodo de incubación de al menos 16-18 h desde su inoculación hasta su lectura.
- •
Mostraban problemas de identificación en todos aquellos microorganismos con dificultades para crecer en los medios líquidos usados para la inoculación de estos paneles, así como en microorganismos con escasa actividad bioquímica y enzimática.
- •
Era necesario considerar el margen de error derivado del hecho de que, individuos de la misma especie, pudieran tener comportamientos distintos frente a diversos sustratos.
Estas limitaciones, si bien eran conocidas y asumidas, han quedado más claramente patentes cuando, ante las discrepancias observadas en algunos estudios entre la EM MALDI-TOF y la identificación convencional, la secuenciación del ARNr 16S demostró que, en la gran mayoría de los casos, la identificación correcta correspondía a la proporcionada por la EM MALDI-TOF2.
A este respecto, la EM MALDI-TOF tiene ventajas evidentes:
- •
Con independencia de la posibilidad de identificar microorganismos directamente a partir de algunas muestras, que se trata en otro apartado, crecimientos en placa incluso muy escasos o precoces permiten obtener una identificación fiable en un corto periodo de tiempo, ahorrando así, como mínimo, esas 16-18 h de crecimiento en los sistemas bioquímicos de identificación.
- •
El análisis del perfil proteico del microorganismo en el espectro de los 2-20kD, que es donde se sitúan la mayor parte de las proteínas ribosómicas, ofrece para la gran mayoría de las especies bacterianas un perfil específico, que permite diferenciarlas del resto con una fiabilidad similar a la ofrecida por la secuenciación del ARNr 16S.
A ello hay que añadir el hecho de que, la introducción de esta tecnología, ha ampliado mucho el abanico de géneros y especies que somos capaces de identificar con fiabilidad, con un método susceptible de ser usado en rutina. Ello ha llevado incluso a una reevaluación del papel como patógenos de microorganismos que, por la dificultad de su identificación por los métodos clásicos, estaban muy probablemente infradiagnosticados3,4.
Ya en 1975, Anhalt y Fenselau proponen la utilización de la EM para la identificación de microorganismos5. Veinte años después se publica el primer estudio que demuestra la eficacia de la EM MALDI-TOF para la identificación de microorganismos a partir de células completas6.
En 2009 se publicó el que probablemente fue un artículo clave para dar a conocer de forma general, a los especialistas implicados en el diagnóstico de las enfermedades infecciosas, las posibilidades de la EM MALDI-TOF7. En él se estudian más de 1600 aislamientos que incluyen bacterias grampositivas y gramnegativas, aerobias y anaerobias, obteniendo un 95,4% de identificaciones correctas. Ya en este artículo se apunta una cuestión que luego se ha demostrado trascendente para la utilidad práctica de esta metodología: la disponibilidad de bases de datos de microorganismos suficientemente amplias, tanto desde el punto de vista cualitativo (número de géneros y especies incluidos) como cuantitativo (los autores demuestran que la probabilidad de identificación correcta es mayor para aquellos microorganismos de los que existan al menos diez perfiles distintos introducidos en la base de datos).
Desde este momento, se produce una rápida expansión del uso de esta tecnología en Microbiología Clínica. La primera publicación en España tiene lugar en 2010, mostrando una correlación con la metodología convencional, a nivel de especie, del 100% en grampositivos y del 87,7% en gramnegativos8. Desde entonces, el número de publicaciones relativas a diferentes aspectos de la utilización clínica de la EM MALDI-TOF, pero sobre todo a la identificación de microorganismos, ha sido exponencial7–12.
Identificación de bacterias gramnegativas mediante MALDI-TOFLos estudios demuestran que la eficacia en la identificación de enterobacterias y otros gramnegativos es excelente, incluyendo tanto los gramnegativos más habituales en clínica como otros menos frecuentes o más complicados de identificar con la metodología clásica (diferentes especies de Yersinia, Aeromonas, Plesiomonas, Brucella, Francisella, Achromobacter, Stenotrophomonas, Burkholderia…). Como en otros casos, los problemas de identificación casi siempre han estado ligados a insuficiencias de las bases de datos más que a limitaciones del método, como ha ocurrido en algunos estudios con géneros como Ralstonia, Elizabethkingia o Sphyngobacterium13. Inicialmente se plantearon algunas limitaciones que podían tener mayor trascendencia clínica, como la incapacidad para diferenciar entre Escherichia coli y Shigella14 o la dificultad para diferenciar serovares de Salmonella enterica. Estudios más recientes sugieren que es posible salvar estas limitaciones. Utilizando programas informáticos como FlexAnalysis y ClinProTools (Bruker Daltonics GmbH, Alemania), se han identificado picos específicos que permiten diferenciar E. coli y Shigella en el 90% de los casos15. Otros autores han demostrado que esta diferenciación es posible incluso sin utilizar software adicional alguno, simplemente incrementando el número y variedad de perfiles de E. coli y de Shigella presentes en la base de datos de referencia16. Con este método, los autores identifican correctamente 60/64 aislamientos de E. coli y 110/116 aislamientos de Shigella.
A todos los que hemos utilizado la EM MALDI-TOF para identificación nos consta que, con la metodología habitual, la identificación de Salmonella a nivel de género es fiable, pero más allá del nivel de género lo es mucho menos. Sin embargo, existen ya desde 2004 publicaciones que sugieren la existencia de picos específicos que podrían permitir identificar serovares con mayor fiabilidad17. Un estudio reciente sugiere la existencia de picos que permiten identificar con fiabilidad el serovar Typhi18. Puesto que parecen existir elementos diferenciales, una ampliación de la base de datos de referencia con un mayor número de espectros de, al menos, los serovares más frecuentes, probablemente mejoraría la resolución en este caso concreto.
En otros casos, como es la diferenciación entre especies del complejo Enterobacter cloacae, la capacidad de la EM MALDI-TOF está en torno al 80%, que sin ser óptima, supone una mejora sensible respecto a la metodología convencional19.
Por otra parte, como ya se ha mencionado, la identificación correcta de microorganismos previamente identificados de manera incorrecta, como el género Raoultella, frecuentemente identificado por los métodos clásicos como Klebsiella o como Enterobacter, está permitiendo una visión más real de su papel como patógenos, previamente infravalorado3.
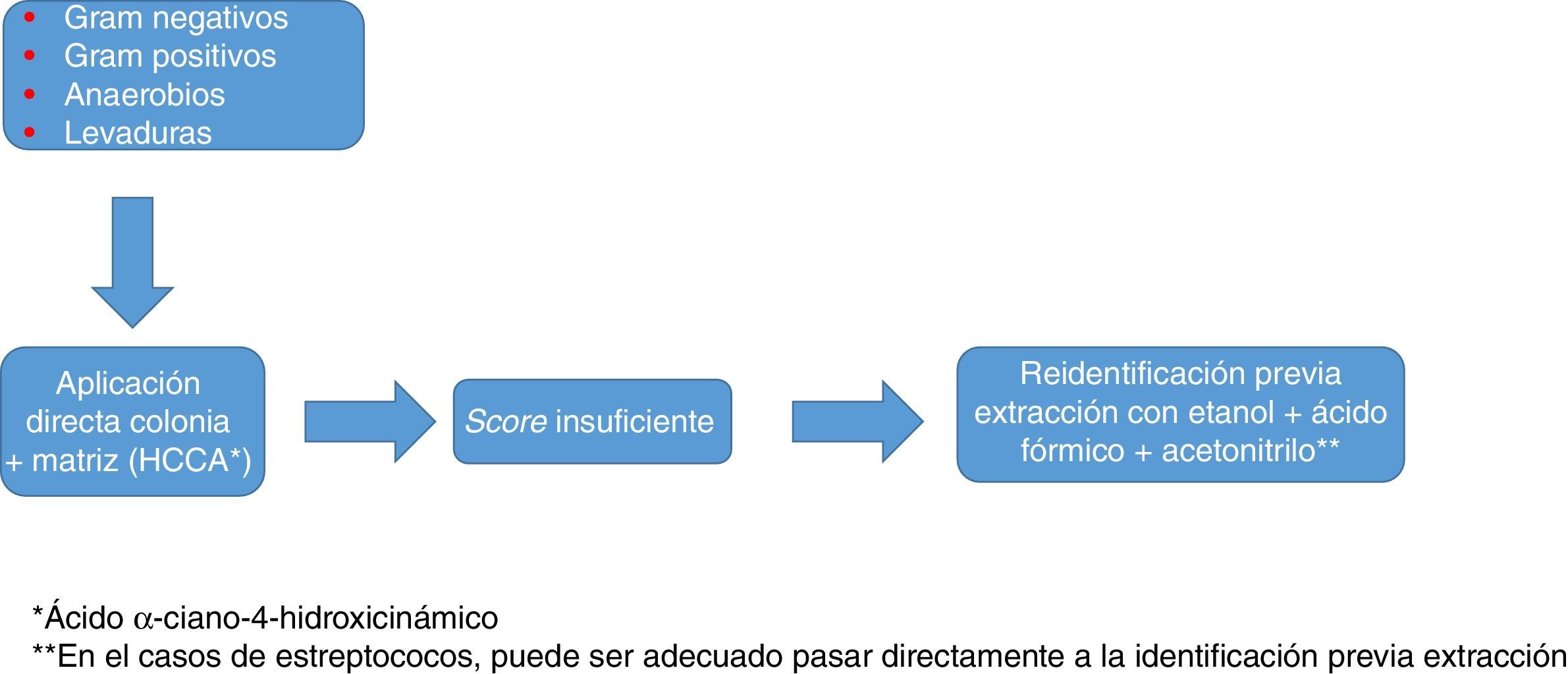
Identificación de bacterias grampositivas mediante MALDI-TOFLa identificación de microorganismos grampositivos suele mostrar unas cifras algo inferiores, debido en algunos casos a la dificultad para lisar la pared, y en otros a la similitud entre especies. En general, el método directo (aplicación de la colonia junto con la matriz sobre la placa de espectrómetro) suele ser suficiente para obtener una buena identificación a partir de colonia, pero en ocasiones puede ser necesaria la extracción previa con etanol/ácido fórmico para alcanzar valores de identificación óptimos. En conjunto, ofrece unos resultados excelentes en la identificación de Staphylococcus aureus y de otras especies y subespecies de estafilococos tanto productores como no productores de coagulasa, así como de enterococos y de otros patógenos menos frecuentes como Micrococcus, Gemella, Rothia, etc. Ha demostrado también su utilidad en la identificación de bacilos grampositivos, incluyendo algunos de identificación compleja por métodos clásicos (Listeria, Lactobacillus, Corynebacterium, Nocardia, Actinotignum…)20.
Además, como en el caso de los gramnegativos, el uso sistemático de esta tecnología está cambiando la consideración como patógenos de algunos microorganismos, como Staphylococcus lugdunensis21. En conjunto, los datos disponibles hacen recomendable, en el caso de estafilococos utilizar, como en cualquier otro caso, las bases de datos que ofrezcan un mayor número de especies y un número razonablemente alto de perfiles por especie, y usar un método de extracción en lugar de la aplicación directa, así como aplicar el microorganismo en la placa por duplicado. La reducción del score requerido para validar una identificación a nivel de especie a valores >1,7, en lugar de >2,0 como es habitual, tal como proponen algunos autores22, puede incrementar el número de identificaciones a nivel de especie, pero a cambio de un descenso en la especificidad cuya pertinencia puede ser discutible.
Probablemente el grupo de los estreptococos es el que presenta mayores problemas entre los grampositivos. Mientras algunas de las principales especies patógenas (Streptococcus pyogenes, Streptococcus agalactiae) se identifican con una alta fiabilidad, Streptococcus pneumoniae (S. pneumoniae) siempre ha sido una fuente de problemas, por su confusión con otras especies del grupo mitis. Los principales equipos en uso en nuestro país identifican S. pneumoniae con fiabilidad. El error más habitual es la identificación errónea de otros estreptococos del grupo mitis como S. pneumoniae, sobre todo con la base de datos de Bruker23. Se han descrito algunos picos como específicos de S. pneumoniae, por lo que algunos autores recomiendan su utilización para corroborar estas identificaciones23. Sin embargo, la última biblioteca de referencia de Bruker Daltonics (MBT 6903 MSP Library) sigue recomendando la utilización de otros test (optoquina, solubilidad en bilis) para diferenciarlos.
Incluso dentro del mismo grupo, la calidad de la identificación de las diferentes especies puede variar considerablemente, como ocurre dentro del grupo Streptococcus anginosus.
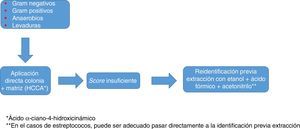
En todo caso, como ocurre con los estafilococos, el uso de una base de datos tan amplia y completa como sea posible, y la identificación por duplicado y con protocolos que incluyan extracción son recomendables para optimizar los resultados (fig. 1).
Identificación de bacterias anaerobias mediante MALDI-TOFLa identificación de bacterias anaerobias, tanto grampositivas como gramnegativas, tiene en conjunto un alto grado de fiabilidad, identificando correctamente las especies más habituales (Bacteroides, Prevotella, Porphyromonas, Fusobacterium, Clostridium, Actinomyces…). La aplicación de la EM MALDI-TOF en este campo ha demostrado, además, que la identificación bioquímica era mucho menos fiable de lo que se pensaba3. La combinación de sencillez, rapidez y fiabilidad convierten en este momento a la EM MALDI-TOF en el método de elección para la identificación rutinaria de bacterias anaerobias en clínica. La extracción o no de la muestra no parece modificar sensiblemente los resultados, como tampoco lo hace el medio de cultivo de origen. Un factor decisivo, como en otros casos, es la amplitud de las bases de datos. Ya uno de los primeros estudios realizados en España3 mostraba que la mayor parte de los errores en la identificación de bacterias anaerobias se debían a la ausencia de perfiles de referencia de la especie correspondiente en la base de datos. Los estudios más antiguos ofrecen una eficacia en la identificación en torno al 75-80%, mientras estudios más recientes, con bases de datos más completas o con bases de datos específicas ofrecen cifras superiores al 90%. La capacidad para identificar ribotipos de Clostridium difficile, sugerida por algunos autores, no ha quedado definitivamente demostrada.
Identificación de levaduras y hongos filamentosos mediante MALDI-TOFLa identificación de levaduras ha mostrado desde el principio resultados excelentes, diferenciando incluso especies complicadas de discriminar por métodos convencionales, como es el complejo Candida parapsilosis. Sin embargo, los resultados en relación con los hongos filamentosos han sido mucho más erráticos. Algunos estudios iniciales mostraban buenos resultados estudiando selectivamente esporas, pero este método es poco práctico en un laboratorio clínico, por lo que la mayoría de los estudios se han dirigido al estudio conjunto de esporas e hifas. En el caso de los hongos, la extracción convencional con ácido fórmico y acetonitrilo sí mejora sensiblemente los resultados. Otros métodos que se han probado, como la utilización de perlas de vidrio para fragmentar las paredes, no parecen ofrecer resultados significativamente superiores a la extracción convencional.
Un problema que plantean los hongos filamentosos es la existencia de diferencias significativas en los perfiles proteicos obtenidos en función de la antigüedad de los cultivos, e incluso entre diferentes subcultivos de la misma cepa24. La solución a esto pasa por la elaboración de bases de datos más amplias y complejas, que incorporen perfiles de un mayor número de cepas y de cultivos de diferente antigüedad. Algunos fabricantes recomiendan la extracción tras un cultivo de un día en caldo, aunque esto repercute en un retraso de 24 h en la emisión del resultado. Un estudio realizado en 201125 demuestra que una base de datos bien elaborada y suficientemente compleja es fundamental, y que puede mejorar la identificación de los hongos filamentosos hasta cifras similares a las obtenidas con bacterias y levaduras. Desafortunadamente, la elaboración y la validación de estas bases de datos son complejas y no están al alcance de muchos usuarios, y pocas de las desarrolladas están disponibles. Por ello, y también a efectos de estandarización, es probablemente más adecuado el uso de las bases de datos de los fabricantes, que deben ser convenientemente ampliadas.
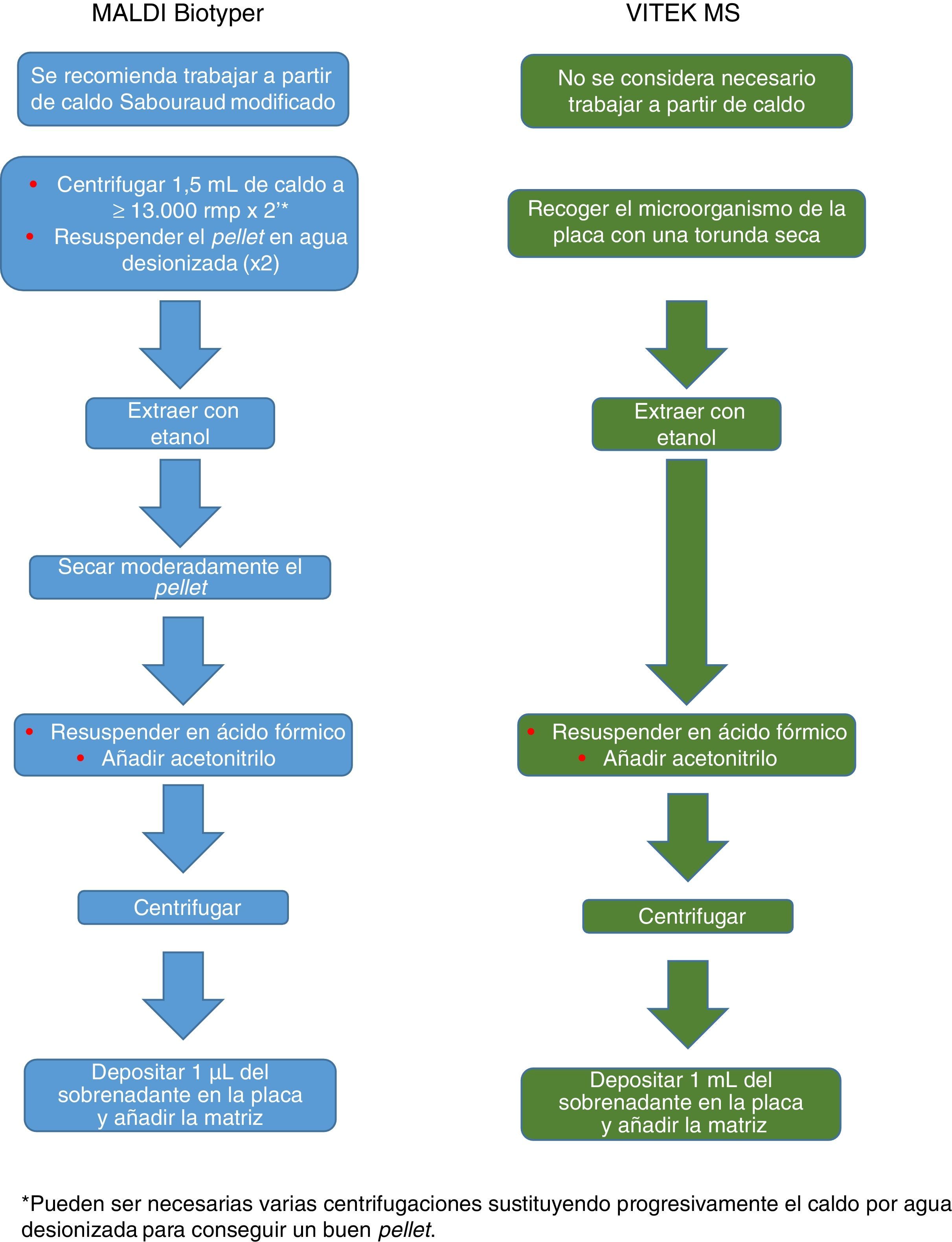
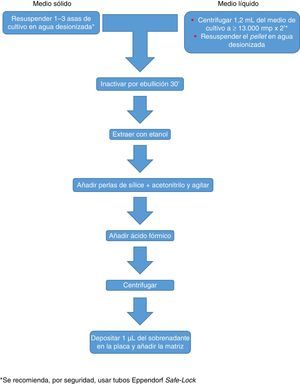
Actualmente Bruker Daltonics, cuya base de datos de hongos filamentosos, en su versión MBT 6903 MSP Library, incluye 25 géneros y 42 especies, recomienda el cultivo en medio líquido durante una noche, seguida de centrifugación, lavado y extracción con etanol, ácido fórmico y acetonitrilo (fig. 2). MS-VITEK, en su versión 3.0, incluye 32 géneros y 81 especies, y SARAMIS en su versión RUO 4.13, incluye una base de datos más amplia, con 45 géneros y 168 especies. bioMérieux no recomienda el cultivo en medio líquido, sino una extracción convencional con etanol, ácido fórmico y acetonitrilo (fig. 2). A pesar de todo, un estudio reciente usando la metodología recomendada por Bruker identifica correctamente, a nivel de especie, solo el 72% de los aislados26, y un estudio muy reciente con VITEK 3.0 identifica correctamente el 66,8% de 318 aislados, debido sobre todo a carencias de la base de datos27. Por tanto hoy por hoy, la identificación de hongos filamentosos mediante EM MALDI-TOF no puede sustituir todavía completamente a la metodología de identificación convencional.
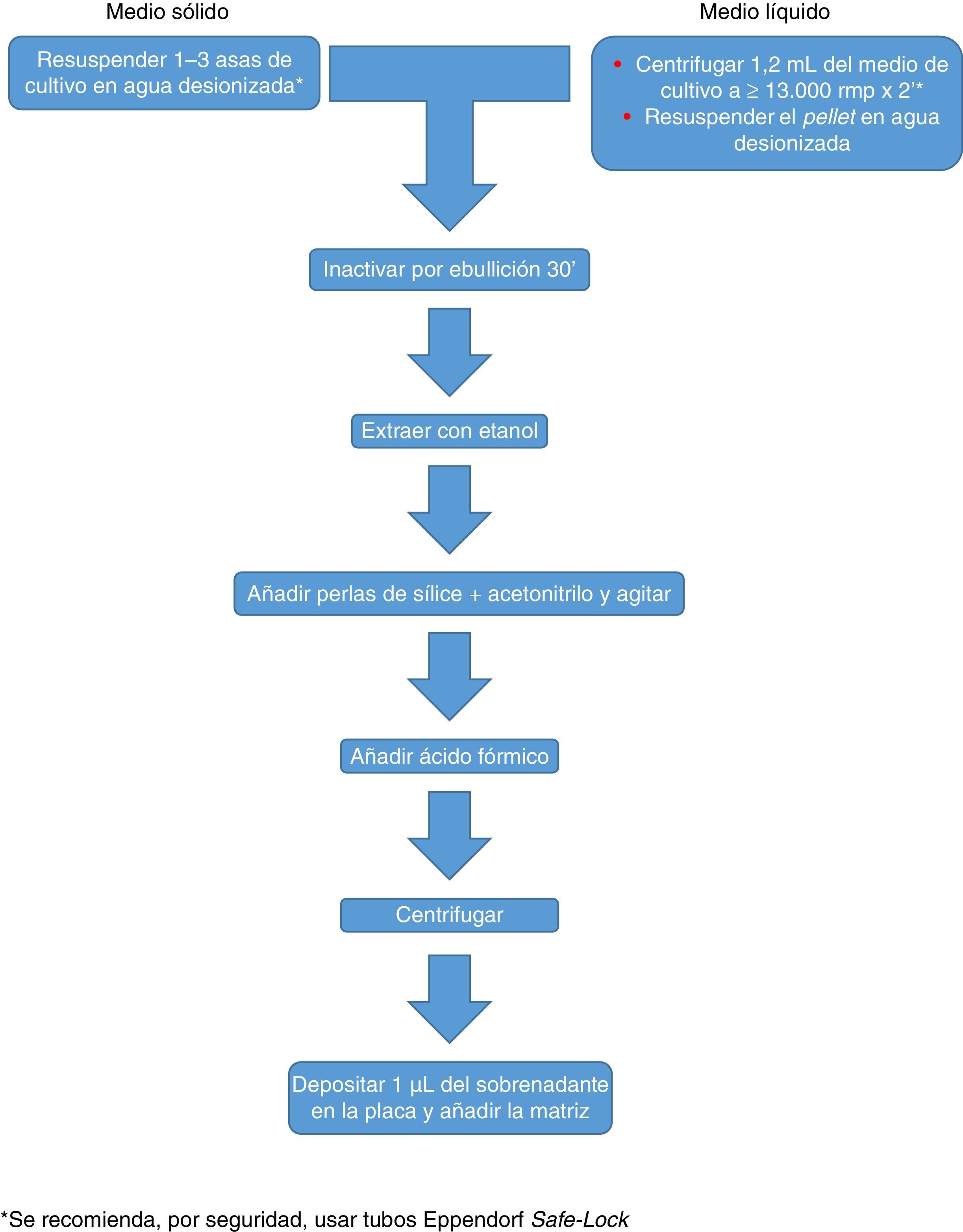
Identificación de micobacterias mediante MALDI-TOFLas limitaciones de la metodología tradicional para la identificación de micobacterias, sobre todo en lo que se refiere al tiempo de respuesta, han hecho que la práctica totalidad de los laboratorios se hayan decantado por los métodos moleculares de identificación. La utilización de la EM MALDI-TOF para la identificación de micobacterias supone una alternativa igualmente rápida y más barata que las técnicas moleculares, pero muestra peculiaridades en varios aspectos. Por una parte, los métodos directos de análisis, o que impliquen una alta concentración o manipulación de los microorganismos previamente a su inactivación, no son recomendables por una cuestión de seguridad biológica. La utilización de un método de extracción que garantice la rotura de las células mejora la calidad de los espectros, e incrementa la seguridad del procedimiento.
Varios factores pueden influir en la eficacia de la identificación de micobacterias mediante EM MALDI-TOF. Como también se ha demostrado con los hongos, las micobacterias pueden presentar perfiles proteicos muy diferentes en función de la antigüedad de los cultivos, por lo que es importante para el diagnóstico incluir en las bases de datos perfiles proteicos de cultivos de diferente antigüedad para cada microorganismo. Se ha hablado también de la posible formación de polímeros que enmascaran el tamaño real de las proteínas características utilizadas para la identificación. Todo ello hace de la identificación de micobacterias un procedimiento menos previsible en cuanto a sus resultados que la de otras bacterias, como demuestra la heterogeneidad de los datos publicados. En las últimas actualizaciones, las bibliotecas han experimentado importantes mejoras. Así, por ejemplo, la biblioteca de micobacterias 3.0 de Biotyper incluye ya 149 especies, y la versión 3.0 de VITEK MS incorpora 48 nuevas especies con respecto a la versión anterior.
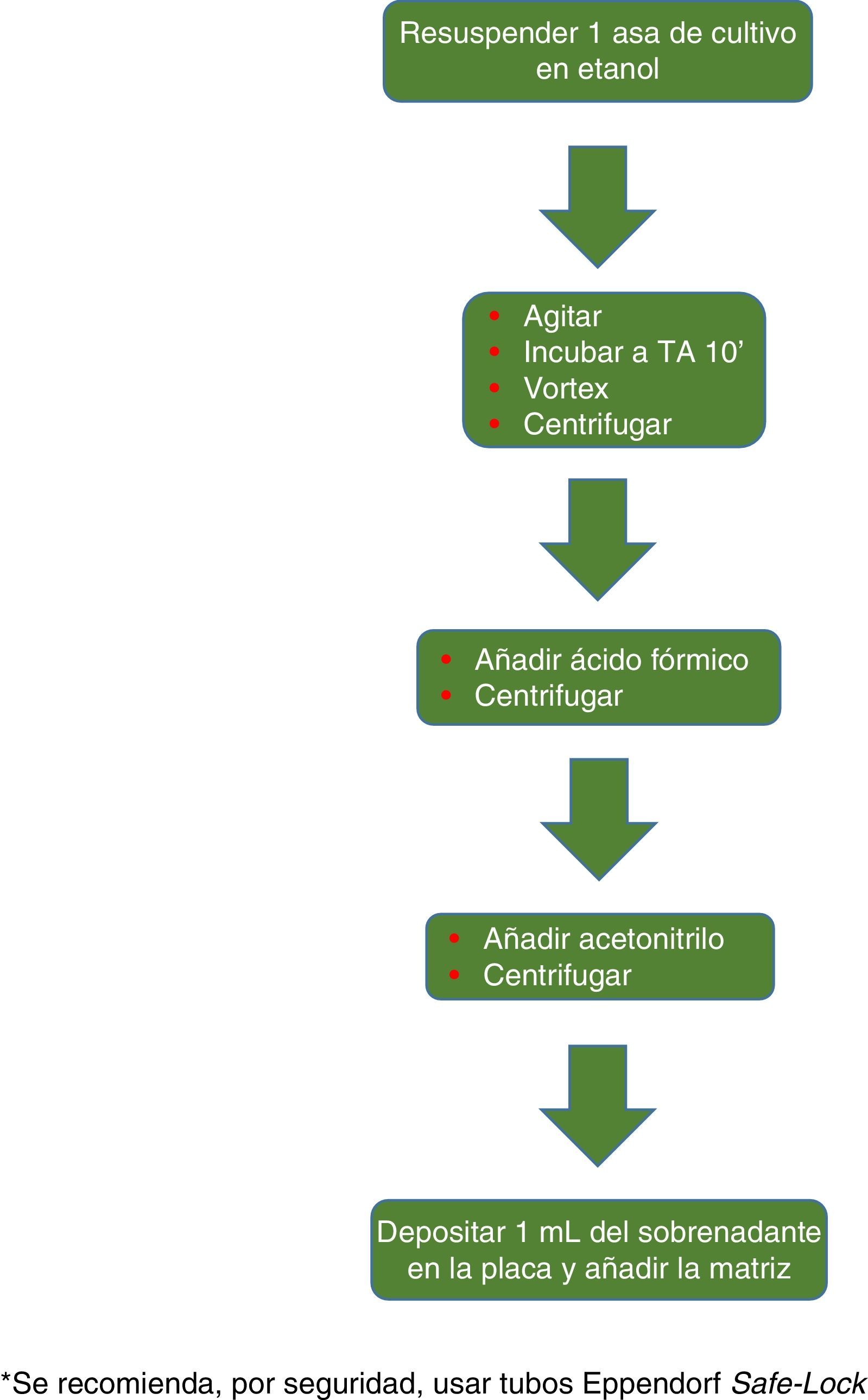
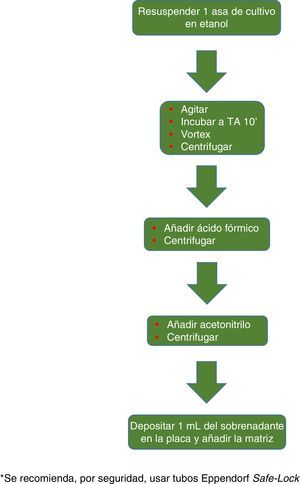
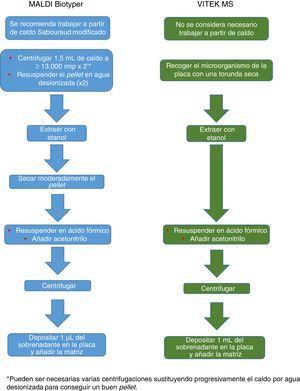
El procedimiento inicialmente recomendado por Bruker incluía una serie de pasos (ajuste a una determinada densidad óptica, varios pasos de centrifugación y resuspensión) que suponían un riesgo biológico probablemente innecesario, por lo que han sido sustituidos por un solo paso de tratamiento con calor. El método recomendado actualmente incluye varios pasos de calentamiento y lavado, seguidos de un tratamiento con bolas de sílice y acetonitrilo. Posteriormente son tratadas con ácido fórmico, aplicadas en la placa y cubiertas con la matriz (fig. 3). Por el contrario, el método propugnado por bioMérieux parte de un tratamiento inicial con etanol al 70% y bolas de sílice, tras el cual la muestra es extraída con ácido fórmico y acetonitrilo, aplicada en la placa y cubierta con la matriz (fig. 4).
Un estudio comparativo entre ambos métodos llevado a cabo recientemente28 muestra que aunque la identificación tiende a ser algo mejor partiendo de medio 7H10 que de Lowenstein Jensen, las diferencias son pequeñas en ambos sistemas. Sin embargo, demuestra que el método de procesamiento de la muestra es crítico, y no es intercambiable entre sistemas. Las muestras obtenidas con el método de extracción de Bruker dan mejores resultados en su espectrómetro y las procesadas con el método de Vitek lo hacen tanto con SARAMIS como en el propio Vitek MS, aunque la diferencia en este caso es menor. En conjunto, Vitek MS parece verse menos afectado por el método de extracción. En cuanto a la eficacia en la identificación, Bruker Biotyper identificó con un score ≥1,8 un 81,5% de los aislados, mientras SARAMIS y Vitek MS 3.0 identificaron con una fiabilidad ≥90% un 85,4 y un 89,2% respectivamente. Utilizando estos mismos criterios, un 22,6% de los aislados que fueron identificados por Bruker Biotyper necesitaron ser extraídos en más de una ocasión para lograr la identificación, lo que ocurrió en un 21,5% con SARAMIS y en un 14,2% con Vitek MS 3.0.
En relación con otros microorganismos con estructura parietal próxima a la de las micobacterias, durante tiempo se ha considerado que su identificación era problemática debido a la composición de su pared. Sin embargo, se ha demostrado que, si se dispone de una base de datos con suficientes referencias, un método de extracción similar al usado con micobacterias puede ofrecer buenos resultados29.
Todos los datos referidos anteriormente sugieren que el espectro de microorganismos susceptibles de identificación mediante EM MALDI-TOF es extraordinariamente amplio y que, con contadas excepciones, el factor limitante de la capacidad de la EM MALDI-TOF asienta casi siempre más en la completitud de las bases de datos de referencia que en la capacidad del método para obtener perfiles fiables de casi cualquier microorganismo bacteriano susceptible de crecer en medios de cultivo. De hecho, las empresas fabricantes parecen ser conscientes del problema; así, la última biblioteca de referencia publicada por Bruker Daltonics (MBT 6903 MSP Library) incorpora 938 nuevos perfiles, de los que un 17,2% van orientados a cubrir nuevos géneros y especies (15 nuevos géneros y 96 nuevas especies en concreto), en especial anaerobios y hongos, pero un 82,8% van orientados a incrementar la diversidad de perfiles dentro de especies ya contempladas en bibliotecas previas. Del mismo modo, la versión 3.0 de VITEK MS incluye 242 nuevas especies bacterianas, entre las que se encuentran 48 nuevas de micobacterias y 15 de Nocardia, y 55 nuevas de hongos, con un promedio global de más de 10 aislados y 25 espectros por especie.
Identificación de microorganismos, por MALDI-TOF, a partir de muestra directaLa posibilidad de identificación de microorganismos previamente al crecimiento de colonias ha sido, desde el principio, un objetivo muy atractivo, por lo que supone a la hora de poder instaurar un tratamiento empírico más orientado, reducir la emergencia de resistencias, y optimizar el gasto30. La EM MALDI TOF, aplicada a muestra directa, se ha convertido en una herramienta de gran utilidad en infecciones graves con alta tasa de morbimortalidad como bacteriemias y fungemias, si bien el trabajo directo sobre muestras presenta inconvenientes que pueden limitar la utilidad de la técnica. En muestras con baja densidad microbiana no es posible realizar una correcta identificación, ya que la cantidad de proteínas bacterianas presentes es insuficiente. Algo parecido ocurre con las muestras polimicrobianas, ya que se puede generar un espectro proteico aberrante, como resultado de la mezcla de varios perfiles, o directamente ignorarse el microorganismo que esté en una proporción menor. Se han desarrollado algunas mejoras del software que en ocasiones pueden detectar, e incluso identificar por separado los microorganismos implicados, pero requiere ser cuidadosamente estudiado y probablemente depurado para tener utilidad real. En la mayor parte de los métodos que trabajan directamente sobre muestra, y ante la posible presencia de microorganismos intracelulares, es recomendable usar métodos que incluyan lisis celular.
No todas las muestras son válidas para este tipo de estudio. La muestra ideal es aquella que proceda de un área habitualmente estéril, que pueda albergar altas concentraciones de microorganismo y en la que no haya limitaciones significativas en cuanto al volumen de muestra. Las muestras procedentes de áreas habitualmente colonizadas (piel, heces, aparato respiratorio alto…) van a generar con toda probabilidad perfiles aberrantes. Una cuestión técnica a tener en cuenta cuando se trabaja directamente sobre muestra es el volumen de muestra disponible, que en casos como orina o hemocultivos no suele ser un problema, pero en otros con líquido cefalorraquídeo sí puede serlo, y que será tanto más importante cuanto menor sea la concentración bacteriana. Además, muestras con alto contenido proteico de origen ajeno al microorganismo también pueden generar problemas a la hora de su interpretación31.
Sin embargo, la ventaja que supone, en especial en los hemocultivos, adelantar 24 h la identificación del microorganismo y orientar de forma más específica el tratamiento, ha hecho que el uso directo de la EM sobre muestra, en especial en estos casos, se haya generalizado.
Identificación directa a partir de muestras de orinaLa orina es una de las muestras que mejor se ajustan a las condiciones ideales para trabajar con EM MALDI-TOF sobre muestra directa. Es un líquido estéril en condiciones fisiológicas, la carga bacteriana en la orina infectada es alta en la mayoría de los casos, y las limitaciones en cuanto al volumen de muestra y a la obtención de nuevas muestras si es necesario, son mínimas.
Se han publicado estudios con resultados excelentes, con concordancias en la identificación que llegan al 90-95%32,33 en comparación con los sistemas automatizados. Se han descrito diferentes protocolos de procesamiento de muestras, coincidiendo todos ellos en la necesidad de un cribado previo (citometría de flujo, tinción de Gram) para discriminar entre muestras positivas y negativas, y limitar el uso de la EM a las muestras inicialmente positivas34.
Con pequeñas variaciones, la preparación de las muestras suele consistir en someterlas a una serie de centrifugaciones y lavados con agua desionizada, llevando a cabo posteriormente el procedimiento convencional de la EM MALDI-TOF. Se han realizado modificaciones incorporando a la alícuota de la muestra SDS al 10%33 para mejorar la liberación de las proteínas, o Tween-8034 para mejorar los resultados. Los resultados obtenidos suelen ser mejores con gramnegativos que con grampositivos y hongos, mejorando mucho la fiabilidad cuando los recuentos son elevados, por encima de 105UFC/mL.
Un inconveniente de este método ha venido siendo la necesidad de llevar a cabo el estudio de sensibilidad mediante la metodología convencional. Un estudio reciente sugiere que, una vez obtenida la identificación por EM MALDI-TOF, el mismo sedimento se puede usar para realizar un antibiograma disco placa con excelentes resultados, lo que permite reducir en 24 h el estudio completo35. El importante cambio organizativo que la introducción de esta metodología supone, y el que se trate de infecciones en las que, con frecuencia, adelantar unas horas el diagnóstico no es crítico para el manejo del paciente, han hecho que el diagnóstico de la infección urinaria mediante EM MALDI-TOF no haya tenido la penetración que ha tenido, por ejemplo, en el caso de los hemocultivos. Es necesario lograr tanto una mayor sensibilidad como una mayor normalización de los métodos para plantearse su introducción en la rutina clínica.
Identificación directa a partir de hemocultivosLa instauración de un tratamiento empírico correcto es decisiva en la evolución de las bacteriemias. La probabilidad de que el tratamiento instaurado sea correcto, será mayor cuanto más exacta y concreta sea la información que seamos capaces de proporcionar con rapidez. En este aspecto, la posibilidad ofrecida por la EM MALDI-TOF de obtener una identificación fiable en un corto periodo de tiempo tras la positivización del hemocultivo supone un avance evidente.
Se han descrito numerosos protocolos de procesamiento del hemocultivo (centrifugación diferencial, lisis celular y extracción de proteínas por métodos químicos, separación mediante gel…). La mayoría de ellos están basados en la lisis y eliminación de los componentes celulares, bien mediante centrifugaciones y lavados, en los que se utilizan diferentes compuestos como saponina, cloruro de amonio o SDS, o bien mediante el uso de tubos con gel separador de suero y activador de la coagulación. En definitiva, lo que se busca con estos métodos es aislar y concentrar los microorganismos hasta alcanzar al menos 105–107UFC/mL, concentración a la cual la cantidad de proteínas es suficiente para generar perfiles adecuados en la EM MALDI-TOF.
En el caso de los hemocultivos existe un procedimiento comercial homologado (Sepsityper, Bruker Daltoniks GmbH, Alemania), consistente en la adición de una solución de lisis a una alícuota de 1ml del hemocultivo, tras lo cual la muestra es sometida a varios pasos de lavado y centrifugación y, finalmente, a una extracción convencional con etanol y ácido fórmico36.
Los resultados del procesamiento directo de hemocultivos son en general buenos, con porcentajes de identificación correcta del 80-90%, aunque con algunos matices. La identificación de los gramnegativos suele ser correcta en el 90-95% de los casos, mientras en grampositivos es mucho más heterogénea, oscilando entre cifras similares a las de gramnegativos y cifras en torno al 50%. Dentro de estos, plantean problemas de fiabilidad sobre todo los estreptococos del grupo viridans y los estafilococos no productores de coagulasa.
Los resultados en casos de fungemia, en los primeros estudios, eran decepcionantes. Una correcta extracción, indispensable en este caso, lleva a cifras de identificación superiores al 90%, homologables con las obtenidas en bacteriemias37. Ha de tenerse en cuenta la posible repercusión de determinados componentes de algunos hemocultivos, como el carbón activo, que puede interferir la identificación. Asimismo, un tiempo de incubación prolongado del hemocultivo puede repercutir negativamente en los espectros obtenidos. No obstante, puesto que el procedimiento habitual es realizar la EM en, como máximo, unas horas a partir de la positivización, y más del 80% de los hemocultivos positivos significativos lo son en las primeras 48 h, el impacto clínico de este aspecto no parece trascendente.
A la hora de valorar la heterogeneidad de los porcentajes de identificación correcta en los hemocultivos, ha de tenerse en cuenta que no existe un criterio homogéneo respecto a cómo valorar los scores obtenidos. Algunos autores consideran una identificación correcta con valores >1,7. Otros disminuyen esta exigencia hasta 1,5, pero incluyen requisitos como que la identificación se repita en las dos o tres primeras posiciones de la lista de identificaciones posibles, o que entre las dos primeras opciones que ofrece la EM MALDI-TOF exista una diferencia de score de al menos 0,3 puntos.
En general, no somos partidarios de reducir el nivel de exigencia en la identificación correcta en los hemocultivos, ya que la existencia de un mayor porcentaje de «no identificaciones» supondrá siempre un menor riesgo, desde el punto de vista clínico, que la proliferación de identificaciones incorrectas, que podrían condicionar tratamientos inadecuados.
En cuanto a la utilización de métodos comerciales de procesamiento, como Sepsityper, los resultados en general son similares en relación con los métodos de procesamiento manuales. Cada laboratorio deberá priorizar entre la mayor normalización y el ahorro en tiempo de procesamiento que propician estos métodos, o el ahorro económico (en torno a 1 €/muestra) que supone la utilización de un método manual.
En conjunto, la EM MALDI-TOF es un método rápido y fiable para la identificación directa de microorganismos en hemocultivos. Su combinación con métodos que permiten detectar mecanismos de resistencia a determinados antimicrobianos, permite ofrecer una información clínica valiosa en un tiempo sensiblemente inferior (24-48 h menos) al requerido por la metodología convencional. Probablemente el principal punto a dilucidar, en este momento, es la forma de integrar esta nueva actividad en los laboratorios de Microbiología, de modo que se optimice la información ofrecida sin suponer una sobrecarga excesiva en servicios, por lo general, no precisamente sobrados de personal técnico y con estructuras horarias muy diversas.
Identificación directa a partir de otras muestrasEl número de estudios relativos a la aplicación directa de la EM MALDI-TOF a otras muestras biológicas es más reducido. Como se comenta en otro apartado, la utilidad de la EM MALDI-TOF en estas muestras viene determinada por varios factores: por una parte, la muestra debe proceder de un área estéril en condiciones fisiológicas, y en la que la infección, cuando aparezca, tienda a ser habitualmente monomicrobiana. Por otra parte, son cruciales tanto la carga bacteriana presente en la muestra, como el volumen de muestra disponible para el estudio. Ello hace que muchas muestras distintas de la orina y el hemocultivo planteen problemas, por el bajo volumen habitualmente disponible (LCR, exudados purulentos), por la baja concentración de microorganismos (LCR, líquido peritoneal en pacientes sometidos a diálisis peritoneal) o por la presencia de cantidades importantes de proteínas no bacterianas que pueden alterar los perfiles o interferir en la ionización de las proteínas bacterianas (exudados purulentos).
La baja sensibilidad en aquellos productos en los que la carga bacteriana no es muy alta, y el volumen de muestra disponible, con frecuencia limitado, hacen que, en general, sean preferibles para el diagnóstico rápido las técnicas moleculares, aun con la limitación de que el abanico de microorganismos detectables en un solo test es mucho menor que con la EM MALDI-TOF. Se han comunicado ocasionalmente diagnósticos etiológicos de meningitis bacterianas por EM MALDI-TOF directa sobre LCR, pero un estudio comunicado al 21erECCMID indicaba que, sobre 183 muestras de LCR, de las que 14 fueron positivas por el método convencional, ninguna fue positiva por EM MALDI-TOF directa sobre muestra38, lo que reafirma el hecho de que, desde el punto de vista de la sensibilidad, la EM MALDI-TOF no puede competir con la técnicas moleculares. En otros tipos de muestra la experiencia es muy limitada, no existen protocolos publicados ni datos respecto a sensibilidad y especificidad, y por tanto la utilización clínica de la EM MALDI-TOF, en este momento, no es pertinente.
Medios cromogénicosDesde la aparición del primer medio cromogénico (MC), hace más de 30 años, se han convertido en una herramienta muy útil para el aislamiento diferencial de microorganismos patógenos. Existen actualmente multitud de MC comercializados para la identificación de bacterias y hongos, así como para el estudio de algunos mecanismos de resistencia a antimicrobianos.
El fundamento de estos medios en la inclusión de un sustrato cromogénico que, al ser hidrolizado por una enzima específica presente en el microorganismo, da lugar a una colonia de coloración característica, permite su diferenciación. Con frecuencia son además medios selectivos, que inhiben en mayor o menor medida el crecimiento de otros microorganismos, favoreciendo la detección de las colonias coloreadas. Los principales sustratos de los MC comerciales son derivados indólicos39 que pueden ser hidrolizados por galactosidasas o glucosidasas, produciéndose derivados poco tóxicos y que no inhiben el crecimiento bacteriano.
Detección de microorganismos grampositivosSe han dedicado grandes esfuerzos al desarrollo de métodos para la detección de S. aureus resistente a meticilina (MRSA), dada su importancia en infección nosocomial. En este contexto, el uso de MC ha cobrado gran importancia para el rápido aislamiento e identificación de esta bacteria. Existen en el mercado numerosos MC para la detección de MRSA. Un estudio reciente40 compara tres de ellos: chromID MRSA SMART (SMART), chromID MRSA first generation (chromID) y Brilliance MRSA (OX2) para el cribado de 1.220 muestras de pacientes hospitalizados. La detección en estos medios se corroboró mediante EM MALDI-TOF, difusión en agar con discos de cefoxitina y PCR comercial para mecA y mecC. La sensibilidad a las 24 h fue mejor para el medio SMART con respecto al medio chromID, pero no hubo diferencias significativas con el medio OX2. No obstante, los autores indican que todavía es necesario, para obtener unos resultados óptimos, el enriquecimiento en caldo durante 24 h antes de la inoculación en cualquiera de los medios estudiados.
Xu et al.41 hacen una revisión de 5 de los medios más usados para la detección de MRSA. El que mejores resultados ofrece en este estudio es el Brilliance MRSA 2 (Oxoid Ltd, Termofisher, EE. UU.), con una sensibilidad del 65,7% y una especificidad del 99,8%. El rendimiento de este medio mejora si se inocula la muestra en un caldo de enriquecimiento antes de sembrarla en el MC, con una sensibilidad del 100% y una especificidad del 99,1%. El medio CHROMagar MRSA, muestra una sensibilidad del 95% después de 24 h de incubación, que alcanza un 100% si se incuba 48 h. La especificidad en ambos casos es del 100%. El medio BBL CHROMO agar MRSA incluye cefoxitina en su composición, lo que simplifica la interpretación de los resultados. Este medio fue estudiado utilizando aislamientos de MRSA obtenidos de hemocultivos, con una sensibilidad y especificidad del 97,6 y el 99% respectivamente. MRSA Select fue evaluado utilizando 652 aislados procedentes de hemocultivos tras 18-24 h de incubación a 35°C, con una sensibilidad del 99% y una especificidad del 98%, que aumentaba al 99% cuando se combinaba con la prueba de la coagulasa. El medio chromID MRSA, que también incorpora cefoxitina en su composición, fue estudiado en aislamientos procedentes de heridas y de hemocultivos. En hemocultivos muestra una sensibilidad del 97,8% a las 24 h, que aumenta a un 100% a las 48 h, y una especificidad del 99,7% que se mantiene (99,6%) a las 48 h. En heridas, la sensibilidad y especificidad fueron del 88,9 y del 100% a las 24 h y del 100% en ambos casos a las 48 h.
El establecimiento de comparaciones entre este tipo de medios de cultivo es complejo, ya que hay muchos factores, aparte del propio medio de cultivo, que pueden influir en los resultados (tipo de muestra, concentración del inóculo, tiempo de incubación…). No obstante, los datos de sensibilidad y especificidad disponibles sugieren que cualquiera de los medios antes mencionados puede ser utilizado de rutina para la detección de MRSA con alta fiabilidad.
Otro grupo de bacterias que ha adquirido importancia en la infección nosocomial son los enterococos resistentes a vancomicina (VRE), hasta el punto de que los CDC recomiendan el estudio de portadores en los centros con alta prevalencia. El estudio de portadores de VRE se ha venido llevando a cabo utilizando agar bilis esculina con azida y vancomicina (BEAV). Su uso implicaba un retraso diagnóstico de 48 h, además de la necesidad de realizar pruebas complementarias para una correcta identificación. Para agilizar este proceso se han desarrollado diversos MC. Un estudio reciente compara cinco MC para VRE usando 400 muestras de heces42. Los medios utilizados fueron InTray Colorex VRE (BioMedDiagnostics, White City, OR), chromID VRE (bioMérieux, Marcyl’Étoile, France), VRESelect (Bio-Rad, Marnes-la-Coquette, France), HardyCHROM VRE (Hardy Diagnostics, Santa Maria, CA) y Spectra VRE (Remel, Lenexa, KS), utilizando agar BEAV y caldo BEAV como método de referencia. La lectura se realizó a las 24 h. Los resultados de los cinco medios muestran una sensibilidad de entre el 89,9 y el 94,9%, más alta en todos los casos que la mostrada por el agar BEAV (84,9%). Los mejores datos de sensibilidad y especificidad los sigue proporcionando el caldo BEAV, pero a costa de un retraso diagnóstico de hasta 48 h con respecto a los MC. El MC más sensible en este estudio fue chromID VR (94,9%), aunque sin diferencias estadísticamente significativas con el resto.
La especificidad y la capacidad para diferenciar especies de Enterococcus fueron similares para los cinco MC. El medio InTray Colorex VR tuvo la especificidad más baja con un 98,8% mientras que las de las otras cuatro placas fueron de 99,7%. Las características de sensibilidad y especificidad de estos medios los hace recomendables para la detección de portadores de VRE cuando esta sea pertinente. Son, cuando menos, perfectamente equiparables al agar BEAV, disminuyendo el tiempo de detección en 24-48 h.
Detección de microorganismos gramnegativosEn 1979, Kilian y Blow describen un nuevo medio de cultivo que utiliza la β-glucoronidasa como sustrato para la detección directa de E. coli en urocultivos. Los estudios de rentabilidad indicaban que este método suponía un ahorro económico del 46% y una reducción del tiempo de identificación del 64% con respecto a los métodos convencionales39. Desde entonces se han desarrollado numerosos MC con diferentes sustratos para la identificación de los principales uropatógenos. Unos de los medios comercializados recientemente es chromID CPS Elite (bioMérieux, Durham, NC) que permite la identificación directa de E. coli, y la identificación presuntiva de Enterococcus spp., de algunas Enterobacteriaceae y de las bacterias del grupo Proteae, aunque en estos casos es necesaria la confirmación de la identificación mediante pruebas bioquímicas o EM MALDI-TOF.
Se ha evaluado la efectividad de este medio para la detección de uropatógenos43, valorando el tiempo requerido para el diagnóstico y los consumibles utilizados en comparación con la utilización de agar sangre y agar MacConkey. La concordancia de este medio con la metodología convencional fue del 88% para orinas clínicamente significativas, del 74% para orinas con crecimiento no significativo, el 69% para las contaminadas y un 95% para placas sin crecimiento bacteriano. Las principales discrepancias se debieron al crecimiento en agar sangre, pero no en el MC de bacterias grampositivas, en general con poca relevancia clínica en urocultivos, como Staphylococcus spp. y Lactobacillus. En cuanto al tiempo transcurrido entre la siembra de la muestra y la identificación de las colonias, la diferencia no fue estadísticamente significativa si se comparan los dos medios de forma global (27,2 h de media para el método convencional frente a 26,6h para el MC), pero sí lo fue en aquellas orinas en las que creció E. coli en cultivo puro (27,1 h en el método convencional frente a 24,4 h en chromID, p<0,0001). Los autores aducen que esta mejora de tiempo es debida a la facilidad y fiabilidad de la identificación por el color de la colonia, que evita la necesidad de métodos de identificación complementarios.
Los medios chromID™ CPS (CPS4) (bioMérieux, St. Laurent, QC) y UriSelect™ 4 (URS4) (Bio-Rad, Montreal, QC), han sido evaluados44, sobre 903 muestras de orina, en comparación también con el método convencional de agar sangre y agar MacConkey. La concordancia con el método convencional fue del 89,3 y 89,5% para URS4 y CPS4 respectivamente. Si se consideran solamente aquellas muestras en las que el crecimiento se consideró clínicamente significativo, esta concordancia aumentó a un 93% en el caso de URS4 y a un 93,1% para el medio CPS4.
En todos los casos, los autores coinciden en que la mayor ventaja del uso de MC es la agilización del proceso de identificación, al reducirse la necesidad de pruebas complementarias, lo que permite realizar un diagnóstico etiológico con mayor rapidez.
Dada la utilidad demostrada de esta herramienta diagnóstica, varios grupos están diseñando nuevos MC, algunos aún no comercializados, que pueden suponer una alternativa interesante en el futuro. Se ha diseñado un nuevo medio45 para la detección de Bacteroides, que incluye en su formulación 3,4-ciclohexanoesculetina-β−D-glucósido, que es la diana de acción de la β-glucosidasa de Bacteroides, y que da lugar a la aparición de colonias de color negro. Este medio se ha comparado con el agar bilis esculina en 100 muestras fecales. El MC permitió la identificación de B. fragilis en 34 muestras, mientras que en agar bilis esculina solo se detectó este microorganismo en 19 muestras. Además, el MC resultó mucho más selectivo, ya que en ningún caso se observó crecimiento de especies no pertenecientes al género Bacteroides, mientras en agar bilis esculina se observó crecimiento de microorganismos pertenecientes a otros géneros (enterobacterias, Clostridium, Candida…) en 34 ocasiones. Los autores estudian además la capacidad de estos MC, suplementados con meropenem y con metronidazol, para detectar la presencia de Bacteroides resistentes a uno u otro antimicrobiano, demostrando que en ambos casos son capaces de detectar cepas resistentes con una sensibilidad del 100% y una especificidad de, al menos, el 85%.
Otro medio novedoso desarrollado es el CHROMOagar Yersinia enterocolitica que presenta como ventaja frente al medio Cefsulodina-Irgasan-Novobiocina la posibilidad de crecimiento de cepas de Y. enterocolitica serovar O3 y de Y. psedotuberculosis, cuyo crecimiento puede verse inhibido en el medio CIN46.
Detección de levadurasAunque los primeros MC desarrollados para hongos, a principios de los 90, tuvieron poco éxito en su aplicación clínica por su limitada capacidad de diferenciación, los MC para micología desarrollados en los últimos 15 años han tenido un éxito mucho mayor, ya que permiten una identificación presuntiva rápida de diferentes especies de levaduras, un mejor estudio de cultivos mixtos, y la identificación precoz de especies asociadas a resistencia a antifúngicos. Los medios desarrollados se basan, como en los casos anteriores, en la presencia de sustratos para una o varias enzimas (β-N-acetil hexosaminidasa, y en algunos casos, β-glucosidasa o fosfatasa), que permiten la identificación rápida, como mínimo, de Candida albicans, y en numerosos casos la identificación definitiva o presuntiva de Candida glabrata, Candida lusitaniae, Candida kefyr, C. parapsilosis, Candida tropicalis y Candida krusei. Los estudios disponibles y la ya prolongada experiencia con este tipo de medios demuestran una alta especificidad de identificación y una buena sensibilidad. No obstante, dada la variabilidad de color y morfología que puede aparecer en aislamientos de la misma especie, la identificación debe siempre ser considerada presuntiva. Un estudio reciente sobre más de 5.600 muestras clínicas positivas para levaduras, demuestra que más del 8% eran cultivos mixtos, cuya detección es mucho más probable si se utilizan MC, y propone un algoritmo que combina MC y EM MALDI-TOF como procedimiento más adecuado de identificación47.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.