Salmonella spp. es un enteropatógeno que se transmite a los humanos a través de alimentos o agua contaminada. En 1997, el Grupo de Microbiología del Instituto Nacional de Salud de Colombia inició el programa de vigilancia de enfermedad diarreica aguda y fiebre tifoidea, que incluye Salmonella spp. Este informe presenta los resultados fenotípicos y genotípicos de los aislamientos recuperados de 2005 a 2011 como parte de la vigilancia.
MétodosUn total de 4.010 aislamientos de Salmonella spp. fueron analizados por serotipificación con el esquema Kauffmann-White-LeMinor, patrones de sensibilidad antimicrobiana y de electroforesis en gel de campos pulsados (PFGE).
ResultadosSe identificaron un total de 93 serovares, con 9 predominantes, Typhimurium, Enteritidis, Typhi, Dublin, Panama, Derby, Braenderup, Saintpaul y Uganda. Salmonella spp. presentó altos porcentajes de resistencia a tetraciclina y ácido nalidíxico. El 52,4% (2.101/4.010) de los aislamientos fueron sensibles a todos los antibióticos. La multirresistencia se observó en el 54,9% de los aislamientos de Typhimurium, representada por 81 combinaciones. Por PFGE se analizaron 51,9% aislamientos (2.083/4.010) de 34 serovares, generando 828 patrones electroforéticos XbaI. De estos, 8 se reportaron en al menos 2 países en Latinoamérica.
ConclusiónLa vigilancia de Salmonella spp. permite conocer la distribución de los serovares, su resistencia y la identificación de clones endémicos en Colombia, aportando bases para un tratamiento óptimo en las infecciones generadas por este patógeno y en el diseño de programas para disminuir la dispersión de aislamientos multirresistentes.
Salmonella is an enteropathogen acquired through contaminated food or water. In Colombia, Salmonella spp. is included in the national surveillance of Acute Diarrhoeal Diseases and typhoid fever initiated in 1997. This report shows the phenotype and genotype results obtained from 2005 to 2011.
MethodsA total of 4010 isolates of Salmonella enterica were analysed by serotyping with Kauffmann-White-LeMinor, antimicrobial resistance patterns, and pulse-field gel electrophoresis (PFGE).
ResultsA total of 93 serovars were identified, of which, Typhimurium, Enteritidis, Typhi, Dublin, Panama, Derby, Braenderup, Saintpaul, and Uganda were prominent. The highest levels of resistance were found for tetracycline and nalidixic acid. Susceptibility was observed in 52.4% (2101/4010) of the isolates. Multi-resistance was recorded in 54.9% of Typhimurium isolates, with 81 different combinations. Using PFGE, 51.9% (2083/4010) isolates were analysed in 34 serovars, and 828 electrophoretic patterns were obtained. From these, 8 patterns were found in at least two Latin-American countries.
ConclusionThe surveillance of Salmonella spp. provides information on the serovar distribution, antimicrobial resistance, and clonal distribution in Colombia, as well as information to treat this disease and control the spread of antimicrobial bacterial resistance.
Salmonella spp. es un enteropatógeno que se transmite a los humanos a través de alimentos o agua contaminada, por lo que se clasifica como una enfermedad transmitida por alimentos (ETA)1. Las personas infectadas con Salmonella spp. presentan síntomas como diarrea, fiebre y dolor abdominal. Afecta principalmente a niños menores de 5años causando enfermedad diarreica aguda (EDA)2. En Estados Unidos, en el año 2013 se reportaron 19.056 casos, 4.200 hospitalizaciones y 80 muertes por ETA3. Es por eso que la reducción de la infección se encuentra dentro de los 5 temas prioritarios para el Departamento de Servicios Humanos y de Salud de Estados Unidos desde 2012. Estas medidas son el resultado de la información generada a partir de la vigilancia por laboratorio que sirve como guía para direccionar los esfuerzos para la prevención de la ETA3,4. En Colombia, la mortalidad por EDA en la población en general es de 1,57 por 100.000 habitantes, con una letalidad del 0,04% para el año 2011. Es más frecuente en menores de 10años, pero el riesgo de muerte es mayor en los pacientes mayores de 80años5.
En Salmonella spp. se han descrito más de 2.600 serovares en 2 especies: S.bongori y S.enterica. Esta última se subdivide en 7 subespecies, y los serovares patógenos para el humano pertenecen a la subespecie enterica. Los serovares Typhimurium y Enteritidis se recuperan a nivel mundial de cuadros de gastroenteritis y tienen un amplio rango de hospederos, mientras que los serovares Typhi (restringido al humano como único hospedero), Sendai y Paratyphi A, B, C causan la fiebre tifoidea6-8.
El Centro de Control y Prevención de Enfermedades de Estados Unidos estima que Salmonella spp. produce un millón de casos de enfermedad, con 19.000 hospitalizaciones y 380 muertes al año1.
En Colombia, la vigilancia nacional de Salmonella spp. se estableció a partir de 1997 como una red de información que incluye el envío de los aislamientos desde los hospitales del país, a los 32 Laboratorios Departamentales de Salud Pública (LDSP) y el Distrito capital, y de ellos, al Grupo de Microbiología del Instituto Nacional de Salud (INS), donde se realiza la caracterización fenotípica por serotipificación y perfil de sensibilidad antimicrobiana y genotípica por electroforesis en gel de campos pulsados (PFGE).
Este informe presenta los resultados obtenidos en la vigilancia fenotípica y genotípica de aislamientos de Salmonella spp. provenientes de muestras clínicas humanas, desde el año 2005 al 2011, dentro del programa de vigilancia de EDA y fiebre tifoidea.
Materiales y métodosCaracterización fenotípicaLos aislamientos de Salmonella spp. remitidos al Grupo de Microbiología-INS fueron confirmados por pruebas bioquímicas9 y serotipificación siguiendo el esquema de Kauffmann-White-Le Minor10.
Se determinaron los perfiles de susceptibilidad antimicrobiana por el método de difusión de disco (Kirby-Bauer) siguiendo las recomendaciones y criterios de interpretación del Instituto de Estándares de Laboratorio Clínico (CLSI)11 a tetraciclina (TE), cloranfenicol (C), ácido nalidíxico (NA), amoxicilina-ácido clavulánico (AMC), aztreonam (AZT), amikacina (AK) y estreptomicina (S); estos 3 últimos antibióticos solo se evaluaron para el serovar Typhimurium. La concentración mínima inhibitoria (CMI) se determinó en el equipo AutoSCAN-4 (Siemens, Alemania) con el panel NC50 a ampicilina (AMP), trimetoprim sulfametoxazol (SXT), ciprofloxacina (CIP), cefotaxima (CTX) y ceftazidima (CAZ). Los aislamientos sensibles a todos los antibióticos fueron definidos como pansensibles y los aislamientos con resistencia a 3 o más familias de antibióticos como multirresistentes (MDR).
Los serovares con un número total de aislamientos mayor o igual a 60 en los 7años de estudio se consideraron como los predominantes en el país.
Caracterización genotípicaLa caracterización genotípica por PFGE se realizó a un porcentaje del total de aislamientos siguiendo el procedimiento y los lineamientos establecidos por la Red PulseNet para América Latina y el Caribe (PulseNet-LA y Caribe)12. Los aislamientos debían cumplir con alguno de estos criterios: ser del serovar Typhi y de serovares frecuentes y poco frecuentes que presentaran características inusuales de tipo de muestra y perfil de resistencia y ser recuperados en brotes. Los serovares Panama y Saintpaul se procesaron siguiendo el protocolo de tio-urea de la Red PulseNet.
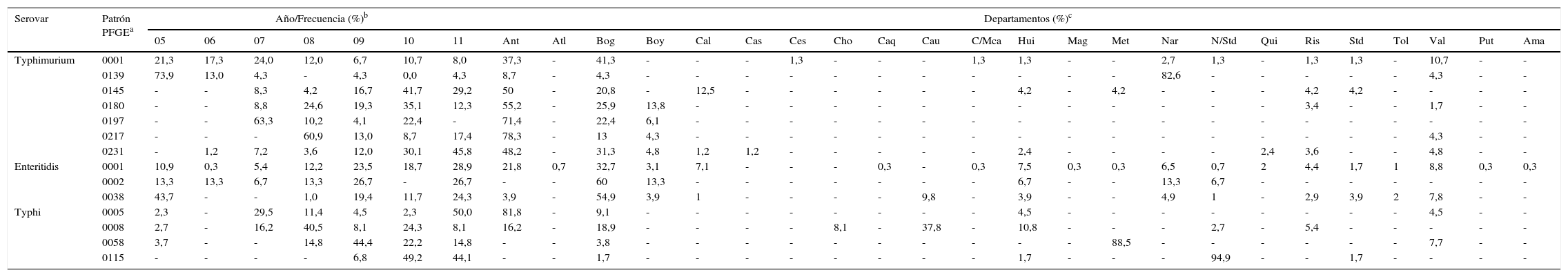
El patrón de PFGE obtenido para cada aislamiento se comparó con la base de datos nacional, conformada por un representante de cada patrón de PFGE encontrado en los diferentes serovares. Los patrones de PFGE se nombraron siguiendo los parámetros establecidos previamente por PulseNet13 (fig. 1). Posteriormente, los patrones de la base de datos nacional se compararon con la base de datos regional (BDR) de la Red PulseNet-LA y Caribe, donde se encuentran los perfiles electroforéticos de Salmonella enterica de los 17 países participantes en la Red14. Esta comparación permite identificar patrones compartidos con uno o más países de Latinoamérica al presentar 100% de similitud genética.
Principales patrones de PFGE en los 9 serovares predominantes de Salmonella spp. en Colombia. Se muestra el dendrograma generado con el algoritmo UPGMA, utilizando el coeficiente de Dice con 1,5% de tolerancia. Los patrones de PFGE son nombrados de acuerdo con los parámetros establecidos previamente por la Red PulseNet13.
CO: Colombia, IN: Instituto Nacional de Salud, 2 números que indican el año de recuperación del aislamiento, 3 letras en mayúscula que identifican cada serovar, 3 caracteres alfanuméricos que corresponden a la enzima de restricción (para XbaI es X01) y por último 4 números que corresponden al patrón asignado consecutivamente. Los patrones marcados con asterisco son los que comparten 100% de similitud con los patrones de la BDR. En la siguiente columna se indica el serovar, seguido del número de aislamientos con el respectivo patrón de PFGE (n). La columna Perfil R muestra el perfil de resistencia predominante en cada patrón de PFGE. La columna n Perfil R (%) muestra el número de aislamientos con el perfil de resistencia predominante, y el porcentaje se calculó n Perfil R/n.
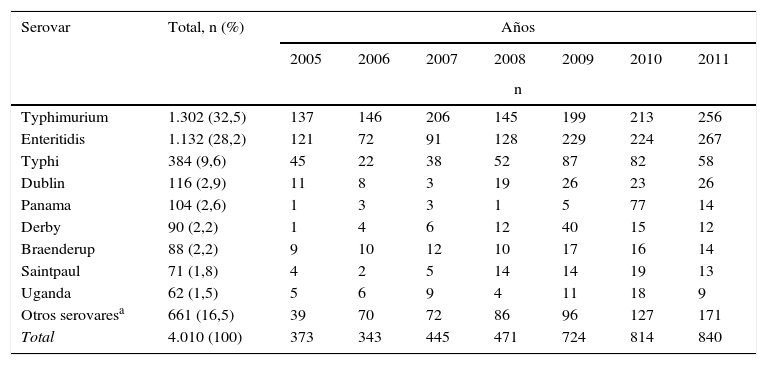
Durante los 7 años analizados se recibieron en el Grupo de Microbiología 4.010 aislamientos de Salmonella spp. en los que se identificaron 92 serovares. Tres serovares agruparon el 70,3% de los aislamientos: Typhimurium con 32,5%, Enteritidis con 28,2% y Typhi con 9,6%. Los siguientes 6 serovares agruparon el 13,2% de los aislamientos, y los restantes 83 serovares agruparon el 16,5%, con menos de 5 aislamientos cada uno (tabla 1).
Serovares de Salmonella spp. recuperados con mayor frecuencia en Colombia de 2005 a 2011
| Serovar | Total, n (%) | Años | ||||||
|---|---|---|---|---|---|---|---|---|
| 2005 | 2006 | 2007 | 2008 | 2009 | 2010 | 2011 | ||
| n | ||||||||
| Typhimurium | 1.302 (32,5) | 137 | 146 | 206 | 145 | 199 | 213 | 256 |
| Enteritidis | 1.132 (28,2) | 121 | 72 | 91 | 128 | 229 | 224 | 267 |
| Typhi | 384 (9,6) | 45 | 22 | 38 | 52 | 87 | 82 | 58 |
| Dublin | 116 (2,9) | 11 | 8 | 3 | 19 | 26 | 23 | 26 |
| Panama | 104 (2,6) | 1 | 3 | 3 | 1 | 5 | 77 | 14 |
| Derby | 90 (2,2) | 1 | 4 | 6 | 12 | 40 | 15 | 12 |
| Braenderup | 88 (2,2) | 9 | 10 | 12 | 10 | 17 | 16 | 14 |
| Saintpaul | 71 (1,8) | 4 | 2 | 5 | 14 | 14 | 19 | 13 |
| Uganda | 62 (1,5) | 5 | 6 | 9 | 4 | 11 | 18 | 9 |
| Otros serovaresa | 661 (16,5) | 39 | 70 | 72 | 86 | 96 | 127 | 171 |
| Total | 4.010 (100) | 373 | 343 | 445 | 471 | 724 | 814 | 840 |
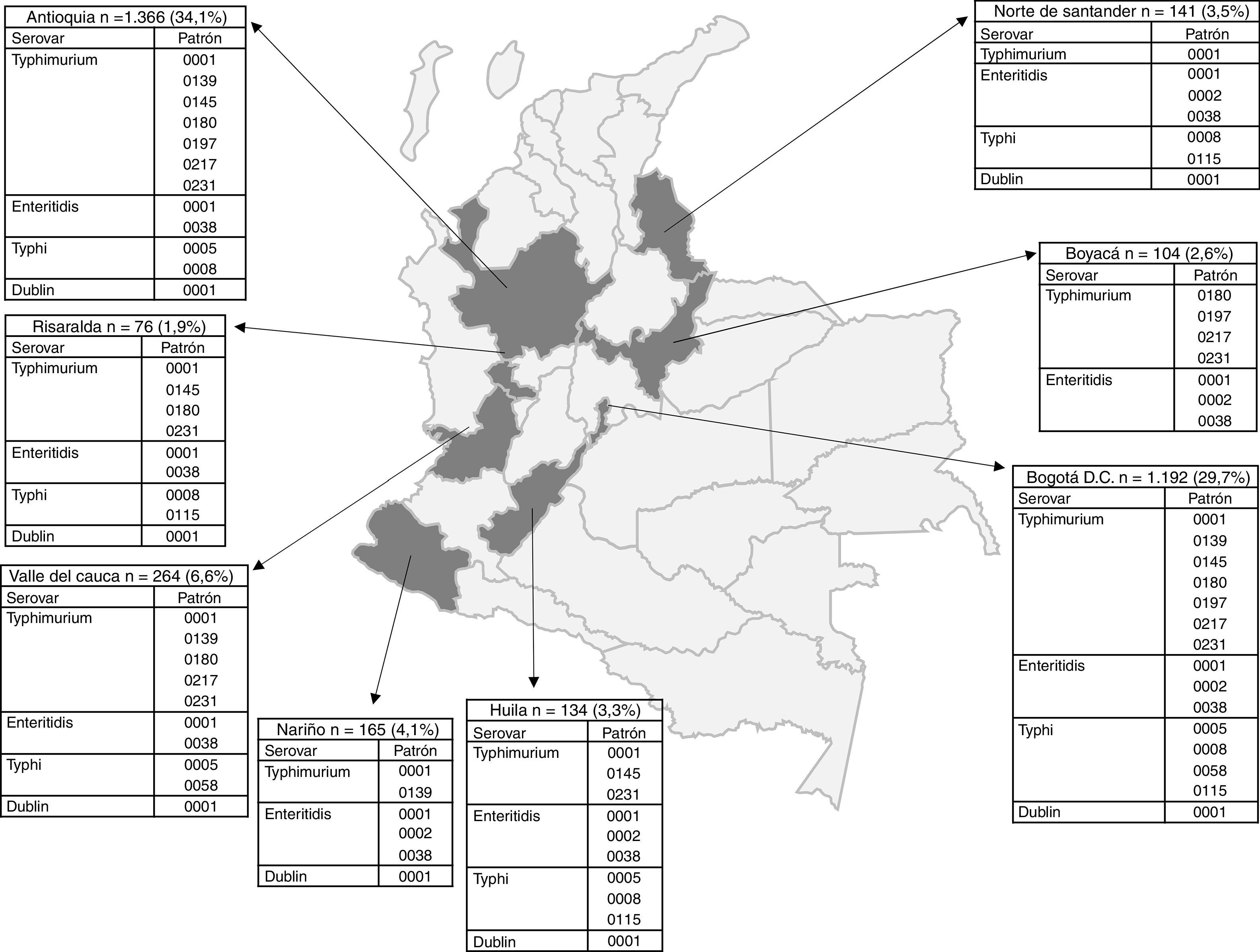
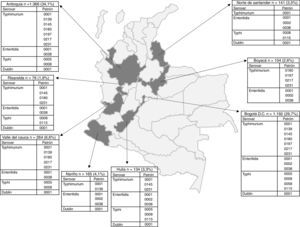
El número de aislamientos de Salmonella spp. recibidos aumentó por año de 373 en 2005 a 840 en 2011 (tabla 1). El departamento de Antioquia y el Distrito capital de Bogotá aportaron el 63,8% de los aislamientos, con 1.366 (34,1%) y 1.192 (29,7%), respectivamente, seguidos por los departamentos de Valle, Nariño y Santander, con 264 (6,6%), 165 (4,1%) y 141 (3,5%) aislamientos (fig. 2). El serovar Typhimurium fue predominante hasta 2008, y fue remplazado por Enteritidis. Durante los años 2008 a 2010 se presentó un incremento en el número de aislamientos del serovar Typhi (tabla 1).
Distribución por departamento de los patrones de PFGE predominantes de Salmonella Typhimurium, Enteritidis y Typhi circulantes en Colombia desde el año 2005 hasta el año 2011. En la figura se muestra la división política de Colombia y en gris oscuro los departamentos de los que se analizaron los datos por presentar el mayor número aislamientos con patrones de PFGE predominantes. En los recuadros se indica el nombre del departamento y el número de aislamientos enviados por el respectivo laboratorio de salud pública entre los años 2005 a 2011 al programa de vigilancia de enfermedad diarreica aguda (EDA) y enfermedad transmitida por alimentos (ETA). El porcentaje se calculó sobre el número de aislamientos totales recuperados durante los 7años de estudio (n=4.010). La nomenclatura completa de los patrones para Typhimurium es COINJPXX01.----; para Enteritidis es COINJEGX01.----; para Typhi es COINJPPX01.----; para Dublin es COINJDXX01,----. Ver figura 1.
Por PFGE se analizaron 2.083 aislamientos (51,9%), de los cuales 1.976 (49,2%) correspondieron a los 9 serovares predominantes (tabla S1).
Salmonella TyphimuriumFue el principal serovar recuperado durante los años analizados, con un total de 1.302 (32,5%) aislamientos (tabla 1). Principalmente se obtuvo de muestras de materia fecal (73,7%) en pacientes de todas las edades, seguido de hemocultivos (19,5%), donde un 78,4% (200/255) eran de mayores de 14años. Los aislamientos restantes se obtuvieron de 10 muestras diferentes de otros procesos invasores.
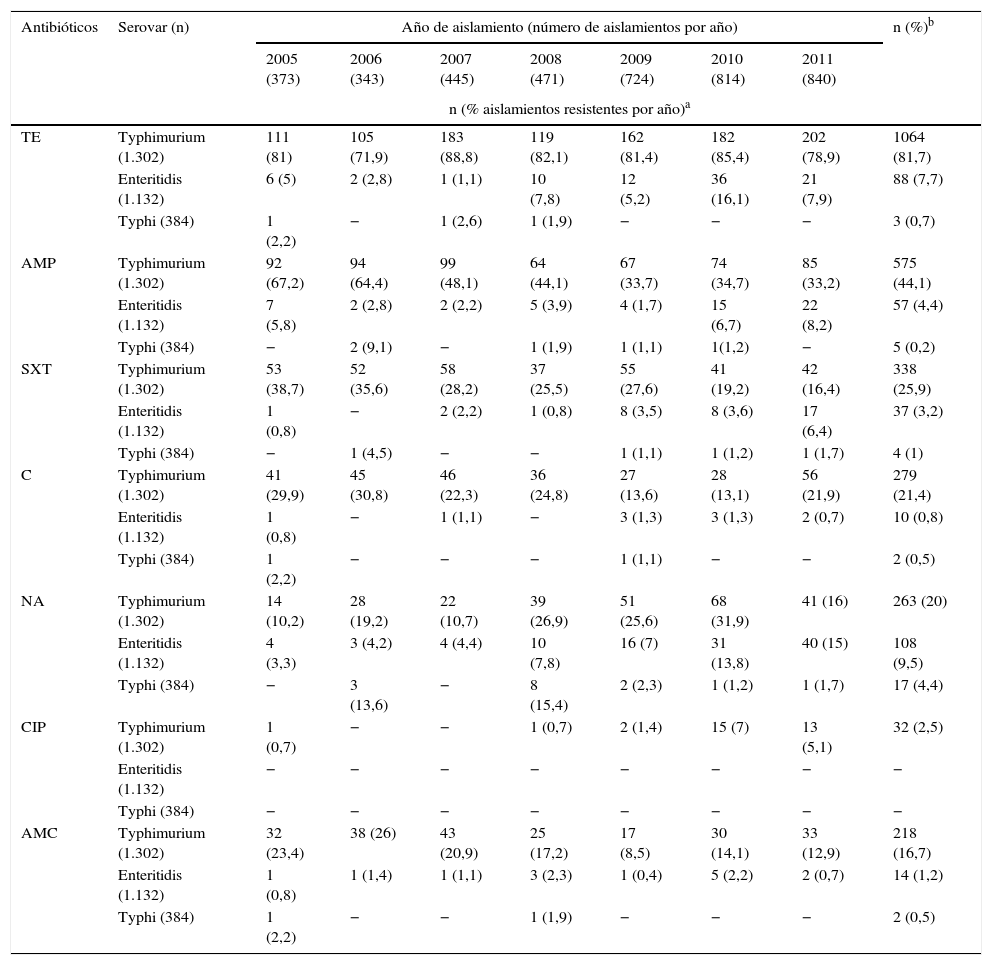
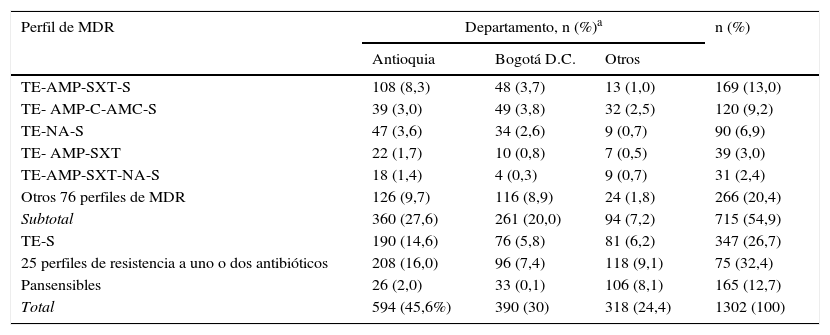
El análisis de la resistencia en este serovar mostró que 1.064 (82%) aislamientos fueron resistentes a TE (tabla 2). La resistencia a AMP, SXT, C y AMC fue del 44, del 26, del 21 y del 17%, respectivamente, siendo más baja en los últimos años del estudio, contrariamente a lo observado con la resistencia a NA, que se incrementó del 10% en 2005 al 32% en 2010 y disminuyó al 16% en el año 2011, mientras que la resistencia a CIP se determinó en el 2,5% de aislamientos. Para las cefalosporinas de tercera generación se presentó una resistencia del 2%. La MDR se observó en el 54,9% de los aislamientos, representada por 81 perfiles, de los cuales el más común fue TE-AMP-SXT-S en 169 (13%) aislamientos, la mayoría provenientes de Antioquia. El segundo perfil más frecuente fue TE-AMP-C-AMC-S, que se encontró en 120 (9,2%) aislamientos, principalmente de Antioquia y Bogotá. El 12,7% de los aislamientos fueron pansensibles (tabla 3).
Porcentaje de resistencia por año en aislamientos de Salmonella Typhimurium, Enteritidis y Typhi recuperados en Colombia de 2005 a 2011
| Antibióticos | Serovar (n) | Año de aislamiento (número de aislamientos por año) | n (%)b | ||||||
|---|---|---|---|---|---|---|---|---|---|
| 2005 (373) | 2006 (343) | 2007 (445) | 2008 (471) | 2009 (724) | 2010 (814) | 2011 (840) | |||
| n (% aislamientos resistentes por año)a | |||||||||
| TE | Typhimurium (1.302) | 111 (81) | 105 (71,9) | 183 (88,8) | 119 (82,1) | 162 (81,4) | 182 (85,4) | 202 (78,9) | 1064 (81,7) |
| Enteritidis (1.132) | 6 (5) | 2 (2,8) | 1 (1,1) | 10 (7,8) | 12 (5,2) | 36 (16,1) | 21 (7,9) | 88 (7,7) | |
| Typhi (384) | 1 (2,2) | − | 1 (2,6) | 1 (1,9) | − | − | − | 3 (0,7) | |
| AMP | Typhimurium (1.302) | 92 (67,2) | 94 (64,4) | 99 (48,1) | 64 (44,1) | 67 (33,7) | 74 (34,7) | 85 (33,2) | 575 (44,1) |
| Enteritidis (1.132) | 7 (5,8) | 2 (2,8) | 2 (2,2) | 5 (3,9) | 4 (1,7) | 15 (6,7) | 22 (8,2) | 57 (4,4) | |
| Typhi (384) | − | 2 (9,1) | − | 1 (1,9) | 1 (1,1) | 1(1,2) | − | 5 (0,2) | |
| SXT | Typhimurium (1.302) | 53 (38,7) | 52 (35,6) | 58 (28,2) | 37 (25,5) | 55 (27,6) | 41 (19,2) | 42 (16,4) | 338 (25,9) |
| Enteritidis (1.132) | 1 (0,8) | − | 2 (2,2) | 1 (0,8) | 8 (3,5) | 8 (3,6) | 17 (6,4) | 37 (3,2) | |
| Typhi (384) | − | 1 (4,5) | − | − | 1 (1,1) | 1 (1,2) | 1 (1,7) | 4 (1) | |
| C | Typhimurium (1.302) | 41 (29,9) | 45 (30,8) | 46 (22,3) | 36 (24,8) | 27 (13,6) | 28 (13,1) | 56 (21,9) | 279 (21,4) |
| Enteritidis (1.132) | 1 (0,8) | − | 1 (1,1) | − | 3 (1,3) | 3 (1,3) | 2 (0,7) | 10 (0,8) | |
| Typhi (384) | 1 (2,2) | − | − | − | 1 (1,1) | − | − | 2 (0,5) | |
| NA | Typhimurium (1.302) | 14 (10,2) | 28 (19,2) | 22 (10,7) | 39 (26,9) | 51 (25,6) | 68 (31,9) | 41 (16) | 263 (20) |
| Enteritidis (1.132) | 4 (3,3) | 3 (4,2) | 4 (4,4) | 10 (7,8) | 16 (7) | 31 (13,8) | 40 (15) | 108 (9,5) | |
| Typhi (384) | − | 3 (13,6) | − | 8 (15,4) | 2 (2,3) | 1 (1,2) | 1 (1,7) | 17 (4,4) | |
| CIP | Typhimurium (1.302) | 1 (0,7) | − | − | 1 (0,7) | 2 (1,4) | 15 (7) | 13 (5,1) | 32 (2,5) |
| Enteritidis (1.132) | − | − | − | − | − | − | − | − | |
| Typhi (384) | − | − | − | − | − | − | − | − | |
| AMC | Typhimurium (1.302) | 32 (23,4) | 38 (26) | 43 (20,9) | 25 (17,2) | 17 (8,5) | 30 (14,1) | 33 (12,9) | 218 (16,7) |
| Enteritidis (1.132) | 1 (0,8) | 1 (1,4) | 1 (1,1) | 3 (2,3) | 1 (0,4) | 5 (2,2) | 2 (0,7) | 14 (1,2) | |
| Typhi (384) | 1 (2,2) | − | − | 1 (1,9) | − | − | − | 2 (0,5) | |
AMC: amoxicilina ácido clavulánico; AMP: ampicilina; C: cloranfenicol; CIP: ciprofloxacina; NA: ácido nalidíxico; SXT: trimetoprim sulfametoxazol; TE: tetraciclina.
El porcentaje de aislamientos resistentes por año para cada serovar, se calculó sobre el n por serovar por año teniendo cuenta los valores registrados en la tabla 1.
Perfiles de MDR observados en los aislamientos de Salmonella Typhimurium recuperados en Colombia de 2005 a 2011
| Perfil de MDR | Departamento, n (%)a | n (%) | ||
|---|---|---|---|---|
| Antioquia | Bogotá D.C. | Otros | ||
| TE-AMP-SXT-S | 108 (8,3) | 48 (3,7) | 13 (1,0) | 169 (13,0) |
| TE- AMP-C-AMC-S | 39 (3,0) | 49 (3,8) | 32 (2,5) | 120 (9,2) |
| TE-NA-S | 47 (3,6) | 34 (2,6) | 9 (0,7) | 90 (6,9) |
| TE- AMP-SXT | 22 (1,7) | 10 (0,8) | 7 (0,5) | 39 (3,0) |
| TE-AMP-SXT-NA-S | 18 (1,4) | 4 (0,3) | 9 (0,7) | 31 (2,4) |
| Otros 76 perfiles de MDR | 126 (9,7) | 116 (8,9) | 24 (1,8) | 266 (20,4) |
| Subtotal | 360 (27,6) | 261 (20,0) | 94 (7,2) | 715 (54,9) |
| TE-S | 190 (14,6) | 76 (5,8) | 81 (6,2) | 347 (26,7) |
| 25 perfiles de resistencia a uno o dos antibióticos | 208 (16,0) | 96 (7,4) | 118 (9,1) | 75 (32,4) |
| Pansensibles | 26 (2,0) | 33 (0,1) | 106 (8,1) | 165 (12,7) |
| Total | 594 (45,6%) | 390 (30) | 318 (24,4) | 1302 (100) |
AMC: amoxicilina ácido clavulánico; AMP: ampicilina; C: cloranfenicol; NA: ácido nalidíxico; SXT: trimetoprim sulfametoxazol; TE: tetraciclina.
Por PFGE, del total de 1.302 aislamientos se procesaron 967 (74%), los cuales se agruparon en 439 (45% de variabilidad genética) patrones electroforéticos (tabla S1). De estos, 7 patrones se observaron en el 34% de los aislamientos. Los patrones ampliamente distribuidos fueron COINJPXX01.0001 (22%), que circuló durante los 7años en 10 departamentos, el COINJPXX01.0231 (24%), que circula desde el año 2006 en 9 departamentos, y el COINJPXX01.0145 (7,2%), que circula desde el año 2007 en 7 departamentos (figs. 1 y 2 y tabla 4). El patrón COINJPXX01.0180 (17,4%) se recuperó principalmente en Antioquia y Bogotá, además de otros 3 departamentos. COINJPXX01.0139 (6,9%) y COINJPXX01.0217 (6,9%) se recuperaron en 4 departamentos, siendo predominantes en Nariño y Antioquia, respectivamente. El patrón COINJPXX01.0197 (14,7%) se recuperó en 3 departamentos (tabla 4, fig. 2).
Distribución de los patrones de PFGE predominantes de Salmonella Typhimurium, Enteritidis y Typhi encontrados en Colombia de 2005 a 2011
| Serovar | Patrón PFGEa | Año/Frecuencia (%)b | Departamentos (%)c | ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 05 | 06 | 07 | 08 | 09 | 10 | 11 | Ant | Atl | Bog | Boy | Cal | Cas | Ces | Cho | Caq | Cau | C/Mca | Hui | Mag | Met | Nar | N/Std | Qui | Ris | Std | Tol | Val | Put | Ama | ||
| Typhimurium | 0001 | 21,3 | 17,3 | 24,0 | 12,0 | 6,7 | 10,7 | 8,0 | 37,3 | - | 41,3 | - | - | - | 1,3 | - | - | - | 1,3 | 1,3 | - | - | 2,7 | 1,3 | - | 1,3 | 1,3 | - | 10,7 | - | - |
| 0139 | 73,9 | 13,0 | 4,3 | - | 4,3 | 0,0 | 4,3 | 8,7 | - | 4,3 | - | - | - | - | - | - | - | - | - | - | - | 82,6 | - | - | - | - | - | 4,3 | - | - | |
| 0145 | - | - | 8,3 | 4,2 | 16,7 | 41,7 | 29,2 | 50 | - | 20,8 | - | 12,5 | - | - | - | - | - | - | 4,2 | - | 4,2 | - | - | - | 4,2 | 4,2 | - | - | - | - | |
| 0180 | - | - | 8,8 | 24,6 | 19,3 | 35,1 | 12,3 | 55,2 | - | 25,9 | 13,8 | - | - | - | - | - | - | - | - | - | - | - | - | - | 3,4 | - | - | 1,7 | - | - | |
| 0197 | - | - | 63,3 | 10,2 | 4,1 | 22,4 | - | 71,4 | - | 22,4 | 6,1 | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | |
| 0217 | - | - | - | 60,9 | 13,0 | 8,7 | 17,4 | 78,3 | - | 13 | 4,3 | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | 4,3 | - | - | |
| 0231 | - | 1,2 | 7,2 | 3,6 | 12,0 | 30,1 | 45,8 | 48,2 | - | 31,3 | 4,8 | 1,2 | 1,2 | - | - | - | - | - | 2,4 | - | - | - | - | 2,4 | 3,6 | - | - | 4,8 | - | - | |
| Enteritidis | 0001 | 10,9 | 0,3 | 5,4 | 12,2 | 23,5 | 18,7 | 28,9 | 21,8 | 0,7 | 32,7 | 3,1 | 7,1 | - | - | - | 0,3 | - | 0,3 | 7,5 | 0,3 | 0,3 | 6,5 | 0,7 | 2 | 4,4 | 1,7 | 1 | 8,8 | 0,3 | 0,3 |
| 0002 | 13,3 | 13,3 | 6,7 | 13,3 | 26,7 | - | 26,7 | - | - | 60 | 13,3 | - | - | - | - | - | - | - | 6,7 | - | - | 13,3 | 6,7 | - | - | - | - | - | - | - | |
| 0038 | 43,7 | - | - | 1,0 | 19,4 | 11,7 | 24,3 | 3,9 | - | 54,9 | 3,9 | 1 | - | - | - | - | 9,8 | - | 3,9 | - | - | 4,9 | 1 | - | 2,9 | 3,9 | 2 | 7,8 | - | - | |
| Typhi | 0005 | 2,3 | - | 29,5 | 11,4 | 4,5 | 2,3 | 50,0 | 81,8 | - | 9,1 | - | - | - | - | - | - | - | - | 4,5 | - | - | - | - | - | - | - | - | 4,5 | - | - |
| 0008 | 2,7 | - | 16,2 | 40,5 | 8,1 | 24,3 | 8,1 | 16,2 | - | 18,9 | - | - | - | - | 8,1 | - | 37,8 | - | 10,8 | - | - | - | 2,7 | - | 5,4 | - | - | - | - | - | |
| 0058 | 3,7 | - | - | 14,8 | 44,4 | 22,2 | 14,8 | - | - | 3,8 | - | - | - | - | - | - | - | - | - | - | 88,5 | - | - | - | - | - | - | 7,7 | - | - | |
| 0115 | - | - | - | - | 6,8 | 49,2 | 44,1 | - | - | 1,7 | - | - | - | - | - | - | - | - | 1,7 | - | - | - | 94,9 | - | - | 1,7 | - | - | - | - | |
Se calculó: n encontrado por año de cada patrón sobre el n total de cada patrón encontrado durante los 7 años de estudio.
Departamentos donde se recuperaron los patrones PFGE predominantes. Ant: Antioquia, Atl: Atlántico, Bog: Bogotá, Boy: Boyacá, Cal: Caldas, Cas: Casanare, Ces: Cesar, Cho: Choco, Caq: Caquetá, Cau: Cauca, C/Mca: Cundinamarca, Hui: Huila, Mag: Magdalena, Met: Meta, Nar: Nariño, N/Std: Norte de Santander, Qui: Quindío, Ris: Risaralda, Std: Santander, Tol: Tolima, Val: Valle del Cauca, Put: Putumayo, Ama: Amazonas. Ver figuras 1 y 2.
En general, en los 7 patrones de PFGE más comunes para este serovar observamos 2 perfiles de resistencia. El perfil MDR TE-AMP-C-AMC-S se encontró en el 50% (38/75) de los aislamientos del patrón COINJPXX01.0001. El perfil de resistencia a TE y S fue el predominante en 5 de los 7 patrones (fig. 1).
La comparación de estos patrones con la BDR encontró un 100% de similitud del patrón COINJPXX01.0139 con el patrón ALJPXX01.0409 de Argentina, del COINJPXX01.0197 con el patrón ALJPXX01.0218 de Brasil y del COINJPXX01.0231 con el patrón ALJPXX01.0326 de Chile (tabla S3).
Salmonella EnteritidisEste serovar fue el segundo en número de aislamientos durante los 7años de estudio (1.132; 28,2%), y a partir del año 2009 desplazó al serovar Typhimurium (tabla 1). Se recuperó principalmente de materia fecal (66,8%) en igual porcentaje de adultos y niños, mientras que los aislamientos de hemocultivos (26,7%) provenían en su mayoría de pacientes mayores de 14años (69%).
La resistencia a TE, AMP, SXT, C y AMC se mantuvo por debajo del 9%. El único antibiótico para el cual se observó un incremento en la resistencia fue al NA: del 3% en 2005 al 15% en 2011 (tabla 2). Todos los aislamientos fueron sensibles a CIP y cefalosporinas de tercera generación.
De 1.132 aislamientos, se analizaron 523 por PFGE (46%), en los cuales se identificaron 130 (25% de variabilidad genética) patrones (tabla S1); de estos, 3 agruparon el 77% de los aislamientos y fueron recuperados principalmente en Bogotá. El principal patrón fue el COINJEGX01.0001 (56%), recuperado en 19 departamentos durante los 7años de estudio; el segundo fue el COINJEGX01.038 (18%), recuperado en 12 departamentos, y el tercero fue el COINJEGX01.002 (2,8%), recuperado principalmente en Bogotá y en 4 departamentos más (tabla 4, fig. 2). En estos 3 patrones de PFGE predominaron aislamientos sensibles (fig. 1).
La comparación con la BDR evidenció que el patrón COINJEGX01.0001 fue 100% idéntico con el patrón ALJEGX01.0010 reportado en Brasil y Paraguay, y el patrón COINJEGX01.0038 con el patrón regional para Latinoamérica ALJEGX01.0001 (tabla S3).
Salmonella TyphiFue el tercer serovar encontrado en la vigilancia, con 384 (9,6%) aislamientos. Se recuperó a partir de hemocultivos en un 82%, donde los mayores de 14años representaron el 71,2% de los pacientes. De materia fecal se recuperó en un 13,8%, donde los mayores de 14años representaron el 60,5%.
De 2008 a 2010 se presentó un incremento en el número de aislamientos de Typhi (tabla 1) en los departamentos de Meta y Norte de Santander.
El 93,4% de los aislamientos fueron pansensibles, la resistencia a NA se observó en el 4% aislamientos, y los porcentajes más altos de resistencia (14 y 15%) se presentaron en los años 2006 y 2008 (tabla 2).
Por PFGE se procesaron 314 aislamientos de 384 (89%), y se agruparon en 133 patrones electroforéticos (42% de variabilidad genética) (tabla S1). De estos, 4 patrones agruparon el 45% de los aislamientos y fueron constantes en los 7años de estudio (tabla 4). El patrón más frecuente fue COINJPPX01.0115 (18,8%), recuperado de 2009 a 2011 en un 95% en Norte de Santander (figs. 1 y 2 y tabla 4). En segundo lugar está el patrón COINJPPX01.0008 (11,8%), distribuido en 7 departamentos. El patrón COINJPPX01.0058 (8,2%) se recuperó principalmente en Meta además de Bogotá y Valle, mientras que el patrón COINJPPX01.0005 (7%) se encontró en mayor proporción en Antioquia, seguido de Bogotá, Huila y Valle (tabla 4, fig. 2).
La comparación con la BDR mostró 100% de similitud del patrón COINJPPX01.0005 con el patrón ALJPPX01.0016 y del patrón COINJPPX01.0058 con el patrón ALJPPX01.0048, ambos reportados en Argentina y Chile (tabla S3).
Serovares Dublin, Panama, Derby, Braenderup, Saintpaul y UgandaEstos serovares ocuparon del cuarto al noveno lugar en número de aislamientos (tabla 1). Se recuperaron principalmente de materia fecal (>74%), a excepción de Dublin, que se encontró en el 72% en hemocultivos. Aunque el número de aislamientos para cada uno de los serovares se mantiene constante a lo largo de los años, llama la atención el incremento observado en el serovar Derby para el año 2009 (tabla 1), relacionado con un brote en la ciudad de Barranquilla, departamento de Atlántico (datos no publicados), así como el incremento en los aislamientos del serovar Panama para el año 2010 (tabla 1), que aunque no estuvieron asociados a brote, el 81,8% (63/77) provenían del departamento de Antioquia de 19 laboratorios diferentes. En general, la resistencia más alta se observó a TE y NA. Para TE, los serovares Derby y Uganda fueron los más resistentes, con 87,5 y 56,5%, respectivamente. Para NA, el serovar Panama fue el más resistente, con 41%, seguido de Braenderup, con 34%. La resistencia a CIP solo se observó en el serovar Braenderup, con un 10% (tabla S2). Se determinaron entre 2 y 20 patrones de PFGE en estos 6 serovares (fig. 1, tabla S1). Al comparar los patrones predominantes de cada uno de estos serovares con la BDR, se encontró que solo el patrón COINJDXX01.0001 del serovar Dublin recuperado en varios departamentos (fig. 2) tuvo un 100% de similitud con el patrón ALJDXX01.0001 recuperado en Argentina y Guatemala (tabla S3).
Otros serovaresLos serovares restantes (n=83) se recuperaron principalmente de materia fecal en 74,1% y de hemocultivos en 14,8%. En la población mayor de 6años el 11,1% de los aislamientos provenían de orina (4,5%), secreciones (1,2%), líquido corporal (0,7%), líquido cefalorraquídeo (LCR) (0,6%), abscesos (0,4%), biopsias y heridas (0,3%), aspirado traqueal, vómito y tejido (0,2%) y no registran dato (2,5%). De estos, el serovar Javiana fue el que más se recuperó de LCR (5,8%). No se encontró un perfil de resistencia predominante. Por PFGE se analizaron 25 serovares relacionados con brotes de ETA o extraintestinales (tabla S1).
DiscusiónEl análisis de los datos obtenidos de la vigilancia de 2005 a 2011 mostró que los principales serovares causantes de salmonelosis en el país son Typhimurium, Enteritidis y Typhi, confirmando lo reportado en años anteriores13,15,16. En este estudio se observó un reemplazo de serovares de 2009 a 2011, cuando Enteritidis, con mayor distribución nacional, desplazó a Typhimurium. De los 32 departamentos del país y el Distrito capital de Bogotá que participan en la vigilancia, Antioquia y Bogotá fueron los que contribuyeron con el mayor número de aislamientos (fig. 2).
Typhimurium y Enteritidis se recuperaron principalmente de materia fecal en cuadros de gastroenteritis, manifestando así la capacidad de estos serovares de causar diarrea17,18. A nivel mundial, Enteritidis es el principal serovar en Europa, Asia y Latinoamérica, mientras que Typhimurium es el predominante en América del Norte y las regiones de Oceanía14,19. En Latinoamérica se ha observado un incremento del serovar Typhimurium del 14,5% en 2001 al 24% en 200719. La infección por estos 2 serovares se atribuye principalmente a la falta de higiene durante la preparación de alimentos como carnes y verduras para el caso de Typhimurium, así como a la falta de cocción en huevos contaminados y en pollo asado para Enteritidis, convirtiéndolos en un principal problema de salud pública3,20-23. A esta problemática se suma la MDR reportada para el serovar Typhimurium en todo el mundo, que se ha asociado con la capacidad de causar enfermedad invasiva en humanos3. En este estudio, identificamos varios perfiles de MDR en Typhimurium, pero ninguno asociado con alguno de los patrones de PFGE predominantes, lo que sugiere que la diseminación de la MDR en este serovar no es por dispersión clonal, sino posiblemente a través de elementos genéticos móviles.
Históricamente, Typhimurium fue el primer serovar de Salmonella en adquirir MDR a 5 familias de antibióticos diferentes, la cual fue reportada en la década de 1960 en el Reino Unido asociada a la producción de ganado y terneros, y se incrementó espectacularmente en la década de 1990, mientras que los reportes de MDR para el serovar Enteritidis se mantuvieron por debajo del 1%24. En general, esta tendencia se ha mantenido desde entonces, convirtiendo a Typhimurium en uno de los principales serovares de Salmonella asociado a MDR a nivel global, mientras que Enteritidis se mantiene con bajos niveles de MDR. Para 2013, en Estados Unidos, Typhimurium se reportó como uno de los serovares predominantes con MDR a 4, 5 y 7 antibióticos diferentes, mientras que Enteritidis fue el serovar más común en los aislamientos resistentes a ácido nalidíxico25. En Colombia, las diferencias en los niveles de resistencia entre estos 2 serovares, además de ser intrínsecas a cada uno de ellos, pueden deberse también al uso de antibióticos como promotores de crecimiento en la producción de porcinos y aves en el país. Estudios locales han documentado la presencia de Salmonella spp. en las etapas de pre-beneficio y beneficio en la producción de porcinos, de los cuales más del 90% son resistentes a 2 o más antibióticos26,27. El serovar Enteritidis se ha reportado en carne de pollo cruda y lista para venta, pero con bajos niveles de resistencia28,29. El serovar Typhimurium portador de genes de resistencia a uno o 5 antibióticos se encontró en muestras de alimentos procesados en puntos de venta30. Estos estudios sugieren una asociación entre los niveles de resistencia observados en los serovares Typhimurium y Enteritidis, recuperados tanto de las muestras clínicas como de algunos alimentos. Sin embargo, faltan más estudios en Colombia que nos permitan establecer la relación entre los serovares de Salmonella que circulan en animales, su resistencia y su relación directa con los aislamientos recuperados en las muestras clínicas. Igualmente, es preocupante la presencia de múltiples genes que confieren MDR a los aislamientos del serovar ParatyphiB variedad Java y Heidelberg en la producción avícola, ya que a pesar de no ser serovares con relevancia clínica en el país, pueden ser una fuente de diseminación de estos a los principales serovares que se recuperan en las muestras clínicas de humanos28,31.
En este estudio, la tipificación por PFGE reafirmó la diversidad genética del serovar Typhimurium, así como la homogeneidad del serovar Enteritidis observada previamente en aislamientos colombianos13,32. En Typhimurium, el patrón electroforético COINJPXX01.0001 se mantuvo como predominante en el país desde 1998 hasta 2011, a excepción del periodo entre 2002 y 2004, en el que fue reemplazado por el COINJPXX01.006213. Con respecto a los otros patrones, se observó un recambio de los predominantes en comparación a los reportados entre 1997 y 2004, reafirmando su plasticidad genética. Este resultado es importante porque generó patrones de PFGE regionales diferentes a los reportados, lo que sugiere un aumento en las fuentes comunes de contaminación, así como la aparición de clonas que tienen un alto potencial de diseminación en la región14. Para Enteritidis, 2 de los 3 patrones de PFGE predominantes en Colombia circulan en la región. Uno de ellos equivale al patrón prevalente en Latinoamérica (ALJEGX01.0001), mientras que el patrón ALJEGX01.0010 se ha reportado en Brasil y Paraguay. Estos resultados sugieren la diseminación de nuevas cepas de Enteritidis en la región14.
Typhi es el tercer serovar más importante en el país, lo que sugiere que la fiebre tifoidea continúa siendo un problema de salud pública para Colombia, a pesar de considerarse como una enfermedad de baja endemicidad33. Se estima que a nivel mundial la fiebre tifoidea causa alrededor de 11,9 millones de casos y alrededor de 129.000 defunciones asociados al consumo de agua contaminada34. En Colombia, para el año 2011 se notificaron al Sistema Nacional de Vigilancia 104 casos de fiebre tifoidea y paratifoidea, de los cuales el 93% (n=96) fueron confirmados por el laboratorio33. Los aislamientos de este serovar continúan siendo pansensibles en el país, lo cual es una ventaja para el tratamiento de la fiebre tifoidea. Por PFGE se ha determinado que los aislamientos de Typhi recuperados en el país son heterogéneos a nivel genético a pesar de que algunos patrones están relacionados con brotes14,35. Previamente se había reportado la circulación de 2 patrones compartidos con Argentina (ALJPPX01.0002 y ALJPPX01.0013)35, y para este estudio se identificaron 2 nuevos patrones (ALJPPX01.0016 y ALJPPX01.0048), compartidos con Argentina y Chile. Estos resultados sugieren la diseminación de cepas de Typhi en el continente, posiblemente a través de viajeros.
Los diferentes serovares no-tifoideos (NTS) recuperados con mayor frecuencia en las muestras clínicas son posiblemente un reflejo de los serovares que circulan en los alimentos para consumo humano. Los 9 serovares predominantes en Colombia difieren de los reportados en Europa y Estados Unidos. Se ha sugerido que los cambios en la frecuencia de serovares específicos puede ser el resultado de movimientos humanos, de animales y de alimentos, y por esta razón es importante la vigilancia constante de este patógeno en las comunidades36. Estos serovares son resistentes a uno, 2 o más antibióticos, siendo la TE y el NA los antibióticos a los que presentan mayor resistencia. Los serovares con altas tasas de resistencia a TE son los que se encuentran asociados con la producción bovina, porcina y avícola (Typhimurium, Derby, Saintpaul y Uganda), lo que sugiere un uso no controlado de este antibiótico en la producción de alimentos23,37-39, aunque en Colombia no tenemos evidencia directa de su uso indiscriminado. La resistencia registrada para NA nos alerta acerca de la posible tendencia de resistencia a fluoroquinolonas, especialmente en los serovares Typhimurium, Panama, Braenderup y Uganda, a pesar de la sensibilidad observada para CIP. Este resultado se debe probablemente a que se evaluó con los puntos de corte establecidos antes de 2012 por el CLSI, los cuales eran muy elevados para CIP40. Además, es importante resaltar que recientemente se ha reportado resistencia a TE y NA mayores al 60% en aislamientos de Salmonella spp. recuperados en carne de pollo en Colombia, lo que podría relacionarse con la resistencia observada en los aislamientos provenientes de humanos analizados en este estudio28. Los porcentajes de resistencia a cefalosporinas de tercera generación están por debajo del 1%, lo cual representa una ventaja de tratamiento ante los cuadros invasivos de salmonelosis, contrario a lo observado en varios países41,42.
Es importante mencionar que los NTS también se recuperaron de otras muestras asociadas con cuadros invasivos, como hemocultivos, LCR y orina. De estos, el serovar Dublin llama la atención porque se recuperó en hemocultivos después del serovar Typhi, lo que concuerda con lo descrito en la literatura acerca de su capacidad de generar cuadros invasivos en humanos43. Se han reportado cambios en la epidemiología de algunos de estos serovares en donde linajes clonales generan cuadros invasivos44. Aunque en Colombia desconocemos si existe una relación entre un linaje específico y los casos invasivos por NTS, los resultados sugieren un incremento de este tipo de aislamientos, similar a lo reportado en Europa y en Estados Unidos, por lo que es importante seguir vigilando el comportamiento de estos serovares en el país45.
LimitacionesEl proceso de vigilancia puede generar un subregistro en el sistema. No obstante, en este reporte se evidencia un incremento en la notificación al laboratorio y envío de aislamientos de Salmonella spp. al Grupo de Microbiología-INS en los años analizados.
ConclusionesEl análisis de los aislamientos de Salmonella spp. recuperados en el programa de vigilancia de 2005 a 2011 nos confirmó que los serovares Typhimurium, Enteritidis y Typhi persisten como los principales causantes de salmonelosis en el país. Se logró establecer la circulación de patrones de PFGE predominantes en cada serovar. En general, Salmonella spp. presenta en Colombia altos porcentajes de resistencia a TE y NA, pero siguen siendo sensibles a CIP y cefalosporinas de tercera generación. La vigilancia de este enteropatógeno permite conocer la dinámica de los serovares de Salmonella spp. y la identificación de clones endémicos en Colombia con el fin de aportar bases para un tratamiento óptimo en las infecciones generadas por este patógeno y poder diseñar programas para disminuir la dispersión de cepas MDR.
FinanciaciónGrupo de Microbiología, Dirección Redes en Salud Pública y Red Nacional de Laboratorios y Dirección de Investigación, Instituto Nacional de Salud, Colombia.
Conflicto de interesesLos autores manifiestan no tener ningún conflicto de intereses. Las personas mencionadas en los agradecimientos conocen el contenido del artículo y están de acuerdo con aparecer en los mismos.
Agradecemos a todos los LSP de Colombia, por su activa participación en el programa de Vigilancia de EDA. A la Dra. Elizabeth Castañeda, Investigadora Emérita del Instituto Nacional de Salud de Colombia, por su revisión crítica y aportes al manuscrito. A la Red PulseNet-LA y Caribe, por la comparación de nuestros patrones con la BDR, así como a las personas encargadas de la red en los diferentes países de Latinoamérica. A Enrique Perez Gutierrez, OPS/OMS del Departamento de Enfermedades Transmisibles y Análisis de la Salud, por el apoyo a la red PulseNet-LA y Caribe.