El programa de vacunación universal con la vacuna antineumocócica conjugada 13-valente (VNC13) se implantó en Andalucía en diciembre de 2016.
MétodosEstudio transversal de colonización nasofaríngea por Streptococcus pneumoniae. Se seleccionó a 397 niños sanos en centros de atención primaria de Sevilla durante los periodos 1/4/2018-28/2/2020 y 1/11/2021-28/2/2022 (periodo VNC13). Se utilizó una colección histórica de un estudio de colonización desarrollado en niños sanos y con infección respiratoria superior entre el 1/01/2006 y el 30/06/2008 (periodo VNC7) para comparar las distribuciones de serotipos/genotipos y las tasas de resistencias antibióticas.
ResultadosUn total de 76 (19%) niños estaban colonizados con S. pneumoniae en el periodo VNC13 y se dispuso de 154 aislamientos del periodo VNC7. La colonización por serotipos incluidos en VNC13 disminuyó significativamente entre los periodos VNC13 y VNC7 (11 vs. 38%; p=0,0001); los serotipos 19F (8%), 3 (1%) y 6B (1%) fueron los únicos serotipos vacunales circulantes. Los serotipos 15B/C y 11A fueron los serotipos no VNC13 más prevalentes durante el periodo VNC13 (14% y 11%, respectivamente); este último se incrementó de forma significativa entre periodos de tiempo (p=0,04). El serotipo 11A solo se asoció en el periodo VNC13 con variantes resistentes a la ampicilina del clon Spain9V-ST156 (ST6521 y genéticamente relacionado ST14698), no detectados en el periodo anterior.
ConclusionesHubo una circulación muy residual de los serotipos vacunales durante el periodo VNC13, con excepción del serotipo19F. El serotipo 11A se incrementó de forma significativa entre los periodos VNC13 y VNC7 por expansión clonal del genotipo resistente a la ampicilina ST6521.
The 13-valent pneumococcal conjugate vaccine (PCV13) universal vaccination program was introduced in December 2016 in Andalusia.
MethodsA cross-sectional study was conducted on the molecular epidemiology of pneumococcal nasopharyngeal colonization. A total of 397 healthy children were recruited from primary healthcare centres in Seville for the periods 1/4/2018 to 28/2/2020 and 1/11/2021 to 28/2/2022 (PCV13 period). Data from a previous carriage study conducted among healthy and sick children from 1/01/2006 to 30/06/2008 (PCV7 period) were used for comparison of serotype/genotype distributions and antibiotic resistance rates.
ResultsOverall, 76 (19%) children were colonized with S. pneumoniae during the PCV13 period and there were information available from 154 isolates collected during the PCV7 period. Colonization with PCV13 serotypes declined significantly in the PCV13 period compared with historical controls (11 vs. 38%, P=0.0001), being serotypes 19F (8%), 3 (1%) and 6B (1%) the only circulating vaccine types. Serotypes 15B/C and 11A were the most frequently identified non-PCV13 serotypes during the PCV13 period (14% and 11%, respectively); the later one increased significantly between time periods (P=0.04). Serotype 11A was exclusively associated in the PCV13 period with ampicillin-resistant variants of the Spain9V-ST156 clone (ST6521 and genetically related ST14698), not detected in the preceding period.
ConclusionsThere was a residual circulation of vaccine types following PCV13 introduction, apart from serotype 19F. Serotype 11A increased between PCV13 and PCV7 periods due to emergence and clonal expansion of ampicillin-resistant genotype ST6521.
Streptococcus pneumoniae es un comensal habitual de la nasofaringe humana. La colonización neumocócica es un proceso universal, con una mayor prevalencia en los niños pequeños y se considera un requisito para el desarrollo de la enfermedad neumocócica (mucosal o invasiva)1. Esta tiene una gran relevancia en la enfermedad infecciosa en todo el mundo por su prevalencia y elevada carga de enfermedad2.
Las vacunas neumocócicas conjugadas (VNC) han reducido de forma significativa la incidencia de enfermedad neumocócica invasiva (ENI) en la población inmunizada y también, aunque en menor escala, entre personas no inmunizadas por «efecto rebaño»3,4. No obstante, su efectividad está limitada por la cobertura parcial de serotipos y puede disminuir con el tiempo por el denominado fenómeno de reemplazamiento (incremento en la incidencia de ENI por serotipos no vacunales [SNV] debido a su mayor presencia en colonización nasofaríngea por la presión inmune vacunal sobre los serotipos vacunales [SV])5. La vigilancia activa de la epidemiología de la ENI tras la introducción de las VNC es fundamental para analizar el impacto vacunal en la incidencia de ENI en todos los grupos etarios, los cambios en la distribución de serotipos y la posible aparición de enfermedad de remplazamiento. Como complemento a la vigilancia epidemiológica en la ENI, los estudios transversales de portación nasofaríngea son de gran importancia para comprender los mecanismos subyacentes del «efecto rebaño» y del reemplazamiento serotípico e informan sobre los cambios en prevalencia, tasas de resistencia y potencial invasivo de los SNV reemplazantes en la nasofaringe.
La vacunación sistemática frente al neumococo con VNC tridecavalente (VNC13) se introdujo en diciembre de 2016 en el calendario de vacunación infantil de Andalucía (España). Diversas formulaciones de las VNC ya habían estado disponibles (7 y 10 valentes y VNC13 desde los años 2001, 2009 y 2010, respectivamente) en el mercado privado con tasas de vacunación antineumocócica variables (41-61%) entre la población pediátrica andaluza6,7. Las coberturas vacunales de VNC13 en la población pediátrica de Andalucía tras el inicio del programa de vacunación infantil tuvieron un rango del 97,2% en 2018 al 98,2% en 2020 (Moreno D. comunicación personal).
Nuestro grupo ha desarrollado 2estudios paralelos de vigilancia epidemiológica molecular de la ENI y de colonización de la nasofaringe en población pediátrica andaluza para una mejor comprensión de los beneficios del programa de vacunación sistemática frente al neumococo y para monitorizar la evolución de los SNV. El objetivo del presente trabajo fue describir la distribución de serotipos y genotipos y las tasas de resistencia antimicrobiana en los aislados neumocócicos obtenidos de la nasofaringe de niños sanos menores de 5 años atendidos en diversos centros de salud de la ciudad de Sevilla tras la introducción de la vacunación universal infantil con VNC13. Además y como objetivo secundario se ha comparado esta información con otra similar obtenida en los últimos 30 meses de un estudio transversal de portación nasofaríngea desarrollado en el mismo ámbito geográfico en un periodo con vacunación parcial con VNC 7 valente (VNC7)7,8.
MétodosDiseño, ámbito y periodo de estudioEstudio descriptivo observacional transversal sobre la epidemiología de la colonización nasofaríngea neumocócica. Se seleccionó a 394 niños sanos de edades comprendidas entre 6 meses y 5 años atendidos en consulta de niño sano o a demanda en 10 centros de atención primaria públicos y privados de la ciudad de Sevilla seleccionados por criterios logísticos. El estudio se extendió durante los periodos 1/4/2018-28/2/2020 y 1/11/2021-28/2/2022 (periodo VNC13).
Procedimientos de toma de muestras y recogida de datosSe seleccionó a los niños participantes sin que hubiera criterios de aleatorización previa. Este estudio se ha realizado teniendo en cuenta la legislación vigente, las normas éticas de la Declaración de Helsinki y las directrices de buenas prácticas de laboratorio clínico. A los padres o tutores legales se les informó de forma verbal y por escrito de las características del estudio y se les solicitó la firma del consentimiento informado. El estudio fue aprobado por el Comité Ético del Hospital Universitario Virgen del Rocío de Sevilla (código 2017/176).
De forma previa a la obtención de la muestra de la nasofaringe se recogió información sobre los siguientes factores de riesgo potenciales de la colonización neumocócica: edad, género, asistencia a guardería, utilización de antibióticos en los 2 meses previos y tipo de antibiótico utilizado, estado de tabaquismo pasivo y estado vacunal frente al neumococo. Las muestras de la nasofaringe las obtuvieron profesionales sanitarios previamente adiestrados en el procedimiento según una metodología descrita con anterioridad9. Las muestras se transportaron en un periodo<24 h al departamento de Microbiología del Hospital Universitario de Virgen del Rocío para su procesamiento microbiológico convencional, tras el cual se congelaron a –80°C hasta completar posteriormente los procedimientos de serotipación y genotipación.
Estudios microbiológicos, serotipación y genotipaciónLas muestras nasofaríngeas se sembraron en placas de agar sangre Columbia, suplementadas con 5μg/ml de gentamicina, y las colonias α-hemolíticas se identificaron como neumococos con base en su morfología, susceptibilidad a la optoquina y solubilidad en bilis. Para la serotipación y genotipación se procedió a la extracción del ADN genómico de las aislados neumocócicos y su secuenciación completa en el Instituto de Biomedicina de Sevilla. La determinación del serotipo capsular se realizó por deducción bioinformática a partir de las secuencias genómicas del locus capsular10. El grupo de SV incluyó todos los serotipos cubiertos por la formulación de la VNC13 (1, 3, 4, 5, 6A, 6B, 7F, 9V, 14, 18C, 19A, 19F, 23F). Los serotipos restantes se consideraron SNV. La genotipación se realizó por tipificación de secuencias multilocus según metodología estándar11. Se definió un complejo clonal (CC) como un secuenciotipo (ST) que compartía al menos 5 de las 7 variantes alélicas.
La sensibilidad antimicrobiana a penicilina, ampicilina, cefotaxima y eritromicina se determinó por E-test con utilización de los puntos de corte recomendados por EUCAST en 2022 para la asignación de las categorías interpretativas (https://www.eucast.org/clinical_breakpoints). Dentro de la categoría de no sensibles se combinaron las cepas sensibles a altas dosis y resistentes.
Controles históricos VNC7Para comparar la distribución de serotipos y genotipos y las tasas de resistencias antibióticas se utilizó una colección histórica de 154 aislados neumocócicos del periodo temporal VNC7 entre el 1/01/2006 y el 30/06/2008, que se habían comunicado de forma parcial (periodo 1/01/2006 al 30/06/2006) en la literatura8. Estas cepas colonizantes fueron obtenidas en un estudio de portación nasofaríngea desarrollado en 4 centros de salud y 2 urgencias hospitalarias de Sevilla, según la metodología descrita7. En este periodo se evaluó la sensibilidad antimicrobiana a la amoxicilina oral, y no a la ampicilina, como en el periodo VNC13.
Análisis estadísticoEl procesamiento estadístico se realizó utilizando el paquete SSPS 26.0. Las variables categóricas se compararon mediante la χ2 y el test exacto de Fisher, según fuera apropiado. Las variables con un valor p<0,10 en el análisis univariante de predictoras del estado de portador neumocócico se introdujeron en un modelo de regresión logística binaria. Se consideró en una prueba significativa cuando el valor de p fue<0,05.
ResultadosCaracterísticas demográficasSe identificaron 76 (19%) cepas colonizantes nasofaríngeas de S. pneumoniae en 397 niños sanos durante el periodo VNC13 y se dispuso de una colección histórica de 154 (35%) aislados nasofaríngeos neumocócicos obtenidos de 443 participantes (89, 20% de niños sanos y 354, 80% de niños con infecciones leves respiratorias de vías altas) durante el periodo VNC7.
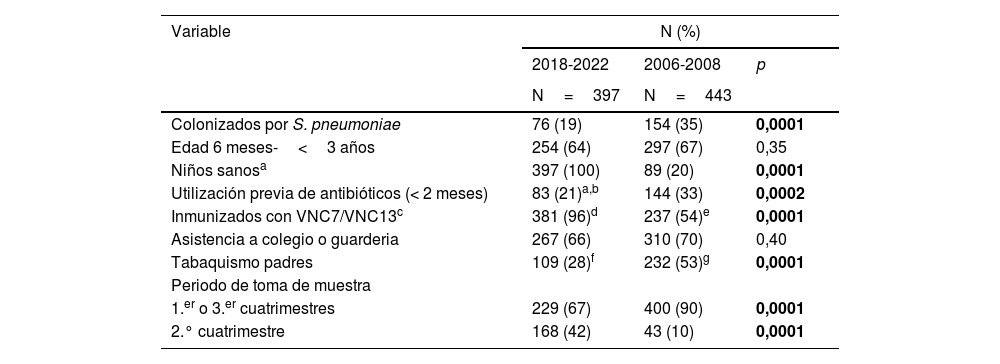
El periodo VNC13 comparado con el periodo VNC7 se asoció con menores tasas de colonización (19 vs. 35%; p=0,0001), de utilización de antibióticos en los 2 meses previos (21 vs. 33%; p=0,0002) y de hábito tabáquico en alguno de los padres (28 vs. 53%; p=0,0001) y con una mayor proporción de niños sanos (100 vs. 20%; p=0,0001) e inmunizados con VNC (96 vs. 54%; p=0,0001) (tabla 1). Por otra parte, no hubo diferencias entre ambos periodos en las proporciones de niños de menor edad (6 meses-3 años) y con asistencia a guardería o colegio. Por último, el reclutamiento de participantes fue significativamente mayor en el segundo cuatrimestre en el estudio contemporáneo que en el histórico (42 vs. 10%; p=0,0001).
Análisis comparativo de las variables demográficas en los estudios de colonización nasofaríngea de los periodos VNC13 y VNC7
| Variable | N (%) | ||
|---|---|---|---|
| 2018-2022 | 2006-2008 | p | |
| N=397 | N=443 | ||
| Colonizados por S. pneumoniae | 76 (19) | 154 (35) | 0,0001 |
| Edad 6 meses-<3 años | 254 (64) | 297 (67) | 0,35 |
| Niños sanosa | 397 (100) | 89 (20) | 0,0001 |
| Utilización previa de antibióticos (< 2 meses) | 83 (21)a,b | 144 (33) | 0,0002 |
| Inmunizados con VNC7/VNC13c | 381 (96)d | 237 (54)e | 0,0001 |
| Asistencia a colegio o guarderia | 267 (66) | 310 (70) | 0,40 |
| Tabaquismo padres | 109 (28)f | 232 (53)g | 0,0001 |
| Periodo de toma de muestra | |||
| 1.er o 3.er cuatrimestres | 229 (67) | 400 (90) | 0,0001 |
| 2.° cuatrimestre | 168 (42) | 43 (10) | 0,0001 |
En negrillas, valores p estadísticamente significativos.
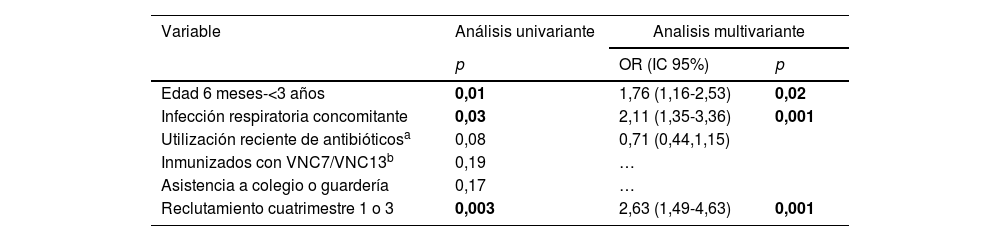
En el análisis univariante, la colonización neumocócica en el global de los 2periodos de estudio se asoció significativamente con edad<3 años, presencia concomitante de infección respiratoria de vías altas (niños enfermos) y reclutamiento en primer o tercer cuatrimestre (tabla 2). Estas variables continuaron asociadas de forma positiva con un mayor riesgo de colonización en el análisis multivariante.
Análisis univariante y multivariante de las variables demográficas asociadas con un riesgo incrementado de colonización neumocócica
| Variable | Análisis univariante | Analisis multivariante | |
|---|---|---|---|
| p | OR (IC 95%) | p | |
| Edad 6 meses-<3 años | 0,01 | 1,76 (1,16-2,53) | 0,02 |
| Infección respiratoria concomitante | 0,03 | 2,11 (1,35-3,36) | 0,001 |
| Utilización reciente de antibióticosa | 0,08 | 0,71 (0,44,1,15) | |
| Inmunizados con VNC7/VNC13b | 0,19 | … | |
| Asistencia a colegio o guardería | 0,17 | … | |
| Reclutamiento cuatrimestre 1 o 3 | 0,003 | 2,63 (1,49-4,63) | 0,001 |
En negrillas, valores p estadísticamente significativos.
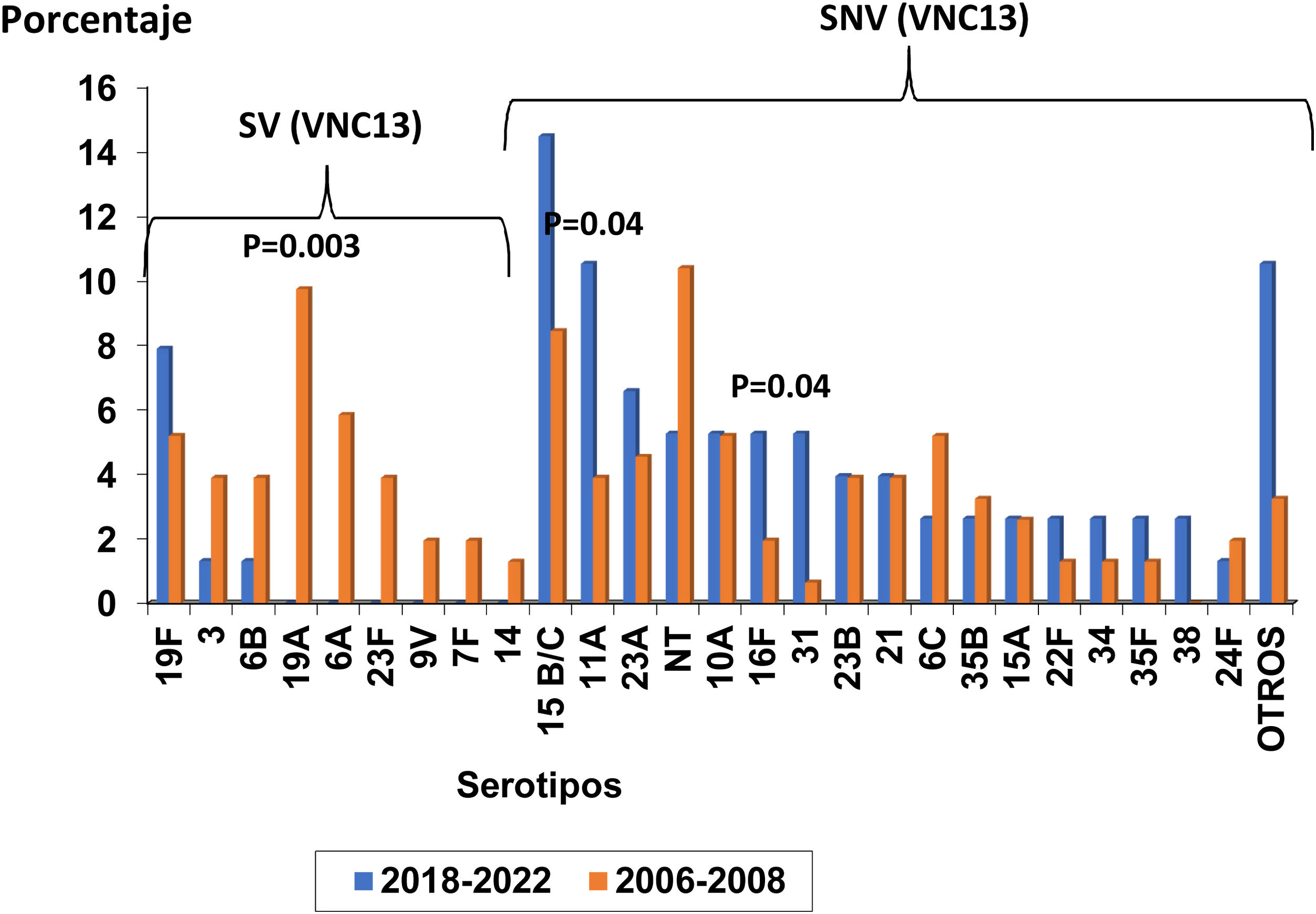
Se identificaron 27 y 32 serotipos individuales diferentes en el total de las 76 y 154 cepas de los periodo de estudios contemporáneo e histórico, respectivamente. La colonización por SV, incluidos en la VNC13, disminuyó de forma significativa entre los periodos VNC13 y VNC7 (n=8, 11% frente a n=58, 38%; p=0,0001).
Durante el periodo del estudio más reciente se identificaron solo los SV 19F (n=6, 8%), 3 (n=1, 1%) y 6B (n=1, 1%) (fig. 1). Estos habían circulado en el periodo VNC7, sin que se observaran diferencias significativas entre ambos periodos. Los restantes SV presentes en el periódico histórico y erradicados en el periodo contemporáneo incluyeron los serotipos: 19A (n=15, 10%), 6A (n=9, 6%), 23F (n=4, 4%), 9V (n=3, 2%), 7F (n=2, 1%), 14 (n=2, 1%), 5 (n=1, 1%) y 18C (n=1, 1%). La proporción de aislados del serotipo 19A disminuyó significativamente entre los 2periodos de tiempo (p=0,003).
Por otra parte, se identificaron en el periodo VNC 13 los siguientes SNV con prevalencia ≥ 3%: 15B/C (n=11, 14%), 11A (n=8, 11%), 23A (n=5, 7%), 10A (n=4, 5%), 16F (n=4, 5%), 31 (n=4, 5%), no tipable (NT) (n=4, 5%), 23B (n=3, 4%), 21 (n=3, 4%), 6C (n=2, 3%), 15A (n=2, 3%), 22F (n=2, 3%), 34 (n=2, 3%), 35B (n=2, 3%), 35F (n=2, 3%) y 38 (n=2, 3%). Otros SNV presentes durante el este periodo temporal se describen en la figura 1. Al comparar los periodos VNC13 y VNC7, se observó un incremento significativo en las proporciones de los serotipos 11A y 31 (p=0,04 para ambos serotipos) y no hubo cambios significativos en los restantes SNV.
Distribución de genotiposSe identificaron por tipificación de secuencias multilocus un total de 40 ST y 67 ST diferentes, de un total de 76 y 152 aislamientos genotipados de los periodos VNC13 y VNC7, respectivamente. Los complejos clonales más comunes en el periodo VNC13 fue CC156 (n=9, 12%), seguido de CC42, CC51 y CC97 (n=4, 5%, todos), mientras que, por otra parte, ST1262 (n=8, 11), ST433, ST1760 y ST2670 (n=3, 4%, todos) fueron los secuenciotipos de mayores frecuencias en este mismo periodo (tabla 3). El CC156 se asoció predominantemente en el periodo VNC13 con el secuenciotipo ST6521, una variante genética del clon global Spain9V-ST156 expresada como serotipo 11A. Este serotipo se asoció exclusivamente con el clon ST62 en el periodo histórico y fue reemplazado por ST6521 en los años más recientes. ST1262 se expresó como serotipo 15B/C y, a diferencia de ST6521, permaneció estable durante ambos periodos de estudio. Otros genotipos identificados en los periodos histórico y contemporáneo se muestran en la tabla 2 y en la tabla del material adicional Appendix B.
Complejos clonales y secuenciotipos con ≥ 3 aislados en los periodos de estudio VNC13 o VNC7 y serotipos asociados
| CC/ST | Serotipo | 2018-2022 | 2006-2008 |
|---|---|---|---|
| N.° aislados (%) | N.° aislados (%) | ||
| N=76 | N=151 | ||
| CC156 | 9 (12) | 5 (3) | |
| ST156 | 14 | 0 | 1 (1) |
| 35B | 1 (1) | 0 | |
| ST162 | 9V | 0 | 2 (1) |
| ST838 | 9V | 0 | 1 (1) |
| ST2944 | 14 | 0 | 1 (1) |
| ST6521 | 11A | 7 (9) | 0 |
| ST14698 | 11A | 1 (1) | 0 |
| CC42 | 4 (5) | 12 (8) | |
| ST42 | 23A | 1 (1) | 6 (4) |
| ST311 | 13F | 1 (1) | 0 |
| ST439 | 23B | 2 (3) | 5 (3) |
| 23F | 0 | 1 (1) | |
| CC51 | 5 (7) | 2 (1) | |
| ST15 | 19F | 1 (1) | 0 |
| ST51 | 19F | 2 (3) | 0 |
| ST179 | 19F | 2 (3) | 2 (1) |
| CC97 | 4 (5) | 8 (5) | |
| ST97 | 10A | 1 (1) | 8 (5) |
| ST1551 | 10A | 3 (4) | 0 |
| CC199 | 1 (1) | 9 (6) | |
| ST199 | 15B/C | 1 (1) | 5 (3) |
| 19A | 0 | 3 (2) | |
| ST2109 | 19A | 0 | 1 (1) |
| CC224 | 1 (1) | 9 (6) | |
| ST224 | 6C | 0 | 1 (1) |
| ST338 | 6A | 0 | 3 (2) |
| 23F | 0 | 2 (1) | |
| ST1150 | 6C | 1 (1) | 2 (1) |
| 6A | 0 | 1 (1) | |
| CC1143 | 0 | 3 (2) | |
| ST1143 | 6A | 0 | 2 (1) |
| ST1876 | 6A | 0 | 1 (1) |
| ST1262 | 15B/C | 8 (11) | 4 (3) |
| ST432 | 21 | 3 (4) | 5 (3) |
| ST1766 | 31 | 3 (4) | 0 |
| ST2670 | 23A | 3 (4) | 0 |
| 23F | 0 | 1 (1) | |
| ST30 | 16F | 2 (3) | 3 (2) |
| ST433 | 22F | 2 (3) | 2 (1) |
| 19A | 0 | 2 (1) | |
| ST1078 | 15B/C | 2 (3) | 0 |
| 15A | 2 (3) | 0 | |
| ST2001 | 34 | 2 (3) | 2 (1) |
| ST2615 | 35F | 2 (3) | 0 |
| 19F | 0 | 1 (1) | |
| ST180 | 3 | 1 (1) | 4 (3) |
| ST392 | 19A | 0 | 1 (1) |
| 15A | 0 | 2 (1) | |
| ST230 | 17F | 1 (1) | 2 (1) |
| ST344 | 24F | 1 (1) | 2 (1) |
| ST62 | NT | 0 | 8 (5) |
| ST558 | 11A | 0 | 5 (3) |
| ST276 | 35B | 0 | 5 (3) |
| ST386 | 19A | 0 | 4 (3) |
| 6B | 0 | 3 (2) | |
| 6C | 0 | 1 (1) |
En negrillas CC y ST identificados únicamente durante el periodo 2018-2022.
CC: complejos clonales; ST: secuenciotipos.
De las 76 cepas del periodo VNC13, 20 (16%) mostraron sensibilidad disminuida a la penicilina, de las que 2 aislados (3%) se clasificaron como resistentes a la penicilina. Las tasas de sensibilidad disminuida y resistencia a los otros 2antibióticos β-lactámicos evaluados en ambos periodos fueron las siguientes: ampicilina (17% y 16%) y cefotaxima (7% y 0%); 15 cepas (20%) presentaron también resistencia a la eritromicina. Las tasas de resistencia antimicrobianas del periodo VNC7 no mostraron diferencias estadísticamente significativas comparadas con las del periodo VNC13 para la penicilina (con sensibilidad disminuida a la penicilina 32 vs. 26%, p=0,34 y resistentes a la penicilina 0 vs. 3%; p=0,11) y la cefotaxima (sensibilidad disminuida 4 vs. 7%; p=0,37 y resistencia 0 vs. 0%), pero fueron, por el contrario, significativamente más elevadas para la eritromicina (38 vs. 20%; p=0,006).
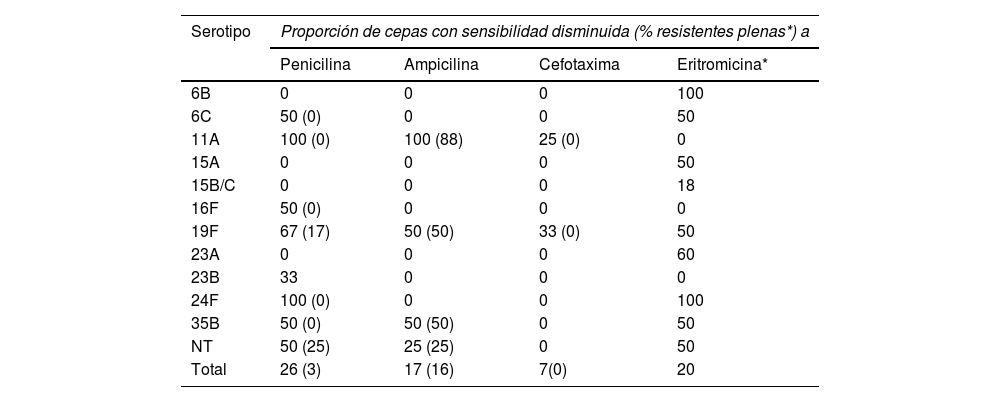
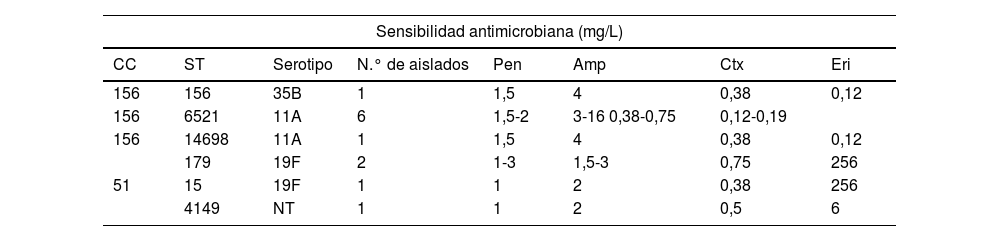
En la tabla 4 se muestra la distribución en el periodo VNC13 de los serotipos con sensibilidad disminuida a los antibióticos β-lactámicos y con resistencia a la eritromicina. Los aislamientos con resistencias a la ampicilina en estos años correspondieron de forma mayoritaria a las cepas del serotipo 11A (clones ST6521 y genéticamente relacionada ST14698) (8/12, 67%), mientras que, por el contrario, los 5 aislados del genotipo ST62 asociado al este serotipo en el periodo VNC7 mostraron sensibilidad plena a la amoxicilina oral (CMI 0,06 mg/L). Los restantes serotipos y genotipos con resistencias a la ampicilina en el periodo VNC13 se describen en la tabla 5.
Serotipos asociados a sensibilidad disminuida a los antibióticos β-lactámicos y eritromicina en los 76 aislados NF del periodo VNC13
| Serotipo | Proporción de cepas con sensibilidad disminuida (% resistentes plenas*) a | |||
|---|---|---|---|---|
| Penicilina | Ampicilina | Cefotaxima | Eritromicina* | |
| 6B | 0 | 0 | 0 | 100 |
| 6C | 50 (0) | 0 | 0 | 50 |
| 11A | 100 (0) | 100 (88) | 25 (0) | 0 |
| 15A | 0 | 0 | 0 | 50 |
| 15B/C | 0 | 0 | 0 | 18 |
| 16F | 50 (0) | 0 | 0 | 0 |
| 19F | 67 (17) | 50 (50) | 33 (0) | 50 |
| 23A | 0 | 0 | 0 | 60 |
| 23B | 33 | 0 | 0 | 0 |
| 24F | 100 (0) | 0 | 0 | 100 |
| 35B | 50 (0) | 50 (50) | 0 | 50 |
| NT | 50 (25) | 25 (25) | 0 | 50 |
| Total | 26 (3) | 17 (16) | 7(0) | 20 |
Serotipos y genotipos asociados con resistencias a ampicilina (CMI>1μg/ml) durante el periodo 2018-2002
| Sensibilidad antimicrobiana (mg/L) | |||||||
|---|---|---|---|---|---|---|---|
| CC | ST | Serotipo | N.° de aislados | Pen | Amp | Ctx | Eri |
| 156 | 156 | 35B | 1 | 1,5 | 4 | 0,38 | 0,12 |
| 156 | 6521 | 11A | 6 | 1,5-2 | 3-16 0,38-0,75 | 0,12-0,19 | |
| 156 | 14698 | 11A | 1 | 1,5 | 4 | 0,38 | 0,12 |
| 179 | 19F | 2 | 1-3 | 1,5-3 | 0,75 | 256 | |
| 51 | 15 | 19F | 1 | 1 | 2 | 0,38 | 256 |
| 4149 | NT | 1 | 1 | 2 | 0,5 | 6 | |
Los genotipos y serotipos en negrilla o bien no se detectaron (ST6521, ST14698, ST15 y ST4149) o bien no estuvieron asociados con resistencia a amoxicilina (CC51, ST179) durante el periodo VNC7.
Amp: ampicilina; CC: complejo clonal; Ctx: cefotaxima; ERI: eritromicina; NT: no tipable; Pen:penicilina; ST: secuenciotipo.
El presente estudio, realizado tras la introducción de la vacunación sistemática con VNC13 en el calendario de inmunización infantil de Andalucía, constituye la primera comunicación nacional sobre la epidemiología molecular de la colonización nasofaríngea neumocócica en niños sanos con coberturas plenas de vacunación con VNC13. Se han publicado previamente diversos estudios de portación neumocócica en niños de diversas comunidades autónomas de España, pero estos se desarrollaron en ámbitos con vacunación privada y coberturas parciales y solo se evaluó la distribución de genotipos en 2estudios del periodo VNC78,9,12-15.
Las VNC ejercen un impacto profundo en la población neumocócica de la nasofaringe. Disminuyen de forma muy importante la circulación de los SV, aunque sin llegar a su completa eliminación. Esta reducción se ve compensada por un incremento en la prevalencia de los SNV. Como consecuencia, se han descrito, tras la introducción de las VNC, tasas de colonización neumocócica similares16,17 o inferiores18,19 a las de la época prevacunal, en función de que el grado de reemplazamiento de los SV por los SNV fuera total o parcial, respectivamente. Nosotros observamos en el periodo VNC13 tasas de colonización inferiores a las registradas durante el periodo VNC7 (19% vs. 35%). Esta diferencia podría relacionarse con la distribución no balanceada entre ambas poblaciones de estudio de algunas de las características demográficas, como infección respiratoria de vías altas concomitante y reclutamiento en el primer o en el tercer cuatrimestres (meses menos cálidos), que fueron predictoras independientes por análisis multivariante de la colonización neumocócica.
Como era previsible, la colonización por SV disminuyó de forma muy marcada, en un 79%, entre los períodos VNC13 y VNC7. Se erradicaron 7 de los 10 SV circulantes durante el periodo histórico, incluyendo al serotipo 19A identificado en el 10% de los aislados de este último periodo, mientras que por el contrario se observó únicamente una persistencia significativa (8%) del serotipo 19F. Por otra parte, los serotipos 3 y 6B, cuyas proporciones disminuyeron entre ambos periodos de tiempo, aunque sin alcanzar la significación estadística, se detectaron de forma muy infrecuente (1%) en el periodo VNC13. Este hallazgo no es insospechado, porque el serotipo 19F se incluye entre los SV detectados con mayor frecuencia en los estudios de colonización realizados tras la introducción de VNC1312,16,20. El serotipo 19F fue genéticamente diverso y asociado con multirresistencias antibióticas. Este serotipo tiene gran capacidad de formar biofilm y, por tanto, alta capacidad de evadir el sistema inmune, lo que junto a las resistencias antibióticas son factores que pueden haber contribuido a su circulación persistente21,22.
Hubo una distribución amplia de SNV en el período VNC13, con identificación de 24 serotipos diferentes. Los SNV dominantes incluyeron los serotipos 15B/C, 11A, 23A, 10A, 16F, 31, NT, 23B y 21, que son SNV habituales en los estudios de colonización del periodo VNC13, aunque con algunas diferencias en el ranking de prevalencia entre los diversos ámbitos geográficos12,16,20,23,24. Estos serotipos se han asociado con bajo potencial invasivo (OR<1 en la comparación de las prevalencias en ENI y portadores) en el periodo VNC13, excepto el serotipo 10A, que presentó OR en rango de 1,2-1,7 en algunos estudios17,19,25. La VNC 20 valente, aprobada para prevención de neumonía y ENI en pacientes adultos y de probable próxima comercialización en población pediátrica, incluye en su formulación este último serotipo y los serotipos 11A y 15B, de elevada prevalencia en el presente estudio.
Las proporciones de los serotipos 11A y 31 aumentaron significativamente entre los periodos VNC13 y VNC7 por la expansión de clones (ST6521 y ST1766, repectivamente) no detectados en el estudio del periodo VNC7. ST6521 surgió por recombinación entre los clones 11A-ST62 y NT-ST344 y el clon global Spain9V-ST156, asociado con los serotipos 9V y 1426. La expansión clonal de ST6521 es preocupante por sus características fenotípicas de resistencia a ampicilina/amoxicilina, mayor capacidad para producir biofilm y elevada capacidad de evadir la activación de la vía clásica del complemento y la fagocitosis en comparación con ST838, que también procede de Spain9V-ST15627. En un contexto de presión antibiótica e inmunidad vacunal, estas propiedades pueden haber contribuido a su relevancia creciente en la etiología de la otitis media aguda en niños y de las exacerbaciones agudas del EPOC en adultos en ciertas localizaciones geográficas de España28,29. Además, este serotipo se puede asociar con ENI en niños y adultos por su elevada prevalencia en portadores y por su marcada letalidad: es incluso más letal que el serotipo 330,31.
Las tasas de sensibilidad disminuida a penicilina y cefotaxima permanecieron estables y se observó una disminución significativa de las tasas de resistencia a eritromicina entre los periodos VNC13 y VNC7. Aunque se erradicaron varios SV (6A, 9V, 19A y 23F) asociados en grado variable con sensibilidad disminuida a los antibióticos β-lactámicos en el periodo VNC7, este efecto beneficioso se vio compensado por la aparición y expansión clonal de ST11A-ST6511. Este es un patrón de evolución de las resistencias antimicrobianas descrito tras la introducción de las VNC, de forma que la disminución de las tasas de resistencia derivada de la erradicación de los SV tiende a amortiguarse con el tiempo por la adquisición progresiva de determinantes de resistencia en los SNV32,33. Debe señalarse, además, que la pandemia por SARS-CoV-2ha provocado un aumento en el porcentaje de cepas resistentes de neumococo. Esto se aprecia de forma clara en el serotipo 11A, que ha pasado de tener en España una CMI90 a penicilina de 2 ug/ml en el periodo prepandémico (2016-2019) a una CMI90 a penicilina de 4 ug/ml en 2020, que es un valor considerado como resistente34.
Esta investigación tiene varias limitaciones. En primer lugar, como ya se ha mencionado, las diferentes características demográficas de las poblaciones de estudio de los periodos VNC7 y VNC13 son un factor de confusión para el análisis comparativo de las tasas de colonización. En segundo lugar, la distribución de algunos serotipos puede diferir entre los niños sanos y aquellos con infección respiratoria superior35. Por último, no se evaluaron las colonizaciones múltiples por técnicas moleculares, ni tampoco se realizó preenriquecimiento en el cultivo, lo que, de haberse llevado a cabo, podría haber incrementado la capacidad de detección de los neumococos en la colonización nasofaríngea.
En conclusión, en los años posteriores a la introducción de VNC13 en el calendario de inmunización pediátrica de Andalucía, se observó una circulación muy residual de los SV en colonizantes de la nasofaringe, excepto para el serotipo 19F, y la expansión clonal entre los SNV de ST11A-ST6511, con capacidad patogénica y asociada a sensibilidad disminuida a la penicilina y resistencia a ampicilina. Sería recomendable mantener la vigilancia epidemiológica de la colonización neumocócica para monitorizar la evolución de los SNV y las tasas de resistencias antibióticas.
FinanciaciónEste trabajo se ha financiado con una ayuda al proyecto de investigación por parte de Pfizer a IO (grant 5323485). No existió ningún tipo de participación del promotor en el diseño del estudio, la recolección, el análisis y la interpretación de los datos, la redacción del artículo ni en la decisión de enviar el artículo para su publicación.
Conflictos de interesesIO recibió ayudas de Pfizer para inscripción a congresos y para la realización de este proyecto. ISC ha recibido becas u honorarios como consultor/asesor y ponente, así como ayudas para la asistencia a congresos y cursos prácticos de Pfizer, GlaxoSmithKline, Sanofi Pasteur, MSD y AstraZeneca. CC ha recibido ayudas para asistencia a congresos de Pfizer, GlaxoSmithKline y MSD. No hay conflictos de intereses en los otros autores.
Los autores desean expresar sus agradecimientos a Ignacio Cruz Navarro, Irene Bullón Durán y Esther Díaz Carrión por su colaboración en la recogida de muestras de los participantes en el estudio.