Hoy conocemos algo más de la participación de la región HLA-DP en la patogénesis de las leucemias gracias a varios estudios demostrando la injerencia temprana de estas moléculas en la modulación de la respuesta inmunitaria frente a patógenos. Para la evaluación de alelos y haplotipos HLA-DPA1*/DPB1* (28 alelos del locus HLA-DPA1* y 123 alelos HLA-DPB1*) se utilizó PCR SSPTM Olerup (Genovision) en 48 pacientes con LLA, 48 con LMC y 48 controles mestizos venezolanos. Para la asociación HLA/leucemias se tomó el riesgo relativo (RR)>3 como positivo y negativo RR < 3, con una p corregida<0,05. Pacientes LLA confirmaron asociaciones positivas con el alelo DPA1*0105 y negativa con el alelo DPA1*010301-010302. Además, resultaron asociados positivamente DPA1*0106 y *0107; DPA1*020101-020106 se asoció negativamente con la LLA. DPA1*0105, *0108 y *0109 resultaron asociados negativamente con la LMC. Las frecuencias observadas de los alelos HLA-DPB1*01:01, 02:01, 03:01, 04:01 y 04:02 en los controles mestizos venezolanos ubicadas entre 7 y 16% fueron superiores a las de pacientes leucémicos. Se confinaron asociaciones negativas de los alelos DPB1*02:01 y DPB1*03:01 con las LLA. No se observaron asociaciones positivas con las LLA. DPB1*99:01 se asoció negativamente con las LMC. Los haplotipos HLA-DPA1*01:03-DPB1*04:01, *02:01, *99:01 resultaron fuertemente asociados positivamente a las LMC. DPA1*01:09-DPB1*02:01, *04:01 se asociaron negativamente con las LMC. DPA1*01:03-DPB1*04:02, DPA1*01:09-DPB1*02:01, *04:01 y DPA1*02:01-DPB1*04:02 se asociaron negativamente con las LLA. La región DPB1* sola no parece estar asociada a las leucemias en esta población venezolana. La fuerte asociación LMC con varios haplotipos DPA/DPB indica importantes diferencias entre las patogénesis de ambas enfermedades.
More is now known of the involvement of HLA-DP region in the pathogenesis of the leukemias through several previousstudies showing interference of these molecules in modulating the immune response to pathogens. For evaluation of HLA alleles and haplotypes DPA1*/DPB1* (28 alleles HLA DPA1* and 123 of HLA- DPB1*) Olerup SSP ™PCR (Genovision) was used in 48 patients with ALL, 48 CML, and 48 Venezuelan twins as controls. For HLA/leukemias, a relative risk (RR) > 3 was considered to be a positive association and negative with an RR<3, with a p corrected P<.05. ALL patients confirmed positive associations with DPA1*0105 allele, and negative with DPA1*010301-010302. In addition, they were positively associated with DPA1*0106 and *0107, with DPA1*020101-020106 being negatively associated with ALL. DPA1*0105, *0108 and *0109 were negatively associated with CML. The observed frequencies of HLA-DPB1* 01:01, 02:01, 03:01, 04:01 and 4:02 alleles in Venezuelan, which twins were between 7 and 16%, were higher than those of leukemic patients. Negative associations of DPB1*2:01, *3:01 and LLA were confined. No positive associations were observed with ALL. Non-confirmed positive associations were observed between DPB1*99:01 and CML. Haplotypes HLA-DPA1*01:03-DPB1*4:01, *2:01, *99:01 were strongly positively associated with CML. DPA1*1:09-DPB1*2:01, *4:01 were negatively associated with the CML. DPA1*1:03-DPB1*4:02; DPA1*01:09-DPB1*2:01, *4:01 and DPA1*02:01-DPB1*04:02 were negatively associated with ALL. The DPB1* single region does not appear to be associated with leukemia in the Venezuelan population. The strong association with several haplotypes DPA*1/DPB1* and LMC suggests massive differences between the pathogenesis of both diseases.
La leucemia linfoide aguda (LLA) es el cáncer más frecuente en niños y adolescentes en la población zuliana. La leucemia mieolide crónica (LMC) es similar en adolescentes, adultos jóvenes y adultos maduros. Los estudios epidemiológicos indican la presencia de varios factores relacionados con la susceptibilidad al padecimientos de ambos tipos de leucemia1,2.
La sustitución individual de los residuos DPβ 9, 11, 35, 55, 56 y 69 eliminan completamente el reconocimiento mediado por 2 tipos de clones de células T HLA-DPw2 aloespecíficas. El análisis detallado del papel del polimorfismo de la cadena HLA-DPB1* en el reconocimiento peptídico por parte de la célula T y la selectividad del péptido podrían ayudar a entender el papel de los isotipos de esta región HLA en la respuesta inmunitaria, además de los mecanismos patogénicos de varios desórdenes inmunes asociados como genes de susceptibilidad al HLA-DP3.
El genotipo DPA1*01-DPB1*02:02:02 fue el único genotipo asociado significativamente con diabetes mellitus insulinodependiente en una población venezolana4. En estudios familiares, el alelo DPB1*03:01 haplotipo DPA1*01:03-DPB1*03:01 se encontró asociado con susceptibilidad, mientras que el DPB1*, haplotipo DPA1*01:03-DPB1*04:02 y el haplotipo DPA1*01:03-DPB1*01:01 resultaron de protección en la diabetes mellitus insulinodependiente5,6. Los genotipos DPA1*02:02-DPB1*05:01 y DPA1*02:02:02-DPB1*03:01, respectivamente, se asociaron fuertemente con el riesgo de infección persistente con el virus de la hepatitis B en asiáticos7.
Es poco lo que se conoce acerca de la relevancia del polimorfismo de la región HLA-DP en la respuesta inmunitaria, sin embargo, se han descrito algunas asociaciones con la leucemia8. Utilizando a 254 pacientes con diferentes tipos de leucemias, se demostró la influencia de la región DP en la susceptibilidad o resistencia a la LLA, mostrando una asociación positiva con el DPw2 y DPw5, ninguna con la LMC, mientras que el HLA-DPw1 solo se observó asociado positivamente a pacientes con leucemia aguda no linfoidea. Otros estudios revelan que la susceptibilidad a leucemia linfoblástica aguda común (LLA-c) en niños estaba asociada con un alelo DPB1*02:01 en el locus HLA-DPB1, apuntando esta susceptibilidad a mecanismos que involucran la presentación de péptidos antigénicos específicos, derivados probablemente de agentes infecciosos, por proteínas alotípicas, relacionadas con DPB1*02:01, que conducen a la activación de células T colaboradoras mediadoras de la proliferación de células preleucémicas9. Se especula que la molécula HLA-DPB1*06:01 puede estar funcionalmente involucrada en leucemias infantiles10. Ciertas hipótesis se han tejido en torno a la posibilidad de participación de estas moléculas del complejo mayor de histocompatibilidad en la patogenia de las leucemias11. Recientemente, el gen HLA-DRB1*15 se señaló como factor de riesgo en niños con LLA12. En investigaciones previas reportamos que el genotipo HLA-DPA1*01:03:01-01:03:02/02:01:01-02:02:06 presentó frecuencias significativamente diferentes (p ≥ 0,05) en los controles (40,4%) comparado con los pacientes LLA (8,5%) y los pacientes LMC (64,6%), entre otras notables variaciones13. Todos estos antecedentes apuntan a que la región DP, conformada por los alelos HLA-DPB1* y HLA-DPA1*, ambas cadenas con diferentes polimorfismos, aparecería involucrada en la patogenia de las leucemias de una manera diferente.
La evaluación completa de los alelos HLA-DPA1/DPB1* de controles sanos mestizos venezolanos de 3 generaciones al igual que en pacientes con LLA y LMC de la misma población, permitirá ubicar ciertos marcadores genéticos de riesgo o de protección además de servir de base para comprender un poco más la complicada patogénesis de estas leucemias.
Materiales y métodosPoblación estudiadaFueron estudiados 28 alelos del locus HLA-DPA1* y 123 alelos HLA-DPB1* usando PCR SSP™ Olerup (Genovision), de acuerdo a las instrucciones del fabricante, en una población representativa de 48 pacientes con LLA, 48 con LMC y 48 controles del Banco de Sangre del Estado Zulia (Venezuela), todos mestizos racialmente no relacionados, con su debido consentimiento.
Análisis estadísticoLas frecuencias alélicas y genotípicas se determinaron empleando el paquete de análisis genético Genepop 1714. El equilibrio de Hardy Weinberg para cada locus se probó con el paquete Arlequín, versión 3,0 el cual emplea una prueba exacta análoga a la prueba Fisher, teniendo en cuenta modificaciones al algoritmo de la cadena de Markov, descrita por Guo y Thompson en 199215. Las frecuencias haplotípicas fueron estimadas igualmente en Arlequín versión 3,0.
Las pruebas de asociación se realizaron utilizando el programa SAS con el procedimiento Prim Comp. Se utilizó un coeficiente de confianza del 95% y se consideró una p corregida≤0,05 como estadísticamente significativa. La asociación HLA-LLA o LMC se consideró positiva si el riesgo relativo (RR) resultaba≥3 y negativa si RR≤3. Los datos obtenidos de la determinación de alelos se registraron en una base de datos computarizada utilizando el paquete estadístico SPSS statitics 17,0 (San Diego, CA, EE. UU.) y el programa Excel para Windows 2007 para cálculo y presentación de datos en tablas y gráficos.
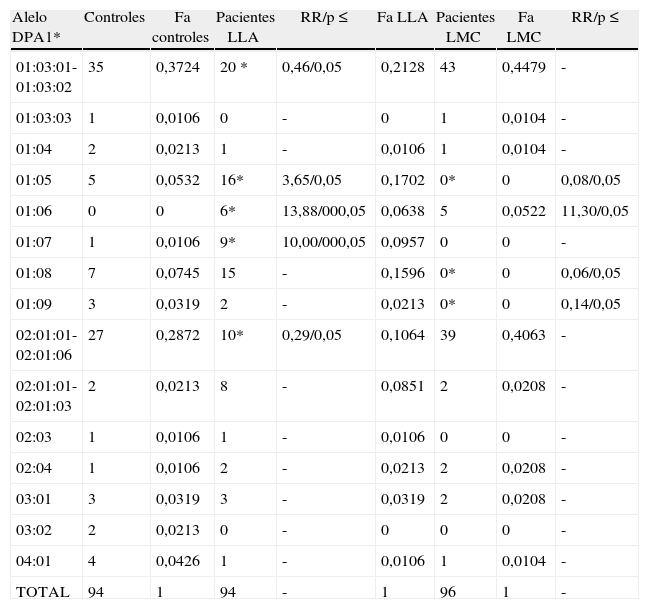
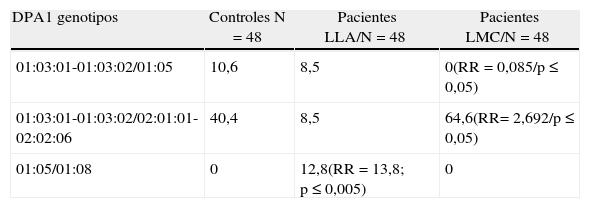
ResultadosMarcador HLA-DPA1*En los 48 pacientes con LLA se confirmaron la asociación positiva con el alelo DPA1*0105 y la negativa con el alelo DPA1*010301-01030216. Adicionalmente, resultaron asociados positivamente los alelos DPA1*0106 y DPA1*0107 a las LLA. El alelo DPA1*020101-020106 se asocia negativamente con la LLA. No hubo asociación positiva entre HLA-DPA1* y la LMC. Los alelos DPA1*0105, DPA1*0108 y DPA1*0109 resultaron asociados negativamente con la LMC (tabla 1). El genotipo HLA-DPA1*010301-010302/020101-020206 cuyas frecuencias significativamente diferentes (p < 0,05) de 40,4% en controles, 8,5% en pacientes LLA y 64,6% en pacientes LMC habían sido reportadas13, se muestran en la tabla 2. Estos patrones disímiles de la distribución de las frecuencias de los alelos y genotipos HLA-DPA1* en pacientes LMC y LLA con respecto a los controles y el escaso polimorfismo de la moléculas DPA1* observado en los pacientes con LMC, se amplían con la evaluación de la región HLA-DPB1* de estos pacientes.
Frecuencias alélicas HLA-DPA1* en contoles y pacientes con LLA Y LMC mestizos venezolanos
| Alelo DPA1* | Controles | Fa controles | Pacientes LLA | RR/p ≤ | Fa LLA | Pacientes LMC | Fa LMC | RR/p ≤ |
| 01:03:01-01:03:02 | 35 | 0,3724 | 20 * | 0,46/0,05 | 0,2128 | 43 | 0,4479 | - |
| 01:03:03 | 1 | 0,0106 | 0 | - | 0 | 1 | 0,0104 | - |
| 01:04 | 2 | 0,0213 | 1 | - | 0,0106 | 1 | 0,0104 | - |
| 01:05 | 5 | 0,0532 | 16* | 3,65/0,05 | 0,1702 | 0* | 0 | 0,08/0,05 |
| 01:06 | 0 | 0 | 6* | 13,88/000,05 | 0,0638 | 5 | 0,0522 | 11,30/0,05 |
| 01:07 | 1 | 0,0106 | 9* | 10,00/000,05 | 0,0957 | 0 | 0 | - |
| 01:08 | 7 | 0,0745 | 15 | - | 0,1596 | 0* | 0 | 0,06/0,05 |
| 01:09 | 3 | 0,0319 | 2 | - | 0,0213 | 0* | 0 | 0,14/0,05 |
| 02:01:01-02:01:06 | 27 | 0,2872 | 10* | 0,29/0,05 | 0,1064 | 39 | 0,4063 | - |
| 02:01:01-02:01:03 | 2 | 0,0213 | 8 | - | 0,0851 | 2 | 0,0208 | - |
| 02:03 | 1 | 0,0106 | 1 | - | 0,0106 | 0 | 0 | - |
| 02:04 | 1 | 0,0106 | 2 | - | 0,0213 | 2 | 0,0208 | - |
| 03:01 | 3 | 0,0319 | 3 | - | 0,0319 | 2 | 0,0208 | - |
| 03:02 | 2 | 0,0213 | 0 | - | 0 | 0 | 0 | - |
| 04:01 | 4 | 0,0426 | 1 | - | 0,0106 | 1 | 0,0104 | - |
| TOTAL | 94 | 1 | 94 | - | 1 | 96 | 1 | - |
Fa: frecuencias alélicas; LLA: leucemia linfoide aguda; LMC: leucemia mieloide crónica; RR: riesgo relativo; X2: chi cuadrado corregido.
*alelos asociados positiva o negativamente.
Distribución de genotipos HLA-DPA1* en controles y pacientes con leucemia linfoide aguda y leucemia mieloide crónica mestizos venezolanos
| DPA1 genotipos | Controles N=48 | Pacientes LLA/N = 48 | Pacientes LMC/N = 48 |
| 01:03:01-01:03:02/01:05 | 10,6 | 8,5 | 0(RR=0,085/p≤0,05) |
| 01:03:01-01:03:02/02:01:01-02:02:06 | 40,4 | 8,5 | 64,6(RR= 2,692/p ≤ 0,05) |
| 01:05/01:08 | 0 | 12,8(RR=13,8; p ≤ 0,005) | 0 |
Datos en porcentaje (%).
LLA: leucemia linfoide aguda; LMC: leucemia mieloide crónica.
Los alelos HLA-DPB1*01:01, 02:01, 03:01, 04:01 y 04:02 son los más frecuentes en los controles sanos de la población mestiza venezolana y oscilan entre el 7 y el 16%. Estos mismos alelos muestran porcentajes inferiores en pacientes con LLA y LMC de esta población.
Se localizaron asociaciones negativas de los alelos HLA-DPB1*02:01 y DPB1*03:01 con las LLA. No se observaron asociaciones positivas de este locus y las LLA (tabla 3). Se observó una asociación positiva entre el alelo HLA-DPB1*99:01 y las LMC, no confirmada luego de la corrección de Yates (tabla 4).
Comparación de las frecuencias alélicas HLA-DPB1* en pacientes con leucemia linfoide aguda y controles mestizos venezolanos
| DPB1* | Controles n=48 | Fa % | LLA n=47 | Fa % | RR | X2 | p ≤ |
| 02:01 | 12 | 12,52 | 3 | 3,19 | 0,2 | 6,19 | 0,01 |
| 03:01 | 6 | 6,26 | 1 | 1,06 | 0,15 | 5,41 | 0,0025 |
Fa: frecuencias alélicas; LLA: leucemia linfoide aguda; RR: riesgo relativo, X2: chi cuadrado corregido.
HLA-DPB1* 99:1 asociado con pacientes con leucemia mieloide crónica en mestizos venezolanos
| DPB1* | Controles n=48 | Fa % | LMC n=48 | Fa % | RR | X2 | p ≤ | X2 Yate | p ≤ |
| 99:01 | 0 | 0 | 5 | 5,21 | 11,5 | 4,30 | 0,0381 | 2,75 | 0,0971 |
Fa: frecuencia alélica; LMC: leucemia mieloide crónica; RR: riesgo relativo; X2: chi cuadrado corregido.
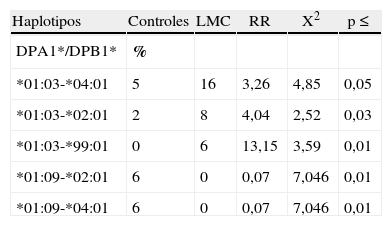
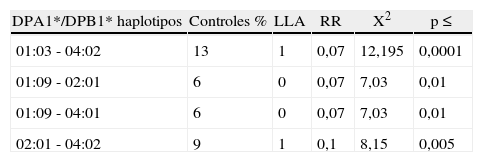
Los haplotipos DPA1*01:03-DPB1*01:01, DPA1*01:03-DPB1*04:02, DPA1*01:03-DPB1*13:01 y DPA1*02:01-DPB1*01:01 se encuentran en desequilibrio de enlace en la población mestiza venezolana. Los haplotipos HLA-DPA1*01:03-DPB1*04:01, *02:01 y *99:01 están fuertemente asociados positivamente a las LMC. Los haplotipos HLA-DPA1*01:09-DPB1*02:01, *04:01 resultaron asociados negativamente con las LMC (tabla 5). Los haplotipos HLA-DPA1*01:03-DPB1*04:02, HLA-DPA1*01:09-DPB1*02:01 y *04:01, y HLA-DPA1*02:01- DPB1*04-02 se asociaron negativamente con las LLA (tabla 6).
Comparación de haplotipos DPA1*/DPB1* en pacientes con leucemia mieloide crónica y controles mestizos venezolanos
| Haplotipos | Controles | LMC | RR | X2 | p ≤ |
| DPA1*/DPB1* | % | ||||
| *01:03-*04:01 | 5 | 16 | 3,26 | 4,85 | 0,05 |
| *01:03-*02:01 | 2 | 8 | 4,04 | 2,52 | 0,03 |
| *01:03-*99:01 | 0 | 6 | 13,15 | 3,59 | 0,01 |
| *01:09-*02:01 | 6 | 0 | 0,07 | 7,046 | 0,01 |
| *01:09-*04:01 | 6 | 0 | 0,07 | 7,046 | 0,01 |
LMC: leucemia mieloide crónica; RR: riesgo relativo; X2: chi cuadrado corregido.
Haplotipos HLA-DPA1*/DPB1* asociados negativamente con leucemia linfoide aguda
| DPA1*/DPB1* haplotipos | Controles % | LLA | RR | X2 | p≤ |
| 01:03 - 04:02 | 13 | 1 | 0,07 | 12,195 | 0,0001 |
| 01:09 - 02:01 | 6 | 0 | 0,07 | 7,03 | 0,01 |
| 01:09 - 04:01 | 6 | 0 | 0,07 | 7,03 | 0,01 |
| 02:01 - 04:02 | 9 | 1 | 0,1 | 8,15 | 0,005 |
LLA: leucemia linfoide aguda; RR: riesgo relativo; X2: chi cuadrado corregido.
Una amplia investigación indica que la modulación inmune apropiada, principalmente a través de la exposición a las infecciones tempranas en el infante, puede tener un impacto significativo en las respuestas posteriores de la vida a los estímulos inmunológicos. El reconocimiento del complejo de DP1-péptido por células T en el contexto de un sistema inmune insuficientemente modulado puede desencadenar una respuesta inmunitaria desfavorable que contribuya al desarrollo de la leucemia. Trabajos anteriores realizados en EE. UU., han demostrado que más del 90% de los alelos DPB1 pueden ser agrupados en 6 supertipos basados en similitudes en sus propiedades funcionales (péptido de unión). El supertipo más común, DP4, estuvo presente en la gran mayoría de los niños blancos no hispanos (79,2%) e hispanos (82,5%) del grupo control. El supertipo DP1, que incluye alelos DPB1 *01:01, *05:01 y *50:01, mostró un OR elevado (1,83) y un aumento del riesgo estadísticamente significativo asociado con la LLA (sub tipo high-hyperdiploid p=0,005). Un fuerte desequilibrio de enlace fue observado en poblaciones infantiles hispanas y no hispanas en EE. UU. entre 2 loci HLA-DPA1*/DPB1* con frecuencias>5% tanto en controles como en pacientes con leucemia, entre ellos se encontraban los DPA1*01:03-DPB1*02:01, DPA1*01:03-DPB1*03:01, DPA1*01:03-DPB1*04:01 y DPA1*01:03-DPB1*04:0216. Solo este último haplotipo DPA1*01:03-DPB1*04:02 coincide con los desequilibrios de enlaces observados en la población mestiza venezolana donde, además, se observaron en desequilibrio los haplotipos DPA1*01:03-DPB1*01:01, DPA1*01:03-DPB1*13:01 y DPA1*02:01-DPB1*01:01.
La variación en las frecuencias de los alelos DP entre poblaciones es una indicación de la diversidad potencial en la estructura genética subyacente y de que las poblaciones pueden haber evolucionado bajo diferentes presiones selectivas. La participación de la molécula HLA como una unidad funcional en la susceptibilidad a la enfermedad es, por lo tanto, dependiente de la presencia del péptido antigénico inmunodominante. En consecuencia, la heterogeneidad de la prevalencia de ciertos agentes infecciosos u otras exposiciones pertinentes de la región geográfica puede contribuir potencialmente a la variabilidad observada. La variación genética HLA-DP combinada con un sistema inmunológico insuficientemente modulado puede desencadenar una respuesta inmune adversa que contribuye al riesgo infantil de sufrir LLA o LMC17.
En esta serie de pacientes LLA y LMC mestizos venezolanos observamos que solo el polimorfismo de la cadena DPA del alelo DP se encuentra asociado fuertemente a ambos tipos de leucemias. Destaca el alelo DPA1*01:06 con frecuencias superiores o iguales al 5% solamente en pacientes LLA o LMC, ausente en todos los controles. Llama igualmente la atención la presencia de varios haplotipos HLA-DPA/DPB asociados de manera importante, positivamente solo a las LMC como el DPA1-DPB1*01:03-*04:01, y sin ninguna asociación positiva de estos con las LLA, observándose especialmente, en esta leucemia, el haplotipo DPA1-DPB1*01:03-*04:02 como protector.
ConclusionesDada la importante participación de las moléculas HLA en la patogénesis de las leucemias descrita por varios autores, especialmente en el abordaje temprano de las infecciones infantiles, se pudiera inferir que existen mecanismos patogénicos diferentes en el origen de ambos tipos de leucemia, LLA o LMC. La dificultad de la presentación de diversos péptidos antigénicos, debido a la ausencia de moléculas presentadoras HLA clase II, de la región DR o DP, codificadas por alelos protectores, pudiera degenerar rápidamente en una LLA de mayor prevalencia en niños. Por el contrario, la generación de células Th1 autorreactivas, con especificidad de secuencias peptídicas de patógenos mimetizadas por moléculas HLA DR y DP, en edades tempranas, pudiera dar lugar a largo plazo a una leucemia en etapas tardías como la LMC.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que los procedimientos seguidos se conformaron a las normas éticas del comité de experimentación humana responsable y de acuerdo con la Asociación Médica Mundial y la Declaración de Helsinki.
Confidencialidad de los datosLos autores declaran que han seguido los protocolos de su centro de trabajo sobre la publicación de datos de pacientes y que todos los pacientes incluidos en el estudio han recibido información suficiente y han dado su consentimiento informado por escrito para participar en dicho estudio.
Derecho a la privacidad y consentimiento informadoLos autores han obtenido el consentimiento informado de los pacientes o sujetos referidos en el artículo. Este documento obra en poder del autor de correspondencia.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
FinanciaciónEstudio financiado por CONDES LUZ Proyecto No.CC-0776-07.