Se evaluó la frecuencia de los polimorfismos Asp-299Gly y Thr-399Ile en el gen de TLR4 en sujetos infectados por VHC y coinfectados por VIH y VHC y se estudió la expresión y función de TLR4 en función de esos polimorfismos.
Material y métodosEl estudio incluyó 53 pacientes con infección por VHC, de los que 28 eran coinfectados además por VIH, más 30 sujetos controles sanos. Los polimorfismos Asp-299Gly y Thr-399Ile se determinaron mediante PCR-RFLP, las poblaciones celulares y la expresión de TLR4 se estudiaron mediante citometría de flujo, mientras que las citocinas proinflamatorias se midieron en suero mediante CBA.
ResultadosSe encontró un descenso significativo de células T CD4+ en los pacientes coinfectados por VHC y VIH. No se demostró una mayor susceptibilidad a sufrir la infección para ninguno de los 2 polimorfismos analizados. TLR4 estaba disminuido en células B, mientras que aumentaba en células T y monocitos. Se comprobó un aumento significativo de las citocinas proinflamatorias que fue el doble en coinfectados que en los portadores de la infección aislada por VHC. Estos cambios no mostraron relación con el polimorfismo estudiado.
DiscusiónLa expresión de TLR4 en células T y monocitos se encuentra aumentada en la infección por VIH y se acompaña de un aumento en suero de citocinas proinflamatorias. Estos hallazgos no guardan relación con los polimorfismos Asp-299Gly y Thr-399Ile de TLR4.
The presence of the Asp-299Gly and Thr-399Ile polymorphisms in the toll-like receptor 4 (TLR4) gene was studied in subjects with HCV infection and HCV+HIV coinfection. The expression and function of TLR4 as regards these polymorphisms is assessed.
Material and methodsThe study included 53 patients infected with HCV, among whom 27 had coinfection HCV+HIV, and 30 healthy subjects. The polymorphisms were studied by PCR-RFLP. The number of lymphocyte subsets, as well as TLR4 expression, was determined by flow cytometry, and the concentration of cytokines was measured in serum by (cytometric bead assay) CBA.
ResultsCD4+ T cells were significantly decreased in patients coinfected with HCV+HIV. There was no association between the presence of any of the two studied polymorphisms and the susceptibility to suffer from infection. TLR4 was less expressed in B cells, whereas it was increased in T cells and, in particular, monocytes. A significant increase in the levels of circulating pro-inflammatory cytokines was found, being two-fold increased in coinfected subjects as compared with patients with isolated HCV infection. None of the findings in cell subsets, TLR4 expression and cytokines was associated with the studied TLR4 polymorphism.
DiscussionThe expression of TLR4 in T cells and monocytes is increased in HCV infection and is accompanied by increased serum levels of proinflammatory cytokines. These findings do not have any relationship with the Asp-299Gly and Thr-399Ile polymorphisms.
Los «Toll-like Receptors» (TLR) juegan un papel esencial, posiblemente el más importante1, en la respuesta inmunitaria innata2 frente a patógenos. No solo inducen la secreción de citocinas proinflamatorias, sino que al tiempo son responsables en gran medida de la conexión entre la respuesta inmune innata y adquirida. El primer TLR identificado fue descrito por Nomura et al. en 19943 y mapeado en un cromosoma posteriormente por Taguchi et al. en 19964. Los TLR son proteínas transmembrana (celular o intracelular), que se caracterizan por un dominio de señalización ubicado en la vertiente citosólica de la membrana citoplasmática o lisosómica y un dominio de reconocimiento de ligando LRR (Leucine-Rich Repeat), ubicado al otro lado de la membrana, formado por repeticiones en tándem de un motivo rico en Leucina de 19 a 25 aminoácidos. El TLR4 reconoce estructuras microbianas conservadas como los lipopolisacaridos (LPS) de las bacterias Gram negativas, además de productos fúngicos y componentes de Mycobacterium tuberculosis5,6. Estudios recientes sugieren que también pueden ser activados por estructuras virales como epítopos de la proteína del core del virus de la hepatitis C (VHC)7. Finalmente, puede reconocer otros ligandos de carácter endógeno, que no son objeto de este estudio.
El virus de la inmunodeficiencia humana (VIH) pertenece a la familia de los retrovirus humanos y a la subfamilia de los lentivirus. Es el agente causante del síndrome de inmunodeficiencia adquirida (sida)8. Este virus infecta principalmente a los linfocitos T que expresan el correceptor CD4, molécula que interacciona con una fuerte afinidad con la glicoproteína gp120 de la cápside viral9. La unión al receptor, junto a la ayuda de correceptores, como los receptores para quimiocinas CCR3 y CCR5 facilitan la entrada del virus en la célula T-CD4+ y potencialmente la muerte de esta, contribuyendo al estado de profunda inmunodeficiencia que induce el virus. Se postulan diversos mecanismos como inductores del efecto citopático del VIH sobre la célula T CD4+10.
Recientemente, se ha descrito la presencia de niveles incrementados de LPS en el plasma de individuos portadores del VIH sin que estos muestren signos de sepsis11. La presencia de este incremento de LPS puede ser consecuencia de una «traslocación microbiana» de productos bacterianos a través del tracto gastrointestinal, puesto que es bien conocido que el VIH induce cambios marcados en la mucosa gastrointestinal12. Independientemente de la fuente, estos niveles incrementados de LPS podrían conducir, a través del TLR4, a una activación del sistema inmune en sujetos con infección crónica de VIH, y de esta forma hacer que la enfermedad progrese13.
Es posible que una expresión defectuosa de los TLR, secundaria o no a variantes génicas o polimorfismos, contribuya a la disfunción inmunitaria asociada a la infección por el VIH, junto al defecto en células CD4+. En estudios recientes, las sustituciones en el dominio extracelular del TLR4 del aspartato en la posición 299 por glicina (rs4986790) y de la treonina en la posición 399 por isoleucina (rs4986791) interfieren en la respuesta a LPS tanto in vivo como in vitro, debido fundamentalmente a una interrupción de la señalización intracelular14.
El objetivo del presente proyecto ha sido evaluar la frecuencia de ambos polimorfismos en el gen que codifica para TLR4 en sujetos con infección por VHC aislada y compararlo con la frecuencia en sujetos coinfectados por VIH. Se estudió así mismo la correlación de estos polimorfismos tanto con el nivel de expresión de la proteína TLR4 en linfocitos y monocitos como con los niveles séricos de citocinas proinflamatorias.
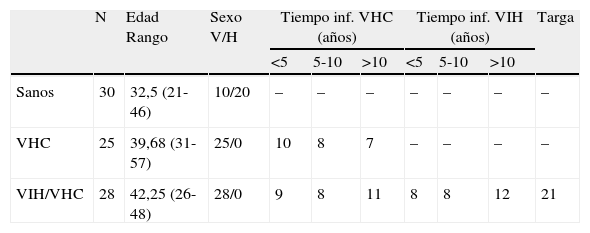
Material y métodosPacientesSe ha llevado a cabo un estudio prospectivo en una población de reclusos del Penal del Dueso (Santoña, Cantabria). Se reclutaron pacientes con infección por VHC (n=25) y coinfección VIH+VHC (n=28). Como control se incluyó una población de sujetos sanos (n=30) de Cantabria. Las características clínico-demográficas de los pacientes y controles se muestran en la tabla 1.
Principales características demográficas y clínicas de los sujetos de estudio
| N | Edad Rango | Sexo V/H | Tiempo inf. VHC (años) | Tiempo inf. VIH (años) | Targa | |||||
| <5 | 5-10 | >10 | <5 | 5-10 | >10 | |||||
| Sanos | 30 | 32,5 (21-46) | 10/20 | – | – | – | – | – | – | – |
| VHC | 25 | 39,68 (31-57) | 25/0 | 10 | 8 | 7 | – | – | – | – |
| VIH/VHC | 28 | 42,25 (26-48) | 28/0 | 9 | 8 | 11 | 8 | 8 | 12 | 21 |
Inf: infección; VHC: virus hepatitis C; VIH: virus inmunodeficiencia humana; TARGA: tratamiento antiretroviral de gran actividad.
En los pacientes se obtuvieron 5ml de sangre venosa mediante venopunción, en EDTA como anticoagulante, y otros 8ml que se dejaron coagular para obtención de suero. En todos los casos se obtuvo previamente consentimiento informado escrito por parte de los participantes incluidos en el estudio. El estudio se llevó a cabo tras la aprobación por parte del Comité de Ensayos Clínicos del Hospital Universitario Marqués de Valdecilla y por Instituciones Penitenciarias.
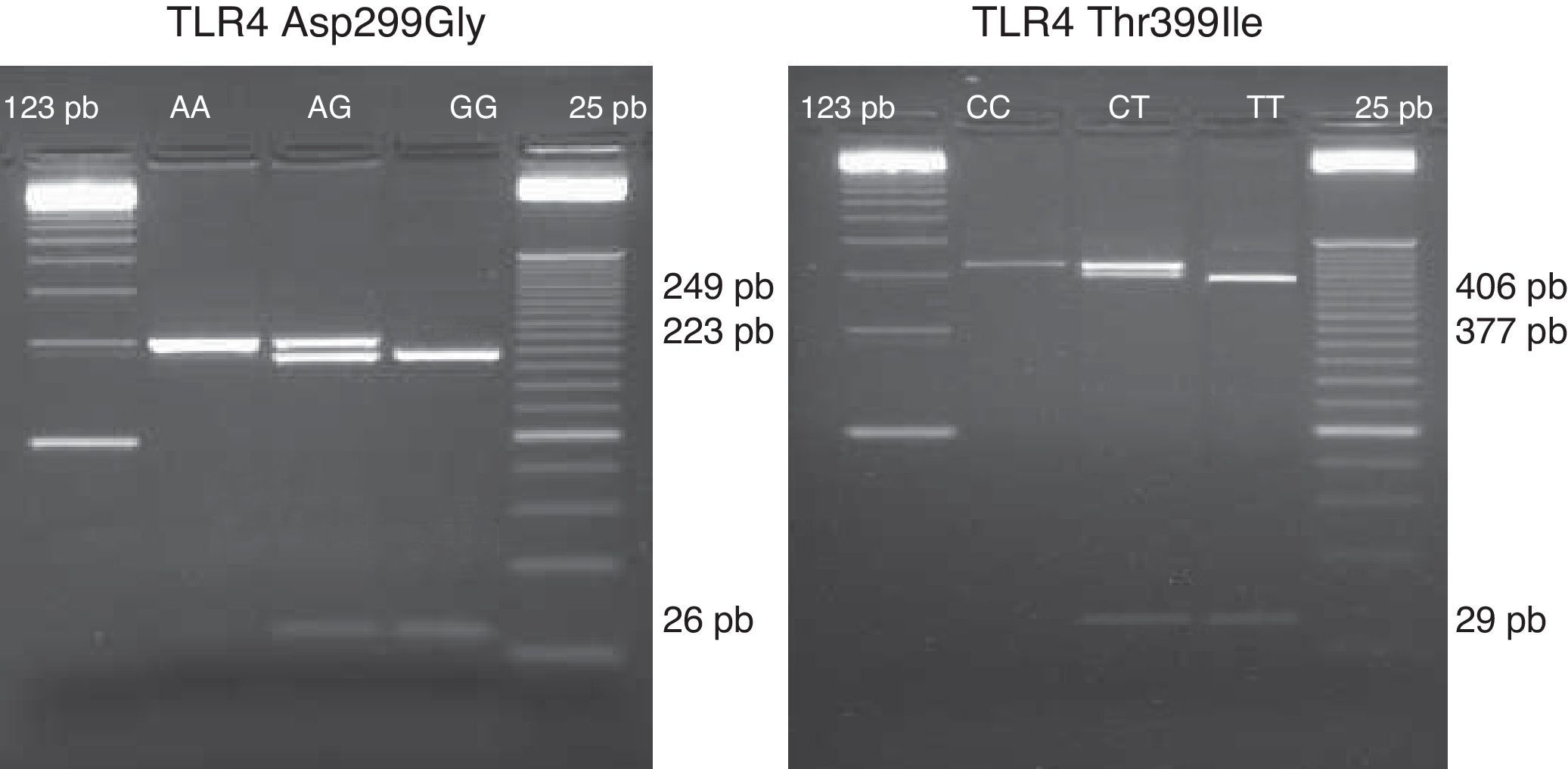
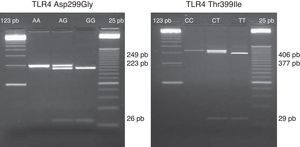
Estudio del polimorfismo de TLR4 Thr-399Ile y Asp-299GlyEl estudio de los polimorfismos de TLR4 se realizó con el DNA extraído a partir de la sangre no coagulada (tubo con EDTA) mediante DNAzol y conservado a -20oC hasta su uso. Los polimorfismos Asp-299Gly (rs4986790) y Thr-399Ile (rs4986791) se estudiaron mediante técnica de PCR-RFLP, descrita previamente15. El DNA genómico fue amplificado usando 1 U de Taq Polimerasa (Promega, Madison, WI) en buffer 10x de PCR (100 mmol/l Tris-HCl, 500 mmol/l KCl y 15 mmol/l MgCl2), 100μM de dNTPs, 2 mM (mmol/l) de MgCl2, y 20pmol de cada primer para Asp-299Gly y Thr-399Ile: sense 5’-GATTAGCATACTTAGACTACTACCTCCATG-3’; antisense 5’-GATCAACTTCTGAAACATTCCCAC-3’ y sense: 5¿-GGTTGCTGTTCTCAAAGTGATTTTGGGAGAA-3¿; antisense: 5¿-ACCTGAAGACTGGAGAGTGAGTTAAATGCT-3¿, respectivamente. Las diferentes fases y condiciones de PCR fueron las siguientes: para Asp-299Gly, a) fase de desnaturalización del DNA a 95°C durante 5 minutos, b) fase de hibridación de 35 ciclos a 95°C durante 30 segundos, 55°C durante 30 segundos y 72°C durante otros 30 segundos, c) fase de elongación final a 72°C durante 5 minutos; para Thr-399Ile, a) fase de desnaturalización del DNA a 95°C durante 5 minutos, b) fase de hibridación de 35 ciclos a 95°C durante 30 segundos, 66°C durante 30 segundos y 72°C durante otros 30 segundos, c) fase de elongación final a 72°C durante 5 minutos. Fueron digeridos 4μl del producto de la PCR usando para ello 2,5μl de NEBuffer 2 con 0,6μl de la enzima de restricción NcoI o HinfI (New England Biolabs), respectivamente, y 17,9μl de agua destilada. El volumen final de 25μl fue digerido durante la noche a 37oC y migrado en gel de agarosa al 3%. Los fragmentos obtenidos para los diferentes alelos del polimorfismo Asp-299Gly fueron: un único fragmento de 249pb para el alelo wild type y 2 fragmentos de 223pb y 26pb para el homocigoto mutante; y para el polimorfismo Thr-399Ile: un único fragmento de 406pb para el alelo wild type, 2 fragmentos de 377pb y 29pb para el homocigoto mutante (fig. 1).
Imagen representativa de los fragmentos obtenidos representativos de los diferentes alelos del polimorfismo Asp-299Gly (rs4986790): un único fragmento de 249pb para el alelo wild type (A/A), 3 fragmentos de 249pb, 223pb y 26pb para el heterocigoto mutante (A/G) y 2 fragmentos de 223pb y 26pb para el homocigoto mutante (G/G). A la derecha se representan un gel de los distintos alelos del polimorfismo Thr-399Ile (rs4986791): un único fragmento de 406pb para el alelo wild type (C/C), 3 fragmentos de 406pb, 377pb y 29pb (calle 2) para el heterocigoto mutante (C/T) y 2 fragmentos de 377pb y 29pb para el homocigoto mutante (T/T).
Las subpoblaciones celulares en sangre y la expresión de TLR4 se determinaron mediante citometría de flujo tras tinción con anticuerpos monoclonales (AcMo) conjugados a fluorocromos, empleando para todo ello una muestra de sangre del tubo con EDTA. Las combinaciones empleadas fueron: CD45 peridin chrorophyll protein (PerCP)/CD3 fluorescein isothyocyanate (FITC)/CD4 phycoerytrin (PE); CD45 PerCP/CD3 FITC/CD8 PE; CD45 PerCP/CD3 FITC/CD19 PE; y CD45 PerCP/CD3 FITC/CD16-56 PE. Brevemente, 100μl de la sangre conservada en EDTA se incubaron a temperatura ambiente con 10μl de cada combinación de estos AcMo. La muestra incubada con los AcMo se adquirió por el citómetro de flujo (FACScalibur) sin lavar, con la solución de FACS lysing (BD Biosciences). Toda la reacción se realizó en la denominada plataforma única, empleando tubos TruCount (BD Biosciences) que contienen unas micropartículas que permiten cuantificar el número de células/mm3 en la muestra, sin necesidad de recurrir al hemograma para realizar los cálculos para obtener el número absoluto de cada tipo de células.
La expresión de TLR4 en linfocitos y monocitos se midió mediante citometría de flujo, tras tinción con anticuerpos monoclonales específicos para CD14-APC (monocitos), CD3-PerCP (células T) y CD19-FITC (células B) junto con un anticuerpo monoclonal anti-TLR4 (eBioscience) conjugado a PE. Los resultados se expresan como porcentaje de células expresando TLR4 o como MFI (mean fluorescence intensity) respecto a la tinción de control de isotipo (tubo de citometría marcado con un anticuerpos de ratón IgG2a conjugado a PE irrelevante).
Cuantificación de citocinas en sueroEl suero se obtuvo mediante centrifugación de la sangre coagulada a 3.000rpm durante 10 minutos, manteniéndose a -80oC hasta su uso. Se cuantificaron los niveles de citocinas proinflamatorias (IL-1β, TNF-α e IL-6), Th1 (IL-12 subunidad p70) y anti-inflamatorias (IL-10) mediante el método BD Cytometric Bead Array (CBA) Human Inflammation kit (BD Biosciences) siguiendo las instrucciones del fabricante. Los límites de detección del ensayo para cada una de las citocinas fueron los siguientes: IL-12p70: 1,9 pg/ml; IL-10: 3,3 pg/ml; IL-1β: 7,2 pg/ml; TNF-α: 3,7 pg/ml; IL-6: 2,5 pg/ml.
Análisis estadísticoTodos los análisis estadísticos de los datos se realizaron con el programa informático SPSS (versión 15.0, Chicago, IL). Para analizar la fortaleza de la asociación entre los alelos y genotipos para los polimorfismos Asp-299Gly y Thr-399Ile, se estimaron los odds ratios (OR) y el 95% de intervalo de confianza (CI). Los niveles de significación se obtuvieron empleando tablas de contingencia mediante análisis de Chi-cuadrado o de Fisher. Para los estudios celulares y de citocinas solubles, los datos se compararon empleando el test de la U de Mann-Whitney, al tratarse de datos no paramétricos. El nivel de significación se indica en cada figura como * (p<0,05), ** (p<0,01) y *** (p<0,001).
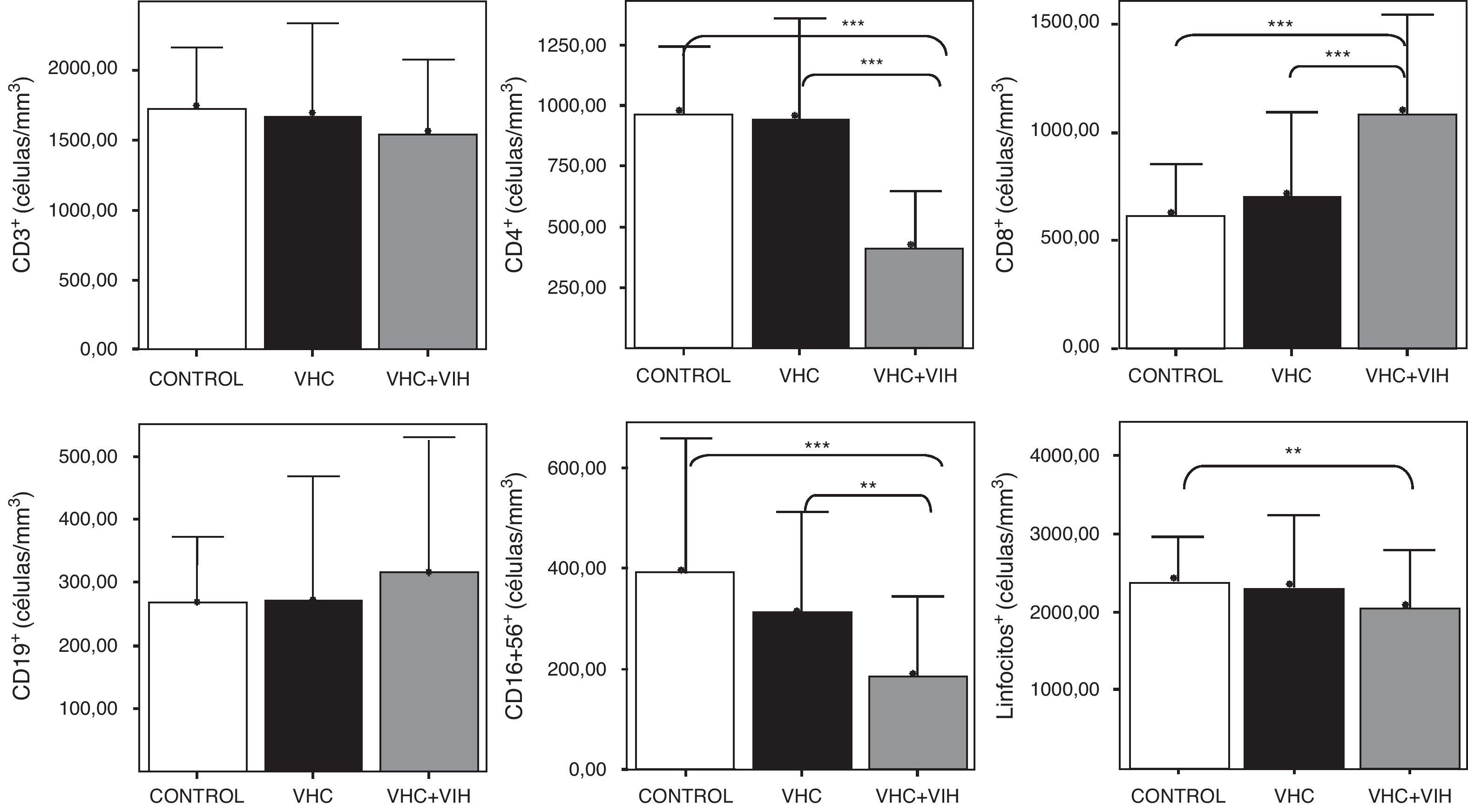
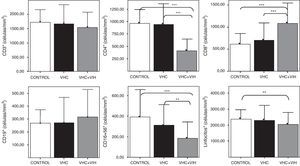
ResultadosNúmeros absolutos de linfocitos sanguíneosLos pacientes con coinfección por el VHC y VIH tuvieron una linfopenia con unos niveles de linfocitos T CD3+ circulantes disminuidos respecto al resto de sujetos infectados y los controles sanos, que tenían unas cifras semejantes (fig. 2). El descenso de los linfocitos T era debido al descenso concreto de los linfocitos T CD4+, aunque se compensaba con un aumento significativo de los linfocitos T CD8+ respecto a los infectados por VHC y los controles sanos (fig. 2). No hubo diferencias entre grupos respecto a las células con fenotipo de células B (CD19+). Las células con fenotipo de células NK (CD3-CD16+CD56+) estaban disminuidas en los pacientes coinfectados por VHC y VIH respecto a los infectados por VHC y a los controles sanos.
Números absolutos en sangre, expresados en células/mm3, de linfocitos y de los distintos subtipos de células CD3+, CD4+, CD8+, CD19+ y CD3-CD16+CD56+. Los histogramas representan la media y las barras la desviación estándar. Se indica la significación estadística solo cuando fue significativa. Se indica la significación estadística de las diferencias entre grupos mediante asteriscos (*: p<0,05; ***: p<0,001).
Los hallazgos obtenidos según porcentajes en sangre fueron prácticamente paralelos a los de los números absolutos, salvo que en la infección VIH (aislada o en coinfección) se observaron porcentajes incrementados de células CD19+ (datos no mostrados).
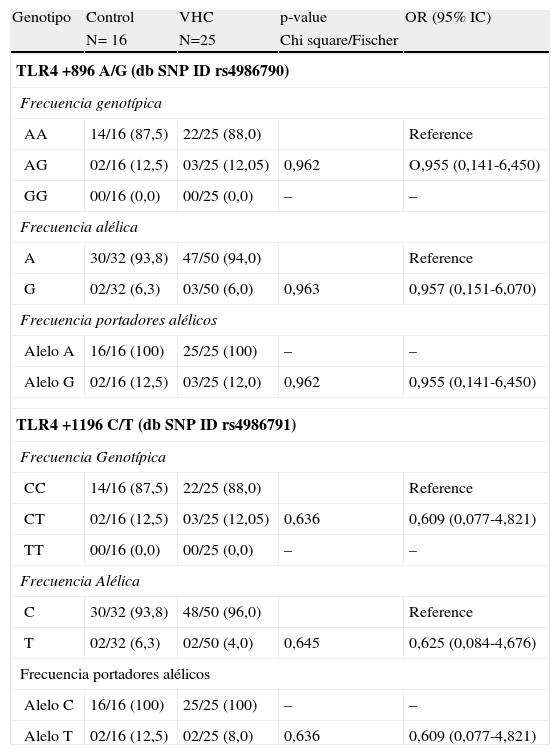
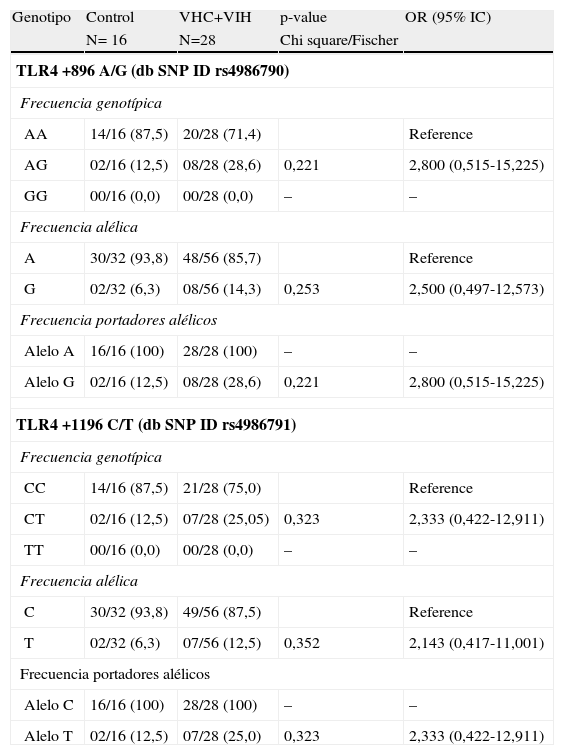
Frecuencia alélica y genotípica de los polimorfismos Asp-299Gly Thr-399Ile del gen TLR4El alelo G del polimorfismo Asp-299Ile y el T del polimorfismo Thr-399Ile estuvieron incrementados en la población coinfectada por el VHC y VIH (tablas 2–4). El aumento de estos polimorfismos, se tradujo en un aumento de los pacientes con los genotipos A/G y C/T para los 2 polimorfismos. Ninguno de los pacientes ni controles de nuestra población de estudio fue homocigoto para las mutaciones G/G ni T/T.
Frecuencias alélicas y genotípicas de los polimorfismos del gen de TLR4 Asp-299Gly Thr-399Ile en pacientes con VHC frente a controles
| Genotipo | Control | VHC | p-value | OR (95% IC) |
| N= 16 | N=25 | Chi square/Fischer | ||
| TLR4 +896 A/G (db SNP ID rs4986790) | ||||
| Frecuencia genotípica | ||||
| AA | 14/16 (87,5) | 22/25 (88,0) | Reference | |
| AG | 02/16 (12,5) | 03/25 (12,05) | 0,962 | O,955 (0,141-6,450) |
| GG | 00/16 (0,0) | 00/25 (0,0) | – | – |
| Frecuencia alélica | ||||
| A | 30/32 (93,8) | 47/50 (94,0) | Reference | |
| G | 02/32 (6,3) | 03/50 (6,0) | 0,963 | 0,957 (0,151-6,070) |
| Frecuencia portadores alélicos | ||||
| Alelo A | 16/16 (100) | 25/25 (100) | – | – |
| Alelo G | 02/16 (12,5) | 03/25 (12,0) | 0,962 | 0,955 (0,141-6,450) |
| TLR4 +1196 C/T (db SNP ID rs4986791) | ||||
| Frecuencia Genotípica | ||||
| CC | 14/16 (87,5) | 22/25 (88,0) | Reference | |
| CT | 02/16 (12,5) | 03/25 (12,05) | 0,636 | 0,609 (0,077-4,821) |
| TT | 00/16 (0,0) | 00/25 (0,0) | – | – |
| Frecuencia Alélica | ||||
| C | 30/32 (93,8) | 48/50 (96,0) | Reference | |
| T | 02/32 (6,3) | 02/50 (4,0) | 0,645 | 0,625 (0,084-4,676) |
| Frecuencia portadores alélicos | ||||
| Alelo C | 16/16 (100) | 25/25 (100) | – | – |
| Alelo T | 02/16 (12,5) | 02/25 (8,0) | 0,636 | 0,609 (0,077-4,821) |
Frecuencias alélicas y genotípicas de los polimorfismos del gen de TLR4 Asp-299Gly Thr-399Ile en pacientes coinfectados por VHC y VIH frente a controles
| Genotipo | Control | VHC+VIH | p-value | OR (95% IC) |
| N= 16 | N=28 | Chi square/Fischer | ||
| TLR4 +896 A/G (db SNP ID rs4986790) | ||||
| Frecuencia genotípica | ||||
| AA | 14/16 (87,5) | 20/28 (71,4) | Reference | |
| AG | 02/16 (12,5) | 08/28 (28,6) | 0,221 | 2,800 (0,515-15,225) |
| GG | 00/16 (0,0) | 00/28 (0,0) | – | – |
| Frecuencia alélica | ||||
| A | 30/32 (93,8) | 48/56 (85,7) | Reference | |
| G | 02/32 (6,3) | 08/56 (14,3) | 0,253 | 2,500 (0,497-12,573) |
| Frecuencia portadores alélicos | ||||
| Alelo A | 16/16 (100) | 28/28 (100) | – | – |
| Alelo G | 02/16 (12,5) | 08/28 (28,6) | 0,221 | 2,800 (0,515-15,225) |
| TLR4 +1196 C/T (db SNP ID rs4986791) | ||||
| Frecuencia genotípica | ||||
| CC | 14/16 (87,5) | 21/28 (75,0) | Reference | |
| CT | 02/16 (12,5) | 07/28 (25,05) | 0,323 | 2,333 (0,422-12,911) |
| TT | 00/16 (0,0) | 00/28 (0,0) | – | – |
| Frecuencia alélica | ||||
| C | 30/32 (93,8) | 49/56 (87,5) | Reference | |
| T | 02/32 (6,3) | 07/56 (12,5) | 0,352 | 2,143 (0,417-11,001) |
| Frecuencia portadores alélicos | ||||
| Alelo C | 16/16 (100) | 28/28 (100) | – | – |
| Alelo T | 02/16 (12,5) | 07/28 (25,0) | 0,323 | 2,333 (0,422-12,911) |
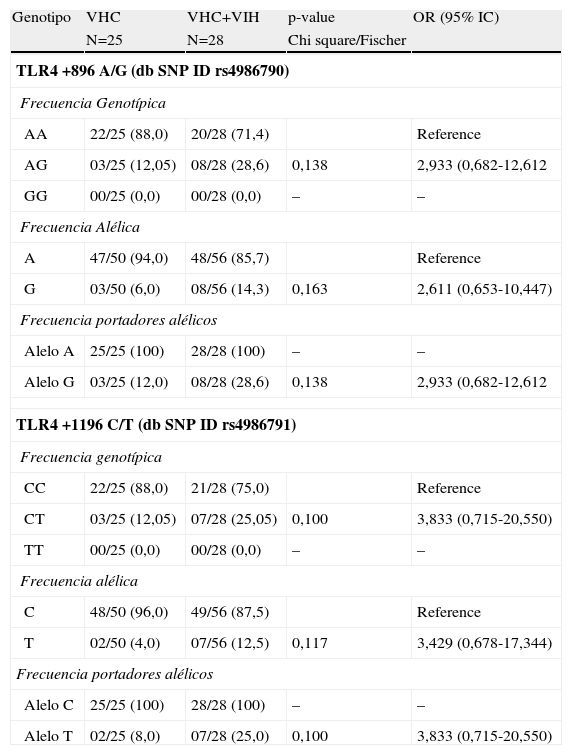
Frecuencias alélicas y genotípicas de los polimorfismos del gen de TLR4 Asp-299Gly Thr-399Ile en pacientes coinfectados por VHC y VIH frente a infectados por VHC
| Genotipo | VHC | VHC+VIH | p-value | OR (95% IC) |
| N=25 | N=28 | Chi square/Fischer | ||
| TLR4 +896 A/G (db SNP ID rs4986790) | ||||
| Frecuencia Genotípica | ||||
| AA | 22/25 (88,0) | 20/28 (71,4) | Reference | |
| AG | 03/25 (12,05) | 08/28 (28,6) | 0,138 | 2,933 (0,682-12,612 |
| GG | 00/25 (0,0) | 00/28 (0,0) | – | – |
| Frecuencia Alélica | ||||
| A | 47/50 (94,0) | 48/56 (85,7) | Reference | |
| G | 03/50 (6,0) | 08/56 (14,3) | 0,163 | 2,611 (0,653-10,447) |
| Frecuencia portadores alélicos | ||||
| Alelo A | 25/25 (100) | 28/28 (100) | – | – |
| Alelo G | 03/25 (12,0) | 08/28 (28,6) | 0,138 | 2,933 (0,682-12,612 |
| TLR4 +1196 C/T (db SNP ID rs4986791) | ||||
| Frecuencia genotípica | ||||
| CC | 22/25 (88,0) | 21/28 (75,0) | Reference | |
| CT | 03/25 (12,05) | 07/28 (25,05) | 0,100 | 3,833 (0,715-20,550) |
| TT | 00/25 (0,0) | 00/28 (0,0) | – | – |
| Frecuencia alélica | ||||
| C | 48/50 (96,0) | 49/56 (87,5) | Reference | |
| T | 02/50 (4,0) | 07/56 (12,5) | 0,117 | 3,429 (0,678-17,344) |
| Frecuencia portadores alélicos | ||||
| Alelo C | 25/25 (100) | 28/28 (100) | – | – |
| Alelo T | 02/25 (8,0) | 07/28 (25,0) | 0,100 | 3,833 (0,715-20,550) |
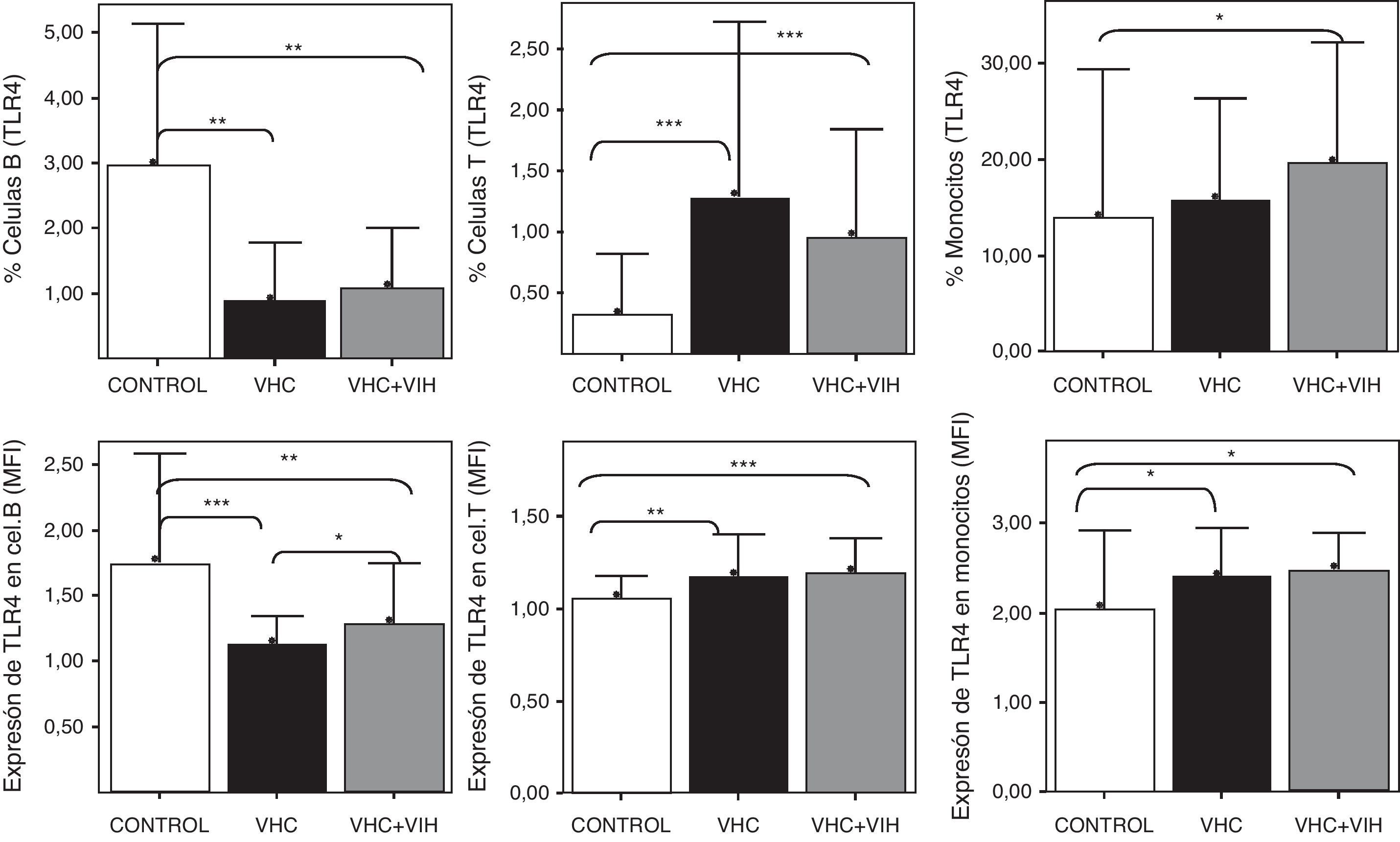
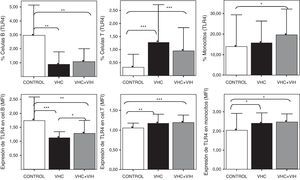
En primer lugar, se analizó el nivel de expresión de TLR4 en conjunto sin atender al polimorfismo genético. Tanto la expresión, cuantificada en MFI de la población TLR4+, como el porcentaje de células B que expresan TLR4 determinado mediante citometría de flujo en células sanguíneas mostró un descenso en las células B CD19+ en pacientes infectados por VHC y coinfectados por VHC+VIH respecto a los controles (fig. 3).
Expresión de TLR4 en células B CD19+, células T (CD3+) y monocitos (CD14+) sanguíneos. Los datos se representan en frecuencias de células que expresan TLR4 (arriba) o en intensidad media de fluorescencia (MFI) para TLR4 en cada población celular. Los datos de MFI corresponden solo a la población TLR4+ y los datos se muestran como el ratio de la tinción obtenida con el anticuerpo anti-TLR4 / tinción obtenida con el anticuerpo de control isotópico. Los histogramas representan la media y las barras la desviación estándar. Se indica la significación estadística solo cuando fue significativa (*: p<0,05; **: p<0,01; ***: p<0,001).
Por el contrario, la expresión aumentó en las células T (CD3+) pero, sobre todo, en los monocitos CD14+ sanguíneos. Este aumento se encontró tanto en frecuencia de células expresando TLR4 como por el nivel de MFI determinado mediante citometría de flujo (fig. 3).
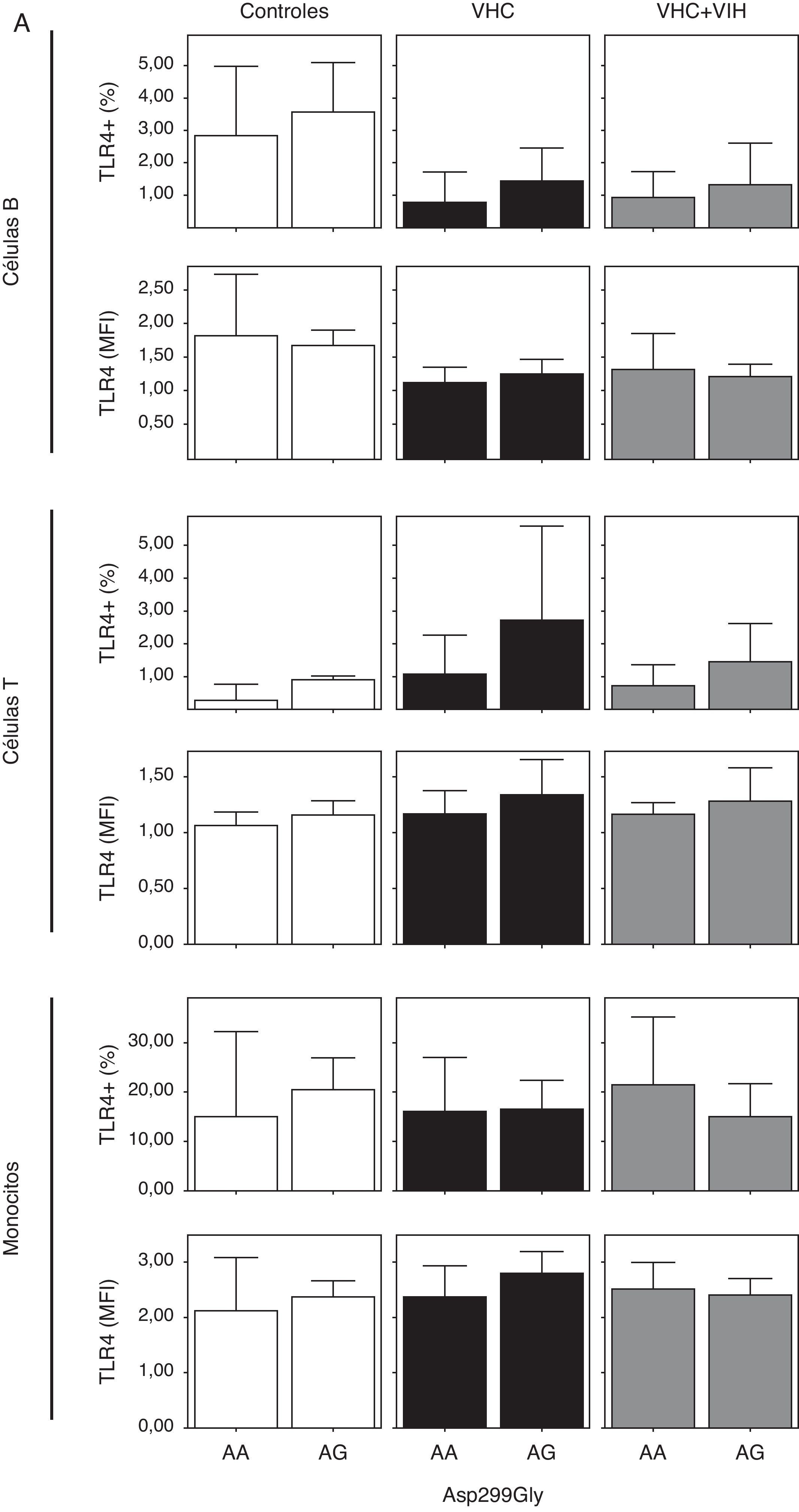
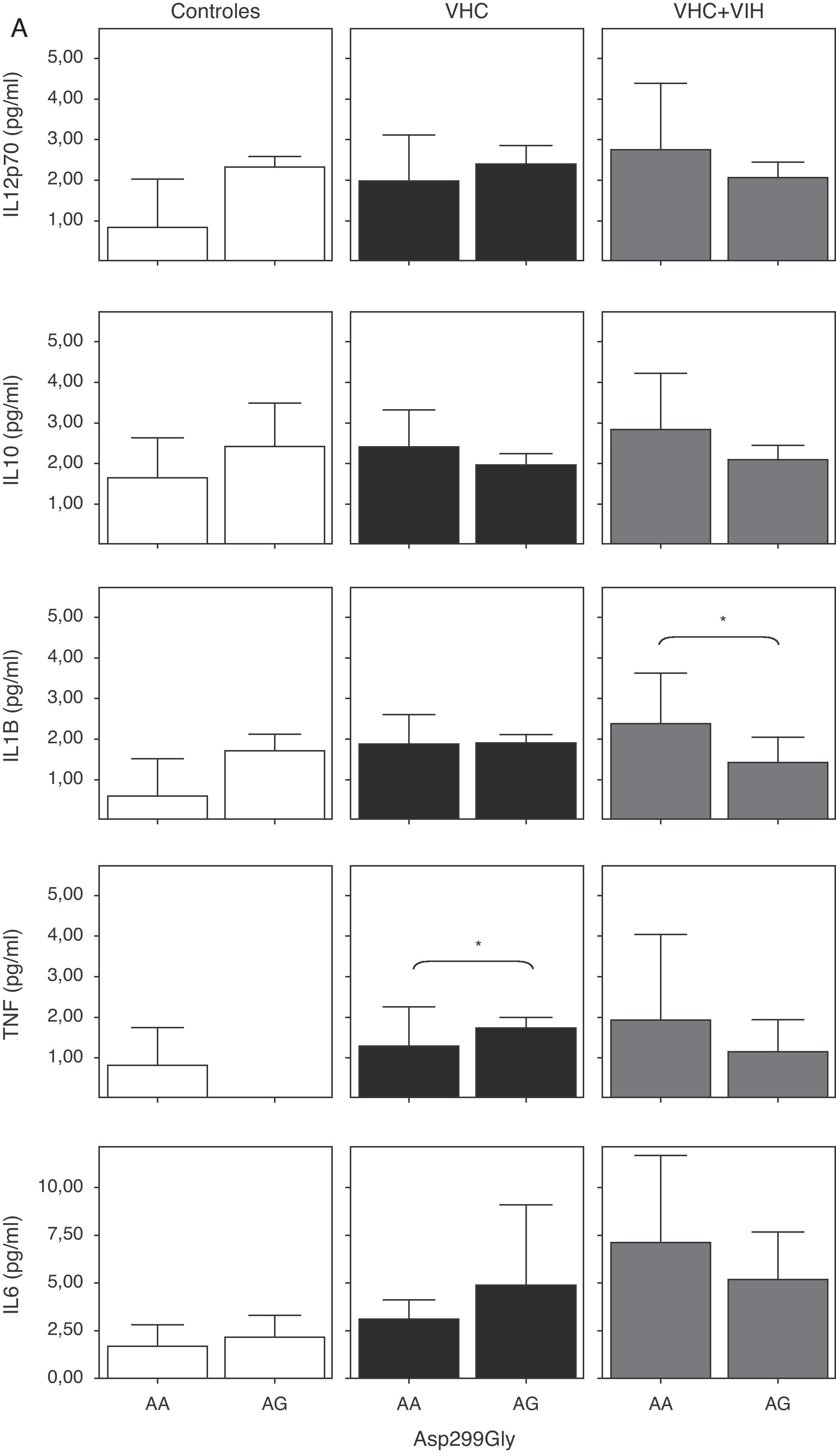
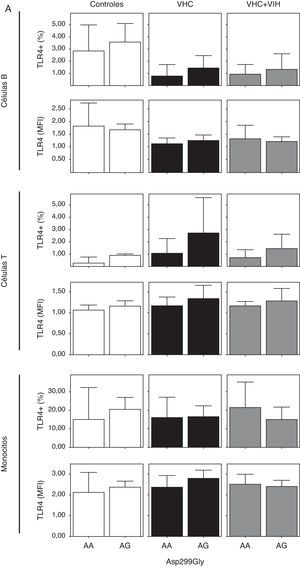
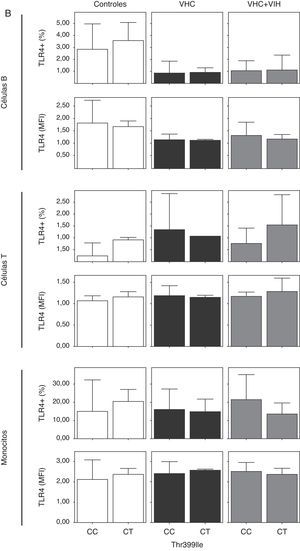
Cuando analizamos la expresión celular según los 2 polimorfismos estudiados, no encontramos diferencias en la expresión, probablemente debido a la baja frecuencia del polimorfismo (fig. 4). Sin embargo, se encontró una tendencia a tener un mayor porcentaje de células T y monocitos que expresan TLR4 en los sujetos con los genotipos A/G y C/T en sujetos controles sanos (fig. 4A y B).
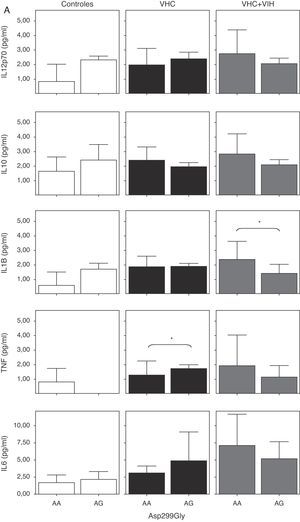
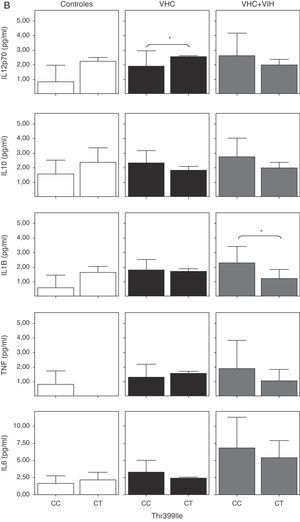
Expresión de TLR4 en células B, células T y monocitos sanguíneos según el genotipo de los polimorfismos Asp-299Gly (A) y Thr-399Ile (B). Los datos, para cada grupo de estudio, se representan en frecuencias de células que expresan TLR4 (arriba) o en intensidad media de fluorescencia (MFI) en cada población celular TLR4+. Los histogramas representan la media y las barras la desviación estándar. No hubo diferencias significativamente estadísticas entre genotipos en ninguno de los grupos de estudio.
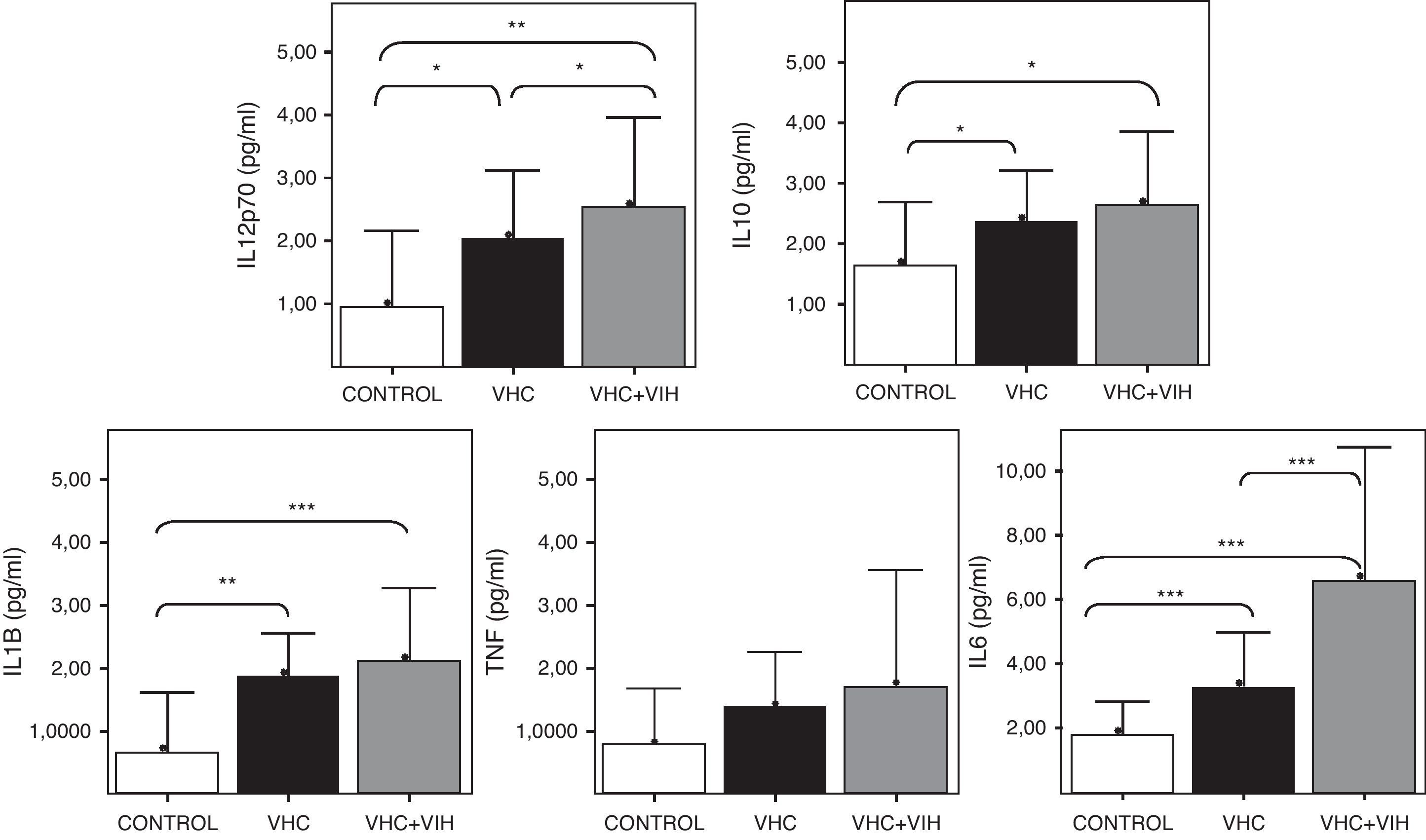
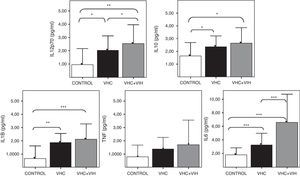
La activación de TLR4 se traduce al final en una activación de las células que lo expresan, como consecuencia de las señales intracelulares que activan NFκB. Esta activación, entre otros efectos, induce la expresión de moléculas de superficie junto con la síntesis y secreción de citocinas proinflamatorias. Por ello, en el presente trabajo se cuantificaron los niveles circulantes de alguna de estas citocinas (fig. 5). En general, se comprobó un aumento significativo de citocinas proinflamatorias (IL-1β, e IL-6) respecto a los individuos sanos. El aumento más claro fue para IL-6, que aumentó significativamente en los coinfectados VHC+VIH. El aumento de IL-6 fue del doble en los coinfectados respectos a los infectados aislados por VHC.
Niveles solubles de IL-12 (subunidad p70), IL-10, IL-1β, TNF-α e IL-6 en los diferentes grupos de estudio. Los histogramas representan la media y las barras la desviación estándar. Se indica la significación estadística solo cuando fue significativa (*: p<0,05; **: p<0,01; ***: p<0,001).
Por otro lado, la figura 5 muestra un aumento de los niveles séricos de la citocina promotora de la respuesta Th1, IL-12, en los pacientes infectados respecto a los sanos, más marcado en los coinfectados por VHC+VIH. Las diferencias fueron significativas en todos los casos. Finalmente, la citocina antiinflamatoria IL-10 estuvo elevada en los pacientes infectados por VHC, tanto de forma aislada como coinfectados, respecto a los controles sanos.
Cuando se analizaron los niveles de citocinas circulantes en función de los polimorfismos de TLR4, se comprobó un descenso de los niveles de citocinas proinflamatorias en los coinfectados VHC+VIH con los genotipos A/G y C/T, aunque solo fue estadísticamente significativa para IL-1β (fig. 6A y B). Además, se comprobó un aumento estadísticamente significativo de TNF-α en los pacientes con el genotipo A/G y de IL-12 en los que tenían el genotipo C/T infectados por VHC.
Niveles solubles de IL-12 (subunidad p70), IL-10, IL-1β, TNF-α e IL-6 en los diferentes grupos de estudio según el genotipo de los polimorfismos Asp-299Gly (A) y Thr-399Ile (B). Los histogramas representan la media y las barras la desviación estándar. Se indica la significación estadística solo cuando fue significativa (*: p<0,05).
Los datos presentados muestran en los pacientes coinfectados por el VHC y el VIH cambios en la expresión de TLR4 en monocitos, pero también en células T y células B. Estos resultados están de acuerdo con otros muy recientes en la literatura que muestran un aumento de expresión de TLR4 en distintos tipos de células T16–18. Por lo tanto, la expresión de TLR4 no se limita a células mieloides, sino que también se ve en linfocitos, sin que hasta el momento se sepa muy bien el mecanismo. En principio, el tratamiento con terapia de alta actividad antiretroviral (TARGA) reduce en cierto modo la expresión de TLR4 en linfocitos T16. En nuestro estudio los pacientes mostraban niveles elevados a pesar de recibir terapia TARGA en la mayoría de los casos. Sin embargo, los pacientes coinfectados mostraban niveles de TLR4 algo disminuidos respecto a los monoinfectados por VHC. Es posible que este hallazgo guarde relación con el tratamiento con IFN-α pegilado y ribavirina en nuestros pacientes, que han demostrado aumentar la expresión de TLR4 en células T19. El tratamiento con TARGA en los coinfectados reduciría en cierto grado dicho efecto. Por otro lado, no hay evidencia en la bibliografía acerca del efecto de la infección por el VHC en la expresión de TLR4 en linfocitos B, aunque no encontramos una explicación plausible para explicar el descenso observado en esta población celular. Es posible que el aumento de expresión de TLR4 en monocitos y células T sea un mecanismo protector frente a la transmisión del virus entre las células T CD4+ puesto que la secreción de interferón tipo i tras la activación de TLR4 inhibe dicha transmisión20. En el presente trabajo no hemos analizado los niveles de IFN-α para poder descartar esta posibilidad.
A pesar de los cambios de expresión de TLR4, no detectamos una relación entre los genotipos A/A y A/G del polimorfismo Asp-299Gly y C/C y C/T del polimorfismo Thr-399Ile, respectivamente, y la expresión de TLR4. Por lo tanto, no parece haber una causa genética que justifique las diferencias de expresión de TLR4. Es posible que los cambios sean debidos a un efecto directo del virus VIH o VHC puesto que recientemente se ha demostrado como, por ejemplo, la secuencia LTR del VIH es capaz de estimular la activación de TLR4 a través de NFκB21. Por otra parte, las variantes polimórficas Asp-299Gly y Thr-399Ile no difirieron entre los grupos de patología y los sujetos sanos, lo cual indica que dichos polimorfismos no confieren mayor susceptibilidad a ninguna de las infecciones del presente estudio. Tampoco tuvo relación con las variables de células CD4+ o carga viral en los pacientes con VIH, lo cual concuerda con otros autores, que no encuentran relación entre estas variables y los polimorfismos de TLR4 estudiado por nosotros, pero sí para el polimorfismo 1635A/G en TLR922. Por otro lado, el alelo G del polimorfismo Asp-299Gly de TLR4 parece ser más frecuente en población negra africana y solo se ha demostrado un efecto de dicho polimorfismo asociado a mayor riesgo de infección por tuberculosis en pacientes VIH23.
Finalmente, la expresión de TLR4 aumentada en nuestra población de estudio se vio acompañada de un aumento de citocinas solubles proinflamatorias y Th1 en suero. Estos datos sugieren una respuesta monocitaria aumentada y Th1 en nuestros pacientes. Es posible que estos datos aporten una evidencia más a favor de una activación del sistema inmunitario en la infección por el VIH, que contribuye a la enfermedad y dificulta la respuesta al tratamiento, al igual que sugieren ciertos autores24. No obstante, nuestros datos solo reflejan de forma indirecta la capacidad de secreción de citocinas por las células de forma individual y no es una prueba clara de hiper o hiporrespuesta de las células a la estimulación a TLR4. Así, otros autores han mostrado una capacidad de respuesta a la estimulación a través de TLR4 y otros TLR por parte de los monocitos de sujetos infectados por el VIH25,26. Más aún, se ha descrito que el VHC es capaz de estimular TLR4 e inducir una respuesta Th2 en lugar de Th1, como un mecanismo de protección desarrollado por el virus frente a la respuesta inflamatoria. Este efecto de reduciría con el tratamiento con interferón27. Por otro lado, llama la atención el aumento de IL-10, no descrito previamente en infección por VHC. Los niveles aumentados de IL-10 podrían tener un efecto antiinflamatorio potenciado por otros factores solubles como LPS aumentados en infecciones virales, tal como se ha descrito in vitro tras activación de TLR4 en monocitos de pacientes con infección crónica por VHC28.
Finalmente, es posible que parte de los cambios observados entre sujetos infectados y los controles sanos se pudieran explicar por diferencias dependientes del sexo, puesto que el grupo de controles sanos se componía de hombres y mujeres, mientras que el de infectados solo tenía hombres. Sin embargo, el análisis estadístico seleccionando únicamente los hombres del grupo control y la regresión univariante no mostraron ninguna dependencia de las variables analizadas respecto al sexo (datos no mostrados).
En resumen, nuestros datos indican una activación del sistema inmunitario en la infección por VHC, incluso en la coinfección con VIH, evidenciada por un aumento de expresión de TLR4 en linfocitos T y monocitos sanguíneos, pero no en linfocitos B, y que se acompaña por un aumento de los niveles séricos de citocinas proinflamatorias y Th1. Los cambios en estos parámetros no parecen ser motivados por los polimorfismos Asp-299Gly y Thr-399Ile en TLR4 que, además, no confieren una mayor susceptibilidad a la infección. Es posible que los cambios en la expresión de TLR4 y en la síntesis de citocinas secundaria sean debidos a factores propios de los virus. El estudio precisaría completarse con una serie amplia de pacientes con infección por VIH aislada, población de infectados difícil de encontrar en nuestro centro de estudio.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.