Introducción
El rechazo agudo continúa siendo la principal causa de fracaso en el trasplante renal de donador cadavérico que causa pérdida del injerto en el primer año post-trasplante con una incidencia de 15% a 25%.1 La función renal es vigilada post-trasplante comúnmente con el seguimiento de las variaciones de las concentraciones de creatinina en suero; sin embargo, éste es un indicador poco sensible ya que está influenciado por cambios en la masa muscular y la secreción tubular.2-5 Actualmente el diagnóstico de rechazo agudo sólo puede confirmarse a través de la biopsia renal, la cual es un método costoso, invasivo y acarrea el riesgo de complicaciones tales como dolor, hematuria, hematomas, fístulas arterio-venosas, sepsis y shock. Además no puede detectar cambios tempranamente porque la función renal no siempre correlaciona con las alteraciones histológicas. Un error de muestreo es otro problema conocido.6,7
En años recientes se ha incrementado el interés en la exploración del proteoma urinario humano, particularmente para el establecimiento de mapas que ayuden en el descubrimiento de biomarcadores auxiliares en el diagnóstico de diversos padecimientos, entre los que se encuentran las enfermedades renales.8,9 Dentro de las herramientas de la proteómica para la detección de proteínas se encuentra la electroforesis bidimensional; que consta de dos procedimientos: el isoelectroenfoque (iEE) en el cual las proteínas contenidas en muestras se separan de acuerdo a su punto isoeléctrico (pI) y la electroforesis en gel de poliacrilamida con dodecil sulfato de sodio (SDS-PAGE) en la cual las proteínas se separan de acuerdo a su peso molecular (PM). Posteriormente se puede realizar la identificación de las proteínas con el empleo de herramientas como la ionización asistida por la matriz mediante desorción con láser - tiempo de vuelo (MALDI-TOF). La concentración y purificación de las proteínas de las muestras es una parte importante en la metodología de la electroforesis bidimensional, para eliminar las interferencias. La orina contiene aniones y cationes que interfieren con el isoelectroenfoque de proteínas cuando se desea realizar la electroforesis bi-dimensional, por lo que es necesario eliminarlos. Se han descrito diferentes métodos de concentración y purificación de proteínas en orina, entre los cuales se encuentra la diálisis, liofilización, filtración, ultrafiltración y la precipitación.10 Reichelt y colaboradores utilizaron SELDI-TOF y encontraron dos proteínas características en un grupo de pacientes con rechazo al injerto renal, una de 25.71 kDa y otra de 28.13 kDa.7 Con esta misma metodología óriordan y colaboradores reportaron la presencia de siete proteínas específicas (2, 2.8, 4.8, 5.9, 7, 19 y 25.7 kDa) en la orina de un grupo de pacientes que presentaron rechazo al injerto renal con rechazo confirmado por histología de acuerdo a la clasificación de Banff.9 Jain y colaboradores utilizaron electroforesis bidimensional, MALDI-TOF y análisis de Western blot, y encontraron que la zinc α2 glucoproteína, α1microglobulina, α1 glucoproteína ácida e inmunoglobulina G estaban presentes en los pacientes con nefropatía diabética (ND) con microalbuminuria, y ausentes en pacientes sin microalbuminuria.11
Objetivo
Desarrollar un método de electroforesis bidimensional para la detección de proteínas en orina que sea útil para el diagnóstico de nefropatías como el rechazo agudo al injerto y la nefropatía diabética.
Métodos
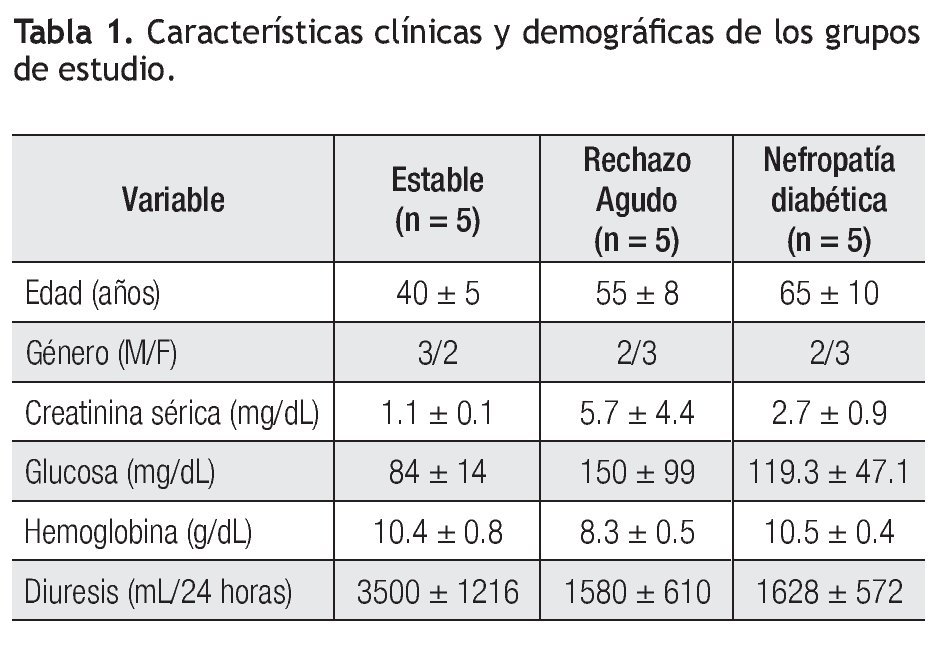
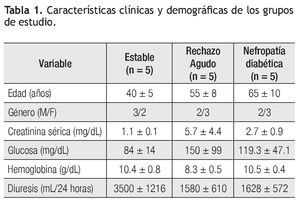
Pacientes: En este estudio se incluyeron pacientes que fueron trasplantados de riñón de donador cadavérico en el Hospital Universitario Dr. José Eleuterio González, durante el periodo junio de 2006 a junio de 2008. Se incluyeron tres grupos de pacientes, un grupo de pacientes trasplantados con evolución estable (n = 5) donde la edad promedio fue de 40 ± 5 años. El valor promedio de diuresis fue 3500 ± 1216 mL/24 horas y creatinina sérica 1.1 ± 0.1 mg/dL; un grupo de pacientes trasplantados con rechazo agudo al injerto renal (RAIR) (n = 5), con edad promedio de 55 ± 8 años, presentó un valor promedio de diuresis de 1580 ± 610 mL/24 horas y creatinina sérica de 5.7 ± 4.4 mg/dL. Se les realizó biopsia del injerto renal para su evaluación histológica de acuerdo a la clasificación de Banff,12 tres pacientes fueron Banff 1, un paciente con Banff 2 y un paciente con Banff 3, y 3) un grupo de pacientes (n = 5) con diagnóstico de nefropatía diabética. La edad promedio fue de 64.5 ± 10 años y presentaron un valor promedio de creatinina de 2.7 ± 0.9 mg/dL, diuresis de 1628 ± 572 mL/24 horas, proteínas en orina de 16.6 ± 2.3 g/24 horas y depuración de creatinina de 27.5 ± 18.3 mL/min (Tabla 1).
Preparación de las muestras: Las muestras de orina de los pacientes se recolectaron, centrifugaron a 3500 rpm durante siete minutos y almacenaron inmediatamente a -70°C en alícuotas de 4 mL. Se evaluaron cuatro procedimientos para el tratamientos de muestra: primero, precipitación con acetona; segundo, purificación de la muestra con un kit comercial (2D Clean up, Amersham) y precipitación con acetona; tercero, eliminación de albúmina con un kit comercial y precipitación con acetona y cuarto, ultrafiltración de muestra, eliminación de albúmina y precipitación con acetona. La precipitación con acetona se consistió en colocar 100 μL de muestra y 1 μL de coctel inhibidor de proteasas (CIP) (Amresco) con 1.5 mL de acetona a -20°C, se dejó precipitar durante 16 h a -20°C. Se centrifugó a 20 °C durante 20 minutos a 3500 rpm. Se eliminó el sobrenadante y se dejó secar el botón de proteínas. Se re-suspendió en 100 μL de agua milliQ. Para el segundo tratamiento se utilizó el kit comercial para la purificación de las muestras, las proteínas de la solución obtenida se precipitaron con acetona. Para el tercer tratamiento se tomaron 100 μL de muestra adicionada con 1 μL de CIP y se usó el kit Montage Albumin Deplete (Millipore) para eliminar la albúmina, se precipitaron las proteínas con acetona. Para el cuarto tratamiento, 4 mL de muestra se ultracentrifugaron con filtros de valor de corte de 10 KDa (Centricon, Millipore) a 9500 rpm a 20°C hasta que el volumen se redujo a 200 μL, se agregaron 2 μL de CIP. Se precipitaron las proteínas de acuerdo al protocolo antes mencionado. Para la determinación de la concentración de proteínas se utilizó el kit 2D Quant (Amersham).
Electroforesis bidimensional: Para la primera dimensión se realizó el iEE con un equipo Protean iEF de Biorad durante cinco horas. Se utilizaron tiras de un gradiente de pH inmovilizado (iPG) de pH 3 a 10 de 7 cm (Amersham), se rehidrataron (n = 3) durante 12 horas con un volumen de muestra equivalente a 150 μg de proteína y buffer de rehidratación constituido por urea 8M, CHAPS 2%, buffer iPG 0.5%, DTT 20 mM y azul de bromofenol 0.002%. Se realizó la incubación de las tiras con un buffer de equilibrio constituido por 50mM Tris-HCl, 6M urea, 30% de glicerol, 2% de SDS y 0.002% de azul de bromofenol. La segunda dimensión se realizó en un gel SDS-PAGE al 12% con un buffer de corrida constituido por 25mM de Tris-HCl, 192 mM de glicerina y 0.1% de SDS a un voltaje constante de 100V. La tinción de los geles se realizó con solución de azul de Coomassie.
Digitalización de geles: Los geles se digitalizaron con el equipo imageScanner ii (Amersham) a 300 dpi con un filtro rojo, y se analizaron con el programa ImageMaster 2D Platinum (Amersham) para verificar la presencia de proteínas características en cada grupo.
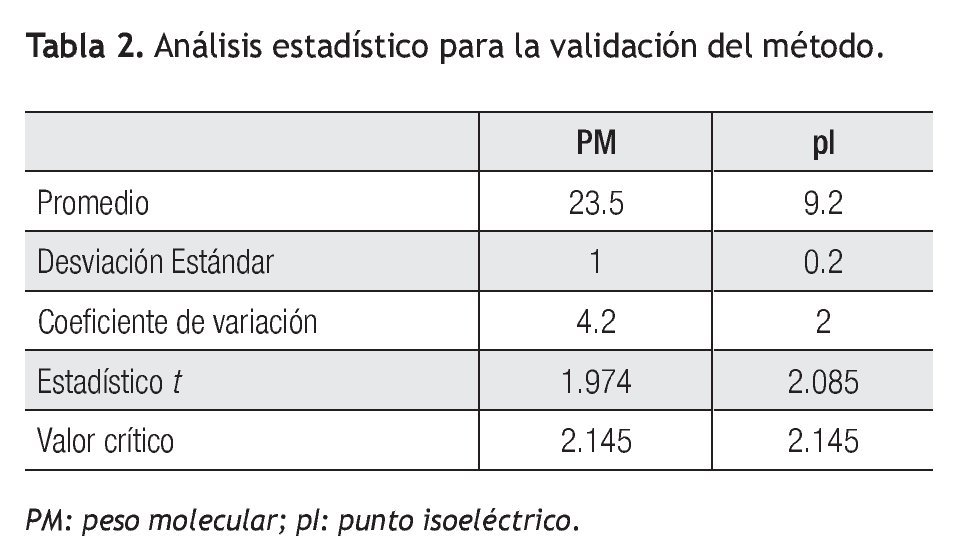
Validación del método: Se revisó la precisión y exactitud del método para el cálculo del PM y pI para lo cual se agregó a 15 muestras 20 μg de calibrador que contiene tripsinógeno, proteína con pI 9.3 y PM de 24 kDa. En el reporte de los geles adicionados, se buscó la mancha que correspondiera al calibrador. Se calculó promedio, desviación estándar y coeficiente de variación (CV) de pI y PM para calcular la precisión del método para el cual se tomó como aceptable un CV ≤5%. Para verificar la exactitud del método para el cálculo de pI y PM se aplicó la prueba t de Student con el programa Excel.
Se calculó sensibilidad, especificidad, valor predictivo positivo y valor predictivo negativo de las proteínas que se catalogaron como características de cada grupo y considerando como grupo de verdaderos negativos a los pacientes con evolución estable.
Resultados
Procedimientos de pre-tratamiento: Pasos 1 y 2: Precipitación con acetona y purificación y precipitación con acetona. Los geles obtenidos no ofrecieron resolución, además presentaron dos o varias proteínas muy abundantes que pudieran enmascarar la presencia de otras proteínas presentes en menor concentración. Debido a la baja resolución de las manchas se descartó para emplearlos con las muestras antes del análisis definitivo de las muestras de los pacientes incluidos en este estudio. Paso 3: Eliminación de albúmina y precipitación con acetona. La concentración final obtenida de proteínas de las muestras, fue la cantidad mínima recomendada para la tinción con azul de Coomassie, de acuerdo al tamaño de tira de iPG que se utilizó en este trabajo, por lo que se decidió no realizar la electroforesis bidimensional.13 Paso 4: Ultrafiltración, eliminación de albúmina y precipitación con acetona. Se pudo apreciar una mayor diversidad de las manchas presentes en la muestra; sin embargo la definición e intensidad de las mismas aún no se consideró suficiente, por lo que se aumentó la concentración de proteína añadida de 100 μg a 150 μg.
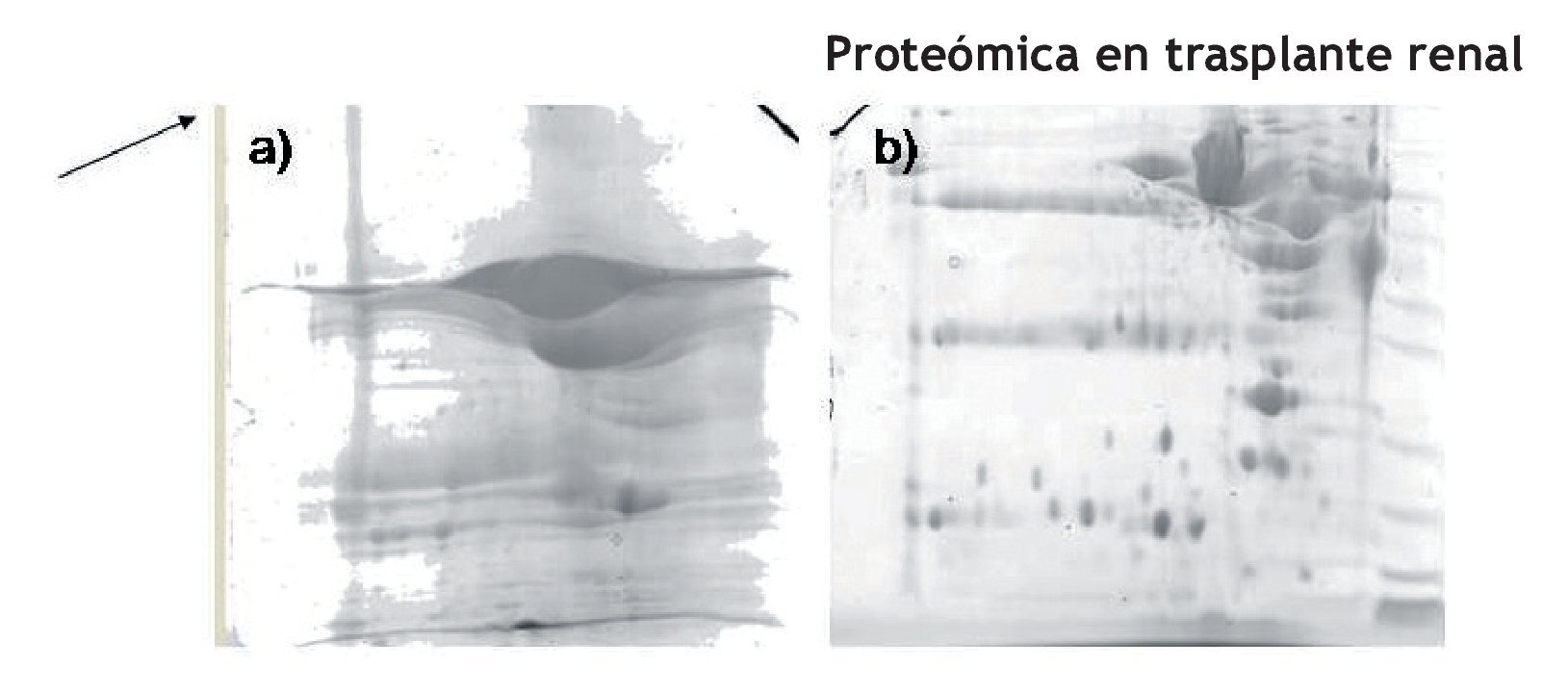
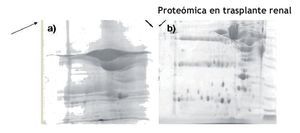
Este pre-tratamiento fue el que se utilizó para procesar todas las muestras incluidas en este proyecto para todos los grupos de estudio (Figura1).
Figura 1. a) Gel 2DE representativo de una muestra tratada con precipitación con acetona, limpieza con kit comercial y precipitación. b) Gel 2DE representativo de una muestra tratada con ultrafiltración para concentración, eliminación de albúmina y precipitación con acetona.
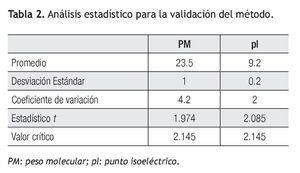
Precisión del método: Los resultados del cálculo de promedio, desviación estándar y coeficiente de variación se presentan en la Tabla 2. Tanto en pI como en PM el coeficiente de variación fue inferior a 5% por lo tanto se consideró que el método tuvo una precisión aceptable.
Exactitud del método: Los resultados de la aplicación de la prueba t de Student para el cálculo de la exactitud de método en la determinación del PM y pI se muestran en la Tabla 2. El valor crítico de t (2.14) fue superior al valor del estadístico t (1.97) lo que significó que el método tuvo la exactitud requerida para un nivel de confianza p = 0.05 para el cálculo del pI y del PM.
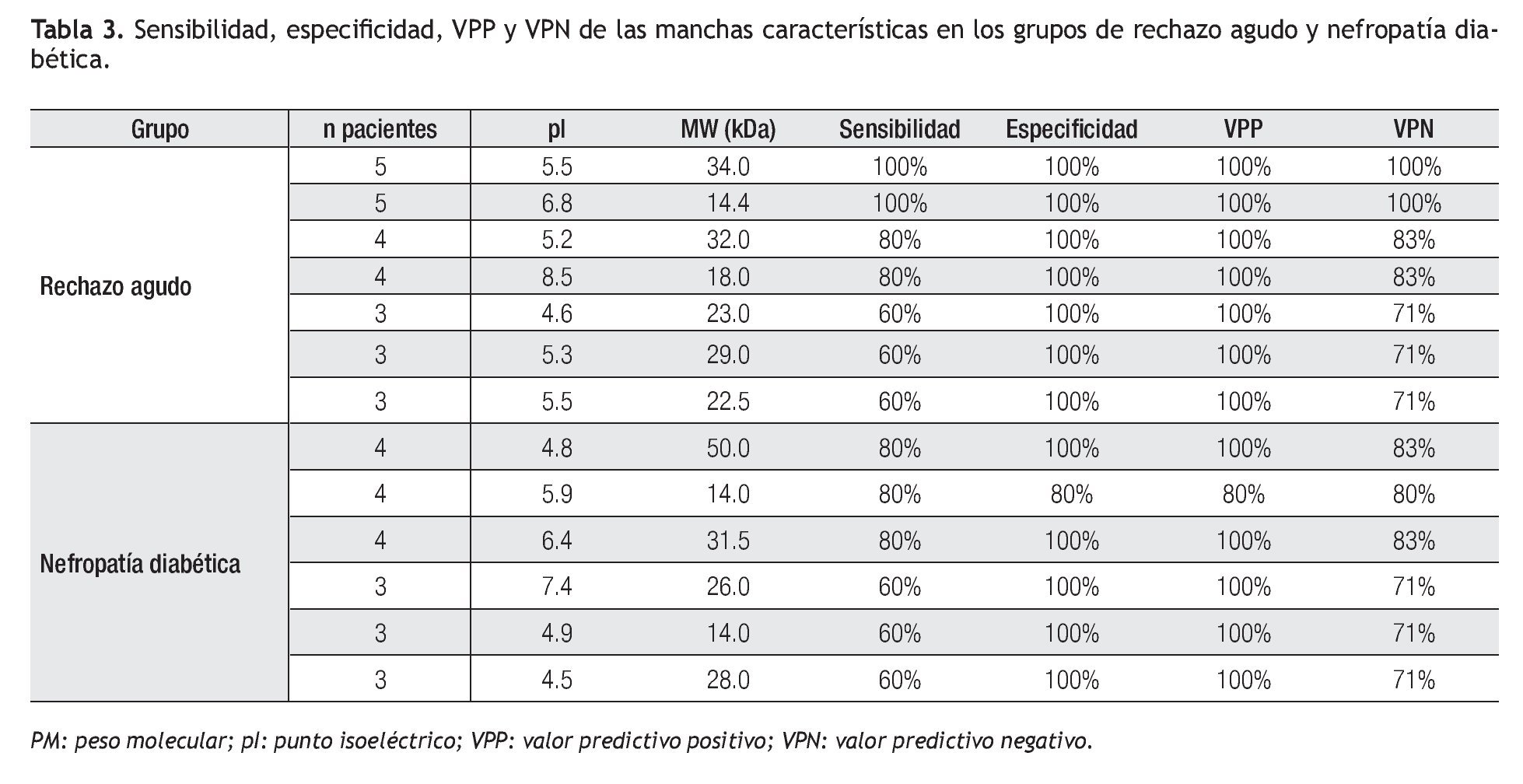
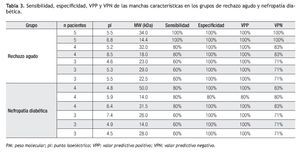
Comparativo entre grupos, sensibilidad, especificidad, valor predictivo positivo (VPP) y valor predictivo negativo (VPN): En los grupos de ND y RAIR se observó mayor resolución de las manchas con PM abajo de 25 kDa; sin embargo, se observó mayor diversidad de proteínas en los geles del grupo de pacientes con RAIR.
En el grupo de pacientes con trasplante renal de evolución estable, un paciente presentó un patrón electroforético diferente al de los otros cuatro pacientes. En tres de los otros cuatro pacientes se puedo detectar la presencia de una mancha muy intensa a un pI de 6.3 y PM de 66.5 aproximadamente, que no puede considerarse como característica del grupo ya que también se presentó en los geles de los pacientes de los grupos de ND y RAIR. Después del análisis de las imágenes se encontraron tres proteínas en el grupo ND que estuvieron presentes en cuatro de cinco pacientes, con 4.8 y 50, 6.4 y 31.5 pI y PM respectivamente, que tuvieron resultados de sensibilidad 80%, especificidad 100%, VPP 100% y VPN 83% , y la otra de 5.9 pI y 14 kDa que mostró una sensibilidad, especificidad, VPP y VPN de 80%, inferior a las otras ya que estuve presente en uno de los pacientes con evolución estable (Tabla 3).
En el grupo de pacientes con RAIR se encontraron dos proteínas que estuvieron presentes en los cinco pacientes con 5.5 y 34, 6.8 y 14.4 pI y PM respectivamente para las cuales los resultados de sensibilidad, especificidad, VPP y VPN fue de 100% y otras dos proteínas estuvieron presentes en cuatro de los cinco pacientes con 5.2 y 32, 8.5 y 18 pI y PM respectivamente en las que tuvieron resultados de sensibilidad 80%, especificidad 100%, VPP 100% y VPN 83% para cada una de ellas (Tabla 3).
Discusión
Se han descrito diversos métodos para el pre-tratamiento de muestras de orina y algunos sugieren la combinación de técnicas de separación y concentración como la diálisis, la ultrafiltración y la precipitación con solventes.10 En este trabajo se probaron diferentes alternativas para el pre-tratamiento. Los mejores resultados se obtuvieron al combinar la ultrafiltración y la precipitación con acetona ya que se pudo obtener mejor resolución de las manchas y con 150 μg de proteína añadida a la tira se pudo apreciar mejor la diversidad de las manchas.
Khan y Packer utilizaron otras alternativas de pretratamiento de muestras de orina de sujetos sanos.8
Es importante hacer notar que el presente trabajo trata sobre pacientes con daño renal, en los que es común la alta excreción de albúmina a través de la orina. Debido a esto, el método de eliminación de albúmina en las muestras antes de someterse a la electroforesis bidimensional fue de vital importancia para poder visualizar las proteínas que se encontraban en menor proporción con respecto a la albúmina. Respecto a la precisión del método electroforético, se obtuvo un CV de 2% en el caso de pI y un CV de 4.2% para el PM; debido a que ambos valores de CV son inferiores a 5%, se estableció que el método es preciso para la determinación de pI y PM con un nivel de confianza de 95%. Y en el caso de la evaluación de la exactitud se aplicó la prueba t de Student a los valores obtenidos. El valor crítico de t (2.14) fue superior al valor del estadístico t (1.97) para el pI, al igual que para el PM se obtuvo que el valor crítico de t (2.14) fue superior al valor del estadístico t (2.08) lo que significó que el método tuvo la exactitud requerida para un nivel de confianza de 95%.
Diversos estudios, han evaluado la utilidad del análisis proteómico en pacientes con diversas nefropatías. Algunos de ellos emplearon SELDI-TOF (Surface Enhanced Laser Desorption Ionization Time of Flight - Mass Spectrometry) en pacientes con trasplante renal que presentaron rechazo agudo, donde identificaron proteínas de 6.5, 6.6, 6.7, 7.1 y 13.4 kDa;14 1915 y de 25.7 kDa y 28.1 kDa7 como posibles biomarcadores de rechazo. En el presente estudio se encontraron proteínas de 14.4 kDa (pI 6.8), 18 kDa (4/5 pacientes) y 29 kDa en los cinco pacientes del grupo de rechazo, lo cual puede sugerir que se trate de las mismas proteínas.
Algunos estudios refieren la presencia en muestras de orina de α-1- microglobulina de pI 5.3 y PM 33 como característica de pacientes con daño tubular.16,17 En el presente trabajo se encontró una mancha de pI 5.5 y 34 kDa que podría corresponder a α-1-microglobulina.
En Rao y colaboradores realizaron el análisis proteómico de muestras de orina de pacientes con diabetes mellitus (DM) tipo 2 con macroalbuminuria, donde encontraron 11 proteínas expresadas diferencialmente por comparación de éste grupo con un grupo de pacientes con DM tipo 2, sin albuminuria. Entre las proteínas que encontraron, se identificó a calgranulina B, la cual tiene pI 6 y 13.2 kDa. En el presente trabajo en el grupo de pacientes con ND se detectó en cuatro pacientes una mancha de pI 5.9 y 14 kDa, es posible que se trate de la misma proteína.18
La biopsia renal es el estándar de oro para el diagnóstico de las nefropatías; sin embargo, su indicación es muy reservada debido a las complicaciones que pueden derivarse del procedimiento de su obtención. Por ello, no es frecuente indicar una biopsia renal en un paciente que clínicamente presenta una evolución satisfactoria en el pos-trasplante, tampoco en el paciente con la historia clínica típica de una ND. El objetivo de incluir a este grupo de estudio fue evaluar si la expresión de las proteínas era diferente con respecto al grupo RAIR, ya que también valdría la pena verificar la expresión de estas proteínas en cada nefropatía pre y pos-trasplante.
En el caso del paciente del grupo de evolución estable que presentó un patrón electroforético distinto del resto del grupo, dicho paciente presentó un proceso infeccioso de vías urinarias una semana posterior a la toma de las muestra de orina utilizada en este estudio. Lo que podría sugerir que esta técnica también puede ser de utilidad en la detección temprana de estos procesos.
Conclusión
La electroforesis bidimensional resultó tener una sensibilidad, especificidad, VPP y VPN superiores a 80% en la detección de proteínas en orina, la cual podría llegar a ser parte de las herramientas de diagnóstico de las enfermedades renales y en el diagnóstico de rechazo al injerto; sin embargo, se requiere aumentar la cantidad de pacientes para definir si estas proteínas son específicas de cada grupo de estudio para poder considerarlos como biomarcadores.
Correspondencia: Dra. C. Paula Cordero Pérez.
Avenida Gonzalitos No. 235, Col. Mitras Centro, Monterrey N.L. CP 64460.
Teléfono: (81) 8329 4205, fax: (81) 8989 4088.
Correo electrónico:paucordero@yahoo.com.mx
Recibido: Marzo 2011.
Aceptado: Julio 2011