El presente artículo revisa la evolución de los estudios en genética del ictus desde la aproximación por gen candidato hasta los recientes estudios de genome wide association. Se destaca la complejidad de esta afección por sus muy variados mecanismos etiopatogénicos, las dificultades que comporta el estudio de su componente genético y las soluciones que se han aportado. Se subraya en especial el valor de las colaboraciones entre distintos centros, ya sea de manera puntual o sobre todo a través de la creación de consorcios estables. Esta estrategia actualmente se hace imprescindible a la hora de realizar estudios de alta calidad científica que permitan seguir avanzando en el conocimiento de las bases genéticas del ictus tanto en etiología, como en tratamiento y prevención.
This article provides an overview of stroke genetics studies ranging from the candidate gene approach to more recent studies by the genome wide association. It highlights the complexity of stroke owing to its different aetiopathogenic mechanisms, the difficulties in studying its genetic component, and the solutions provided to date. The study emphasises the importance of cooperation between the different centres, whether this takes places occasionally or through the creation of lasting consortiums. This strategy is currently essential to the completion of high-quality scientific studies that allow researchers to gain a better knowledge of the genetic component of stroke as it relates to aetiology, treatment, and prevention.
El ictus representa hoy en día en España la segunda causa de muerte en la población general y la primera en mujeres (http://www.ine.es/prensa/np703.pdf). Además, es la primera causa de discapacidad física en adultos y la segunda de demencia1.
Sin embargo, y a pesar de las importantes mejoras en prevención, diagnóstico y tratamiento del ictus que se han realizado en las últimas 2 décadas, la Organización Mundial de la Salud prevé un incremento de su incidencia del 27% hasta el 20252. Se han descrito múltiples factores de riesgo asociados a esta afección, aunque a la hora de entender sus causas parece necesario contar con la participación tanto de factores externos como de la predisposición de cada individuo3. En este sentido, se ha demostrado un componente hereditario de la enfermedad y la carga genética desempeñaría un papel muy significativo al respecto4. El desarrollo tecnológico ha favorecido en los últimos años llevar a cabo estudios de genotipificación masiva, lo que ha permitido realizar estudios genéticos no sesgados y el descubrimiento de nuevos genes y vías metabólicas asociadas al ictus.
Los objetivos de este trabajo son revisar el estado actual y las perspectivas futuras de la genética en el campo de la enfermedad cerebrovascular.
DesarrolloConsideraciones genéticas del ictusA diferencia de las enfermedades monogénicas, las enfermedades complejas se caracterizan por presentar una gran diversidad etiopatológica. Se sabe que la historia familiar es uno de los factores de riesgo más importantes en muchas de las enfermedades complejas, como ocurre en enfermedades cardiovasculares, cáncer o enfermedades autoinmunes5. Esto indica que, a pesar de no tratarse de enfermedades mono/oligogénicas, existen variaciones genéticas heredadas. Se cree que la combinación de variantes alélicas con efecto de riesgo pequeño, pero aditivo o multiplicativo y su interacción con el ambiente, pueden desempeñar un papel crucial en el desarrollo de una afección compleja5,6.
En el caso del ictus isquémico, se ha descrito una heredabilidad de entre el 30 y el 50%4. Debido a sus múltiples causas, a los numerosos factores de riesgo implicados y a las diferentes formas de manifestación, el ictus podría considerarse como la manifestación final de múltiples enfermedades complejas. Aparte de la clasificación más simple de los ictus entre isquémicos y hemorrágicos, podemos diferenciar varios subtipos etiopatogénicos completamente diferentes. Varios estudios publicados recientemente han demostrado la existencia de heterogeneidad genética, en cuanto a la heredabilidad entre los ictus lacunares, cardioembólicos y ateroescleróticos, siendo estos últimos los más fuertemente asociados a historia familiar4,7,8.
Lo mismo ocurre en las hemorragias intracerebrales (HIC), que representan el 15% de todos los ictus. Las vías patogénicas implicadas en la hemorragia lobar, muy asociada a la angiopatía amiloide cerebral (AAC), pueden diferir de forma sustancial con las implicadas en las hemorragias hipertensivas9,10.
Así pues, el planteamiento de los estudios genéticos en el ictus debe contemplar estas particularidades en su diseño y contar con las dificultades que plantea la investigación de una enfermedad heterogénea.
Estudios genéticos de asociación en enfermedades complejas. Diferentes técnicas y abordajesLa investigación sobre las causas genéticas del ictus de forma global, prescindiendo de aquellas que siguen un patrón de herencia mendeliana clásica, se fundamenta sobre todo en los estudios de asociación caso-control. Estos se basan en estudiar la diferente frecuencia de variantes genéticas en enfermos respecto a no enfermos. El estudio de variantes comunes o polimorfismos de nucleótido simple (SNP) se basa principalmente en diseños de «candidate-gene» y de genome wide association, mientras que el reciente desarrollo de técnicas de secuenciación completa del exoma permite estudiar variantes infrecuentes. Otros abordajes, como el análisis de la variación de número de copias o el estudio del epigenoma, son tratados también más adelante.
Estudios mediante «candidate-gene»Durante varias décadas, y hasta hace pocos años, el abordaje más extendido fue el de «candidate-gene» o estudios de gen candidato. Esta aproximación consiste en seleccionar aquellos genes que podrían asociarse a una enfermedad basándose en la fisiopatología de esta y en las vías que se supone están implicadas. Se identifican los SNP previamente descritos en la zona de estos genes candidatos a ser analizados y se escogen los más representativos (conocidos como TagSNP), generalmente por hallarse en desequilibrio de ligamiento11. Estos TagSNP candidatos son genotipificados en una población y, mediante análisis estadísticos, se determina si existe alguna asociación entre las variantes genéticas y el espectro clínico.
Estos estudios presentan varios problemas. Por un lado, el hecho de que la selección de estos genes se sustente en hipótesis generadas a priori puede implicar una inclinación del investigador a buscar diferencias espurias basadas en sus ideas preconcebidas. Por otro lado, deja de analizar posibles genes potencialmente implicados en la enfermedad.
En relación con los estudios de gen candidato, se han asociado múltiples genes con el riesgo de presentar un ictus isquémico. Sin embargo, un estudio reciente demuestra que tras aplicar una corrección estadística adecuada ajustando por el número de SNP analizados, ninguna de estas posibles asociaciones alcanza la significación estadística4. Por otro lado, ninguno de los resultados obtenidos mediante este abordaje ha sido replicado posteriormente de forma clara, por lo que esta aproximación ha demostrado ser poco consistente y fiable en cuanto al ictus isquémico4,12.
Aun así, a través de esta aproximación se ha demostrado que mutaciones en los genes COL4A1 y COL4A2 se asocian a HIC. Los heterotrímeros compuestos por colágeno tipo iv alfa 1 (COL4A1) y alfa 2 (COL4A2) son uno de los principales componentes de casi todas las membranas basales del organismo y se han asociado a gran número de enfermedades no solo cerebrovasculares, sino también oculares, renales y musculares13. Inicialmente, mediante estudios de herencia familiar, se encontró que las mutaciones en COL4A1 causaban porencefalia tipo i, demostrando posteriormente causar HIC subcorticales, tanto esporádicas como recurrentes, y también ictus lacunares y leucaraiosis14-16.
En relación también con la HIC, se ha demostrado recientemente que los pacientes portadores de los haplotipos ¿2 y ¿4 del gen APOE tienen un riesgo mayor de HIC lobar (odds ratio [OR] 1,82 [IC del 95%, 1,50 a 2,23]; p=6,6×10−10) y (OR 2,20 [IC del 95%, 1,85 a 2,63]; p=2,4×10−11, respectivamente), probablemente por el efecto de estas variantes en el riesgo de desarrollar una AAC17. Posteriormente, el mismo grupo demuestra también que la variante APOE ¿2 se asocia a un mayor volumen de la HIC y, como consecuencia del mayor volumen, a un peor pronóstico funcional y mayor mortalidad18.
Estudios de asociación en genoma completo o genome wide association studiesEl desarrollo de los llamados estudios de asociación de genoma completo (GWAS) que permiten determinar la frecuencia alélica de cientos de miles de SNP en casos versus controles, ha sido posible por 2 grandes motivos. En primer lugar, por la creación del International HapMap Project, que determinó características, equilibrio de ligamiento y frecuencias alélicas de los SNP hallados en el genoma de 270 individuos de 4 orígenes ancestrales diferentes, con el objetivo de crear una base genome-wide pública, de acceso abierto a todos los investigadores5,19. Y, en segundo lugar, gracias a los importantes avances técnicos y al desarrollo de plataformas que permiten genotipificar entre 50.000 y 2,7 millones de SNP por individuo a un precio cada vez menor. Los estudios mediante GWAS representan una forma mucho más exhaustiva de estudiar el genoma que los diseños de gen candidato20. Evitan los sesgos de hipótesis preconcebidas y además permiten identificar nuevas asociaciones aun cuando el gen todavía no ha sido implicado en la enfermedad, a diferencia de los estudios anteriores. Pero esta aproximación conlleva otro tipo de limitaciones. Las 2 principales son los grandes tamaños muestrales necesarios en estos estudios y la dificultad en la interpretación de los posibles resultados positivos21. Además, implican la necesidad de realizar estrictos controles de calidad, con el fin de detectar errores de genotipificación y valorar la posible estratificación de poblaciones22. Esta última se basa en que hay diferencias en las frecuencias alélicas de un SNP dependiendo de los orígenes étnicos ancestrales de cada individuo (aunque fenotípicamente sean de la misma etnia). Así, se pueden generar resultados falsos si las poblaciones de casos y controles no son comparables en su origen genético y, por tanto, es necesario realizar los ajustes pertinentes. Actualmente, la mejor forma de evitar estas inconsistencias es mediante la replicación de los resultados obtenidos en muestras independientes de gran tamaño23. Debido a la gran cantidad de análisis a realizar (al menos uno por cada SNP genotipificado), se deben aplicar penalizaciones estadísticas por comparaciones múltiples, que obligan a establecer la significación en valores de p de hasta 10−8 (en el caso de analizar un millón de SNP)6. Así pues, para alcanzar la potencia estadística necesaria, estos estudios requieren aumentar significativamente el número de individuos analizados.
Los primeros resultados relacionados con enfermedades complejas aparecieron en 2005, descubriendo la implicación del complemento H en la degeneración macular relacionada con la edad24. Se han conseguido descubrimientos interesantes en relación con la diabetes mellitus de tipo 1 (PPARG25, KCNJ1126 y TCF7L227), la artritis reumatoide y la diabetes mellitus de tipo 2 (PTPN2228,29) y la enfermedad de Crohn (NOD230), entre muchos otros, descubriendo así un gran número de loci implicados en la predisposición o riesgo de presentarlas.
En contraposición a los resultados desalentadores obtenidos con otras aproximaciones, varias asociaciones a ictus isquémicos han sido replicadas consistentemente. Es el caso de algunas asociaciones descubiertas mediante GWAS para otras enfermedades asociadas a ictus isquémicos, que han demostrado ser también factores de riesgo para ictus31. Así, 2 variantes, rs1906591 (OR 1,48 [IC del 95%, 1,28 a 1,71]; p=4,7×10−6) y rs7193343 (OR 1,22 [IC del 95%, 1,10 a 1,35]; p=0,00021), en los cromosomas 4q25 y 16q22, en regiones cercanas a los genes PITX2 y ZFHX3, respectivamente, inicialmente identificadas por contribuir al riesgo de presentar una fibrilación auricular, han demostrado también asociarse al riesgo de presentar un ictus de etiología cardioembólica32,33.
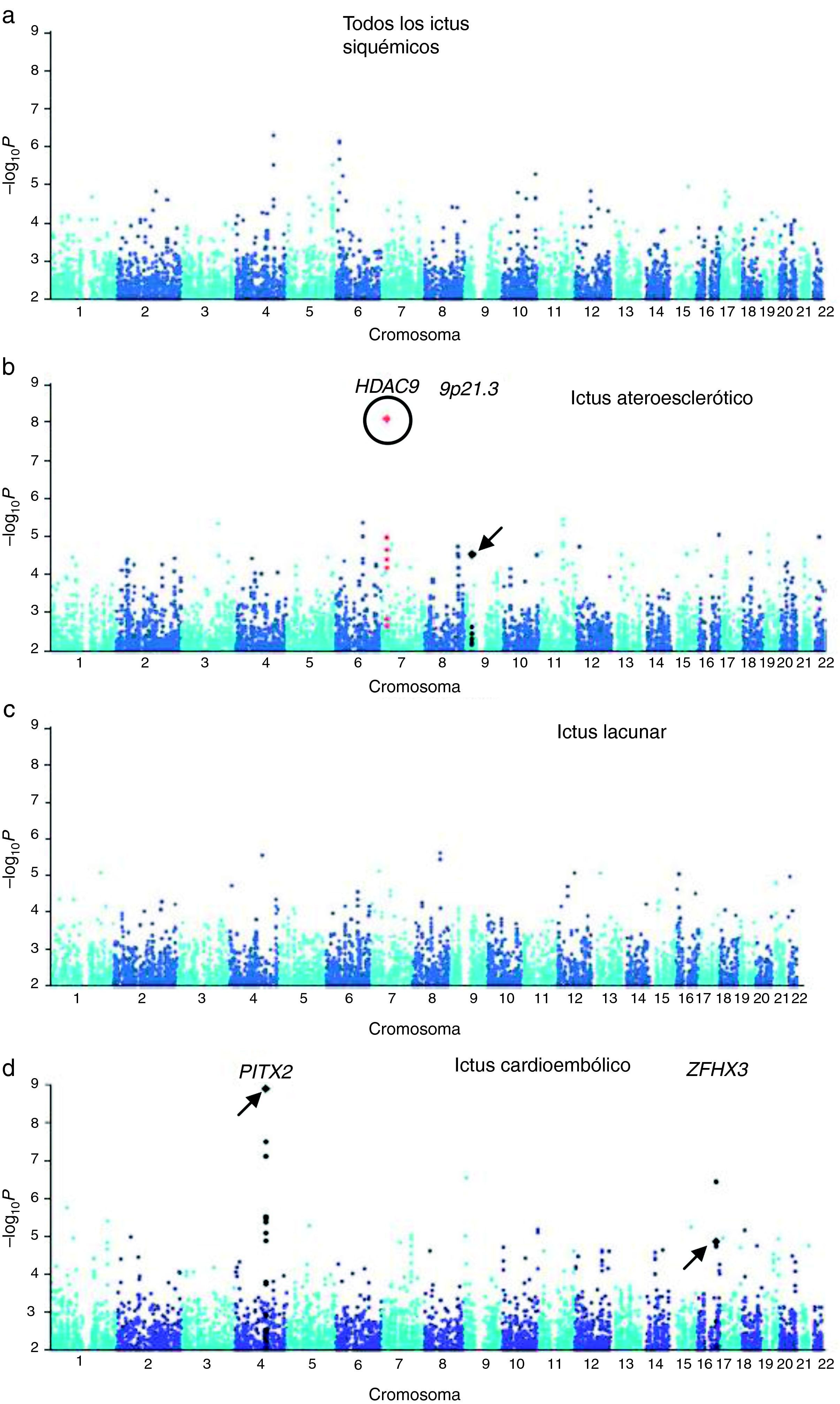
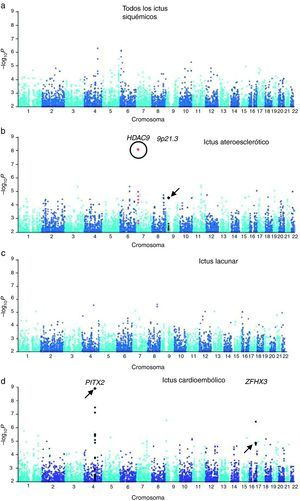
Se identificó una región en el cromosoma 9p21, próxima a los genes CDKN2A/B, asociada a infarto de miocardio y enfermedad coronaria. Examinando posteriormente estos loci mediante un estudio de gen candidato, se vio la asociación del SNP rs1537378 (OR 1,21; [IC del 95%, 1,07-1,37]; p=0,002) con ictus de etiología aterotrombótica34. También, en relación con los ictus isquémicos de etiología aterotrombótica, una nueva asociación mediante GWAS ha sido identificada: el polimorfismo rs11984041 (OR 1,42 [IC del 95%, 1,28 a 1,57]; p=1,87×10−11) en la región del gen HDAC9 en el cromosoma 7p21.1, aunque se desconoce el mecanismo patogénico35 (fig. 1).
Resultados del GWA de las muestras de Reino Unido y Alemania. a) Resultado de todos los ictus isquémicos. b) Ictus ateroescleróticos. c) Ictus lacunares. d) Ictus cardioembólicos. Rodeado por un círculo aparece el nuevo locus HDAC9. Señalados con una flecha aparecen los loci previamente descritos en la literatura.
Publicado con el permiso de Macmillan Publishers Ltd: Nature Genetics34, copyright 2012.
Adaptada con el permiso de Macmillan Publishers Ltd: Nature Genetics34, copyright 2012.
Como asociaciones nuevas, un grupo japonés ha identificado una relación significativa entre el polimorfismo rs2230500 (OR 1,66 [IC del 95%, 1,33 a 2,09]; p=9,84×10−6) del gen PRKCH en el cromosoma 14 con ictus de etiología lacunar, con resultados en replicación dispares en otras etnias4,36.
Secuenciación del exomaUna de las debilidades de los GWAS es que únicamente analizan variantes comunes (frecuencia poblacional > 1%) y no contemplan las variantes infrecuentes (rare variants). Es posible que existan múltiples variantes infrecuentes que contribuyan al riesgo genético de las enfermedades complejas. Para intentar identificarlas, se están desarrollando nuevas técnicas de secuenciación masiva del exoma o exome sequencing. Estas nuevas técnicas analizan la presencia de todo tipo de polimorfismos o mutaciones que se encuentren en las regiones codificantes del genoma, por lo tanto, son capaces de detectar estas variantes infrecuentes que los estudios de GWAS no identifican. Se trata de una aproximación prometedora, aunque su metodología está en desarrollo y todavía están por consensuar las formas más adecuadas de manejar estadísticamente las cantidades ingentes de datos que generan estos análisis37.
Estudios de variación en el número de copiasLa variación en el número de copias o copy number variation (CNV) es una variación estructural del genoma, que puede producirse en forma de (micro) deleciones, duplicaciones o inversiones, generando un número anómalo de copias de un fragmento del ADN. Esta variación puede influir tanto en la expresión como en la conformación de las proteínas para las que codifica esa región38-40. Poco se ha publicado hasta la fecha con respecto al ictus. En un estudio con pacientes con ictus isquémicos no se detectó ninguna CNV asociada, aunque el tamaño muestral era reducido y no se tuvieron en cuenta los distintos subtipos de ictus41. Sin embargo, hay proyectos de genotipificado masivo en marcha que incluyen en sus plataformas el análisis de múltiples CNV.
EpigenéticaOtro campo emergente es el de la epigenética, modalidad que estudia los cambios en la regulación génica heredables que no afectan a la secuencia del ADN. A pesar de que el genoma de cada célula de nuestro organismo es igual, la transcripción génica en cada tejido viene regulada por los distintos mecanismos epigenéticos. Entre los principales, destacan la metilación del ADN, las modificaciones en las histonas y el ARN no codificante42. De estos, la metilación del ADN es el más estudiado y conocido hasta el momento. Los hallazgos más destacables se han obtenido en el estudio de la epigenética de tejidos tumorales. Así, se ha visto que en el caso del glioblastoma multiforme la metilación del gen MGMT inactiva al enzima O (6)-metilguanina-ADN metiltransferasa (MGMT), lo que se ha asociado a una mejor respuesta al tratamiento quimioterapéutico con temozolamida. Esta determinación es aplicada ya en la práctica clínica43. Actualmente, los estudios epigenéticos se están ampliando a otras enfermedades, incluido el ictus. Se ha descrito una asociación entre el ictus y una hipometilación global del ADN, aunque está en fase de replicación44. En el marco del Consorcio Español de Genética del Ictus, están en marcha varios estudios en este campo de los que todavía no se dispone de resultados.
Escalas de riesgo y algoritmos genéticos (genetic networks)De cara a integrar la información y la capacidad predictiva de cada uno de los SNP que se han asociado a un fenotipo, están tomando relevancia el diseño de scores y genetic networks. Los métodos más usados para crear estas escalas o scores genéticos son: mediante la suma de alelos de riesgo (1 punto por cada alelo presente), o bien mediante un riesgo ponderado (la puntación para cada alelo depende de la razón de oportunidades u OR reportada para ese alelo). En un estudio muy reciente, se ha demostrado una asociación entre un score conformado por alelos de riesgo para hipertensión arterial y el riesgo de presentar una HIC profunda45.
Otra forma de englobar el conocimiento es mediante la creación de redes o algoritmos46. Un ejemplo, es la red bayesiana o Bayesian network, una herramienta estadística que representa todo un conjunto de variables asociadas teniendo en cuenta las relaciones de dependencia condicionales que se establecen entre ellas. Lo que permite este modelo es estimar la probabilidad de que sucedan las variables no conocidas en función de las que sí conocemos. Las principales ventajas de estas redes bayesianas son, por un lado, que permiten las inferencias bidireccionales (es decir, de los efectos a las causas y de las causas a los efectos) y, por el otro, la rápida actualización de conocimientos47.
Genomic convergenceEn el estudio genético de las enfermedades complejas está tomando relevancia el concepto de genomic convergence. Este se fundamenta en sumar y concentrar la información y los datos que aportan cada uno de los diferentes abordajes para alcanzar así una mayor potencia de descubrimiento y una mayor solidez en los resultados48. Como descubrimiento reciente, varios polimorfismos del gen TTC7B han demostrado conferir susceptibilidad en el ictus isquémico49.
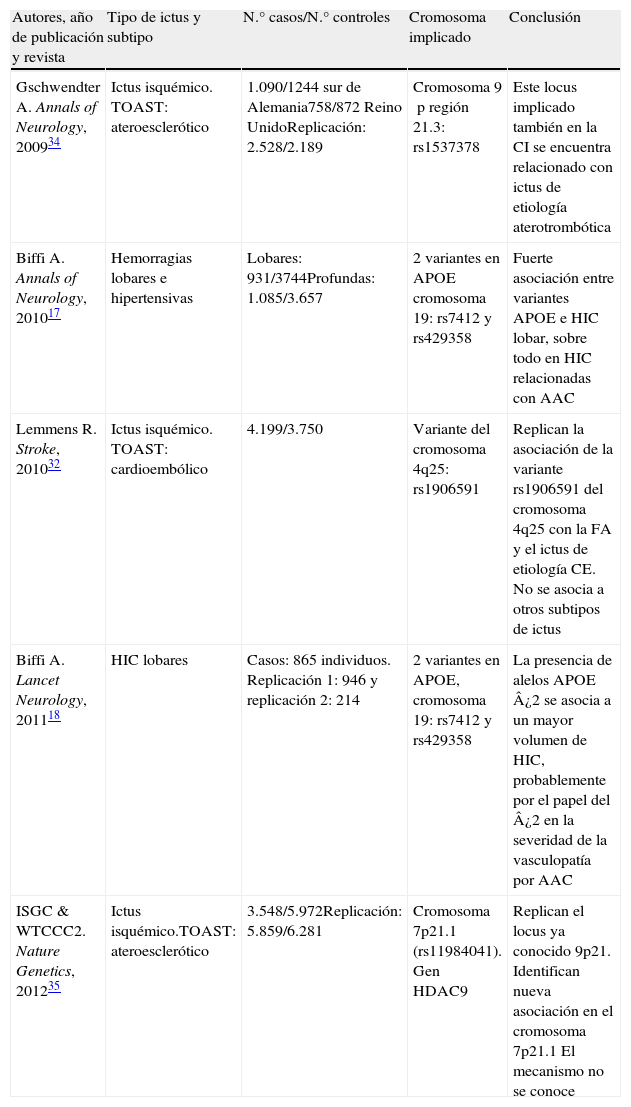
La creación de consorcios. Consorcio Español de Genética del ictus (GeneStroke)A partir de la formación de grandes consorcios internacionales, como el Wellcome Trust Case Control Consortium del Reino Unido, se crea en el año 2007, el International Stroke Genetics Consortium (ISGC) (http://www.strokegenetics.org). Se han identificado y replicado diversas asociaciones en el marco de este consorcio (ISGC) (tabla 1) y se han generado varios de los proyectos, actualmente en marcha, más ambiciosos en este campo a nivel mundial.
Principales artículos publicados en revista de gran impacto en el marco del International Stroke Genetics Consortium
| Autores, año de publicación y revista | Tipo de ictus y subtipo | N.° casos/N.° controles | Cromosoma implicado | Conclusión |
| Gschwendter A. Annals of Neurology, 200934 | Ictus isquémico. TOAST: ateroesclerótico | 1.090/1244 sur de Alemania758/872 Reino UnidoReplicación: 2.528/2.189 | Cromosoma 9p región 21.3: rs1537378 | Este locus implicado también en la CI se encuentra relacionado con ictus de etiología aterotrombótica |
| Biffi A. Annals of Neurology, 201017 | Hemorragias lobares e hipertensivas | Lobares: 931/3744Profundas: 1.085/3.657 | 2 variantes en APOE cromosoma 19: rs7412 y rs429358 | Fuerte asociación entre variantes APOE e HIC lobar, sobre todo en HIC relacionadas con AAC |
| Lemmens R. Stroke, 201032 | Ictus isquémico. TOAST: cardioembólico | 4.199/3.750 | Variante del cromosoma 4q25: rs1906591 | Replican la asociación de la variante rs1906591 del cromosoma 4q25 con la FA y el ictus de etiología CE. No se asocia a otros subtipos de ictus |
| Biffi A. Lancet Neurology, 201118 | HIC lobares | Casos: 865 individuos. Replicación 1: 946 y replicación 2: 214 | 2 variantes en APOE, cromosoma 19: rs7412 y rs429358 | La presencia de alelos APOE ¿2 se asocia a un mayor volumen de HIC, probablemente por el papel del ¿2 en la severidad de la vasculopatía por AAC |
| ISGC & WTCCC2. Nature Genetics, 201235 | Ictus isquémico.TOAST: ateroesclerótico | 3.548/5.972Replicación: 5.859/6.281 | Cromosoma 7p21.1 (rs11984041). Gen HDAC9 | Replican el locus ya conocido 9p21. Identifican nueva asociación en el cromosoma 7p21.1 El mecanismo no se conoce |
AAC: angiopatía amiloide cerebral; CE: cardioembólico; CI: cardiopatía isquémica; FA: fibrilación auricular; HIC: hemorragia intracerebral; TOAST: Trial of Org 10172 in Acute Stroke Treatment.
El Consorcio Español de Genética del Ictus (GeneStroke) (http://www.genestroke.com), se crea en el año 2009 con el objetivo prioritario de estudiar las bases genéticas del ictus y promover la asociación entre grupos con intereses comunes en este campo. GeneStroke ha puesto en marcha diversos proyectos relacionados con la etiología, la evolución y el pronóstico del ictus, en los que colaboran distintos centros. Los detalles de los proyectos y sus participantes se pueden consultar en su página web (www.genestroke.com).
Desde el Consorcio Español de Genética del Ictus se fomenta el modelo de grandes estudios colaborativos para afrontar los retos que plantea la investigación en el campo de la genética de las enfermedades cerebrovasculares.
ConclusionesEl ictus es, en nuestro entorno, una de las enfermedades más prevalentes y con mayor impacto en morbimortalidad. Presenta una alta heredabilidad, por lo que el conocimiento de sus bases genéticas podrá contribuir a identificar a los individuos de riesgo y a diseñar estrategias de prevención y terapias más eficientes. El estudio de este sustrato genético debería ser una prioridad, así como la colaboración entre diferentes grupos y profesionales con el objetivo de realizar proyectos de alta calidad científica que puedan traducirse en un futuro beneficio para la sociedad.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Agradecer, en primer lugar, a todos los miembros del Consorcio Español de Genética (GeneStroke), que forman parte del Consorcio de forma estable, o bien por su contribución puntual en algún proyecto. Un agradecimiento especial a los miembros del International Stroke Genetics Consortium.