El glioblastoma multiforme es el tumor cerebral primario más común y con el pronóstico más desfavorable del sistema nervioso central. A pesar de los numerosos estudios y avances en medicina, este sigue siendo letal, con una esperanza de vida promedio de 15 meses posteriores a la quimiorradioterapia.
DesarrolloRecientemente, se han estudiado diversos factores asociados al diagnóstico y el pronóstico de pacientes con glioblastoma, como la localización tumoral, principalmente la zona subventricular; una de las áreas neurogénicas más activas del cerebro humano adulto. Los pacientes con glioblastoma asociados a esta zona en particular presentan generalmente una mayor agresividad, lo que resulta en un pronóstico desfavorable y una menor esperanza de vida. Actualmente, se ha profundizado en el estudio de los microARN, los cuales reflejan patrones de expresión distintos en condiciones fisiológicas o fisiopatológicas. Está reportado que los niveles de expresión de ciertos microARN, principalmente aquellos relacionados a procesos neurogénicos, se ven desregulados en eventos oncogénicos, favoreciendo así la gliomagénesis y la agresividad tumoral. En la presente revisión se discuten algunos de los microARN más importantes implicados en procesos neurogénicos de la zona subventricular y su asociación con la agresividad del glioblastoma.
ConclusionesLa regulación y función de los microARN desempeña un rol importante en el desarrollo y la progresión del glioblastoma; en consecuencia, la comprensión de las alteraciones de los microARN implicados en la diferenciación, así como en la maduración neural y glial, podrían ayudar a entender mejor las características malignas del glioblastoma.
Glioblastoma multiforme is the most common primary brain tumour, with the least favourable prognosis. Despite numerous studies and medical advances, it continues to be lethal, with an average life expectancy of 15 months after chemo-radiotherapy.
DevelopmentRecent research has addressed several factors associated with the diagnosis and prognosis of glioblastoma; one significant factor is tumour localisation, particularly the subventricular zone, which represents one of the most active neurogenic niches of the adult human brain. Glioblastomas in this area are generally more aggressive, resulting in unfavourable prognosis and a shorter life expectancy. Currently, the research into microRNAs (miRNA) has intensified, revealing different expression patterns under physiological and pathophysiological conditions. It has been reported that the expression levels of certain miRNAs, mainly those related to neurogenic processes, are dysregulated in oncogenic events, thus favouring gliomagenesis and greater tumour aggressiveness. This review discusses some of the most important miRNAs involved in subventricular neurogenic processes and their association with glioblastoma aggressiveness.
ConclusionsMiRNA regulation and function play an important role in the development and progression of glioblastoma; understanding the alterations of certain miRNAs involved in both differentiation and neural and glial maturation could help us to better understand the malignant characteristics of glioblastoma.
Los gliomas son un grupo heterogéneo de tumores primarios del sistema nervioso central (SNC), que se distinguen según su linaje celular (astrocitos, microglía, oligodendrocitos)1. De este grupo de tumores del SNC, el glioblastoma multiforme (GBM) es el tumor con el pronóstico más desfavorable, con una expectativa media de vida de aproximadamente 15 meses posterior a su diagnóstico2. Actualmente, el GBM es considerado un importante problema de salud pública, debido a su desalentador pronóstico y baja tasa de supervivencia3. A pesar de los avances en el uso de marcadores moleculares, como la hipermetilación de la metil guanina metil transferasa y la mutación o estado silvestre de la isocitrato deshidrogenasa 1 (IDH1), actualmente no se cuenta con marcadores específicos para el diagnóstico oportuno del GBM.
En los últimos años se han realizado diversos estudios en la búsqueda de biomarcadores para mejorar el diagnóstico y el pronóstico del GBM4. Sin embargo, los patrones moleculares que determinan la agresividad del tumor no han sido del todo esclarecidos, por lo que es esencial la exploración de nuevos factores asociados a este padecimiento. Como lo señalado en el estudio realizado en el 2014 por Shuang et al., donde se enfatiza el papel que desempeñan los microARN (miARN) en la regulación de la biogénesis del GBM5. El estudio de los miARN en distintos tipos de cáncer los han colocado como importantes reguladores de vías de señalización relacionadas con el control del ciclo celular, invasión, tumorogénesis, resistencia farmacológica, entre otras6-8. Asimismo, cabe señalar que la expresión de múltiples miARN con frecuencia se encuentran alterados en el GBM9.
Por ello, el estudio de la desregulación de los perfiles de miARN resulta relevante, ya que podría representar el desarrollo de nuevas estrategias terapéuticas en la medicina personalizada, especialmente del GBM asociado a la zona subventricular (SVZ), un área neurogénica en la que se ha demostrado que los tumores asociados a esta zona presentan propiedades que favorecen la agresividad tumoral, como una mayor capacidad proliferativa, invasividad y recurrencia10; estas características podrían estar fuertemente relacionadas con la expresión de ciertos miARN asociados a la neurogénesis derivada de la SVZ.
En el presente trabajo se aborda la función de aquellos miARN implicados en la neurogénesis de la SVZ y que, a su vez, se encuentran desregulados en el GBM, encontrándose fuertemente asociados con el grado de malignidad.
Una de las características más representativas de los GBM es su composición celular heterogénea, desde células poco diferenciadas como células troncales neurales (CTN), células progenitoras neurales (PN) y progenitoras gliales (PG), hasta células gliales bien diferenciadas, como astrocitos, microglía, oligodendrocitos e inclusive neuronas11,12; esta composición de células le confiere al GBM propiedades pleomórficas, migratorias, proliferativas e infiltrantes13.
Clasificación histopatológica y molecular de GBM glioblastoma multiformeBasado en la conformación celular del GBM, estos se clasifican clínicamente en GBM primario o GBM secundario. Alternativamente, algunos estudios han evaluado la correlación del tipo de GBM con alteraciones moleculares, en el caso del GBM primario se destacan la mutación y sobreexpresión de los genes EGFR y MDM2, además de la deleción de p16 y la pérdida de la heterocigosidad del cromosoma 10q, para el caso del GBM secundario se destacan la sobreexpresión de PDGFA/PDGFRa, la pérdida de heterocigosidad del cromosoma 19q y las mutaciones en los genes de IDH1/2 y PT533. Adicionalmente, se ha realizado una nueva subclasificación de GBM, mediante metaanálisis de datos del «The Cancer Genome Atlas», la cual, los subdivide en 4subtipos diferentes, denominados: proneural, neural, mesenquimal y clásico; asociados con la alteración de los genes PDGFR, IDH1/2, NF1 y EGFR, respectivamente14. Recientemente, con base en estudios previos, la Organización Mundial de la Salud generó una nueva clasificación del GBM, en GBM IDH/silvestre y GBM IDH/mutante, combinando marcadores histológicos y moleculares como el estado de la IDH1 (silvestre o mutante)15.
Sin embargo, esta clasificación no puede determinar el comportamiento ni el pronóstico del GBM, por lo que, adicionalmente a estos marcadores moleculares, actualmente se investigan los mecanismos de la regulación epigenética de diversos miARN implicados en procesos biológicos como gliomagénesis, renovación y proliferación celular, invasión y farmacorresistencia16. Además, se ha planteado que estos miARN desempeñan un papel importante en la biología tumoral17, regulando la expresión de múltiples blancos como oncogenes, genes supresores tumorales y factores de crecimiento como K-RAS, CDK-4/6, BCL2, NOTCH, p53, EGFR, VEGF, entre otros, implicados en múltiples funciones biológicas como el control del ciclo celular, la proliferación y diferenciación celular, la infiltración, la angiogénesis y la apoptosis16,18.
Localización tumoral: zona subventricularComo se mencionó anteriormente, la topografía tumoral resulta de gran importancia para el pronóstico del GBM19 puesto que refleja otro aspecto importante de la heterogeneidad tumoral. Debido a que la SVZ es una de las regiones donde se desarrolla la neurogénesis en adultos, y que posee un nicho estructurado, con una organización celular que incluye las CTN, PN, PG, células gliales y células vasculares, todas en constante e intrínseca comunicación dentro de este nicho celular. También se ha hipotetizado que este nicho neurogénico representa el origen y la expansión del GBM20, debido a la transformación maligna de las CTN y PN a células troncales de glioblastoma (CTG), implicadas en la génesis y propagación del GBM21 y a que una pequeña población del tumor presenta características propias de CTN, como multipotencia, autorrenovación y marcadores celulares tales como nestina, Sox2, bmi-1 o CD13322. Estas características apoyan la hipótesis de «tumor jerárquico», la cual establece que las células troncales cancerosas son las únicas con verdadera capacidad de autorrenovación y propiedades de diferenciación multipotentes23.
Diversos estudios indican que los tumores que contactan con la SVZ muestran un patrón de crecimiento tumoral distinto, mostrando una mayor progresión y un comportamiento clínico más agresivo10,20,24. Así, el desarrollo de GBM en la SVZ representa un factor potencial para un pronóstico desfavorable, con una mayor reincidencia e infiltración, así como un incremento en la resistencia farmacológica. Se ha hipotetizado que las CTN presentes en esta zona le confieren a los tumores estas características malignas20,25,26. Tal como se ha sugerido anteriormente, un factor determinante de la malignidad tumoral podría ser la regulación aberrante de ciertos miARN, puesto que modulan aspectos importantes del comportamiento y las capacidades malignas del GBM27. Por lo tanto, resulta de gran interés el estudio de estos componentes y su participación en la agresividad de los GBM localizados en la SVZ.
MicroARN: importancia y actividadLos miARN son secuencias cortas de ARN no codificante de aproximadamente 20 a 25 nucleótidos de longitud con la capacidad de modular la expresión postranscripcional mediante su unión a la región 3-UTR de ARN mensajeros (mARN) blanco, induciendo la degradación del mARN y, en la mayoría de los casos, la inhibición de la traducción proteica28. Por consecuencia, los miARN pueden modificar la fisiología de la célula, modulando procesos celulares tan importantes como proliferación, diferenciación y supervivencia29.
Debido a su extensa implicación en diversos procesos celulares, se ha estimado que los miARN regulan hasta un tercio de los genes humanos30, por lo que, indudablemente, están implicados en la regulación de la traducción de oncogenes y genes supresores tumorales31. Por ejemplo, se ha detectado la sobreexpresión de miR-26a en aproximadamente el 12% de los GBM, el cual reprime la expresión de PTEN y promueve la gliomagénesis32. También se ha descrito que miR-21, miR-17 y miR-221/222 se encuentran sobreexpresados en GBM, principalmente relacionados con funciones antiapoptóticas y procesos de proliferación celular16. Por otra parte, los miR-34a, miR-24 y miR-7 pueden inhibir el crecimiento tumoral8, mientras que los miR-195, miR-455-3p y miR-10a están implicados en la resistencia celular a temozolomida (TMZ) en GBM33.
Estos estudios resaltan la importancia que implica el estudio de los miARN en los procesos oncológicos, ya que, a pesar del progreso en el entendimiento de la regulación de las vías patogénicas del GBM, resulta relevante determinar los mecanismos epigenéticos regulados por miARN y cómo la alteración en la expresión de estos puede potenciar las características malignas del tumor (fig. 1), especialmente con relación en la posición anatómica del mismo, principalmente en la SVZ, la cual confiere propiedades que favorecen el crecimiento tumoral.
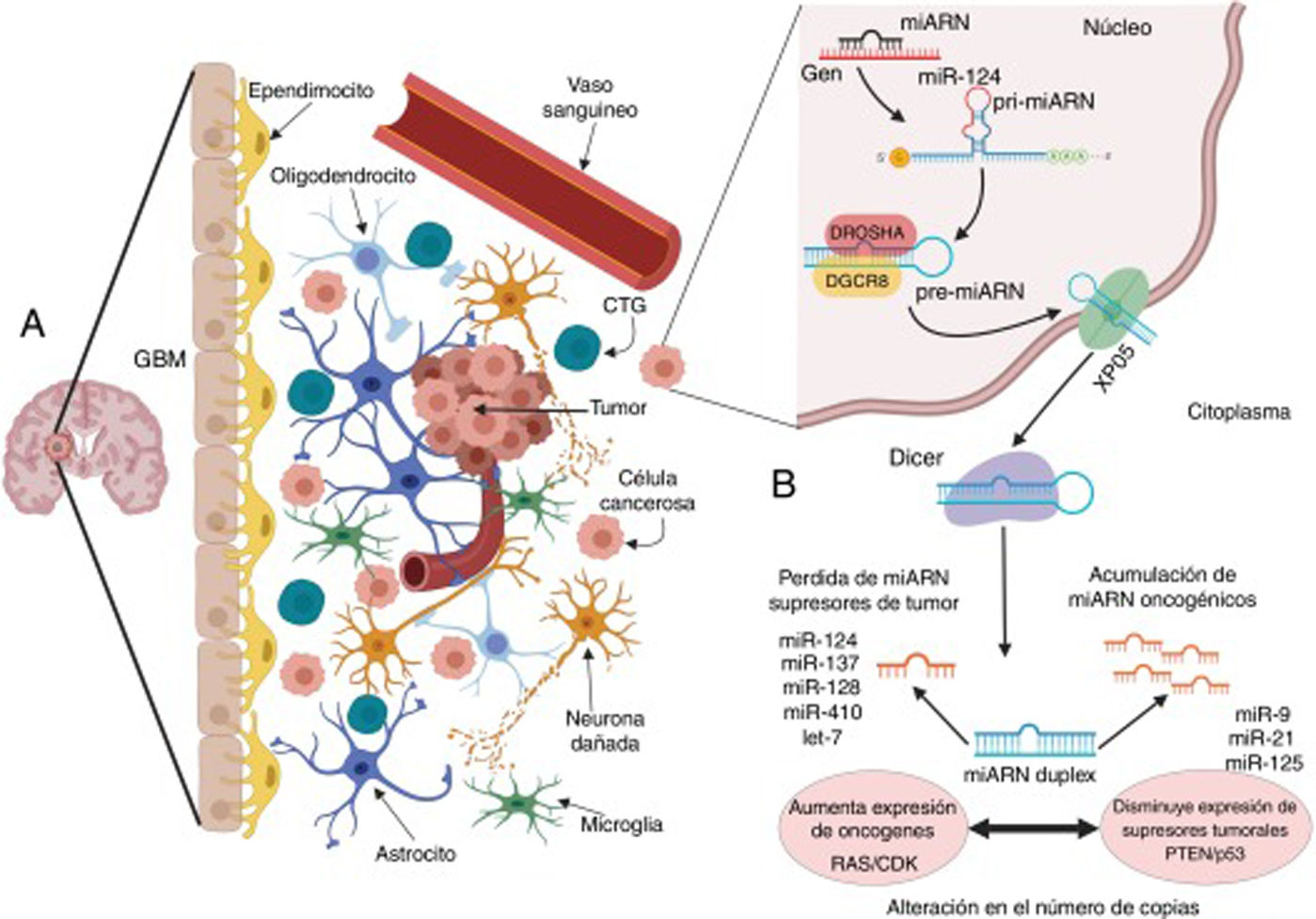
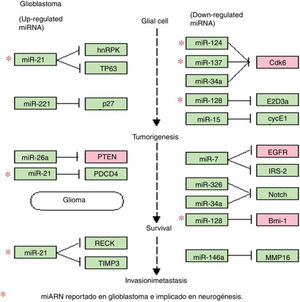
Microambiente del glioblastoma y alteraciones en la biogénesis de miARN que conducen a su crecimiento y propagación. A) El GBM es un tumor compuesto por diversos tipos de células; este nicho tumoral posee tanto células diferenciadas como indiferenciadas que contribuyen al inicio, el crecimiento y la expansión del tumor, a través de múltiples alteraciones en las células cancerosas, que modifican el entorno y a las células que las rodean para su beneficio; una de las alteraciones más importantes es la modificación y la transferencia de moléculas y material genético como los miARN. B) En las células cancerosas como el GBM, la biogénesis de los miARN se ve alterada por distintos procesos comúnmente desregulados por mutaciones y desórdenes en el procesamiento, la exportación y la maduración de los miARN; en el GBM estas modificaciones pueden alterar el número final de copias de miARN, propiciando la disminución o la pérdida de aquellos miARN supresores, como miR-124, miR-137, miR128, miR-410 y let-7, lo que se traduce en un amento en la expresión de oncogenes como los de las familias RAS y CDK, entre otros, o, por el contrario, se genera un aumento en el número de miARN oncogénicos como miR-9, miR-21 y miR-125, que actúan como silenciadores de supresores tumorales como PTEN y p53.
Gracias a múltiples análisis transcriptómicos realizados para el estudio de los miARN y de las CTG, se ha observado que estas células confieren a los tumores una extraordinaria agresividad, posiblemente atribuida a la desregulación de distintos miARN, los cuales confieren, entre otros aspectos, un incremento en la resistencia a la terapia convencional con TMZ34,35; no obstante, otros miARN que generalmente se encuentran suprimidos en GBM, como miR-124, miR-137, miR-128, miR-125b y miR-181a/b, sensibilizan las CTG, reduciendo su resistencia36,37. Por otro lado, también se ha reportado la relación de diversos miARN específicos a la topología cerebral como la SVZ, los cuales modulan funciones neurogénicas como la determinación celular, la regulación del desarrollo y la maduración de CTN y PN38.
En la siguiente sección se describen algunos de los miARN más ampliamente reportados y relevantes en neurogénesis, que a su vez están desregulados en GBM (tabla 1).
miARN asociados con neurogénesis en la SVZ y agresividad en GBM
| miARN | Función en SVZ | Referencias | Función en GBM | Expresión en GBM | Referencias |
|---|---|---|---|---|---|
| miR-124 | Diferenciación neural, proliferación de PN, neurogénesis | 39-43 | Promueve la diferenciación de CTG. Disminuye la troncalidad tumoral, la proliferación, la angiogénesis, la invasividad y la farmacorresistencia | Disminuido o ausente | 44-47 |
| miR-137 | Neurogénesis, diferenciación, maduración | 37,48, 49 | Promueve la diferenciación de CTG, arresto del ciclo celular y la apoptosisDisminuye la troncalidad tumoral, la proliferación, la migración y la invasividad | Disminuido | 37, 48, 50-54 |
| miR-9 | Diferenciación de CTN, proliferación, neurogénesis | 55-57 | Promueve la farmacorresistenciaDisminuye la angiogénesis, laproliferación y la invasividad | Incrementado | 58-62 |
| miR-128 | Desarrollo del SNC, maduración y migración neuronal, proliferación de PN | 63-65 | Promueve la apoptosisDisminuye la troncalidad de CTG, la proliferación, angiogénesis y la fármaco y radiorresistencia | Disminuido | 44, 66-69 |
| miR-21 | Desarrollo, mantenimiento y proliferación de CTN | 70-72 | Promueve la farmacorresistencia, la troncalidad y la proliferación de CTG y la angiogénesis e invasividadDisminuye la apoptosis | Incrementado | 73-78 |
| miR-410 | Diferenciación neuronal y astroglial de CTNMaduración y alteración de la morfología neuronal | 79-84 | Disminuye la proliferación, la angiogénesis, la invasividad y la farmacorresistencia de CTG | Disminuido | 85-87 |
| miR-125 | NeurogénesisDiferenciación y proliferación de CTN y PNNeurogénesis adulta, diferenciación y maduración neuronalRegulación de la función sináptica | 88-90 | Promueve la invasividad y la proliferaciónDisminuye o inhibe la apoptosisPromueve la diferenciación de CTGDisminuye la troncalidad y proliferación de CTG | Incrementado | 36, 91, 92 |
| Let-7 | Disminución de la troncalidad y proliferación de CTN y PNDiferenciación neuronal y astroglialMigración radial de PN | 93-97 | Disminuye la troncalidad de CTG, la proliferación e invasividad, la quimio y farmacorresistencia, induce arresto celular y disminuye el crecimiento tumoral | Disminuido | 8, 98-100 |
En la presente tabla se resume la función neurogénica normal y la función oncogénica en GBM de los 8miARN previamente descritos. También se muestra la expresión aberrante de estos miARN en el GBM, en su mayoría disminuidos (miR-124, miR-137, miR-128, miR-410 y let-7) y otros incrementados (miR-9, miR-21 y miR-125). Estas alteraciones en la regulación están mediadas por distintos componentes moleculares, lo que les confiere cualidades malignas.
CTG: célula troncal de GBM; CTN: célula troncal neural; PN: progenitor neural.
El miR-124 es uno de los miARN más estudiados y abundantes del cerebro39; este se expresa en distintas etapas del linaje de las CTN en la SVZ, como en las CTN, células de amplificación transitoria, neuroblastos y neuronas maduras40. El miR-124ha sido implicado en eventos neurogénicos, como la determinación de neural; se ha reportado que la disminución de miR-124 en las células progenitoras de la SVZ puede inhibir la diferenciación neuronal, mientras que la sobreexpresión conduce a la determinación neuronal41; regula la transición de células de amplificación transitoria a neuroblastos40, regulado por Sox9, PTBP1 y CDK642. Por otro lado, miR-124 regula negativamente Sox9, como consecuencia se reduce la proliferación de precursores y estimula la diferenciación neural, al promover la expresión de Tuj1 y de MAP237,43. También se ha reportado que miR-124 se encuentra disminuido o ausente en GBM, debido en parte a que los factores de crecimiento EGF, FGF y PDGF promueven su supresión44; miR-124 posee funciones supresoras tumorales y de diferenciación celular, por lo que puede inhibir la proliferación de CTG mediante la regulación de SOS145, reducir la angiogénesis y aumentar la quimiosensibilidad a TMZ regulando negativamente a oncogenes de la familia RAS46. Por último, cuando miR-124 se encuentra incrementado, puede disminuir la invasividad de CTG, así como promover la diferenciación de estas mediante el silenciamiento de SNAI2, un gen incrementado en glioma y que regula funciones de las células troncales47.
Otro de los miARN más estudiados y abundantes del cerebro es el miR-13748. Su regulación sobre diversos genes blanco ha sido relacionada con eventos neurogénicos de la SVZ, como la diferenciación neuronal de las CTN37; además, miR-137 desempeña un importante papel en la regulación de la maduración de neuronas jóvenes48,49. Por otro lado, miR-137 se encuentra disminuido en GBM, debido en parte, a que, bajo condiciones tumorales, este se encuentra hipermetilado48, aunado al hecho de que, al igual que miR-124, se ha reportado que los factores de crecimiento EGF, FGF y PDGF promueven su supresión37. También se ha demostrado que miR-137 actúa como un supresor tumoral, el cual puede reducir la troncalidad de las CTG, al disminuir su capacidad de autorrenovación e inducir su diferenciación mediante el silenciamiento de LSD1, que junto con TLX, regulan la autorrenovación y el estado indiferenciado de las CTN adultas50,51; esto, en parte, mediante la activación de la vía de Wnt/β-catenina52 y la represión transcripcional de los genes blanco de TLX, como p21 y PTEN, que estimulan la proliferación y autorrenovación de las CTN50. Además, miR-137 está implicado en la inhibición de la proliferación, la migración y la invasividad, así como en la inducción del arresto del ciclo celular en la fase G0/G1, mediante el silenciamiento de CDK637. De igual forma, el miR-137 se ha implicado en la diferenciación de las CTG y la inducción de apoptosis en GBM37. Adicionalmente, se identificó RTVP-1 como un gen blanco de miR-13753, regulando negativamente su expresión en GBM, puesto que RTVP-1 se ha relacionado con el incremento de la resistencia a la apoptosis54. Debido a su baja expresión es que se lo podría relacionar con el mantenimiento de las células troncales tumorales como las CTG, siendo así uno de los mayormente asociados a la diferenciación de células neuronales.
El miR-9 también ha sido reportado en diversos estudios relacionados a la neurogénesis; este es un miARN específico de tejido cerebral, tanto en etapas embrionarias como adultas55, resaltando su expresión en áreas neurogénicas en etapas tempranas, siendo ampliamente expresado en la SVZ56. Además, se han identificado diversos reguladores transcripcionales como blancos directos de miR-9, entre los que figuran: TLX, FoxG1 y Hes1, los cuales forman un complejo regulador de proliferación y diferenciación de CTN y PN57. Recientemente, se ha reportado que miR-9 se encuentra incrementado en GBM, actuando sobre la inhibición de la proliferación y la migración celular, así como en la disminución de la capacidad invasiva y de renovación58. Esta función antagonista tumoral, se asocia al silenciamiento de FOXP159, un oncogén que inhibe el crecimiento tumoral dependiente del EGFR y está incrementado en GBM60. Aunado a lo anterior, se ha reportado que la inhibición de miR-9 promueve la expresión de VEGF-A, lo que resulta en un incremento de la angiogénesis61. Sin embargo, existen reportes contradictorios que indican que miR-9 se encuentra aumentado en GBM resistente a TMZ, indicando que miR-9 induce una disminución de PTCH1, lo cual promueve la quimiorresistencia a TMZ en GBM, por lo que la expresión incrementada de miR-9 es asociada a un pronóstico adverso62.
El miR-128 es un miARN abundante en tejido cerebral; su expresión se ha relacionado con el desarrollo del SNC, contribuyendo en la diversidad y plasticidad celular, así como en la maduración de células neuronales63. Asimismo, miR-128 regula la proliferación de PN dentro de la SVZ64 y junto a la proteína PHF6 controla la migración de las neuronas a través de la corteza cerebral65. Cabe destacar que el miR-128 es uno de los miARN más disminuidos en GBM44; esta disminución de miR-128 se ha relacionado con el potencial maligno de GBM, ya que actúa como supresor tumoral, al disminuir la capacidad troncal de las CTG, a través de la inhibición del regulador epigenético PRC y el factor de renovación BMI1, los cuales actúan como oncogenes en GBM, potenciando las capacidades de proliferación, mantenimiento, resistencia y renovación de las CTN como las CTG66; también se ha demostrado que miR-128 promueve la sensibilización de las CTG a la quimio y radioterapia, mediante la regulación negativa de BMI167. Por otro lado, existen reportes que indican el incremento de la expresión de miR-128 en GBM promueve una disminución en su capacidad de crecimiento, renovación e invasión celular, además de mostrar efectos antiproliferativos, antiangiogénicos y proapoptóticos, esto mediante la represión de factores de crecimiento como RTK, EGF y PDGFRa68,69. Debido a lo anteriormente reportado, es que se ha sugerido que la pérdida de expresión de miR-128 corresponde a un evento temprano de la gliomagénesis66, resultando en la disminución de la diferenciación y el aumento en la capacidad de renovación y mantenimiento de las CTG, resultando en tumores más agresivos.
El miR-21 es expresado durante el desarrollo cerebral embrionario y neonatal; además, es altamente expresado en áreas con gran cantidad de células PN y PG como la SVZ70. El miR-21 coexpresa con SOX2, el cual se ha demostrado que participa en la proliferación y el mantenimiento de las CTN, lo que sugiere que miR-21 tiene funciones similares71. Además, miR-21 promueve la señalización de neurotrofinas como Ngf, que participan en el control de la diferenciación neuronal, regulando así el desarrollo, la supervivencia, la sinapsis y la plasticidad neuronal72. El miR-21 se encuentra sobreexpresado en la mayoría de los gliomas malignos y es asociado a un mal pronóstico. Se ha reportado que la sobreexpresión de miR-21 está asociada a la angiogénesis, la apoptosis, la proliferación y la invasión tumoral73, ya que, al expresarse, favorece el aumento en la expresión de AKT y ERK, lo que regula positivamente la expresión de HIF-1a y VEGF, resultando en la promoción de la angiogénesis tumoral74. Por otra parte, la inhibición de miR-21 en cultivos de GBM resulta en la activación de las caspasa-3 y caspasa-7, lo que conduce al aumento de muerte celular por apoptosis, además del aumento en la expresión de Bcl-2 y BAX; esto sugiere que la expresión incrementada de miR-21 actúa como un factor antiapoptótico, que promueve la adquisición de quimiorresistencia75,76. También se han identificado algunos supresores tumorales como PDCD4, TPM1 y PTEN como blancos silenciados por miR-2177, lo cual desencadena un incremento en la proliferación y la invasión. Además, el grupo de Hermansen et al., en 2016, ha sugerido que la sobreexpresión miR-21 comienza en las primeras etapas de desarrollo del tumor, confiriéndole así a las CTG capacidad de autorrenovación y proliferación aumentadas78.
El miR-410 es otro importante miARN; este es ampliamente encontrado en células del SNC, donde fue inicialmente descrito y restringido al SNC en desarrollo79. miR-410 forma parte del grupo miR-379-410, implicados en diversos procesos del desarrollo neurológico, actuando como reguladores de la diferenciación, proliferación y migración neuronal80,81. Específicamente, miR-410 se expresa en la SVZ; además, transitoriamente es expresado en células troncales mesenquimales. También se sabe que miR-410 es regulado negativamente por el factor de transcripción Noggin, el cual es sintetizado por células ependimarias y que antagoniza con BMP-4, que es sintetizado por astrocitos, permitiendo que las nuevas células se determinen hacia el linaje neural y no hacia el glial82. Se ha observado que cuando miR-410 se encuentra disminuido, se promueve la diferenciación neuronal y disminuye la diferenciación astroglial de las CTN; a su vez, se observó que este miARN también participa en el proceso de maduración y alteración de la morfología neuronal83,84. Por otro lado, miR-410 se encuentra muy disminuido en GBM en comparación con gliomas de grado bajo de malignidad y el cerebro normal85. Trabajos previos del grupo de Chen et al., en 2012, demostraron que la expresión positiva de miR-410 puede inhibir significativamente la proliferación y reducir la capacidad invasiva en células de glioma, actuando como un supresor tumoral, mientras al inhibir miR-410 el efecto era el contrario. De igual modo, demostraron que sus niveles de expresión están inversamente regulados por los niveles de expresión de MET, el cual es un blanco de miR-41086. Cabe destacar que la proteína MET ha sido asociada a GBM mesenquimal y cáncer metastásico, así como a niveles de expresión elevados en diversos tumores, propiciando la evasión de la apoptosis, el aumento de la proliferación celular, la invasividad, la angiogénesis y la farmacorresistencia, proporcionando una vía de rescate para que los tumores reaparezcan de una forma más agresiva87.
Otro mi-ARN que se encuentra ampliamente expresado en tejido cerebral normal es el miR-125b; este se encuentra sobreexpresado en CTN y neuroblastos de la SVZ durante la neurogénesis, promoviendo su proliferación y diferenciación88; es también un indicador de neurogénesis adulta y maduración neuronal, ya que es expresado por neuronas adultas89. Este miARN regula el linaje celular de las CTN, al promover la diferenciación neuronal y astroglial; a su vez, regula parte de la función sináptica, pues se ha visto que la mayoría de sus genes blanco están relacionados con esta función90. El papel de la familia de miR-125 en GBM resulta algo controversial, ya que algunos estudios han indicado que miR-125b se encuentra incrementado en gliomas36 y es relacionado directamente con el grado de malignidad y la supervivencia general, pues miR-125 actúa como un oncogén, reprimiendo a supresores tumorales y vías de señalización apoptóticas como p53 y p38MAPK, promoviendo así la invasividad y la proliferación, e inhibiendo la apoptosis en células de GBM91. Contradictoriamente, otros estudios han informado que miR-125a se encuentra disminuido en GBM y actúa como supresor tumoral, promoviendo la diferenciación y reduciendo la proliferación de las CTG mediante la regulación negativa del mediador TAZ y sus oncogenes diana, como CTGF y survivina, implicados en la iniciación y progresión tumoral92.
Finalmente, let-7 es un miARN muy abundante en CTN y las neuronas inmaduras93, participando activamente en distintos procesos como la migración radial de neuroblastos94 y en la regulación del destino celular en CTN y PN, desempeñando un papel clave en la decisión del desarrollo neural o glial95. Cuando let-7 se encuentra sobreexpresado, induce una disminución de la autorrenovación y proliferación de CTN, promoviendo así la diferenciación neuronal y astroglial, mientras que la disminución de let-7 refleja el efecto contrario96. Dicha disminución o inhibición de let-7 puede ser regulada por Lin28, una proteína de unión a ARN, promoviendo que los genes blanco de let-7, como c-Myc, Hmga2, RAS, Ciclina D1 y TLX, puedan ser mayormente expresados; dichos genes están implicados positivamente en la proliferación de CTN y PN97. Por otra parte, let-7 también ha sido señalado como un supresor tumoral en gliomas, encontrándose generalmente disminuido en GBM8; let-7 es capaz de reducir la agresividad de GBM, ya que se ha reportado que posee capacidades antiproliferativas y antimigratorias, mediante el silenciamiento de oncogenes como MYC, N-RAS y K-RAS, implicados en la proliferación, invasión y supervivencia del GBM98. La baja expresión de miembros de la familia let-7 en diversos tipos de cáncer puede deberse a la activación de Lin28A y Lin28B; esto se ha asociado a una respuesta deficiente a la quimio y radioterapia99, por lo que el aumento en la expresión de let-7 en células de GBM podría sensibilizarlas a las terapias dirigidas. Adicionalmente, Song et al., en el 2016, indicaron que la sobreexpresión de Let-7b inhibió el crecimiento tumoral y la troncalidad de las CTG in vitro100.
Herramientas bioinformáticas para análisis de miARNEl amplio uso de predicciones computacionales ha sido muy útil en la identificación de los blancos de miARN, ya que esta es la acción más importante para comprender sus funciones regulatorias101. Actualmente, se han desarrollado diversos programas y algoritmos que permiten estudios multivariados a gran escala de los miARN como la identificación de miARN, sus niveles de expresión, la predicción de sus precursores y blancos, la visualización de sus funciones, la creación de redes transcripcionales de miARN, entre otras102,103.
El uso de herramientas bioinformáticas como «miRNet» permite vincular fácilmente los miARN a sus blancos y vías de señalización para asociarlos con funciones celulares más concretas104, puesto que, incluso cuando se han identificado los transcritos blancos para un miARN específico, no es sencillo comprender o visualizar sus implicaciones, además del hecho de que los miARN pueden tener cientos de blancos potenciales distintos y, por lo tanto, pueden afectar una gran variedad de procesos.
Por su parte, la generación y el uso de distintas bases de datos especializadas, apoyan el estudio y la caracterización de sistemas biológicos, como la llamada «miRBase», la cual tiene registro de más de 2.600 secuencias maduras de miARN en humanos105, de las cuales, en la base de datos de «miRTarBase»106, se han reportado al menos 118 miARN distintos junto con sus genes blanco para GBM.
En los últimos años, se ha generado una gran cantidad de información sobre nuevos biomarcadores de pronóstico y diagnóstico basados en miARN y sus blancos funcionales, tanto en procesos celulares normales como en el desarrollo de enfermedades107. Sin embargo, los mecanismos de regulación de estos miARN en conjunto con sus genes blancos y las implicaciones fisiológicas de estos no están bien esclarecidas102, por lo que el uso de bases de datos en conjunto con herramientas bioinformáticas podría facilitar los procesos de compilación y análisis a gran escala, proporcionando la información necesaria para construir redes de interacciones miARN-blanco y comprender mejor estos mecanismos y procesos. En el presente artículo se utilizó la herramienta online miRNet104,108 en conjunto con las bases de datos Kyoto Encyclopedia of Genes and Genomes (KEGG)109-111 y miRBase112-115, además de artículos publicados, para construir mapas de interacción entre miARN y sus blancos, que sean expresados en la SVZ y estén desregulados en GBM, para poder asociarlos con la agresividad de GBM y la neurogénesis de la SVZ.
Análisis in silico de los marcadores de miARNEs importante identificar los genes blanco de los miARN y su interacción con las vías de señalización de distintos procesos celulares, para comprender las funciones de los miARN, para lo cual se realizó un análisis visual in silico utilizando la plataforma online «miRNet». Esta herramienta hace uso de la base de datos de miARN «miRBase», la base de datos del National Center for Biotechnology Information (NCBI) para los genes blanco de los miARN y la base de datos KEGG para visualizar las vías de señalización. Se realizó la selección de los 8miARN descritos anteriormente para generar un mapeo o red de interacciones entre los miARN con sus respectivos genes blanco, con esto se pudo visualizar las vías de señalización potencialmente alteradas en GBM por el efecto de estos 8miARN. El análisis fue dividido en 2partes; la primera muestra los 4miARN más ampliamente reportados en glioblastoma, mediante un diagrama de interacción realizado con la herramienta online KEGG Mapper, perteneciente a la plataforma de KEGG (fig. 2). Posteriormente, se hizo el análisis de interacción con los 8miARN descritos; para ello se hizo uso de la plataforma online miRNet, con los 8miARN y se generó un mapa de interacción miARN-gen que muestra todos aquellos genes que son regulados por estos miARN en el GBM (fig. 3).
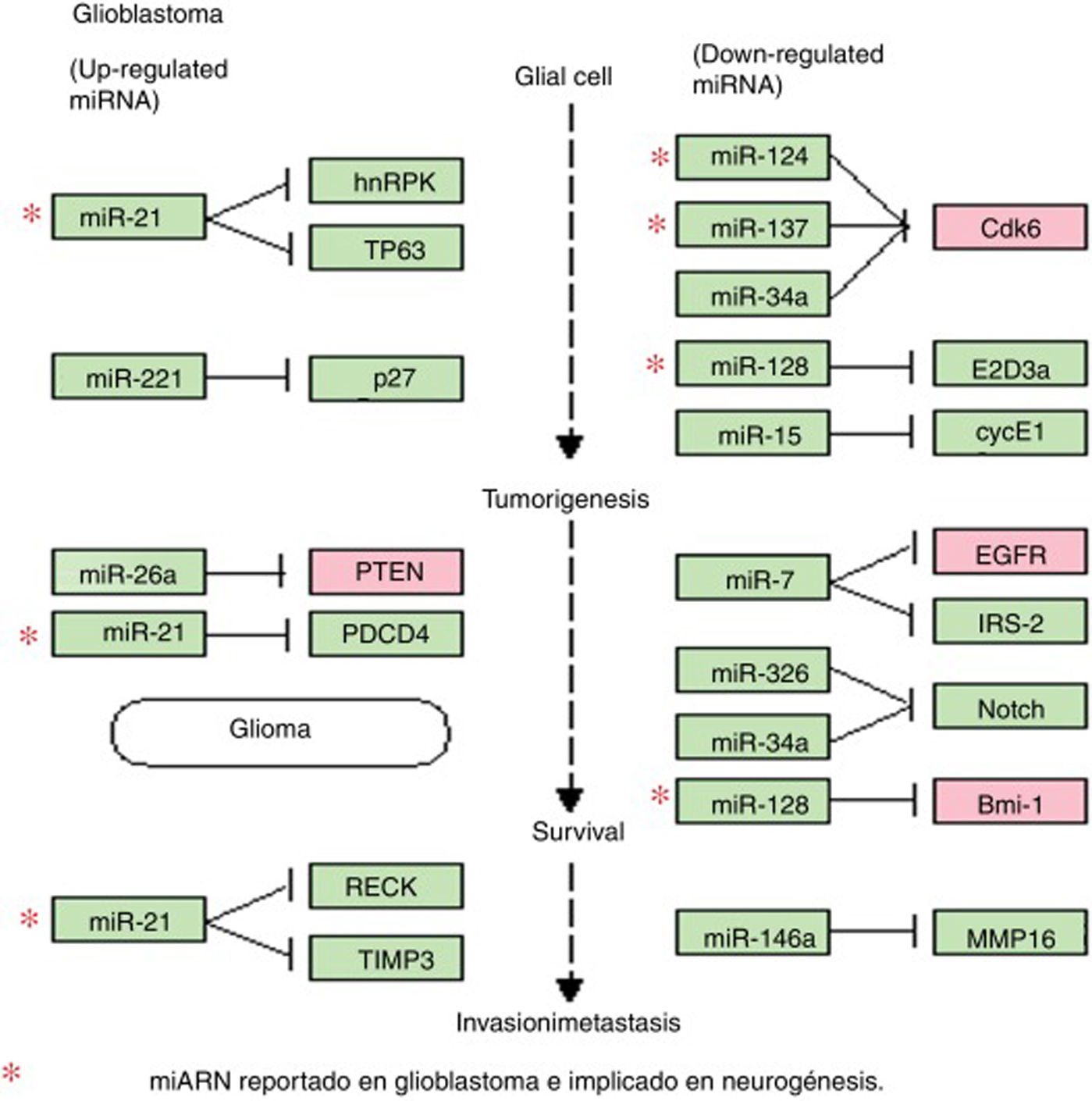
Diagrama de la regulación de miARN y sus genes blanco en glioblastoma. El diagrama de miARN de glioblastoma, obtenido en la plataforma en línea de KEGG, muestra los miARN desregulados más ampliamente observados en el glioblastoma, así como sus principales genes blanco. En el diagrama se incluyen 4de los 8miARN previamente descritos (miR-21, miR-124, miR-137 y miR-128), todos implicados en distintos procesos de la gliomagénesis y la agresividad del GBM. Este diagrama evidencia la importancia del miR-21 (incrementado en GBM), potenciando aspectos como la inhibición de la apoptosis y el aumento de la resistencia farmacológica, así como el mantenimiento y proliferación de las CTG. Por otro lado, se destaca el papel de los miR-124, miR-137 y miR-128 (disminuidos en GBM), los cuales actúan sobre procesos de diferenciación celular, inducción de la apoptosis, así como el arresto del ciclo celular y la inducción de quimio-sensibilidad.
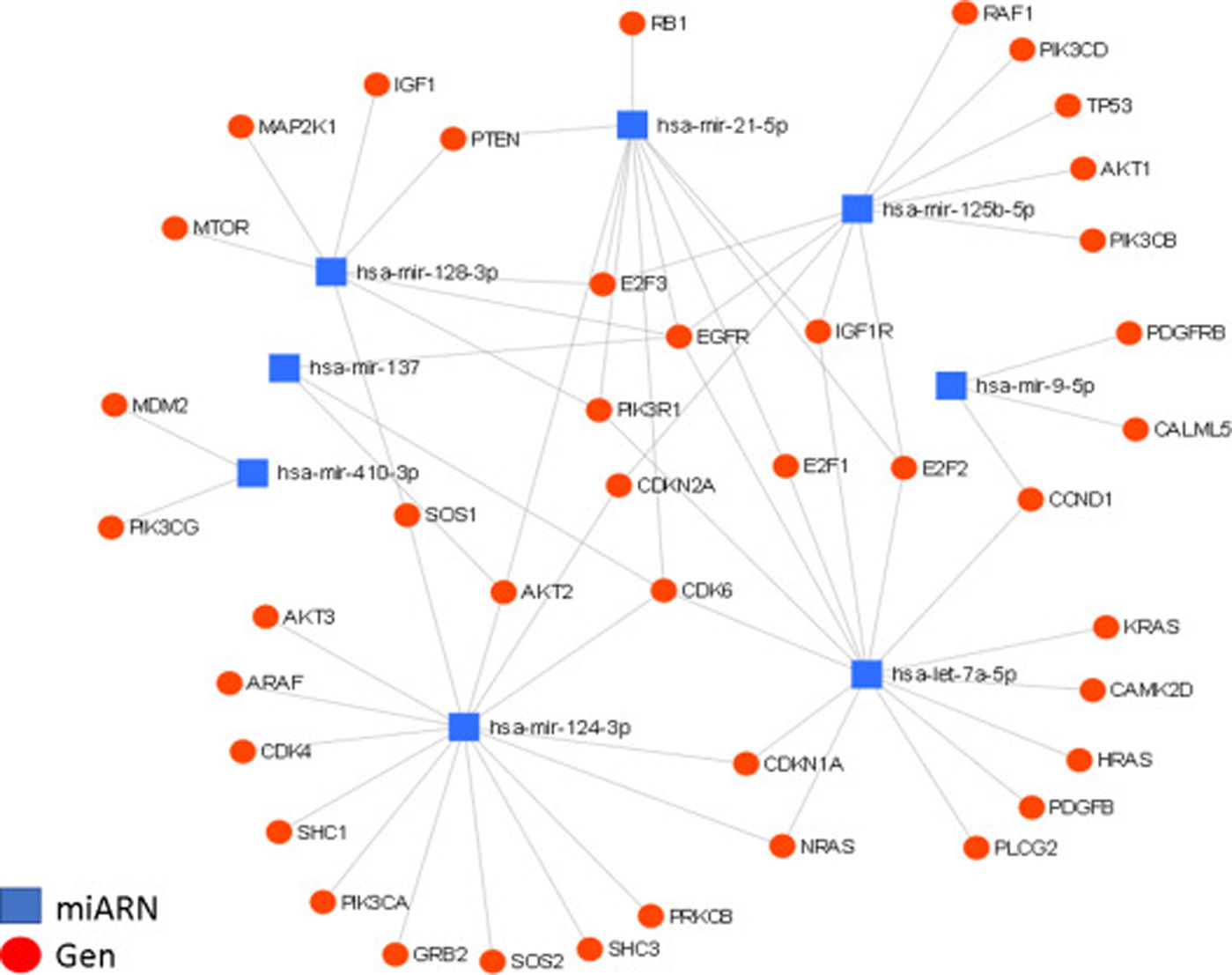
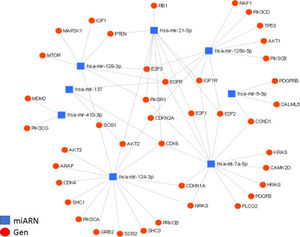
Interacción de los miARN con sus genes blanco. Diagrama de interacción de miARN y su regulación de genes blanco, generado mediante la plataforma online miRNet; en él se muestran las interacciones entre los 8miARN descritos y sus genes blanco. Se presentan un total de 36 genes blanco para los 8miARN en glioma, todos agrupados en nodos de interacción miARN-gen, con un valor de afinidad de 8.31e-11.
Destacamos la regulación de los genes EGFR y CDK6. En el caso del gen EGFR, se encuentra regulado por al menos 5de los miARN aquí descritos (miR-21, miR-125, miR-137, miR-128 y let-7); este factor de crecimiento se encuentra generalmente sobreexpresado en GBM y es, en parte, responsable del aumento en la proliferación celular, así como de la evasión de la apoptosis. En el caso del gen CDK6, está siendo regulado por 4miARN (miR-124, miR-137, miR-21 y let-7); este gen también se encuentra incrementado en GBM y es asociado a un aumento en la proliferación, la migración y la invasividad tumoral.
En el diagrama de regulación de los miARN se evidencia la importancia de algunos de los miARN previamente descritos, como el miR-21 (incrementado en GBM), relacionado con aspectos malignos del GMB; asimismo, se destaca la regulación de negativa de otros miARN, como miR-124, miR-137 y miR-128 (disminuidos en GBM), los cuales actúan como supresores tumores que, al encontrarse disminuidos o ausentes, ven fuertemente menguadas sus capacidades antitumorales, resultando en el aumento del potencial maligno en el GBM. Por otro lado, observamos que la regulación de algunos genes es mediada por múltiples miARN, en donde destacamos la regulación de los genes EGFR y CDK6, los cuales están regulados, respectivamente, por 5y 4de los miARN aquí descritos. Ambos genes se encuentran sobreexpresados en GBM y son fuertemente implicados en procesos malignos, actuando directamente sobre la progresión tumoral, ya que ambos se relacionan y potencian eventos oncogénicos como la génesis, el crecimiento, la proliferación, la evasión de la apoptosis y la quimiorresistencia en el GBM.
Aplicaciones terapéuticas de los miARN en GBM glioblastoma multiformeLa capacidad de los miARN para interactuar con diferentes objetivos y vías de señalización los colocan como importantes blancos terapéuticos, teniendo 2enfoques distintos, ya sea reducir o estimular su expresión con distintas estrategias debido a la capacidad de estos para actuar como oncogenes o supresores tumorales, lo que ha derivado en el desarrollo de terapias basadas o dirigidas contra miARN en el tratamiento del GBM8,116. Esto puede ser posible gracias a la relativa facilidad actual para obtener la información genómica y un perfil de miARN personalizado, debido al creciente avance y uso de microarreglos y de herramientas computacionales y bioinformáticas117,118. Así, el uso de un perfil basado en la expresión de los miARN propios proporcionaría la base clínica para un mejor diagnóstico, pudiendo discernir entre tumores que compartan el mismo grado histopatológico, un pronóstico más acertado de la supervivencia general y la estimación de respuesta al tratamiento para cada paciente119.
ConclusionesEl GBM es un importante problema de salud médica y social a nivel mundial, pues es el tumor más común y con el peor pronóstico del SNC, cuya etiología no ha sido del todo dilucidada. Se ha descrito que el GBM puede surgir a partir de mutaciones en células troncales o derivados de tumores de menor malignidad como los astrocitarios. Pero aun cuando la etiología puede ser multifactorial, uno de los principales aspectos a considerar para su evolución y pronóstico es su localización, destacando la principal región neurogénica, rica en células troncales neurales con capacidad multipotente y proliferativa, llamada SVZ, donde el desarrollo tumoral en esta región ha sido relacionado a una mayor agresividad.
A pesar de los avances significativos en el estudio de esta enfermedad, actualmente se conocen solo algunos de los factores moleculares asociados a esta patología, destacando principalmente a los miARN, los cuales han sido relacionados a múltiples procesos, tanto fisiológicos como patológicos. En el caso del GBM, se ha visto que dichos miARN muestran una desregulación en sus perfiles de expresión, confiriéndoles capacidades que influyen tanto en el origen como en el desarrollo del tumor, lo cual los hace candidatos interesantes en el estudio del procesamiento patológico de dicha enfermedad.
Aquí, mediante una revisión bibliográfica y el uso de bases de datos, se identificó la relación que guardan los distintos miARN con el desarrollo del GBM. Donde destacamos que entre los miARN neurogénicos más importantes se encuentran: miR-124, miR-137, miR-9, miR-128, miR-21, miR-410, miR-125 y let-7; todos ellos cumplen diversas funciones en células troncales de la SVZ, actuando sobre el mantenimiento, la diferenciación y el destino de las CTN, así como la proliferación y migración de PN y PG, entre otras.
Es evidente la importancia de los miARN en un nicho neurogénico como lo es la SVZ, así como sus implicaciones en los eventos de diferenciación y maduración neural y glial; así también, la importancia de las alteraciones de estos miARN en el cáncer, actuando sobre el desarrollo y la progresión del GBM, como el mantenimiento de las CTG, la inhibición de la diferenciación neural y glial, así como una mayor proliferación, migración, angiogénesis y resistencia farmacológica, todas cualidades representadas en tumores asociados a la SVZ. Por lo tanto, una mayor comprensión de las alteraciones en la función y regulación de estos miARN podría esclarecer el campo de las posibles causas de la agresividad mostrada por los GBM vinculados a la SVZ, a la vez que proporcionarían importantes indicadores de diagnóstico y pronóstico para los pacientes con GBM.
Revisión bibliográficaSe realizó la búsqueda de revisión bibliográfica en la plataforma de NCBI y el buscador de PubMed —utilizando los términos: microRNA, miRNA, neurogenesis, differentiation, glioma, glioblastoma, bioinformatics, therapeutic—, donde se eligieron los artículos a revisar y se resumieron datos mostrados.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Agradecemos al Consejo de Ciencia y Tecnología (CONACyT) de México, por el apoyo de la beca de maestría (N.° 488270), a L.J. Reséndiz-Castillo y la beca de doctorado (N.° 487713) a E.E. Reza-Zaldivar.