Suele aceptarse que las infecciones viral de vías respiratorias altas (IvVRA) tienen una relación causal con el desarrollo de nuevos episodios de otitis media1-3 (OM). Sin embargo, hay desacuerdos sobre la importancia relativa de las diferentes especies de virus que ocasionan esta complicación, y también sobre la verdadera tasa de complicaciones y el porcentaje relativo de todos los episodios de OM que son una complicación de las Iv-2,4-8. La resolución de estas cuestiones es importante si tenemos en cuenta la opinión muy compartida de que puede evitarse la OM si se reduce la carga de IvV-RA y/o se separa la OM como forma de presentación9-11.
Típicamente, la presencia de una IvVRA se reconoce basándose en una serie de síntomas y signos auto-apre-ciados y/o secundariamente asignados que definen a una enfermedad de tipo resfriado12 (ETR). Debido a que las IvVRA sin expresión de síntomas y signos no aparecen clínicamente, las valoraciones de coincidencia para las diferentes complicaciones de las IvVRA se refieren habitualmente a las ETR. Para la OM, las estimaciones anteriores sugerían que aproximadamente el 20-40% de las ETR en niños se complican por una OM, y que cerca del 50% de todos los nuevos episodios de OM son atribuibles a las ETR3,8,13. Estudios recientes muestran que estas estimaciones se elevan al incrementar la densidad de las valoraciones otológicas14 (es decir, al captar más fenómenos de OM). Además, la infección experimental de adultos con diferentes virus de vías respiratorias altas muestra que la mitad de quienes se considera infectados por eliminación y/o seroconversión no expresan signos o síntomas de ETR15-19. En un análisis de estos datos, la frecuencia de las complicaciones otológicas en los sujetos no enfermos fue significativa18, y otros estudios han mostrado que los anticuerpos preexistentes y el tratamiento antiviral influían de modo diferente en las ETR y las complicaciones otológicas20,21. Así mismo, la presencia de virus en la nariz y nasofaringe sin una ETR concomitante se documentó en poblaciones de control de lactantes, niños y adultos22-25, aunque no se valoró el significado de esta observación en la presentación de complicaciones como la OM.
El objetivo del presente estudio consistió en determinar si las IvVRA subclínicas se complican por una OM en un grupo de niños de corta edad controlados con un diario para registrar los signos de enfermedad, con exploraciones mediante otoscopio neumático realizadas por personal del estudio y con recogida periódica de secreciones nasales para estudio de virus por PCR. Las hipótesis nulas investigadas son que la tasa de complicaciones de OM para una IvVRA estimada por la presencia de ETR constituye una estimación precisa de la verdadera tasa de complicaciones para las IvVRA, y que los porcentajes de coincidencia de la OM con los episodios totales de OM son similares cuando la coincidencia se define por las ETR y las IvVRA. La aceptación o el rechazo de estas hipótesis es importante para diseñar estrategias destinadas a prevenir la OM mediante interferencia en la adquisición de virus, eliminación de virus o presentación de la enfermedad durante una IvVRA, al aportar estimaciones de la máxima eficacia y eficiencia de las diferentes intervenciones propuestas11.
MATERIALES Y MÉTODOSLos datos para este informe se resumieron de los disponibles para el primer año de nuestro estudio de 5 años, que se halla en curso, titulado “Papel de los virus y susceptibilidad genética en la otitis media”. Mediante anuncios en la prensa, cada año se ofrece la participación a familias con 2 niños de 1 a 5 años. Los criterios de exclusión incluyen: afecciones médicas importantes en cualquiera de ellos; afecciones médicas que predispongan a la OM persistente; tímpanos no intactos o estructuralmente anormales; pérdida auditiva neurosensorial previa, o incapacidad para cooperar de un modo suficiente en los procedimientos y las pruebas de exploración. Después de manifestar su voluntad de participar y de otorgar por escrito un consentimiento informado, las familias ingresaron en el estudio en octubre y se controlaron hasta abril del año de estudio correspondiente. Las familias recibieron 100 dólares mensuales por su participación. El protocolo del estudio fue aprobado por las Institutional Review Boards de la University of Pittsburg y la University of Virginia.
Los datos para este estudio incluyen la información demográfica general de la familia, obtenida al ingresar, y los resultados recogidos en un diario, cumplimentado por la madre y donde se recoge la presencia o ausencia de 6 signos (rinorrea, congestión nasal, dolor de garganta, tos, fiebre e irritabilidad) y la presencia o ausencia de un resfriado o gripe, según los criterios diagnósticos usuales, en cada niño. Cada semana se realizaron valoraciones del estado del oído medio por otoscopia neumática, o bien en visitas domiciliarias (Pittsburg) o en la clínica del estudio (Charlottesville), por parte del personal de la investigación. Además, se recogieron periódicamente las secreciones nasales para la identificación de virus por PCR, en una visita domiciliaria o en la clínica del estudio. Los objetivos de la presente investigación consistieron en determinar la tasa de complicaciones de OM en una ETR y los porcentajes de aislamiento de un virus de vías respiratorias altas; y también, por el contrario, las frecuencias de todos los episodios de OM que son complicaciones de una ETR, o de aislamiento de un virus de vías respiratorias altas.
Para definir una ETR se utilizó un algoritmo desarrollado previamente que reduce el sesgo interobservadores en la asignación de un resfriado26. Específicamente, las diferentes madres utilizaron una amplia variedad de constelaciones de signos para asignar un resfriado o una gripe, y el algoritmo estandariza la definición de una ETR en las distintas familias. El algoritmo asigna primeramente un día de enfermedad si el informe materno para ese día incluye dos o más signos como rinorrea, congestión nasal y tos, o si el informe parental incluye la rinorrea o la congestión nasal si el día anterior se designó en el algoritmo como un día de enfermedad. Luego, los días de enfermedad/no enfermedad se unieron en tiras específicas en cada niño para observaciones secuenciales que comenzaron en octubre y se prolongaron hasta el 30 de abril; los elementos de la tira se codificaron como 0 (día sin enfermedad) o 1 (día con enfermedad). En las escasas ocasiones en que la secuencia de la tira quedó interrumpida por ausencia de valores, se introdujo un valor por defecto de 0, a menos que las observaciones inmediatas indicaran días de enfermedad, en cuyo caso se introdujo un valor de 1. Todos los valores de 0 se eliminaron de cada tira para obtener unas secuencias agrupadas de variable longitud de unos, que si duraban al menos 3 días se consideraban como ETR, cuyo comienzo se definió como la primera observación de un día de enfermedad, y su final, como el primer día sin enfermedad, en una secuencia de, al menos, 5 días consecutivos sin enfermedad.
Los datos de la otoscopia neumática bilateral realizada en cada exploración se clasificaron dicotómicamente en cada oído como OM ausente/presente, basándose para ello en las puntuaciones del tímpano, otorgadas por el otoscopista con arreglo a los parámetros de visibilidad, estado, posición, aspecto, color, vascularización, reflejo luminoso y movilidad14. Los datos se codificaron para los oídos izquierdo y derecho como OM ausen-te/presente (0 = ausente, 1 = presente), sin tener en cuenta la expresión concomitante de los signos y síntomas otológicos. En cada niño, los días de observación para los dos oídos se unieron en forma de tiras temporales; los valores inter-observaciones se asignaron según la observación inicial del intervalo. Un episodio de OM se definió como una serie secuencial de unos, limitada por ceros, en un determinado oído.
Se intentó recoger periódicamente muestras de la secreción nasal, a intervalos mensuales, y otras muestras suplementarias durante los períodos de resfriado “identificado por los padres” y/o de OM diagnosticada otoscópicamente. Sin embargo, ello fue imposible por la resistencia de algunos niños a la toma de muestras en ausencia de rinorrea libre (usualmente en períodos sin ETR), y porque en ciertos casos los padres no notificaron enseguida al personal del estudio un diagnóstico de resfriado, por la imposibilidad de que los padres acudieran para una visita durante un resfriado identificado por ellos (p. ej., días festivos, vacaciones) y por el número de resfriados identificados por los padres, mayor del esperado, que obligó por motivos de presupuesto a reducir el número de tomas de muestras durante los períodos sin ETR. Se recogieron en total 513 muestras, que se analizaron en busca de virus.
La técnica de recogida de las secreciones nasales y nasofaríngeas se ha descrito anteriormente25. En síntesis, en cada vestíbulo nasal se colocó secuencialmente un dispositivo de aspiración Yankauer (Conmed, Utica, NY) conectado a un frasco Lukens para recogida de muestras (Sherwood Medical, St. Louis, MO); se aplicó una presión negativa (–90 mmHg) mediante una bomba de aspiración. Si no había secreción visible que llegara al tubo y el sujeto lo toleraba, se instilaron 1-2 ml de suero salino no bacteriostático en las ventanas nasales, con aspiración en la ventana contralateral. La secreción visible en el tubo se lavó con solución salina para recogerla en el frasco.
Todas las muestras recogidas se congelaron a –70 °C y se enviaron por lotes a nuestro laboratorio de virología en la University of Virginia, para análisis por PCR para detectar adenovirus, coronavirus, virus gripales y paragripales, rinovirus (enterovirus) y virus respiratorio sincitial (VRS). Estos virus se investigaron siguiendo un protocolo adaptado del procedimiento comercial Hexaplex27 (Prodesse, Inc., Waukesha, WI). Se extrajo el ácido nucleico viral de 280 µl de la muestra de aspira-do/lavado, mediante el QIAmp® Viral RNA Mini-Kit (Qiagen, Valencia, CA). Se sintetizó el ADNc mediante hexámeros aleatorios y transcripción inversa. Se purificó el ácido nucleico viral y se transcribió inversamente al ADNc al descongelar las muestras originales por primera vez. La cantidad de ADNc fue impuesta por el número planificado de diferentes amplificaciones PCR, dado que la investigación de muestras originales recongeladas o del ARN purificado puede dar resultados falsos negativos25. La purificación del ADN adenoviral con esta técnica (incluida la transcripción inversa superflua) fue equivalente a la extracción por un método para ADN (QIAmp® DNA blood minikit; Qiagen) que se ha utilizado28.
La PCR se realizó con una mezcla patrón y unos parámetros de amplificación específicos para cada virus. Los cebadores no incorporados y los trifosfatos de desoxinucleósido se eliminaron de los productos PCR con el QIA Quick PCR Purification Kit (Qiagen). El producto amplificado se detectó con una sonda de oligonucleóticos específica para cada virus, marcada con peroxidasa del rábano picante, en placas recubiertas de avidina y con el amortiguador de hibridación incluido en el kit comercial Hexaplex (Prodesse). Después de la hibridación a 50 °C durante 1 hora, se lavaron las placas, se añadió el sustrato, se detuvo la reacción al cabo de 10 minutos y se midió la densidad óptica de cada pocillo a 450 nm en un espectrofotómetro. El valor límite positivo fue de 3 desviaciones estándar por encima de la media de los controles con agua.
PicornavirusSe han detallado anteriormente25 (Adapted Protocol) los ceba-dores, la composición de la mezcla patrón de PCR, los parámetros de amplificación y la sonda para la hibridación. La prueba RT-PCR con este método en diluciones logarítmicas de un acúmulo titulado de rinovirus tipo 39 demostró que se detectaba µ 1 dosis infecciosa de cultivo de tejidos50/0,1 ml de muestra.
CoronavirusLa misma mezcla patrón de PCR25 se utilizó para los corona-virus OC43 y 229E; los cebadores, la sonda y los parámetros de amplificación para una secuencia del gen M de los dos virus se utilizaron del modo descrito con anterioridad29. La sensibilidad del análisis para 229E fue de 0,01 DICT50/0,1 ml, con titulación en células MRC-5.
Virus paragripales 1-3, virus gripales A y BEl análisis para estos virus se efectuó con reactivos Hexaplex suministrados por el fabricante (Prodesse). La amplificación del ADNc con una mezcla patentada para PCR que contiene ceba-dores para los virus (Supermix, Prodesse) se realizó utilizando los parámetros cíclicos detallados por Prodesse. El valor límite positivo fue el de una densidad óptica µ 0,4 a 450 nm, de acuerdo con las instrucciones del fabricante.
VRSLos cebadores, la sonda y los parámetros cíclicos de PCR se basaron en los descritos previamente30. La sensibilidad del análisis, determinada por un acúmulo de VRS titulado en células MRC-5 (suministradas amablemente por el Dr. Ron Turner), fue de 0,28 dosis infecciosas de cultivo de tejidos50/280 µl.
AdenovirusLa prueba PCR para adenovirus se basa en la PCR genérica desarrollada por Echavarria et al para la detección de todas las cepas de adenovirus clínicamente relevantes28,31. La mezcla patrón para PCR contenía 0,8 µM de cada cebador (cebadores biotinilados Hex 3 y Hex 4) en amortiguador 1X AmpliTaq Gold con 2,7 mM de Cl2Mg y polimerasa AmpliTaq Gold (Applied Biosystems, Foster City, CA). Los parámetros de amplificación corresponden a los detallados en una publicación previa32. En el paso de hibridación se utilizó la sonda de oligonucleóticos Hex 30 marcada con peroxidasa del rábano picante. La sensibilidad de la PCR para adenovirus fue < 1 DICT50/0,1 µl, basada en la titulación de una cepa clínica de adenovirus no numerada en células A549 (suministradas amablemente por el Dr. Mike Ison).
Los resultados de los 513 análisis se expresaron en tiras, y el número de análisis descrito se redujo a 425 análisis independientes; para ello se unieron las detecciones de virus idénticos en un período de 20 días, pero sin observación intermitente de “no hay virus detectable” como detección de un virus (análisis), y por asignación de múltiples análisis a la misma ETR y/o tira de OM al virus detectado durante esa tira o a la ausencia de virus detectable.
En cada niño, las tiras de ETR, las tiras de virus y las tiras de OM izquierda y derecha se alinearon en el eje del tiempo. En primer lugar, las tiras de ETR se examinaron con respecto a su relación con los virus, y se clasificaron en 3 categorías: ETR asociadas a virus (los virus precedían inmediatamente o estaban incluidos en la tira), ETR sin virus (con observaciones negativas de virus incluidas), y ETR de etiología desconocida (tiras en las que no había muestras de virus asociadas temporalmente), y se procedió a contarlas. Las ETR con virus asociados se etiquetaron según la especie viral, y las ETR que incluían recuperaciones virales positivas y negativas se etiquetaron según el virus recuperado.
Los episodios de OM (uni o bilaterales) se clasificaron en 4 grupos: OM como complicación de una ETR (episodios incluidos o inmediatamente posteriores a la ETR con asignación de etiología primaria al virus asociado); OM como complicación de una infección viral, pero sin que se demostrara una ETR coexistente (episodio con una muestra positiva a virus durante el período existente de OM); OM independiente de una ETR/etiología viral (OM no asociada con una ETR y en la que los análisis para virus incluidos fueron negativos), y OM de etiología desconocida (episodio sin una ETR coexistente y sin toma de muestras para virus durante todo el episodio).
A efectos de uniformidad, el formato resumido para la presentación de los datos fue siempre el de la media ± desviación estándar.
RESULTADOSEn el año 1 participaron en el estudio 31 familias, 7 de las cuales lo abandonaron antes de finalizarlo. Los motivos del abandono fueron: las obligaciones impuestas eran excesivas para la familia (n = 5), o la familia cambió de domicilio (n = 2). Por lo tanto, los resultados se refieren a los datos disponibles para 60 niños (3 de raza negra, 57 de raza blanca) de 4,4 ± 2,0 años (1,5-9,3 años) en las 24 familias que completaron el estudio: 2 niños en 13 familias, 3 en 10 familias y 4 en una. Durante el período de estudio, desde el 1 de octubre hasta el 30 de abril, se recogieron 513 muestras nasales y se analizaron por PCR, con un promedio de 9,6 ± 2,5 (límites 2 a 16) muestras por niño (425 análisis independientes, v. Métodos). Se realizaron 1.513 otoscopias neumáticas bilaterales, con un promedio de 25,2 ± 4,0 (límites 9 a 32) exploraciones bilaterales por niño. Los diarios parentales se cumplimentaron para 12.296 días-niño, con un promedio de 205 ± 6 (límites 197 a 213) días por niño. Estos resultados indican una excelente colaboración con las exploraciones otoscópicas planificadas y con otras recogidas de datos, atribuible principalmente a las visitas domiciliarias realizadas por personal del estudio.
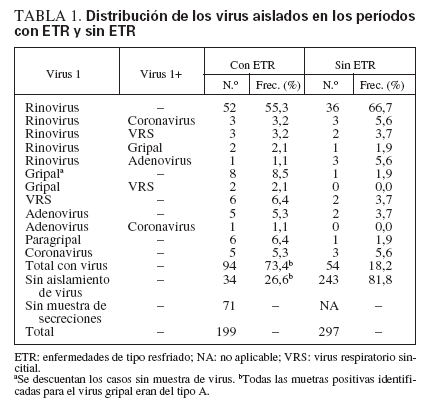
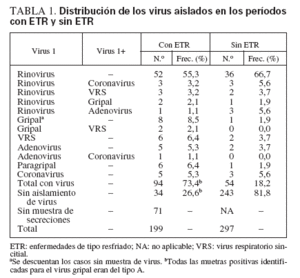
El algoritmo asignó 2.077 de todos los días-niño (17%) a un día de enfermedad; la carga media por resfriado, definida por el porcentaje de días de observación asignados a un día de enfermedad, fue de 17 ± 15% (límites 0 a 68%) por niño. Estos días se vincularon como 199 ETR, con una tasa media de 3,3 ± 2,0 (límites 0 a 7) ETR/niño. En 71 casos (36%) no se recogieron frotis nasales durante la ETR, debido a la ausencia de secreciones libres en niños que rehusaron el lavado nasal, o bien a que la familia no se hallaba disponible durante el período de ETR (vacaciones, días festivos) o a que la madre no comunicó el resfriado al personal del estudioen las ocasiones en que la ETR se identificó retrospectivamente por el algoritmo. De las 128 ETR con recogida de frotis nasal (64%), se identificó un virus en 94 (73%). En los distintos niños se detectó un virus durante un promedio de 2,1 ± 1,3 (límites 0 a 5) de las ETR con frotis, con una tasa media de detección de 77 ± 27%. La distribución de virus y combinaciones de virus en asociación temporal con una ETR se presenta en la tabla 1. Como era de esperar, en las ETR con identificación del virus, el rinovirus se asoció con más del 50% de los episodios.
TABLA 1. Distribución de los virus aislados en los períodos con ETR y sin ETR
De los 297 análisis para virus realizados durante los períodos sin ETR, 54 (18,2%) fueron PCR-positivos para un virus o combinación de virus. En la tabla 1 se expone la distribución de especies de estos virus. En todos los sujetos, la tasa media de aislamiento positivo de virus en un período sin ETR fue de 16 ± 17% (límites 0 a 67%). La distribución de frecuencias relativas de los virus y combinaciones de virus es similar a la de las ETR, el más frecuente fue el rinovirus, seguido del VRS y el virus gripal, y luego el adenovirus, el virus paragripal y el coronavirus. En 243 muestras obtenidas en períodos sin ETR no se aislaron virus (81,8%), y en los distintos sujetos, la frecuencia media de falta de aislamiento de virus en ausencia de ETR fue de 84 ± 17% (límites 33 a 100%).
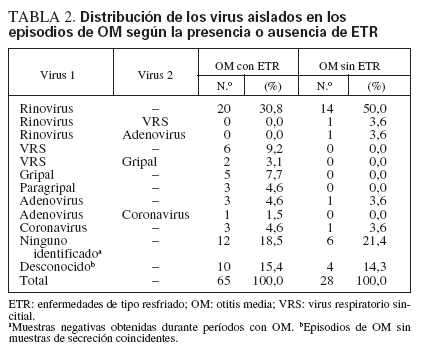
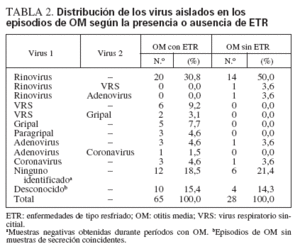
En total se diagnosticaron 93 episodios de OM (uni o bilateral). El promedio de episodios por sujeto fue de 1,6 ± 1,3 (límites 0 a 4). En la tabla 2 se muestra la distribución de los virus asociados con estos episodios cuando se diagnosticó una ETR y cuando se identificó el virus en ausencia de ETR. Sesenta y cinco de todos los diagnósticos de OM (70%) estuvieron relacionados con una ETR concomitante (independientemente de aislamiento simultáneo de virus). De ellos, no se obtuvieron muestras de la secreción en 10 casos (15%), y en otros 12 casos (18%) no se identificaron virus a pesar del frotis practicado. El orden relativo de frecuencias de aislamiento de virus en la OM no difirió del observado en las ETR (v. tabla 1).
TABLA 2. Distribución de los virus aislados en los episodios de OM según la presencia o ausencia de ETR
De mayor interés es la frecuencia relativa de OM durante los períodos sin ETR con identificación de virus en el frotis nasal. En estos casos, otros 18 episodios de OM (19%) se asociaron con el aislamiento de un virus en el frotis nasal y nasofaríngeo, 6 episodios de OM (6%) no se asociaron con virus a pesar de tomar la muestra durante el episodio de OM, y 4 episodios de OM (4%) no pudieron asignarse debido a que no se recogieron muestras de la secreción durante el episodio. Al igual que en las ETR, la mayoría de estos episodios de OM se asociaron con una minoría de adenovirus y coronavirus, pero el virus gripal y el VRS estuvieron llamativamente ausentes.
En esta población, la tasa de la complicación de OM en una ETR se estimó en 65 episodios de OM/199 ETR, o 33%. De los 79 episodios de OM con frotis nasal, se aislaron 1 o más virus en 61 (77,2%). De las 94 ETR con aislamiento de virus, 43 (45,7%) se complicaron con un nuevo episodio de OM. Finalmente, un nuevo episodio de OM se asoció con 18 (33,3%) de las 54 ocasiones en que se aisló un virus en un frotis nasal en ausencia de ETR.
DISCUSIÓNLos resultados presentan una serie de coincidencias temporales entre el aislamiento de virus de las vías respiratorias altas, la expresión de una ETR y la OM como complicación otológica que sugiere una relación causal. En estudios anteriores realizados en voluntarios humanos se apoya esta interpretación al mostrar que la infección experimental de la nariz y nasofaringe por rinovirus, virus gripal A o VRS provoca una expresión variable de signos y síntomas de enfermedad que se cataloga de ETR en aproximadamente dos tercios de los expuestos, y complicaciones otológicas incluida la OM en algunos sujetos, pero no en todos15-17,19. Otros análisis de estos datos mostraron que la expresión de enfermedad y la expresión otológica no guardan relación; por ejemplo, una ETR no es un prerrequisito de las complicaciones otológicas, y las dos muestran respuestas diferentes al título de anticuerpos previo y al tratamiento an-18,20,21. Debido a que se aíslan virus de las vías respiratorias altas en frotis nasales y nasofaríngeos practicados a lactantes, niños y adultos en ausencia de enfermedad manifiesta22-25, estos resultados sugieren que la tasa de complicaciones de una IvVRA subestima la verdadera tasa cuando se basa en una ETR concomitante; de modo similar, el porcentaje total de episodios de OM explicados por una IvVRA es mucho mayor de lo que se había publicado anteriormente.
La distribución de especies de virus durante las ETR es similar en este estudio a la descrita anteriormente: los rinovirus son responsables de la mayoría (cerca del 60%) de enfermedades, seguidos por el virus gripal A y el VRS con un 10%, y luego adenovirus, coronavirus y virus paragripal, cerca de un 5% cada uno1,3,8. Tiene interés señalar que se observó una distribución de frecuencias relativas de estos virus similar en los períodos sin ETR con aislamiento positivo de virus. Es de esperar que esta distribución refleje el muestreo proporcional para toda la “temporada de resfriados” y que la frecuencia de recuperación de virus quede sesgada hacia una determinada especie al concentrar el muestreo durante un período endémico.
Aproximadamente el 70% de los episodios de OM se asociaron temporalmente con una ETR, y cuando fue posible comprobarlo, no hubo evidencia de sesgo a favor de determinados virus. En vez de ello, las ETR complicadas por OM mostraron la misma distribución de virus que la población fuente de la que se obtuvieron. Esta observación contrasta con algunos informes previos que señalaron una frecuencia mayor de la esperada de los episodios de OM con una determinada especie de virus1,2,4-6. Como nuestros resultados concuerdan con unas investigaciones más globales en distintas temporadas3,8, sugerimos que estos informes estuvieron sesgados por diversos factores: la dependencia de signos y síntomas de enfermedad para identificar la OM; la población fuente (p. ej., niños hospitalizados), y la temporada del estudio.
Finalmente, los datos del estudio rechazan nuestra hipótesis nula. Ambas hipótesis requieren que el aislamiento de virus durante los períodos sin ETR no se asocie con OM, una formulación que no coincide con nuestros resultados. En vez de ello, la OM ocurre como complicación de la presencia de virus en la nariz y/o nasofaringe independientemente de la presentación de una ETR. Además, aunque la serie es pequeña, no parece haber un claro orden preferente en que los virus se asocien con OM en ausencia de enfermedad. A este respecto, nuestra valoración de la presencia o ausencia de una ETR se realizó mediante un algoritmo y es posible que el código establecido no fuera sensible a patrones de signos específicos de un determinado virus o a otras presentaciones de signos. Para comprobar esta posibilidad, se examinó un período de 20 días centrado en cada observación de una asociación de ausencia de ETR y aislamiento positivo de virus, en busca de signos concomitantes. Los resultados fueron compatibles con una serie nula típica o una regresión esporádica de signos que no estaban claramente relacionados con el virus (datos no mostrados).
CONCLUSIONESEn resumen, el 70% de todos los episodios de OM fueron atribuibles a un episodio concomitante de ETR, y en esta población de niños mayores, la OM fue una complicación en el 33% de todos los episodios de ETR. El agente etiológico primario asociado con los episodios de ETR y OM fue el rinovirus. Los virus respiratorios se aislaron en un gran número de períodos sin ETR, y también se asociaron con el desarrollo de OM. Más recientemente se han identificado otros virus respiratorios, como metaneumovirus y bocavirus, como causantes de ETR y desencadenantes de OM; en el presente estudio no se analizó su presencia, dado que las técnicas necesarias no se hallaban todavía disponibles en nuestros labo-ratorios33,34. Es posible que éstos y otros virus fueran agentes causales en los episodios de ETR y OM en que nuestro panel de análisis no pudo identificar una etiología viral. Nuestra conclusión es que la mayoría de episodios de OM son la complicación de una IvVRA, y que las anteriores estimaciones de OM como complicación de una IvVRA, ajustadas a la edad, están sesgadas a la baja al depender de la presentación de una ETR.
AGRADECIMIENTOSSubvencionado parcialmente por una beca de National Institutes of Health DC005832.
Los autores desean expresar su agradecimiento a Kathleen Ashe por su ayuda con los análisis virológicos; a Margaretha L. Casselbrant, Harriette Wheatley, Kathy Tekely y Ellen Reynolds por las exploraciones otoscópicas y la obtención de muestras, y a Julianne Banks y James T. Seroky por la introducción de los datos.
Correspondencia: Cuneyt M. Alper, MD, Children’s Hospital of Pittsburgh, 3705 Fifth Ave. en De Soto St., Pittsburgh, PA 15213, Estados Unidos.
Correo electrónico: alperc@pitt.edu