Escherichia coli O157 es un patógeno emergente asociado a diarrea, colitis hemorrágica y síndrome urémico hemolítico. Los productos cárnicos constituyen una importante fuente de contaminación con este microorganismo. Los objetivos de este estudio fueron establecer la frecuencia de detección de E. coli O157 en productos cárnicos y media res en la provincia de Tucumán, caracterizar los factores de virulencia de los aislamientos obtenidos, establecer la relación clonal entre cepas regionales mediante electroforesis de campo pulsado y comparar con lo consignado en la base de datos nacional. Desde 2004 hasta 2013 se analizaron 169 muestras de carne picada, 35 embutidos y 216 esponjados de media res. Se identificaron 13 aislamientos de E. coli O157; 6 de ellos fueron O157:H7 productores de toxina Shiga y se caracterizaron como stx2c(vh-a)/eae/ehxA (n = 5) y stx2/eae/ehxA (n = 1); los 7 aislamientos de E. coli O157 no toxigénicos fueron O157:NT(n = 4),O157:NM (n = 1),O157:ND (n = 1) y O157:H16 (n = 1). Los patrones de PFGE fueron diferentes entre sí y de los registrados en la base de datos nacional. Se concluye que existe gran diversidad genética en los aislamientos de E. coli O157 circulantes en nuestra región.
Escherichia coli O157 is an emergent pathogen associated with diarrhea, hemorrhagic colitis and hemolytic uremic syndrome. Meat products constitute an important transmission source of this microorganism. The aims of this study were to characterize E. coli O157 isolated from cattle and meat products collected from abattoirs and retail stores, to establish the clonal relatedness among regional isolates and to compare them with those in the national database. Between 2004 and 2013, 169 minced meat, 35 sausage and 216 carcass samples were analyzed. Thirteen E. coli O157 isolates were identified; 6 of which were O157:H7 and characterized as stx2c(vh-a)/eae/ehxA (n = 5) and stx2/eae/ehxA (n = 1). The 7 remaining isolates were non-toxigenic E. coli strains, and serotyped as O157:NT (n = 4), O157:NM (n = 1), O157:ND (n = 1) and O157:H16 (n = 1). The strains yielded different XbaI-PFGE patterns. Compared to the E. coli O157 isolates in the National Database, none of these patterns have been previously detected in strains of different origin in Argentina.
Escherichia coli O157:H7/NM es un patógeno emergente transmitido por alimentos. Se asocia a casos esporádicos y brotes de diarrea, colitis hemorrágica y síndrome urémico hemolítico (SUH). También ocasiona enfermedad extraintestinal severa, caracterizada por anemia hemolítica microangiopática, trombocitopenia y falla renal aguda.
Los bovinos constituyen el principal reservorio de E. coli productor de toxina Shiga (STEC) O157 y no-O157, y los alimentos de origen cárnico son la fuente de infección más importante para el hombre10.
Las cepas patógenas humanas y las aisladas de bovinos u otros rumiantes, tanto sanos como con diarrea, poseen factores de virulencia comunes a ambas especies, en tanto que las cepas patógenas para el cerdo tienen características de virulencia propias11. Pistone Creydt et al. (2005) demostraron que cepas STEC aisladas de terneros con diarrea sanguinolenta tienen efecto citopático sobre el colon humano in vitro13. Por este motivo es que los rumiantes, en particular los bovinos, son señalados como el principal reservorio de STEC y la fuente de la infección para el hombre. En particular, el serotipo O157:H7, mundialmente asociado a brotes de enfermedad intestinal, es el principal responsable de complicaciones sistémicas graves.
La infección por STEC es considerada por la Organización Mundial de Sanidad Animal (originalmente Oficina Internacional de Epizootias) como una zoonosis de origen alimentario4. Dentro del espectro de cepas STEC que son capaces de colonizar el tracto intestinal del bovino, solo un número limitado de serotipos son patógenos para esta especie y ocasiona diarrea en terneros4.
La frecuencia de detección de E. coli O157 en bovinos es relativamente similar en diferentes países, dependiendo del número de animales estudiados y la metodología empleada para la detección; sin embargo, la incidencia de la enfermedad humana es variable. Las estrategias de intervención para evitar la contaminación fecal de los productos cárnicos se han concentrado en la planta faenadora, con la aplicación de prácticas que evitan la transmisión y proliferación de este patógeno7.
El alerta clínico-epidemiológico y de laboratorio debe ser sistemática a fin de detectar aislamientos relacionados, que permitan encontrar la fuente de contaminación y tomar las acciones de control pertinentes. Un elemento clave para el reconocimiento de los patógenos es poder identificarlos en las probables fuentes de contaminación, hasta llegar, en lo posible, a establecer su reservorio y las vías de transmisión.
Las metodologías de biología molecular, como subtipificación y electroforesis de campo pulsado (pulsed-field gel electrophoresis [PFGE]), permiten establecer la relación epidemiológica existente entre cepas aisladas de distintos orígenes y realizar una vigilancia de los clones circulantes en una región determinada9. En nuestro país se ha establecido la relación clonal de cepas provenientes de diferentes orígenes y los datos se encuentran en la Base de Datos Nacional de la Red Regional de Subtipificación para la Vigilancia de las Enfermedades Transmitidas por Alimentos (red PulseNet).
En Tucumán, E. coli productor de toxina Shiga O157 y no-O157 se detecta en pacientes desde 19985; los productos cárnicos constituyen una importante fuente de contaminación con este microorganismo6. El presente trabajo tuvo como meta evaluar la frecuencia de detección de E. coli O157 en medias reses bovinas y en productos cárnicos expendidos en dicha provincia en un período de 10 años, así como determinar la calidad microbiológica en las muestras de media res procesadas. Los objetivos planteados fueron aislar E. coli O157 en muestras de carne picada, embutidos y media res recolectadas en esta provincia, caracterizar sus factores de virulencia, establecer la relación clonal entre las cepas aisladas en la región y comparar los patrones de PFGE obtenidos con los existentes en la base de datos de la Argentina.
Recolección de muestras: en el marco de convenios realizados por la Dirección de Bromatología de la Provincia de Tucumán, la Dirección de Ganadería de esta misma provincia y la Municipalidad de San Miguel de Tucumán con la Universidad Nacional de Tucumán (UNT) para la prevención de SUH y el control de STEC, desde 2004 se realizan muestreos en carnicerías de la capital provincial y localidades del interior, y desde 2011 se lleva a cabo el asesoramiento y control de todos los frigoríficos categoría B habilitados, en el cumplimiento de las normativas establecidas por el Servicio Nacional de Sanidad y Calidad Agroalimentaria (SENASA, Circular 3496/02). Es por ello que se realiza la investigación de E. coli O157 en productos cárnicos y medias reses bovinas.
Las muestras de productos cárnicos fueron recolectadas por personal de la Dirección de Bromatología provincial en conjunto con personal municipal de carnicerías habilitadas y siguiendo las normativas del Código Alimentario Argentino. Se establecieron como zonas de muestreo San Miguel de Tucumán y 3 localidades del interior de la provincia: Concepción, Trancas (zona ganadero-lechera por excelencia) y Tafí Viejo.
Medias reses bovinas: las muestras fueron obtenidas por la Secretaría de Agricultura y Ganadería de animales faenados en frigoríficos de la provincia de Tucumán: Capital, Bella Vista, Famaillá, Santa Bárbara y San Isidro de Lules. Con esponjas estériles se tomaron muestras de 4 regiones corporales: cuadril, vacío, pecho y cogote; la frecuencia de muestreo estuvo ajustada a las normativas nacionales de SENASA (Circular 3192/96-Anexo II).
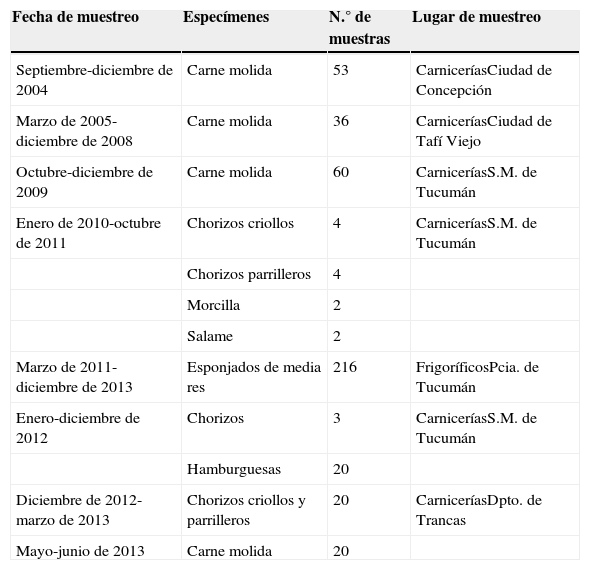
Muestras: se analizaron 169 muestras de carne picada tomadas en los años que se indican entre paréntesis: 53 de la ciudad de Concepción (2004), 36 de Tafí Viejo (8 [2005], 12 [2006], 9 [2007], 7 [2008]); 60 de San Miguel de Tucumán (2009) y 20 del Departamento de Trancas (2013). También se analizaron 35 embutidos. De estos, 15 se tomaron en S.M. de Tucumán y eran de elaboración casera (12 [2010/2011]; 3 [2012]); los otros 20 se tomaron de puestos habilitados del Departamento de Trancas (2012/2013). Por último, se analizaron 216 esponjados de media res (2011/2013) procedentes de 6 frigoríficos de la provincia distribuidos del siguiente modo: Capital, frigoríficos A y B; Bella Vista, frigorífico C; Famaillá, frigorífico D; Santa Bárbara, frigorífico E, y San Isidro, frigorífico F.
La procedencia de las muestras, el tipo de material del cual provenían y el número investigado en cada caso se detallan en la tabla 1.
Detalle de las muestras recolectadas para la detección de E. coli O157 en productos cárnicos en la provincia de Tucumán
| Fecha de muestreo | Especímenes | N.° de muestras | Lugar de muestreo |
|---|---|---|---|
| Septiembre-diciembre de 2004 | Carne molida | 53 | CarniceríasCiudad de Concepción |
| Marzo de 2005-diciembre de 2008 | Carne molida | 36 | CarniceríasCiudad de Tafí Viejo |
| Octubre-diciembre de 2009 | Carne molida | 60 | CarniceríasS.M. de Tucumán |
| Enero de 2010-octubre de 2011 | Chorizos criollos | 4 | CarniceríasS.M. de Tucumán |
| Chorizos parrilleros | 4 | ||
| Morcilla | 2 | ||
| Salame | 2 | ||
| Marzo de 2011-diciembre de 2013 | Esponjados de media res | 216 | FrigoríficosPcia. de Tucumán |
| Enero-diciembre de 2012 | Chorizos | 3 | CarniceríasS.M. de Tucumán |
| Hamburguesas | 20 | ||
| Diciembre de 2012-marzo de 2013 | Chorizos criollos y parrilleros | 20 | CarniceríasDpto. de Trancas |
| Mayo-junio de 2013 | Carne molida | 20 |
Todas las muestras fueron procesadas por duplicado en los laboratorios de Bacteriología de la Facultad de Bioquímica de la UNT y de la Dirección de Bromatología de la provincia.
Aislamiento y cuantificación de E. coli en media res: la cuantificación de E. coli genérico se realizó mediante recuento de coliformes y de E. coli (Petrifilm®). Se determinó por cuenta viable la carga de E. coli en las muestras (esponjas diluidas en 25ml de agua peptonada). Se consideró como valor marginal un conteo de 5 UFC/cm2.
Aislamiento y caracterización de E. coli O157: se siguieron las especificaciones de las normativas United States Department of Agriculture-Food Safety Inspection Services (USDA/FSIS)17 vigentes en las diferentes fechas de procesamiento de las muestras (años 2004 a 2013).
En 2004, las muestras se procesaron teniendo en cuenta la normativa USDA-FSIS 2002, el enriquecimiento se realizó en caldo EC modificado (ECm) (BectonDickinson, Le Pont de Claix, Francia) adicionado con 20mg/l de novobiocina (Sigma Chemical Co. St. Louis, EE. UU.); las muestras se incubaron a 37°C durante 20-24h, no se realizó tamizaje, por lo tanto, la separación inmunomagnética (SIM) se efectuó en todas las muestras. La norma USDA-FSIS 2008 reemplazó el caldo ECm por caldo tripteína soja con novobiocina y casaminoácidos (mTSB+n, Neogen, EE. UU.). Se incubó a 42°C, 15 a 22h; como se advierte, se modificaron el intervalo y la temperatura de incubación, no se realizó tamizaje, por lo tanto, la SIM se ensayó en todas las muestras. En la norma USDA/FSIS 2010, utilizamos TSBm+n con novobiocina, 20mg/l (Sigma Chemical Co. St. Louis, EE. UU.), a 42°C, 15 a 22h y luego se hizo el tamizaje y se realizó SIM en las muestras con tamizaje positivo para E. coli O157. En la norma USDA/FSIS 2012, la concentración de novobiocina se disminuyó de 20 a 8mg/l (mTSB+n, Neogen, EE. UU.) la temperatura de incubación fue de 41,5°C durante 15 a 22h y se realizó SIM en las muestras con tamizaje positivo para E. coli O157.
Resumiendo, se cumplieron las siguientes etapas metodológicas según las normas USDA/FSIS vigentes: a) enriquecimiento en los diferentes caldos estandarizados; b) tamizaje a elección previamente validado a partir de la norma USDA/FSIS 2010; c) SIM; d) siembra en medios selectivos y diferenciales, y e) caracterización molecular de los aislamientos.
a) Enriquecimiento. Carne molida y embutidos: se colocaron 65g de muestra en 585ml de caldo de enriquecimiento; esponjados de medias reses: se colocaron las esponjas en 50ml de caldo de enriquecimiento.
b) Tamizaje: a todas las muestras se les realizó el tamizaje a partir del caldo de enriquecimiento, se utilizó el kit comercial RapidChek®E. coli O157 Test Kit (Strategic Diagnostics Inc., Newark, EE.UU.).
c) Separación inmunomagnética: se utilizaron perlas inmunomagnéticas (Dynabeads® Dynal Brown Deer [EE. UU.]) y Beijing (China) pertenecientes a Dynal Biotech. El producto inmunoconcentrado (100μl) se sembró en 2 alícuotas de 50μl cada una en agar MacConkey sorbitol (Becton Dickinson, EE. UU.) adicionado con cefixima-telurito (Biomérieux, Marcy-l’Étoile, Francia) y CHROM agar (CHROM agar, París, Francia). Las placas fueron incubadas a 37°C durante 24h. De cada placa se seleccionaron las colonias sospechosas, las cuales fueron identificadas mediante pruebas bioquímicas convencionales.
d) Identificación fenotípica y serotipificación: la identificación bioquímica de las colonias que presumiblemente podrían ser E. coli O157 se realizó mediante la evaluación de la fermentación de celobiosa, el crecimiento en cianuro de potasio y la producción de pigmento y de lisina descarboxilasa (Britania, Buenos Aires, Argentina). La detección de movilidad se realizó en medio de Craigie14. La determinación del biotipo se realizó mediante la fermentación de sorbitol, dulcitol, rafinosa y ramnosa (ICN Biomedicals, Aurora, Ohio, EE. UU.). La serotipificación se realizó con los antisueros somáticos O157 (Oxoid, Ltd., Hampshire, Reino Unido) y flagelar H7 (Instituto de Producción de Biológicos-ANLIS «Dr. Carlos G. Malbrán»).
e) Caracterización molecular: los aislamientos de E. coli O157 fueron caracterizados por PCR múltiple para amplificar los genes stx1, stx2 y rfbO157, la caracterización de los marcadores de virulencia accesorios eae, ehxA y fliCH7 se realizó por PCR simple mediante protocolos previamente estandarizados14.
Subtipificación: la variante de la toxina Shiga fue evaluada mediante el estudio del polimorfismo utilizando PCR-RFLP16.
Electroforesis en campo pulsado. PFGE: la subtipificación empleando la técnica de macrorrestricción con la enzima XbaI y separación por electroforesis en gel de campo pulsado (XbaI-PFGE) se realizó utilizando el protocolo estandarizado de 24h de PulseNet para E. coli O1573. La restricción enzimática del ADN inmovilizado en los plugs se realizó con 25 U de XbaI (Promega Corporation, Madison, EE. UU.) a 37°C. El análisis de los patrones electroforéticos se efectuó mediante el programa Bionumerics Software Package 4.0 (Applied Maths, Bélgica) utilizando el coeficiente de Dice y UPGMA para generar los dendrogramas. Para la comparación con cepas STEC O157 de distintos orígenes (humano, animal, alimentos y medio ambiente) aisladas en Argentina durante el período 1988-2012, se utilizó la base de datos de PulseNet Argentina, que consta de 2318 cepas de STEC O157: 1799 humanas, 247 de alimentos, 198 de animales y 74 de medio ambiente. Dicha base incluye, además, los datos epidemiológicos de los casos y los resultados de las técnicas de caracterización y subtipificación.
El recuento de E. coli genérico fue mayor de 5 UFC/cm2 de superficie (evaluación marginal) en el 10% de las muestras de media res, obtenidas de 6 frigoríficos diferentes.
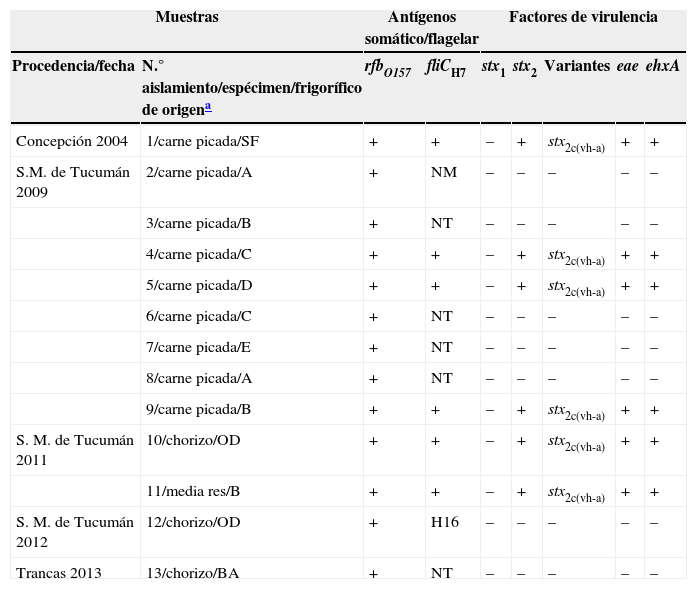
E. coli O157 se aisló en 1/53 muestras de carne picada obtenidas de carnicerías de la ciudad de Concepción, abastecidas por un frigorífico de la ciudad de Santa Fe y en 8/60 muestras obtenidas de carnicerías de San Miguel de Tucumán, abastecidas por 5 frigoríficos diferentes de Tucumán. Al discriminar estas últimas según su origen, se determinó que en 3 casos compartían el frigorífico de origen (6 muestras) y en 2 provenían de frigoríficos diferentes (2 muestras) (tabla 2). En los muestreos de carne picada restantes no se obtuvo E. coli O157.
Caracterización molecular de los aislamientos de E. coli O157 recuperados de productos cárnicos en la provincia de Tucumán
| Muestras | Antígenos somático/flagelar | Factores de virulencia | ||||||
|---|---|---|---|---|---|---|---|---|
| Procedencia/fecha | N.° aislamiento/espécimen/frigorífico de origena | rfbO157 | fliCH7 | stx1 | stx2 | Variantes | eae | ehxA |
| Concepción 2004 | 1/carne picada/SF | + | + | – | + | stx2c(vh-a) | + | + |
| S.M. de Tucumán 2009 | 2/carne picada/A | + | NM | – | – | – | – | – |
| 3/carne picada/B | + | NT | – | – | – | – | – | |
| 4/carne picada/C | + | + | – | + | stx2c(vh-a) | + | + | |
| 5/carne picada/D | + | + | – | + | stx2c(vh-a) | + | + | |
| 6/carne picada/C | + | NT | – | – | – | – | – | |
| 7/carne picada/E | + | NT | – | – | – | – | – | |
| 8/carne picada/A | + | NT | – | – | – | – | – | |
| 9/carne picada/B | + | + | – | + | stx2c(vh-a) | + | + | |
| S. M. de Tucumán 2011 | 10/chorizo/OD | + | + | – | + | stx2c(vh-a) | + | + |
| 11/media res/B | + | + | – | + | stx2c(vh-a) | + | + | |
| S. M. de Tucumán 2012 | 12/chorizo/OD | + | H16 | – | – | – | – | – |
| Trancas 2013 | 13/chorizo/BA | + | NT | – | – | – | – | – |
BA: frigorífico de la ciudad de Buenos Aires; gen eae: intimina; gen ehxA: enterohemolisina; gen fliC: antígeno flagelar; gen rfb O157: serogrupo O157; gen stx1: toxina Shiga 1; gen stx2: toxina Shiga 2; NM: no móvil; NT: antígeno flagelar no tipificable; OD: frigorífico de origen desconocido; SF: frigorífico de ciudad de Santa Fe.
De los embutidos analizados, en 3/29 se aisló E. coli O157, 2 de ellos eran de San Miguel de Tucumán (elaboración casera con materia prima de origen desconocido) y el restante fue obtenido de una carnicería de Trancas y estaba elaborado con materia prima provista por un frigorífico de la ciudad de Buenos Aires.
En medias reses, solo de 1/216 muestras se recuperó E. coli O157, esta muestra había sido obtenida del frigorífico B de San Miguel de Tucumán en marzo del 2011 (tabla 2).
De los 13 aislamientos de E. coli O157 provenientes de carne molida, embutidos y media res, 6 fueron O157:H7 productores de toxina Shiga caracterizados como stx2c(vh-a)/eae/ehxA (n = 5) y stx2/eae/ehxA (n = 1). Se identificaron como E. coli O157 no toxigénicos 7/13 aislamientos serotipificados como O157:NT (n = 4), O157:NM (n = 1), O157:ND (n = 1) y O157:H16 (n = 1).
En las cepas aisladas en Tucumán, los patrones de PFGE fueron diferentes entre sí y también diferentes de los 1026 patrones XbaI-PFGE registrados en la Base de Datos Nacional. La relación clonal de los aislamientos obtenidos en este estudio se observa en la figura 1.
En este estudio se investigó E. coli O157 en carne picada y embutidos, alimentos muy consumidos en nuestra provincia e importantes vehículos de transmisión, y también en medias reses bovinas, ya que no existen en nuestra región estudios previos efectuados en las plantas faenadoras. Cabe aclarar que el origen de este microorganismo, al ser aislado de embutidos, pudo haber sido la carne de cerdo. En Argentina, Alustiza et al. (2012) publicaron un trabajo que asocia E. coli O157 con el reservorio porcino1.
El porcentaje de aislamientos de E. coli O157 en productos cárnicos es variable en diferentes países. En Latinoamérica, Uruguay reporta 1,8% de STEC O157 en carne picada fresca18. En Lima, en un estudio de 102 muestras de carne de bovino molida, Mora et al. encontraron un 22,55% positivo para E. coli O15712. En Argentina, Brusa et al. aislaron en 2013 STEC O157 en el 12,2% de las muestras de carne picada analizadas2. En nuestro trabajo, E. coli O157 se aisló en el año 2004 en 1,9% (1/53) y en el año 2009 en 13,3% (8/60) de las muestras de carne picada analizadas. En los muestreos realizados en 2005, 2006, 2007, 2008, 2012 y 2013 los resultados fueron negativos.
En embutidos, Roldán y Chinen comunicaron un 4,8% de E. coli O15715; nuestros resultados fueron variables: 8% (1/12), 33% (1/3) y 7% (1/14) en los diferentes muestreos realizados.
En media res, Masana et al. describen un 2,6% de STEC O1579, en nuestro trabajo, sobre un total de 216 muestras de media res provenientes de 6 frigoríficos (2011/2013), se aisló STEC O157 en el 0,46% de las muestras (1/216) y su aislamiento guardó una estrecha relación con el recuento marginal para E. coli genérico.
En el estudio de Roldán y Chinen antes citado15, estos autores demostraron que las cepas toxigénicas aisladas de productos cárnicos portaron los genes de los marcadores de virulencia accesorios necesarios para causar enfermedad severa en el hombre: ehxA y eae. Li et al. aislaron a partir de productos cárnicos cepas de E. coli O157 productoras de Stx2 y que no estaban asociadas con los factores de virulencia eae o ehxA8.
En Tucumán, la frecuencia de aislamiento de E. coli O157:H7/NM en algunos muestreos de carne picada y media res fue más baja respecto de otros estudios realizados en Argentina, por lo tanto, el reservorio bovino y los alimentos cárnicos no demostrarían en este estudio ser la principal fuente de transmisión ni el principal reservorio.
En nuestro trabajo, los aislamientos de E. coli O157:H7 productores de toxina Shiga se asociaron en su totalidad a los factores de virulencia accesorios eae y ehxA.
Al realizar la subtipificación por PFGE en nuestros aislamientos, no se encontró un patrón semejante entre ellos, ni aun entre cepas provenientes de carnicerías abastecidas por un mismo frigorífico. El porcentaje de similitud fue de 67% y los patrones XbaI-PFGE detectados no se correspondieron con aquellos ampliamente diseminados en diferentes años y en diferentes regiones del país. Por lo tanto, uno de los principales hallazgos de este trabajo es que no hubo relación clonal entre los aislamientos y que a lo largo del período 2004-2013 no fue posible asociar ninguno de estos aislamientos con los casos humanos registrados en la Base de Datos Nacional, compuesta de 2318 cepas de STEC O157.
Consideramos que es importante conocer los reservorios de E. coli O157 en la región del noroeste argentino, caracterizar genotípicamente los factores de virulencia de las cepas de origen animal, evaluar su diseminación y conocer las vías de transmisión para brindar apoyo a los departamentos sanitarios locales. Este trabajo es el primer estudio realizado en la provincia de Tucumán que permitió caracterizar cepas de E. coli O157 regionales y determinar la diversidad genética de aquellas entre sí y comparadas con las que están registradas a nivel nacional.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datosLos autores declaran que en este artículo no aparecen datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores declaran que en este artículo no aparecen datos de pacientes.
FinanciaciónEste trabajo fue financiado por la Secretaría de Ciencia y Técnica de la Universidad Nacional de Tucumán. Programa CIUNT 26/D417.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Los autores agradecen a la Bioqca. Silvia Vega por su colaboración en la recolección, recepción y conservación de muestras.