Se aislaron del contenido intestinal del mejillón patagónico dos cepas de bacterias ácido lácticas y se caracterizaron por pruebas fenotípicas y moleculares. Los aislamientos se identificaron como Enterococcus hirae y fueron denominados E. hirae 463Me y 471Me. Por técnicas de PCR se identificó el gen de la enterocina P en ambas cepas, mientras que solamente en la cepa 471Me se detectó la enterocina hiracin JM79. Ambas cepas resultaron sensibles a los antibióticos clínicamente importantes y entre los rasgos de virulencia investigados mediante amplificación por PCR solo se pudieron detectar los genes cylLl y cylLs, sin embargo, no se observó actividad hemolítica en la prueba de agar sangre. Los sobrenadantes libres de células resultaron activos contra todas las cepas de Listeria y Enterococcus ensayadas, contra Lactobacillus plantarum TwLb 5 y contra Vibrio anguilarum V10. En óptimas condiciones de crecimiento, ambas cepas mostraron actividad inhibitoria contra Listeria innocua ATCC 33090 después de 2h de incubación. E. hirae 471Me alcanzó una actividad inhibitoria máxima de 163.840UA/ml después de 6h de incubación, mientras que el mismo valor se registró para E. hirae 463Me después de 8h. En ambos casos, la actividad antagonista alcanzó su máximo antes de lograr la fase estacionaria y permaneció estable hasta las 24h de incubación. En nuestro conocimiento, este es el primer informe de aislamiento de cepas bacteriocinogénicas de E. hirae de mejillón patagónico. La alta actividad inhibitoria y la ausencia de rasgos de virulencia indican que estos microorganismos podrían aplicarse en áreas biotecnológicas como la biopreservación de alimentos o las formulaciones probióticas.
Two bacteriocin-producing lactic acid bacterial strains were isolated from the intestinal content of the Patagonian mussel and characterized by phenotypic and molecular tests. The isolates were identified as Enterococcus hirae and named E. hirae 463Me and 471Me. The presence of the enterocin P gene was identified in both strains by PCR techniques, while enterocin hiracin JM79 was detected only in the 471Me strain. Both strains were sensitive to clinically important antibiotics and among the virulence traits investigated by PCR amplification, only cylLl and cylLs could be detected; however, no hemolytic activity was observed in the blood agar test. Cell free supernatants were active against all Listeria and Enterococcus strains tested, Lactobacillus plantarum TwLb 5 and Vibrio anguilarum V10. Under optimal growth conditions, both strains displayed inhibitory activity against Listeria innocua ATCC 33090 after 2h of incubation. E. hirae 471Me achieved a maximum activity of 163840AU/ml after 6h of incubation, while the same value was recorded for E. hirae 463Me after 8h. In both cases, the antagonist activity reached its maximum before the growth achieved the stationary phase and remained stable up to 24h of incubation. To our knowledge, this is first report of the isolation of bacteriocinogenic E. hirae strains from the Patagonian mussel. The high inhibitory activity and the absence of virulence traits indicate that they could be applied in different biotechnological areas such as food biopreservation or probiotic formulations.
Las bacterias ácido lácticas (BAL) constituyen un grupo de microorganismos gram positivos, no esporulados, catalasa y oxidasa negativos, aerotolerantes, que producen ácido láctico como principal metabolito derivado de la fermentación de azúcares10. Las BAL son ampliamente utilizadas como cultivos iniciadores en la industria láctea para la fabricación de quesos, yogures y leches cultivadas, y han ganado notoriedad como probióticos, término utilizado para referirse a aquellos microorganismos que componen un suplemento alimentario microbiano vivo que beneficia al huésped animal con una mejoría del balance microbiano intestinal14.
Las BAL han sido extensamente estudiadas no solo por sus características biotecnológicas, sino debido a su capacidad de sintetizar bacteriocinas, es decir, péptidos de origen ribosomal que representan un mecanismo de defensa contra grupos bacterianos filogenéticamente relacionados32. Se han identificado algunas BAL con actividad contra bacterias gram negativas, sin embargo, se trata de casos aislados18,32. Las bacteriocinas pueden ser aplicadas como conservantes naturales para proteger la calidad y la vida útil de alimentos y bebidas. Esta tendencia se ve impulsada por la exigencia cada vez mayor de los consumidores, que requieren alimentos exentos de conservantes químicos y con nuevas propiedades benéficas para la salud, más allá de la nutrición básica24,29.
Existen numerosos estudios que demuestran los beneficios del uso de bacteriocinas para la conservación de alimentos. Sin embargo, en la actualidad solo la nisina y la pediocina PA-1 son permitidas como aditivos en la industria alimentaria29.
Durante las últimas décadas ha aumentado en forma exponencial el número de investigaciones dirigidas al aislamiento de cepas de BAL bacteriocinogénicas y a la purificación de nuevas bacteriocinas a partir de alimentos fermentados obtenidos en hábitats terrestres9,21. En forma contraria, las iniciativas para lograr el aislamiento de microorganismos de hábitats acuáticos o marinos con potenciales aplicaciones biotecnológicas son realmente escasas16.
Una de las posibles causas de este fenómeno es la presunción de la baja probabilidad de desarrollo de BAL en medios acuáticos debido a sus altos requerimientos nutricionales. Sin embargo, aún en bajo número pero con frecuencia creciente, comienzan a conocerse comunicaciones no solo del aislamiento de BAL de medios acuáticos, sino de su potencial aplicación en conservación de alimentos o como probióticos, gracias a sus características bioquímicas particulares9,17.
En la última década se han reportado aislamientos de BAL bacteriocinogénicas de peces, moluscos, bivalvos, crustáceos y alimentos derivados de productos del mar7,22,43; algunas de estas cepas han sido aplicadas con éxito en modelos de control de flora deteriorante o contaminante y contra microorganismos patógenos comunes en acuicultura7,33,34.
Se ha planteado, como estrategia general, el aislamiento de cepas de hábitats que ejercen una presión selectiva en virtud de sus condiciones de temperatura, salinidad o baja disponibilidad de nutrientes, porque estas resultarían más eficientes como biopreservadoras de alimentos o probióticos en acuicultura.
En nuestro país son escasos los grupos de investigación dedicados al estudio de BAL provenientes del medio marino; sin embargo, las publicaciones referidas a las cepas aisladas demuestran que estas exhiben un gran potencial desde el punto de vista biotecnológico. En el litoral marítimo de la provincia del Chubut se han logrado aislar del contenido intestinal de distintas especies nativas cepas de los géneros Lactococcus, Lactobacillus y Enterococcus con capacidad de sintetizar metabolitos antimicrobianos31,37,39,44.
En este trabajo se exhiben los resultados de las pruebas fenotípicas y genotípicas efectuadas en dos cepas de Enterococcus hirae bacteriocinogénicas aisladas de mejillones de la costa marítima de la provincia del Chubut. Se incluyen también los resultados de su actividad antimicrobiana contra bacterias contaminantes o deteriorantes de alimentos y contra patógenos comunes en acuicultura.
Materiales y métodosOrigen e identificación de los microorganismosLas cepas de E. hirae 463Me y 471Me se obtuvieron de la colección perteneciente al Laboratorio de Biotecnología Bacteriana (Facultad de Ciencias Naturales y Ciencias de la Salud, Sede Trelew - Universidad Nacional de la Patagonia, Argentina). Ambas cepas habían sido aisladas del contenido intestinal del mejillón patagónico (Mytilus edulis platensis) utilizando como medio selectivo el caldo púrpura de bromo-cresol azida (Merck, Alemania), con posterior repique a agar bilis esculina (Merck, Alemania), hasta obtener cultivos puros. Las cepas se conservaron en caldo tripteína soya (TS, Britania, Argentina) suplementado con glicerol al 10% a −30°C y se reactivaron mediante sucesivos cultivos en caldo y en agar de Man, Rogosa y Sharp (MRS, Biokar, Francia).
La identificación fenotípica se realizó mediante pruebas bioquímicas y de fermentación de azúcares, según las recomendaciones de Manero y Blanch27.
Determinación de factores de virulenciaActividad de la gelatinasaSe evaluó en TSA suplementado con 0,8% (m/v) de gelatina. La placa se incubó durante 48h a 37°C y se reveló con una solución de ácido tricloroacético al 20% (v/v). La aparición de zonas claras alrededor de los puntos de siembra se consideró un resultado positivo25.
Actividad hemolíticaLa producción de hemolisinas se evaluó en agar cerebro-corazón (Biokar, Francia) suplementado con sangre desfibrinada de conejo al 5%, luego de una incubación a 37°C durante 48h. Los resultados se interpretaron como positivos cuando se observó un halo de hemólisis completa alrededor de las colonias (β-hemólisis)38.
Sensibilidad a antibióticosSe determinó mediante la prueba de difusión en disco en agar Mueller-Hinton (Britania, Argentina) por el método de Bauer et al.4. Se utilizaron los siguientes discos de sensibilidad a los antibióticos (Laboratorios Britania, Argentina): ampicilina (10μg), amoxicilina-clavulánico (20/10μg), cefalotina (30μg), eritromicina (15μg), rifampicina (5μg), trimetoprima/sulfametoxazol (1,25/23,75μg) y vancomicina (30μg). Las placas se incubaron a 37°C durante 24h. Las cepas Enterococcus faecalis ATCC 29212 y Staphylococcus aureus ATCC 25923 se utilizaron como microorganismos de control de calidad. Luego de la incubación se midieron los diámetros de los halos de inhibición de crecimiento y los aislamientos de interés fueron categorizados como sensibles (S), intermedios (I) o resistentes (R), de acuerdo a las recomendaciones del National Committee for Clinical Laboratory Standards8.
Detección de factores de virulencia mediante PCRLas cepas de E. hirae se incubaron en caldo MRS a 35°C durante 18h. Posteriormente, los cultivos se centrifugaron a 12.000g a 4°C durante 5min y el ADN genómico se extrajo utilizando el kit comercial de purificación Wizard Genomics (Promega, Madison, Wisconsin, EE.UU.), siguiendo las instrucciones del fabricante. Mediante reacciones de PCR se amplificaron los genes agg (proteína de agregación), gelE (gelatinasa), esp (proteína de superficie), hylEfm (hialuronidasa), IS16 (elemento de inserción) y cylLl/Ls (citolisinas) utilizando los cebadores y las condiciones propuestas por los autores que se indican en la tabla 1.
Determinantes genéticos de factores de virulencia investigados por PCR en aislamientos de Enterococcus hirae obtenidos de mejillón patagónico
| Factor de | Secuencia (5‘- 3‘) | Fragmento (pb) | Referencia |
|---|---|---|---|
| virulencia | |||
| Agg | f: AAGAAAAAGAAGTAGACCAAC | 1553 | |
| r: AAACGGCAAGACAAGTAAATA | Eaton y Gasson12 | ||
| GelE | f: ACCCCGTATCATTGGTTT | 419 | |
| r: ACGCATTGCTTTTCCATC | Eaton y Gasson12 | ||
| Esp | f: TTGCTAATGCTAGTCCACGACC | 933 | |
| r: GCGTCAACACTTGCATTGCCGAA | Eaton y Gasson12 | ||
| HylEfm | f: GAGTAGAGGAATATCTTAGC | 661 | |
| r: AGGCTCCAATTCTGT | Rice et al.35 | ||
| IS16 | f: CATGTTCCACGAACCAGAG | 547 | |
| r: TCAAAAAGTGGGCTTGGC | Werner et al.45 | ||
| CylLl | f: GATGGAGGGTAAGAATTATGG | 253 | |
| r: GCTTCACCTCACTAAGTTTTATAG | Semedo et al.38 | ||
| CylLs | f: GAAGCACAGTGCTAAATA AGG | 240 | |
| r: GTATAAGAGGGCTAGTTTCAC | Semedo et al.38 |
Agg: proteína de agregación; GelE: gelatinasa; Esp: proteína de superficie; HylEfm: hialuronidasa; IS16: elemento de inserción; cylLl/Ls: citolisinas.
Se realizó la amplificación por PCR empleando cebadores específicos para especie (HI1 5́-CTTTCTGATATGGATGCTGCT-3́ y HI2 5́-TAAATTCTTCCTTAAATGTTG-3́), en las condiciones descritas por Jackson et al.23, y los cebadores específicos para género rrs (5́-CTTTGGATTAGATACCCTGGTAGTCC-3́ y 5́-TCGTTGCGGGACTTAACCCAAC-3́), según las sugerencias de Kariyama et al.26. Ambas reacciones se llevaron a cabo utilizando un termociclador Multigene Gradient (Labnet International Inc., Woodbridge, NJ, EE.UU.). En el caso de las cepas en estudio se realizó una PCR multiplex con ambos pares de cebadores26. Como control positivo de los cebadores específicos para género y especie se amplificó el material genético de las cepas de referencia E. faecalis ATCC 29212 y E. hirae F6 (N.° de acceso GenBank: FJ892740), respectivamente.
Actividad inhibitoria del sobrenadanteLa actividad antibacteriana se determinó luego de cultivar las cepas en caldo MRS a 30°C durante 18-20h. Terminado el período de incubación, los cultivos se centrifugaron en refrigeración (4°C) a 8.000g durante 5min. El sobrenadante libre de células (SLC) se ajustó a pH 6,8-7 con NaOH 0,5M (Anedra, Argentina) y se sometió a un calentamiento a 100°C durante 5min. Posteriormente, los SLC se filtraron utilizando membranas Sartorius de 0,22μm de diámetro de poro (Sartorius, Alemania) y se almacenaron a −30°C hasta la realización de los ensayos. La actividad de los SLC contra Listeria innocua ATCC 33090 se determinó por el método de difusión en placa, según lo descrito por Vallejo et al.44. Con el propósito de evaluar el espectro de inhibición de las cepas E. hirae 463Me y 471Me, se utilizaron los microorganismos indicadores en las condiciones de crecimiento mencionadas en la tabla 2.
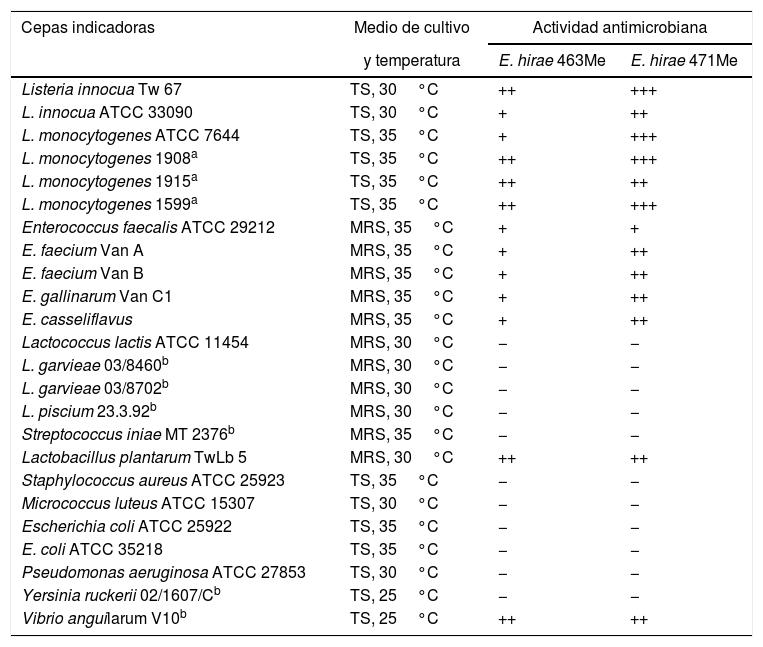
Actividad antimicrobiana de aislamientos de Enterococcus hirae obtenidos de mejillón patagónico
| Cepas indicadoras | Medio de cultivo | Actividad antimicrobiana | |
|---|---|---|---|
| y temperatura | E. hirae 463Me | E. hirae 471Me | |
| Listeria innocua Tw 67 | TS, 30°C | ++ | +++ |
| L. innocua ATCC 33090 | TS, 30°C | + | ++ |
| L. monocytogenes ATCC 7644 | TS, 35°C | + | +++ |
| L. monocytogenes 1908a | TS, 35°C | ++ | +++ |
| L. monocytogenes 1915a | TS, 35°C | ++ | ++ |
| L. monocytogenes 1599a | TS, 35°C | ++ | +++ |
| Enterococcus faecalis ATCC 29212 | MRS, 35°C | + | + |
| E. faecium Van A | MRS, 35°C | + | ++ |
| E. faecium Van B | MRS, 35°C | + | ++ |
| E. gallinarum Van C1 | MRS, 35°C | + | ++ |
| E. casseliflavus | MRS, 35°C | + | ++ |
| Lactococcus lactis ATCC 11454 | MRS, 30°C | − | − |
| L. garvieae 03/8460b | MRS, 30°C | − | − |
| L. garvieae 03/8702b | MRS, 30°C | − | − |
| L. piscium 23.3.92b | MRS, 30°C | − | − |
| Streptococcus iniae MT 2376b | MRS, 35°C | − | − |
| Lactobacillus plantarum TwLb 5 | MRS, 30°C | ++ | ++ |
| Staphylococcus aureus ATCC 25923 | TS, 35°C | − | − |
| Micrococcus luteus ATCC 15307 | TS, 30°C | − | − |
| Escherichia coli ATCC 25922 | TS, 35°C | − | − |
| E. coli ATCC 35218 | TS, 35°C | − | − |
| Pseudomonas aeruginosa ATCC 27853 | TS, 30°C | − | − |
| Yersinia ruckerii 02/1607/Cb | TS, 25°C | − | − |
| Vibrio anguilarum V10b | TS, 25°C | ++ | ++ |
Halo de inhibición:+, ≥10mm;++, ≥15mm;+++, ≥20mm; −, sin inhibición.
Los cebadores y protocolos utilizados en las reacciones de PCR para la amplificación de los genes estructurales de las enterocinas A, B, P, LB50A/LB50B, bact 91, bact 31, 1071 A/B, ent Q, hiracin JM79 y mun KS se mencionan en la tabla 3.
Cebadores específicos empleados para detectar en aislamientos de Enterococcus hirae obtenidos de mejillón patagónico genes estructurales de enterocinas
| Enterocinas | Secuencia (5́-3́) | Fragmento (pb) | Referencia |
|---|---|---|---|
| Ent A | f: GGTACCACTCATAGTGGAAA | 138 | De Vuyst et al.11 |
| r: CCCTGGAATTGCTCCACCTAA | |||
| Ent B | f: CAAAATGTAAAAGAATTAAGTACG | 201 | De Vuyst et al.11 |
| r: AGAGTATACATTTGCTAACCC | |||
| Ent P | f: GCTACGCGTTCATATGGTAAT | 87 | De Vuyst et al.11 |
| r: TCCTGCAATATTCTCTTTAGC | |||
| Ent LB50A | f: ATGGGAGCAATCGCAAAATTA | 274 | De Vuyst et al.11 |
| r: TTTGTTAATTGCCCATCCTTC | |||
| Ent LB50B | f: ATGGGAGCAATCGCAAAATTA | 274 | De Vuyst et al.11 |
| r: TAGCCATTTTTCAATTTGATC | |||
| Bact 96 | f: GTGGAGAGGACGAAAGGAGA | 291 | Henning et al.21 |
| r: TTGATTAGTGGAGAGGACGGATTA | |||
| Bact 31 | f: CCTACGTATTACGGAAATGGT | 130 | Özdemir et al.30 |
| r: GCCATGTTGTACCCAACCATT | |||
| 1071 A/B | f: GGGGAGAGTCGGTTTTTAG | 273 | Martín et al.28 |
| r: ATCATATGCGGGTTGTAGCC | |||
| EntQ | f: ATGAATTTTCTTCTTAAAAATGGTATCGCA | 105 | Belgacem et al.5 |
| r: TTAACAAGAAATTTTTTCCCATGGCAA | |||
| mun KS | f: TGAGAGAAGGTTTAAGTTTTGAAGAA | 379 | Zendo et al.46 |
| r: TCCACTGAAATCCATGAATGA | |||
| HirJM 79 | f: ATGAAAAAGAAAGTATTAAAACATTGTGTTATTCTAGG | 408 | Almeida et al.2 |
| r: ATAAGTTAAGCTTGTACTACCTTCTAGGTGCCCATGGACC |
Ent A: enterocina A; Ent B: enterocina B; Ent P: enterocina P; Ent LB50A/50B: enterocina LB50A/B; Bact 96: bacteriocina 96; Bact 31: bacteriocina 31; 1071 A/B: enterocina 1071 A/B; EntQ: enterocina Q; mun KS: mundticina KS; HirJM 79: hiracina JM 79.
Se determinó la curva de crecimiento y la actividad de las bacteriocinas producidas a 35°C durante 24h. Se tomaron muestras en forma aséptica para determinar la actividad antimicrobiana y la densidad óptica (DO) a 600nm en un espectrofotómetro Jenway 6400. La actividad se definió como la recíproca de la dilución más alta que exhibió inhibición completa de L. innocua ATCC 33090 y se expresó en unidades arbitrarias (UA) por mililitro de SLC.
Resultados y discusiónLas dos cepas en estudio se aislaron del contenido intestinal de ejemplares de mejillón patagónico (Mytilus edulis platensis) capturados en las costas de la ciudad de Comodoro Rivadavia (latitud −45,83; longitud −67,46) y Rada Tilly (latitud −45,92; longitud −67,55, respectivamente). Ambos microorganismos resultaron ser cocos gram positivos, catalasa y oxidasa negativos, que exhibieron crecimiento a 10°C y a 45°C, a pH 9,6; en caldo MRS suplementado con NaCl al 6,5% y mostraron actividad de leucina aminopeptidasa y de pirrolidonil arilamidasa.
La identificación fenotípica se completó mediante las pruebas sugeridas por Manero y Blanch27, un esquema de reacciones bioquímicas que permite la clasificación de los miembros del género Enterococcus a nivel de especie. Ambas cepas fermentaron la arabinosa, el manitol y el metil-α-D glucopiranósido, además presentaron actividad enzimática de arginina deshidrolasa y α-galactosidasa, reacciones que permitieron clasificarlas como E. hirae.
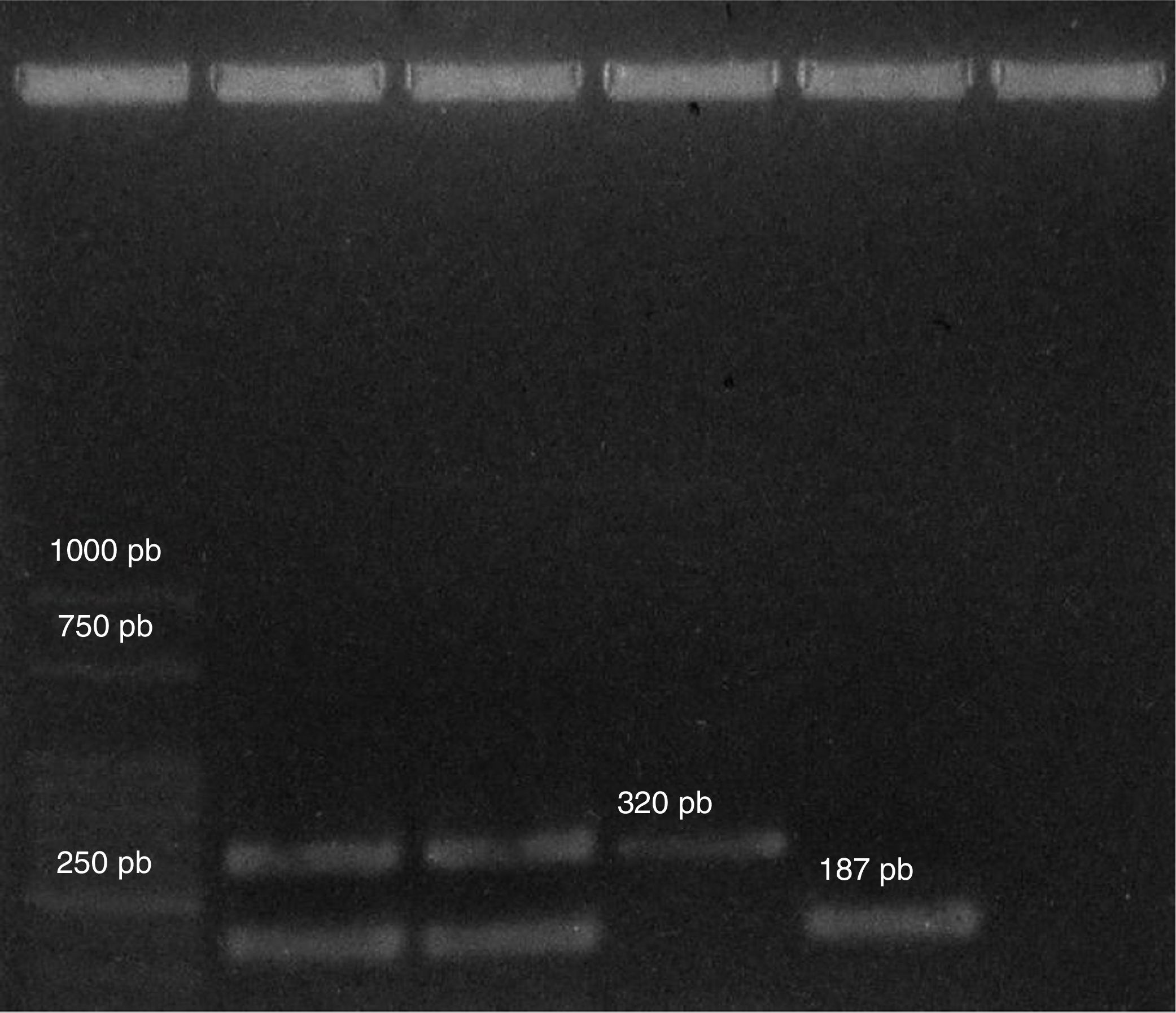
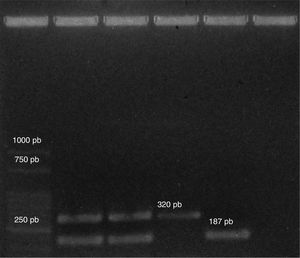
En la figura 1 se pueden observar los productos obtenidos tras la amplificación del material genético por PCR multiplex utilizando los cebadores específicos para género y especie. Se obtuvieron dos bandas, una de aproximadamente 320pb, en correspondencia con la obtenida con E. faecalis ATCC 29212, que fue utilizada como control de género, y la otra de aproximadamente 187pb, coincidente con la banda obtenida con E. hirae F6 (N.° de acceso GenBank: FJ892740), utilizada como control de especie. Los resultados obtenidos mediante las técnicas moleculares descritas permitieron confirmar los obtenidos mediante las pruebas bioquímicas.
E. hirae ha sido descrito como patógeno zoonótico, raramente aislado en infecciones humanas. La revisión bibliográfica permite encontrar un número escaso de reportes que señalan el aislamiento de este microorganismo a partir de humanos y, en todos los casos, asociado a la presencia de enfermedades subyacentes6,40,42. El primer informe de infección humana causada por E. hirae fue publicado por Gilad et al.15 en 1998; este describía un caso de septicemia en un hombre de 49 años con insuficiencia renal tratada con hemodiálisis.
Las cepas cuyo aislamiento se informa en trabajos previos fueron sensibles a los antibióticos de uso común en la clínica diaria, datos coincidentes con los obtenidos en este estudio. Las dos cepas aisladas del contenido intestinal del mejillón patagónico fueron sensibles a ampicilina, amoxicilina-clavulánico, trimetoprima-sulfametoxazol, rifampicina y vancomicina (datos no mostrados). La resistencia observada contra cefalotina y eritromicina, por tratarse de microorganismos de origen ambiental, podría ser interpretada como un fenómeno intrínseco, no vinculado con la transmisión de elementos móviles. En el futuro y dado el potencial uso biotecnológico de estas cepas, se deberán realizar estudios complementarios para confirmar la ausencia de genes relacionados con resistencias transmisibles.
La prueba de hemólisis en placa resultó negativa en ambas cepas (datos no mostrados), lo que podría ser contradictorio con los obtenidos por amplificación de los genes cylLl y cylLs, que resultaron positivos en ambos casos. La presencia de uno o más genes es un rasgo ampliamente distribuido dentro del género Enterococcus12; sin embargo, no es una condición suficiente para lograr la expresión de la toxina. El operón cyl está constituido por 6 genes vinculados con la expresión, la maduración, la secreción, la activación y la inmunidad. Las pruebas fenotípicas y la detección de genes cyl revelan que la actividad de la toxina está directamente vinculada con la presencia del operón completo38. El ensayo en placa continúa siendo la «prueba de oro» para detectar la presencia de la citolosina38.
No se obtuvieron productos de amplificación por PCR de los genes agg (proteína de agregación), gelE (gelatinasa), esp (proteína de superficie), hylEfm (hialuronidasa) e IS16 (elemento de inserción). No se detectó actividad gelatinasa en la prueba en placa (datos no mostrados), hecho congruente con el de PCR.
El primer reporte de E. hirae productor de bacteriocina fue el de Siragusa, en 199241; este investigador aisló una cepa con estas características del intestino bovino. Lamentablemente no hay datos adicionales en este trabajo que describan qué tipo de bacteriocina era la responsable de la actividad antagonista observada. El autor la denominó hiraecina S y en el trabajo solo se reportan datos sobre su caracterización fisicoquímica.
En este sentido, en los bancos de datos específicos de bacteriocinas, hasta la fecha hay un solo registro confirmado sobre una cepa de E. hirae productora de hiraecina JM7936. En 2016, Gupta et al.19 publicaron un trabajo en donde se sugiere la purificación de una nueva bacteriocina producida por una cepa de E. hirae denominada enterocina LD3. Sin embargo, no han sido publicados datos adicionales sobre su secuenciamiento completo o datos genéticos que corroboren dicho hallazgo19.
Se han publicado otros trabajos donde se reportan aislamientos de cepas de E. hirae bacteriocinogénicas identificadas por técnicas moleculares1,3,20, pero no hay precisiones sobre las características del principio activo y, en consecuencia, no es posible realizar afirmaciones concluyentes.
El material genético de las cepas E. hirae 463Me y E. hirae 471Me se amplificó usando cebadores específicos (tabla 3) a fin de determinar la presencia de genes relacionados con la producción de bacteriocinas. Los resultados obtenidos revelaron la presencia de los genes estructurales de la enterocina P y de la hiraecina JM79 en la cepa 471Me y solo del gen de la enterocina P en la cepa 463Me. Ambas bacteriocinas pertenecen a la clase IIa, tipo pediocina, y exhiben actividad inhibitoria contra cepas del género Listeria como principal característica13.
En la tabla 2 se presenta el espectro de actividad de los sobrenadantes de ambas cepas. Como en otros casos de bacteriocinas de BAL tipo IIa, se observó actividad contra todas las cepas de L. innocua y L. monocytogenes ensayadas. En este sentido, también los SLC exhibieron antagonismo contra Lactobacillus plantarum TwLb5 y todas las cepas de Enterococcus utilizadas como blanco. Resulta un hecho especialmente llamativo la sensibilidad a estas bacteriocinas de las tres cepas resistentes a vancomicina (Van A, Van B y Van C). Sin embargo, los principios activos no resultaron efectivos contra Lactococcus garvieae 03/8460, L. garvieae 03/8702, Lactococcus piscium 23.3.92 y Streptococcus iniae MT 2376, cocos gram positivos y patógenos habituales en acuicultura. Además, los SLC no inhibieron a la cepa productora de nisina Lactococcus lactis ATCC 11454.
Dentro del espectro de bacilos gram negativos ensayados, solo Vibrio anguilarum V10, un patógeno de peces de mar, resultó sensible a la actividad de ambos sobrenadantes. En todos estos casos, exceptuando el de la cepa E. faecalis ATCC 29212, E. hirae 471Me desarrolló una actividad más intensa que E. hirae 463Me.
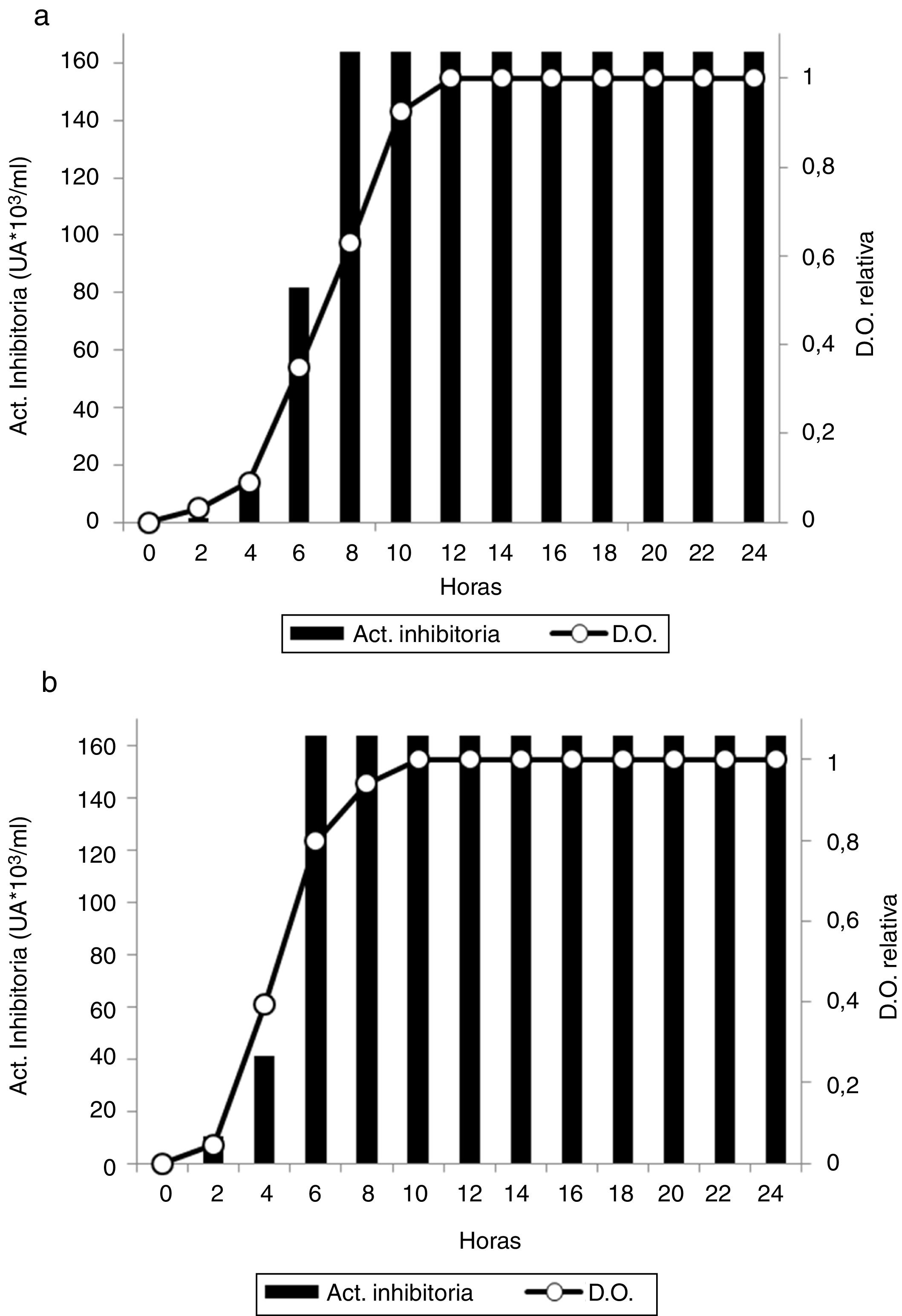
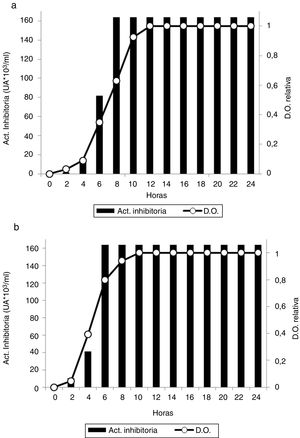
En la figura 2a se puede observar la curva de crecimiento y la actividad inhibitoria de la cepa E. hirae 463Me en condiciones óptimas de crecimiento (35°C en caldo MRS). Se determinó una fase lag de aproximadamente 4h y la máxima densidad poblacional se alcanzó a las 12h de incubación, sin que se registraran variaciones hasta las 24h. La producción de metabolitos antagónicos se detectó a las 2h y alcanzó el máximo a las 8h de incubación; dicho valor se mantuvo estable hasta las 24h de ensayo.
La cepa 471Me (fig. 2b), bajo las mismas condiciones de cultivo, exhibió una fase lag más breve y alcanzó el máximo de población a las 10h de comenzado el ensayo; asimismo, se mantuvo estable hasta las 24h. Como en el caso anterior, la actividad inhibitoria se detectó a las 2h de incubación y el máximo a las 6h, sin registros de variación hasta las 24h.
Utilizando L. innocua ATCC 33090 como bacteria blanco, en ambos casos se determinó una actividad máxima de 163.840UA/ml, un valor nunca antes logrado en nuestra experiencia con cepas de Enterococcus aisladas del medio ambiente. Resulta difícil comparar la actividad antagonista con otras cepas de E. hirae bacteriocinogénicas reportadas en la literatura científica debido a diferencias en la metodología empleada y el espectro de cepas indicadoras.
ConclusionesLa búsqueda bibliográfica efectuada permite afirmar que son escasas las cepas de E. hirae productoras de bacteriocinas aisladas hasta este momento. En consecuencia, creemos que son importantes los hallazgos aquí documentados, ya que sería este el primer reporte en el que se informa el aislamiento de estos microorganismos del medio marino y, concretamente, del contenido intestinal de mejillón patagónico.
El gen de la enterocina P se detectó en ambas cepas y el de la hiraecina JM79 solo en la cepa 471Me, pero dado lo particular del nicho ecológico del que provienen estos microorganismos, sería conveniente realizar estudios genéticos complementarios que pudieran dar información sobre la potencial presencia de una nueva bacteriocina.
Ambas cepas exhibieron una marcada actividad antagonista y con las pruebas fenotípicas y genotípicas efectuadas no revelaron la presencia de factores de virulencia; estas propiedades las convierten en potencialmente útiles para su uso en la conservación de alimentos o como probióticos para acuicultura.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
FinanciaciónEste trabajo se financió con fondos otorgados por la Agencia Nacional de Promoción Científica y Tecnológica (PICT-2014-0575) y UNPSJB (SCT PI N.° 1341).