Staphylococcus aureus resistente a la meticilina adquirido en la comunidad (SARM-AC) es uno de los principales patógenos causantes de infecciones de piel y partes blandas, aunque también se lo implica en infecciones graves, como osteomielitis y neumonía. El objetivo de este estudio descriptivo fue determinar el tipo de cassette SCCmec, el perfil de virulencia y la variabilidad genética de 21 aislamientos de SARM-AC que infectaron a niños paraguayos en el año 2010. Se determinó por PCR el tipo de cassette SCCmec y los factores de virulencia, en tanto que la variabilidad genética se determinó por la técnica multiple locus variable analysis (MLVA). Todos los aislamientos (100%) presentaron cassette SCCmeciv; algunos portaron factores de virulencia como hla, hlb y sea (el 28,6, el 9,5 y el 4,8%, respectivamente). El análisis MLVA mostró gran variabilidad genética, con datos de antibiotipo y perfil de virulencia congruentes. Este trabajo pone de manifiesto por primera vez en Paraguay la presencia de SARM-AC portador del cassette SCCmeciv con elevada diversidad genética.
Community-acquired methicillin-resistant Staphylococcus aureus (CA-MRSA) is one of the first causes of skin and soft tissue infections, and can also produce severe diseases such as osteomyelitis and pneumonia. The aim of this descriptive study was to determine the SCCmec type and virulence profile and to study the genetic diversity by MLVA analysis of 21 CA-MRSA isolates that infected Paraguayan children in 2010. The SCCmec type and virulence factors were performed by PCR and genetic diversity by MLVA (multiple locus variable analysis). All the isolates carried SCCmec cassette iv. hla, hlb and sea genes were detected in 28,6%, 9,5% and 4,8% respectively. The MLVA analysis showed high genetic diversity with congruent antibiotic resistance and virulence profiles. This study revealed the presence of CA-MRSA harbouring SCCmeciv with high genetic diversity, providing information not available in our country.
Staphylococcus aureus resistente a la meticilina adquirido en la comunidad (SARM-AC) es uno de los principales patógenos causantes de infecciones de piel y partes blandas, aunque también puede producir infecciones graves, como bacteriemia, osteomielitis y neumonía necrosante3. Los aislamientos resistentes a la meticilina se caracterizan por portar los genes mecA, mecB o mecC, que son transportados por un elemento genético móvil denominado staphylococcal cassette chromosome element (SCC). Algunos tipos de SCCmec son más frecuentes en aislamientos de origen comunitario, lo que los convierte en una importante herramienta para el estudio de la epidemiología de los aislamientos4.
La capacidad de S. aureus de portar genes de resistencia a antibióticos y una amplia gama factores de virulencia, así como su gran variabilidad genética, le confieren una epidemiología compleja, por lo que es necesario contar con metodologías que permitan la monitorización y la vigilancia de estos aislamientos en tiempo real.
La técnica conocida como multiple locus variable analysis (MLVA) es una metodología que permite discriminar entre diferentes aislamientos clínicos de S. aureus empleando una reacción de PCR múltiple, que amplifica 7 regiones intragénicas con número variable de repeticiones en tándem. Este método, de bajo costo, rápida ejecución y fácil interpretación, ha sido validado a nivel internacional y tiene un poder discriminatorio comparable con la pulse field gel electrophoresis (PFGE), considerada como el gold standard para la tipificación molecular de S. aureus11,13.
En Paraguay, los datos publicados sobre S. aureus se limitan a un reporte de circulación del clon cordobés/chileno y del clon brasileño en aislamientos que provenían de adultos6, y al estudio de un brote epidémico asociado al consumo de leche contaminada con S. aureus portador de enterotoxinas15.
En referencia a la población pediátrica, un estudio de 113 aislamientos de S. aureus asociado a la comunidad mostró un 18% de resistencia a la meticilina y generó los primeros datos de perfil de virulencia, entre los que se destacó la portación de la leucocidina de Panton-Valentine (PVL) como principal factor de virulencia en el 58% del total de las muestras analizadas12.
El objetivo de este trabajo consistió en determinar el tipo de cassette SCCmec y el perfil de virulencia de 21 aislamientos de SARM-AC que infectaron a niños paraguayos menores de 15 años y en estudiar por MLVA su variabilidad genética.
Los aislamientos fueron colectados de 4 hospitales de referencia, que dan cobertura de servicios de salud a más del 50% de la población de Asunción y del Departamento Central durante el año 2010. Estos aislamientos se encontraban preservados en el biobanco del Instituto de Investigaciones en Ciencias de la Salud (IICS) como resultado de proyectos iniciales limitados a caracterizaciones clínicas y microbiológicas. El protocolo de estudio contó con la aprobación del Comité Científico y el Comité de Ética del IICS, UNA (P44/2012).
Se definió como infección por SARM-AC cualquier infección por SARM diagnosticada a un paciente ambulatorio o a uno internado si el diagnóstico fue dentro de las 48 h de hospitalización, teniendo en cuenta que el paciente no presentara otra enfermedad de base o crónica que hubiera requerido su exposición previa al ambiente hospitalario7. Se excluyeron los aislamientos provenientes de puntas de catéter y secreción traqueal, para evitar fenómenos de portación.
Se contaba con datos clínicos y demográficos de los niños infectados. La sensibilidad a antibióticos se evaluó por el método de Kirby Bauer, según el criterio del CLSI2. Los aislamientos criopreservados fueron cultivados en agar sangre de carnero al 5% e incubados a 35°C durante 24-48 h en atmósfera del 5% de CO2. La extracción de ADN se realizó empleando el kit Wizard Genomics (Wizard Genomic DNA, Purification Kit, Promega, Madison, EE. UU.) y se cuantificó con espectrofotómetro Biowave DNA (Biochrom WPA Biowave DNA, Life Science Spectrophotometer, Biochrom, Cambridge, Reino Unido).
La PCR para la detección de mecA y lukS/F-PV se realizó según Carpinelli et al.1. Los genes codificantes de enterotoxinas (sea, seb, sec, sed, seh) y el control interno 16S ARNr de hemolisinas (hla y hlb) y toxinas exfoliativas (eta y etb) se amplificaron según lo describen Rodríguez et al.12. La identificación del tipo de cassette SCCmec se realizó con las condiciones de Kondo et al.4.
El análisis MLVA se realizó con las condiciones descritas por Sabat et al.13, con algunas modificaciones11. El tamaño de los productos se obtuvo por digitalización con el software UN SCAN IT GEL 6.1; estos datos se emplearon para realizar un dendrograma mediante el uso del software Treeview versión X 0.5.1.
Del total de muestras del biobanco (n=113), 21 aislamientos SARM-AC cumplieron los criterios de inclusión antes descritos. Esos aislamientos infectaron a 8 niñas y 13 niños, cuyas edades se encontraban comprendidas entre 1 mes y 15 años (mediana: 4 años; rango intercuartílico: 1 a 11 años).
La distribución de los aislamientos correspondió al Hospital General Pediátrico (HGP) (n=10), el Hospital de Clínicas (n=6), el Instituto de Previsión Social (IPS) (n=3) y el Hospital Nacional de Itauguá (n=2). La mayor frecuencia de aislamiento se vio en el verano (57,1%). El 86% (n=18) de los aislamientos causó infecciones de piel y partes blandas, mientras que el 14% (n=3) restante causó cuadros invasivos. El 100% de los aislamientos fue sensible a vancomicina, tetraciclina, rifampicina, ciprofloxacina, trimetroprima-sulfametoxazol, teicoplanina y novobiocina. Los niveles de resistencia a cloranfenicol, eritromicina, gentamicina y clindamicina fueron, respectivamente, del 3,3, el 19,0, el 14,3 y el 9,5%.
El gen mecA dentro del cassette SCCmec de tipo iv fue detectado en el 100% de los aislamientos y el gen codificante de la PVL en el 33,3%.
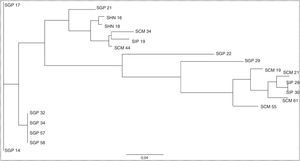
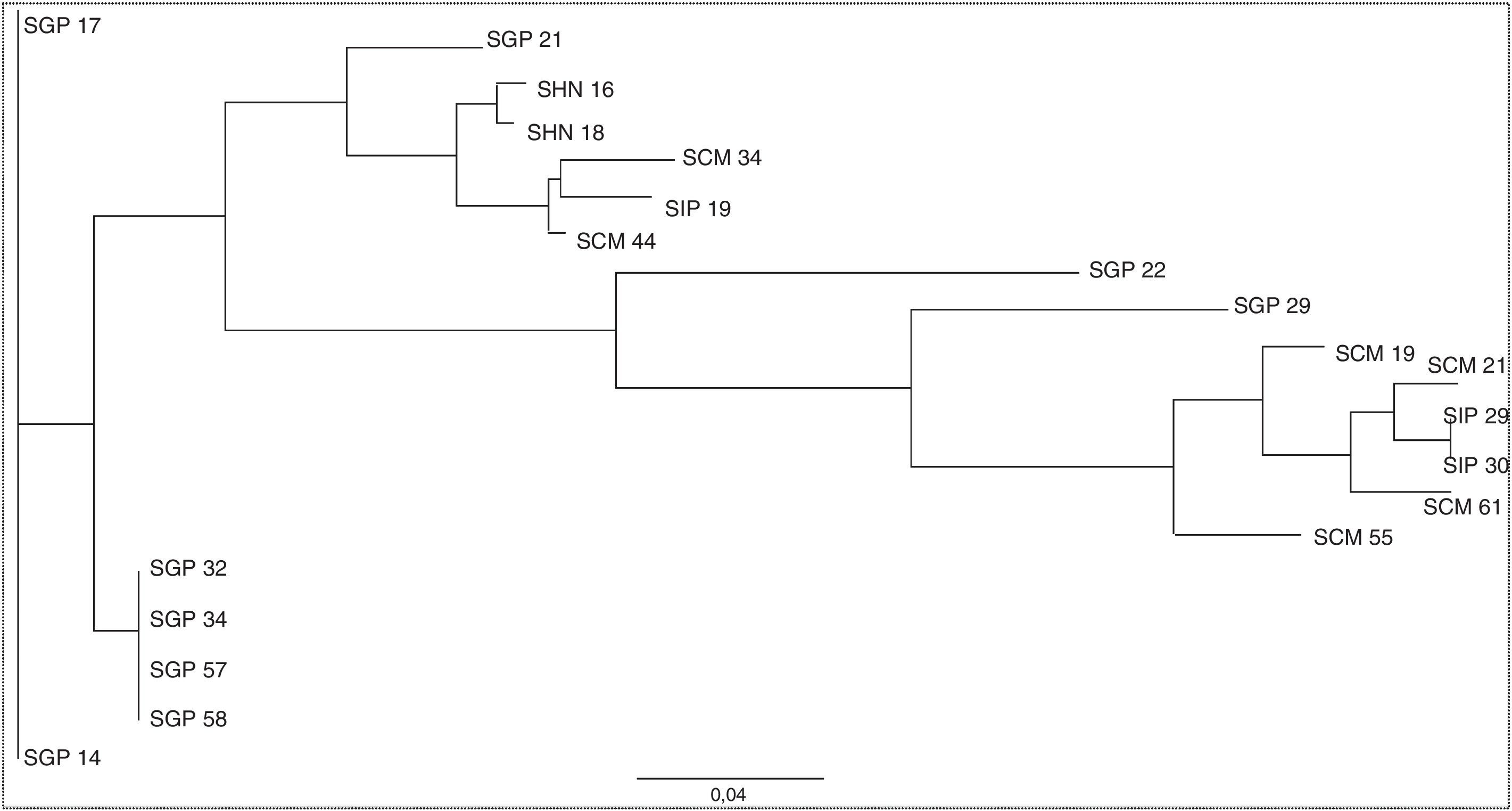
El análisis MLVA de 20 aislamientos mostró 15 perfiles distintos (fig. 1). Un aislamiento no pudo ser tipificado debido a la falta de amplificación en un locus. En el perfil 2 se observaron 4 aislamientos con el mismo patrón de bandas, los aislamientos presentes en este grupo fueron obtenidos en el HGP en los meses de febrero, julio y octubre del 2010; estos no portaban factores de virulencia y eran resistentes a la meticilina.
Perfil MLVA de aislamientos SARM-AC estudiados (n=21). Electroforesis en gel de poliacrilamida con tinción de nitrato de plata de algunos (n=10) de los productos obtenidos por la técnica MLVA. Carril: 1) SGP14; 2) SGP17; 3) SGP11, el cual no pudo ser tipificado (se observa amplificación parcial de los locus estudiados); 4) SGP21; 5) SGP22; 6) SGP29; 7) marcador de PM de 50 pb Fermentas (GeneRuler 50 bp DNA Ladder, Fermentas [ThermoFisher], Waltham, Massachusetts, EE. UU.); 8) SGP32; 9) SGP34; 10) SGP57 y 11) SGP58.
Los perfiles 1 y 15 agruparon 2 aislamientos cada uno. Los aislamientos componentes del perfil 1 provenían del HGP y se obtuvieron entre enero y octubre del 2010; eran resistentes a meticilina, uno de ellos presentaba PVL y ambos originaron infecciones de piel y partes blandas. Los del perfil 15 fueron aislados en el IPS en enero y marzo, y se caracterizaron por ser resistentes a la meticilina, y el cloranfenicol; a su vez, presentaron PVL y hemolisina A y B. Los perfiles restantes incluían un único aislamiento cada uno; las características de los mismos se describen en la tabla 1.
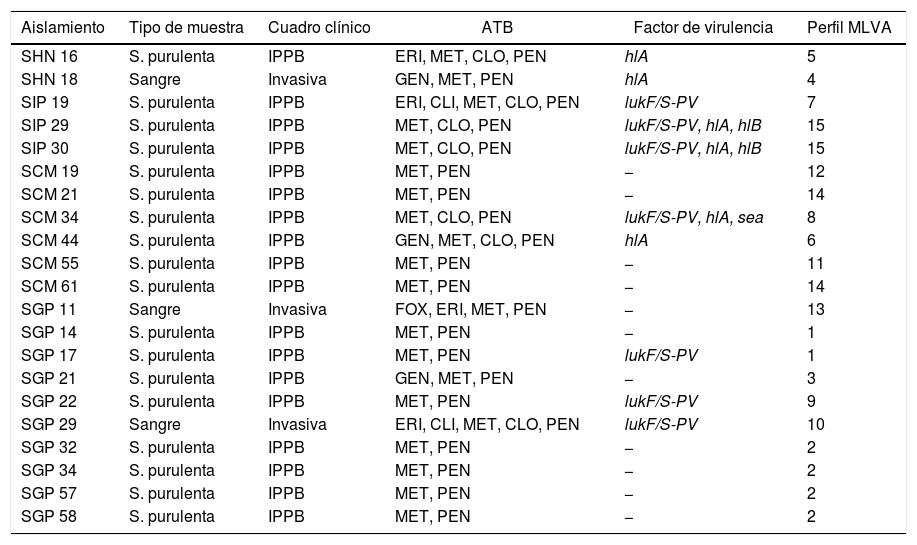
Características de los aislamientos SARM-AC en estudio (n=21)
| Aislamiento | Tipo de muestra | Cuadro clínico | ATB | Factor de virulencia | Perfil MLVA |
|---|---|---|---|---|---|
| SHN 16 | S. purulenta | IPPB | ERI, MET, CLO, PEN | hlA | 5 |
| SHN 18 | Sangre | Invasiva | GEN, MET, PEN | hlA | 4 |
| SIP 19 | S. purulenta | IPPB | ERI, CLI, MET, CLO, PEN | lukF/S-PV | 7 |
| SIP 29 | S. purulenta | IPPB | MET, CLO, PEN | lukF/S-PV, hlA, hlB | 15 |
| SIP 30 | S. purulenta | IPPB | MET, CLO, PEN | lukF/S-PV, hlA, hlB | 15 |
| SCM 19 | S. purulenta | IPPB | MET, PEN | − | 12 |
| SCM 21 | S. purulenta | IPPB | MET, PEN | − | 14 |
| SCM 34 | S. purulenta | IPPB | MET, CLO, PEN | lukF/S-PV, hlA, sea | 8 |
| SCM 44 | S. purulenta | IPPB | GEN, MET, CLO, PEN | hlA | 6 |
| SCM 55 | S. purulenta | IPPB | MET, PEN | − | 11 |
| SCM 61 | S. purulenta | IPPB | MET, PEN | − | 14 |
| SGP 11 | Sangre | Invasiva | FOX, ERI, MET, PEN | − | 13 |
| SGP 14 | S. purulenta | IPPB | MET, PEN | − | 1 |
| SGP 17 | S. purulenta | IPPB | MET, PEN | lukF/S-PV | 1 |
| SGP 21 | S. purulenta | IPPB | GEN, MET, PEN | − | 3 |
| SGP 22 | S. purulenta | IPPB | MET, PEN | lukF/S-PV | 9 |
| SGP 29 | Sangre | Invasiva | ERI, CLI, MET, CLO, PEN | lukF/S-PV | 10 |
| SGP 32 | S. purulenta | IPPB | MET, PEN | − | 2 |
| SGP 34 | S. purulenta | IPPB | MET, PEN | − | 2 |
| SGP 57 | S. purulenta | IPPB | MET, PEN | − | 2 |
| SGP 58 | S. purulenta | IPPB | MET, PEN | − | 2 |
S. purulenta: secreción purulenta; IPPB: infección de piel y partes blandas; ATB: antibiograma; FOX: cefoxitina; ERI: eritromicina; GEN: gentamicina; CLI: clindamicina; MET: meticilina; CLO: cloranfenicol; PEN: penicilina; hlA: hemolisina alfa; hlB: hemolisina beta; lukF/S-PV: leucocidina de Panton-Valentine; sea: enterotoxina A.
En la figura 2 se muestra el dendrograma obtenido; se observan las 3 agrupaciones descritas (perfiles 1, 2 y 15), que engloban 2 (SGP 14 y GP 17), 4 (SGP 32, SGP 34, SGP 57 y SGP 58) y 2 (SIP 29 y SIP 30) aislamientos, respectivamente.
Dendograma de aislamientos SARM-AC estudiados (n=21).
Dendograma obtenido utilizando software Treeview versión X 0.5.1. Aislamientos con mismos perfiles MLVA: 1) SGP 14 y SGP 17; 2) SGP 32, SGP 34, SGP 57 y SGP 58; 15) SIP 29 y SIP 30. Se observa el dendrograma con el total de aislamientos tipificables por MLVA (95%, n=20).
Este es el primer reporte de SARM-AC con cassette SCCmec de tipo iv en Paraguay. Los datos publicados hasta el momento hacían referencia al clon cordobés/chileno (cassette SCCmec tipo i) y al clon brasileño (cassette SCCmec tipo iii)6. Si bien no contamos con datos de spa-tipo y secuenciotipo MLST para poder definir a qué clones pertenecen estos aislamientos (lo que constituye una limitante de este trabajo), los aislamientos estudiados muestran diferencias genotípicas con respecto a los anteriormente descritos en Paraguay, por la portación del cassette SCCmec tipo iv. Países como Argentina y Uruguay ya han reportado la circulación de los clones SARM-AC emergentes ST30/t019 y ST5/t311, portadores del cassette SCCmec tipo iv3,10,14.
La tasa de resistencia a los antimicrobianos de los aislamientos SARM-AC estudiados en este trabajo fue en general baja y similar a la informada en otros estudios realizados en la región8,9.
La frecuencia de portación de genes codificantes de factores de virulencia, como la PVL y las hemolisinas, fue comparable con la de otros estudios similares. Varios autores señalan la importancia de identificar aislamientos portadores de estos genes, por la capacidad que tienen de generar cuadros graves5,15. Los datos obtenidos amplían la escasa información sobre los factores de virulencia que caracterizan a los SARM-AC que circulan en Paraguay.
Los resultados de la MLVA mostraron una variabilidad genética alta, con 15 perfiles diferentes, de los cuales solo 3 contenían 2 o más aislamientos. La concordancia del antibiotipo y del perfil de virulencia de los aislamientos agrupados fue buena.
Varios trabajos señalan como ventajas del MLVA su capacidad para manejar un alto flujo de muestras y la similitud de las agrupaciones que se obtienen con respecto a las generadas por el MLST, lo que, sumado a la fácil interpretación de los resultados, la convierten en una técnica de alto valor en estudios epidemiológicos11,13. Es importante señalar que en Paraguay, el acceso a métodos de análisis como PGFE es muy restringido, con lo cual el uso del MLVA puede ser una alternativa para estimar la diversidad genética de los aislamientos SARM-AC.
Este trabajo pone de manifiesto la presencia de SARM-AC portadores de cassette SCCmec de tipo iv, con perfiles de virulencia variables y una elevada diversidad genética, como agentes causales de infecciones en niños que viven en Asunción y en el Departamento Central del Paraguay. De esta manera, provee información no disponible hasta el momento en esta región. La identificación de los clones a los que pertenecen estos aislamientos es nuestra prioridad. Esperamos poder profundizar los análisis moleculares de estos aislamientos en un futuro próximo.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datosLos autores declaran que han seguido los protocolos de su centro de trabajo sobre la publicación de datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores declaran que en este artículo no aparecen datos de pacientes.
FinanciaciónEl presente estudio fue llevado a cabo con fondos propios de los investigadores.
Conflicto de interesesLos autores declaran no tener conflictos de interés.





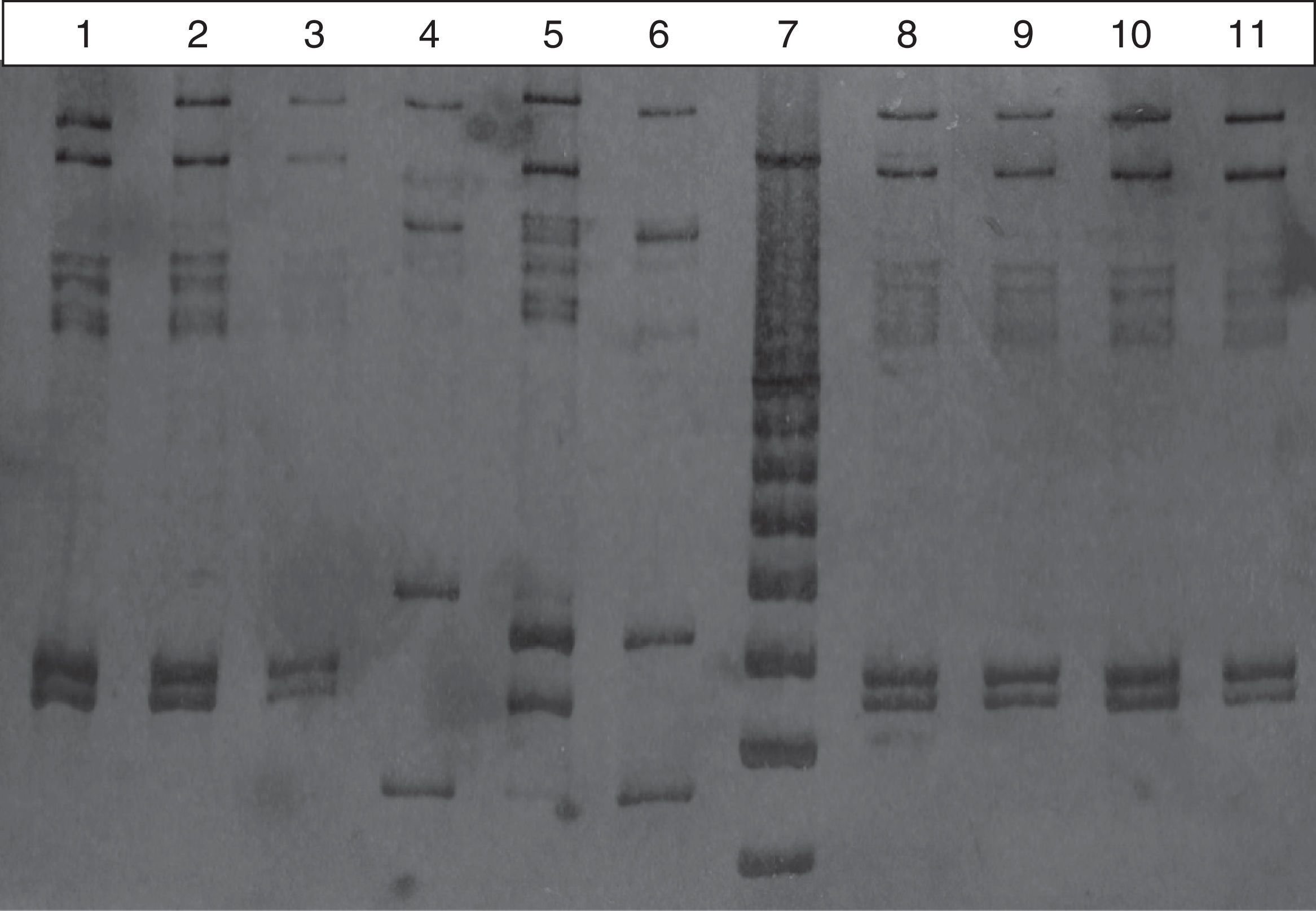
![Perfil MLVA de aislamientos SARM-AC estudiados (n=21). Electroforesis en gel de poliacrilamida con tinción de nitrato de plata de algunos (n=10) de los productos obtenidos por la técnica MLVA. Carril: 1) SGP14; 2) SGP17; 3) SGP11, el cual no pudo ser tipificado (se observa amplificación parcial de los locus estudiados); 4) SGP21; 5) SGP22; 6) SGP29; 7) marcador de PM de 50 pb Fermentas (GeneRuler 50 bp DNA Ladder, Fermentas [ThermoFisher], Waltham, Massachusetts, EE. UU.); 8) SGP32; 9) SGP34; 10) SGP57 y 11) SGP58. Perfil MLVA de aislamientos SARM-AC estudiados (n=21). Electroforesis en gel de poliacrilamida con tinción de nitrato de plata de algunos (n=10) de los productos obtenidos por la técnica MLVA. Carril: 1) SGP14; 2) SGP17; 3) SGP11, el cual no pudo ser tipificado (se observa amplificación parcial de los locus estudiados); 4) SGP21; 5) SGP22; 6) SGP29; 7) marcador de PM de 50 pb Fermentas (GeneRuler 50 bp DNA Ladder, Fermentas [ThermoFisher], Waltham, Massachusetts, EE. UU.); 8) SGP32; 9) SGP34; 10) SGP57 y 11) SGP58.](https://static.elsevier.es/multimedia/03257541/0000005000000002/v1_201806010416/S0325754117301104/v1_201806010416/es/main.assets/thumbnail/gr1.jpeg?xkr=ue/ImdikoIMrsJoerZ+w96p5LBcBpyJTqfwgorxm+Ow=)