Las pérdidas reproductivas constituyen una causa importante de pérdida económica en el ganado bovino, aunque en más del 50% de los casos la etiología es desconocida. Las especies de la familia Chlamydiaceae han sido asociadas con abortos en bovinos y otras especies animales, pero no existen datos al respecto en la República Argentina. El objetivo de este trabajo fue determinar la presencia de Chlamydia spp. y de Chlamydia abortus en pérdidas reproductivas de ganado bovino en La Pampa, Argentina. Se estudiaron 251 muestras provenientes de abortos y mortinatos. Se realizó PCR en tiempo real para la detección de la familia Chlamydiaceae y ArrayTube para la identificación de las especies presentes. Se detectó ADN de la familia Chlamydiaceae en 12 muestras (4,78%); el 83,33% (10/12) correspondió a abortos y el 16,66% (2/12) a mortinatos. El análisis por ArrayTube detectó C. abortus en 5 muestras (1,99% del total, 41,67% de las muestras con detección de Chlamydiaceae). Este trabajo presenta la primera confirmación de la presencia de ADN de diversas especies de Chlamydiaceae (incluida C. abortus) en muestras de pérdidas reproductivas de ganado bovino en Argentina. El valor de prevalencia hallado (4,78%) debe ser tomado como un valor basal, debido al tipo de muestras estudiadas. Se halló material genético de Chlamydiaceae que no coincidió con ninguna de las especies conocidas; esto podría deberse a variantes intraespecie o a especies autóctonas aún no descriptas. Es necesario avanzar en el estudio de la infección por estas bacterias en el ganado bovino de Argentina para conocer su dimensión y analizar su impacto económico y zoonótico, y también para planear medidas de prevención y control.
Reproductive losses linked to an infectious etiology in bovine cattle are a major economic concern worldwide. In Argentina, more than 50% of abortion cases have unknown causes. Species belonging to Chlamydiaceae family are frequent etiologic agents of abortion around the world; however, there is yet no information on their prevalence in Argentina. The objective of this work was to identify Chlamydia spp., and particularly C. abortus in reproductive losses from bovine cattle in La Pampa, Argentina. Real time PCR targeting Chlamydiaceae-specific DNA fragments was performed on 251 samples obtained from bovine abortions and stillborns, and ArrayTube was used for species identification on positive samples. Chlamydiaceae DNA was detected in 12 samples of aborted fetuses (4.78%), 83.33% (10/12) accounting for abortions and 16.66% (2/12) for stillborns. C. abortus was detected by ArrayTube in 5 cases (1.99% of all samples, and 41.67% of Chlamydiaceae positive samples). This study shows the first detection of Chlamydiaceae and C. abortus DNA on reproductive losses of bovine cattle in Argentina, and the described prevalence value (4.78%) should be taken as baseline value due to the type of samples analyzed. Detection of genetic material from Chlamydiaceae not matching any of the studied species could be due to intraspecies variants or local species not yet described. Further research on Chlamydia infections in bovine cattle in Argentina is imperative to describe their range, to analyze their economic and zoonotic implications and to make recommendations about prevention and control measures.
La ganadería bovina como fuente de producción de carne y leche es una de las principales actividades económicas de la República Argentina, donde se cuenta con un stock de 52 millones de cabezas34. Las pérdidas reproductivas representan una causa sustancial de perjuicio económico. Se estima que las enfermedades infecciosas reproductivas reducen en alrededor del 10% la tasa anual de destete18.
En la provincia de La Pampa, la cría de bovinos es de tipo extensiva. Esta provincia cuenta con un stock ganadero de 3 millones de cabezas y las razas que predominan en la región que comprendió este estudio son Aberdeen Angus, Hereford y Holando Argentino. La eficiencia del rodeo en esta región (relación ternero/vaca) es menor que el promedio nacional34.
La proporción de pérdidas reproductivas atribuidas a un agente específico varía según la región, la condición climática, el tipo de producción, las prácticas de manejo y los programas de vacunación y de control. El éxito del diagnóstico etiológico se sustenta en el tipo y la calidad de la muestra analizada. Entre los agentes más frecuentemente asociados a abortos bovinos en la Argentina se encuentran virus (herpesvirus bovino y el virus de la diarrea viral bovina), bacterias (Brucella abortus, Campylobacter fetus, Lesptospira spp.) y protozoos (Tritrichomonas foetus y Neospora caninum); sin embargo, el 50-60% de los abortos estudiados son de etiología indeterminada6,10,12,21.
Recientemente se han publicado estudios locales sobre la situación epidemiológica de agentes infecciosos específicos1,13,20 y de los principales agentes infecciosos involucrados en pérdidas reproductivas del ganado28; sin embargo, no existen datos fehacientes con respecto a la participación local de bacterias de la familia Chlamydiaceae en animales enfermos o en abortos.
Todos los miembros de la familia Chlamydiaceae son bacterias de vida intracelular obligada, presentan un genoma muy pequeño (1,04-1,23Mb), son capaces de infectar una amplia variedad de organismos8,19 y pueden causar enfermedades, tanto en animales como en humanos36.
Las técnicas de amplificación de ADN han permitido reclasificar el orden Chlamydiales varias veces en las últimas décadas. La revisión más reciente ha llevado a la reunificación de todas las especies de la familia Chlamydiaceae conocidas en un solo género: Chlamydia29,35. Actualmente son reconocidas 11 especies: Chlamydia abortus, C. pecorum, C. psittaci, C. pneumoniae, C. felis, C. caviae, C. trachomatis, C. suis, C. muridarum y las recientes C. avium y C. gallinacea32.
Los datos epidemiológicos indican que la infección de rumiantes por Chlamydia spp. ocurre con frecuencia en todas partes del mundo26, en la mayoría de los casos sin síntomas clínicos registrados, pero con perjuicio de rendimiento24.
Las especies que pueden infectar el ganado bovino son C. pecorum (infecciones intestinales, articulares y oculares), C. abortus (infecciones genitales, ocasionalmente con abortos) y C. psittaci (infecciones respiratorias)8,18,22,24,26,36.
El único trabajo publicado sobre la infección por clamidias en bovinos en la Argentina indicó que el 23% de los animales estudiados presentaba anticuerpos específicos27. Estos resultados demuestran que esta infección está subdiagnosticada y que el efecto de estas bacterias sobre la producción animal es desconocido.
El diagnóstico de la infección por Chlamydia spp. requiere de técnicas sensibles y específicas9,33. Actualmente, las técnicas de amplificación de ADN son las más útiles para el diagnóstico de la infección. El uso de PCR, PCR en tiempo real y microarray permite la detección y la tipificación de las especies involucradas4,30,31.
El objetivo de este trabajo fue determinar la presencia de Chlamydia spp. y de C. abortus en particular en pérdidas reproductivas del ganado bovino en la provincia de La Pampa, Argentina.
Materiales y métodosCriterios utilizadosPara definir las pérdidas reproductivas se tomaron los criterios del Comité de Nomenclatura de Reproducción Bovina7. Se consideró «aborto» la expulsión de un feto entre los 42 y 260 días de gestación. Se clasificó como «parto prematuro» la eliminación de fetos cuya edad varía desde el día 260 de preñez hasta el final de la misma. Se consideraron «mortinatos» los nacimientos de terneros a término (280 días) muertos. Como «pérdidas neonatales» se consideró al recién nacido hasta los primeros 28 días de vida.
MuestrasSe incorporaron al estudio las muestras de órganos provenientes de 251 pérdidas reproductivas ocurridas en la provincia de La Pampa, Argentina, que ingresaron durante el período 2004-2011 a la Estación Experimental Agropecuaria Anguil del Instituto Nacional de Tecnología Agropecuaria, para la realización del diagnóstico histológico e infectológico habitual. Los órganos seleccionados fueron cerebro, pulmón, corazón, hígado y riñón; estos estaban fijados en formol al 10% e incluidos en bloques de parafina.
Extracción de ADNLas muestras se procesaron como lo describieron Borel et al.4. Por cada animal se preparó una única muestra con partes proporcionales de cada uno de los 5 órganos. Del bloque parafinado correspondiente a cada órgano, se cortaron secciones de 30 a 60μm y se colocaron agrupadas en un tubo cónico estéril. La parafina se removió con 1,2ml de xileno. Posteriormente se centrifugó a 10.000g durante 5min. El xileno residual se eliminó por doble extracción con 1,2ml de etanol. El etanol se retiró cuidadosamente luego de centrifugar las muestras 2 veces a 10.000g durante 5min.
El ADN para PCR se extrajo a partir del sedimento del tejido utilizando un kit comercial (DNeasy® Tissue Kit, Qiagen, Hilden, Alemania), siguiendo las instrucciones del fabricante.
PCR en tiempo real para la detección de ChlamydiaceaeTodas las muestras fueron examinadas en un equipo PCR Real-Time ABI 7500 Fast (Applied Biosystems, Singapur, Singapur), usando un protocolo validado por el laboratorio de referencia de la Oficina Internacional de Epizootias para clamidiosis11. Se utilizaron cebadores específicos para la familia Chlamydiaceae dirigidos al gen 23S del ARNr, para obtener un producto de 111pb específico de los miembros de la mencionada familia11: cebador 1 (5’-CTG AAA CCA GTA GCT TAT AAG CGG T-3’) y cebador 2 (5’-ACC TCG CCG TTT AAC TTA ACT CC-3’; la sonda empleada fue la siguiente: 5’-FAM/CT CAT CAT GC AAA AGG CAC GCC G/BHQ-3’. Se adicionaron 2,5μl del extracto de ADN a una solución madre comercial (TaqMan® Fast Universal PCR Master Mix, Applied Biosystems, Foster City, CA, EE. UU.), con una concentración final de 5pmol/μl de cada primer y de la sonda (Microsynth, Balgach, Suiza), y 12,5μl de solución tampón 2x para llegar a un volumen final de 25μl. La amplificación se realizó con una desnaturalización inicial a 95°C por 10min, seguida de 45 ciclos de 94°C por 15s y 60°C por 60s11. Como control positivo se utilizó la cepa de C. abortus 09DC76 y como control negativo, agua bidestilada estéril.
Todas las muestras se procesaron por duplicado. Se consideraron positivas aquellas que presentaron un ciclo umbral menor o igual a 42, de acuerdo con el protocolo utilizado en el Friedrich-Loeffler-Institut. La presencia de inhibidores se evaluó mediante la amplificación de un control interno.
ArrayTube para la identificación especies del género ChlamydiaSe utilizó el sistema de microarrays en tubo ArrayTube (Alere Technologies GmbH, Jena, Alemania).
El chip de ADN incluía sondas para la identificación de las 11 especies reconocidas. Para C. trachomatis, C. pneumoniae, C. suis y C. psittaci se utilizaron 4 sondas para cada especie; 3 sondas para C. avium, C. muridarum y C. caviae, y 2 sondas para C. abortus, C. felis y C. gallinacea. El chip contó con 4 controles positivos (sondas consenso) y un control interno (marcador de biotina). El procedimiento se realizó como describieron Borel et al.4. Brevemente, se realizó una PCR de un fragmento del gen 23S del ARNr utilizando los cebadores biotinilados U23F-19 (5’-ATTGAMAGGCGAWGAAGGA-3’) y 23R-22 (5’-biotin-GCYTACTAAGATGTTTCAGTTC-3’). Los productos de la reacción fueron transferidos a un tubo cónico de 1,5ml, de base plana. Se incubaron dichos productos con el chip de ADN que contiene las sondas específicas y que se encuentra en la base plana del tubo. El resultado de la hibridización se reveló con estreptavidina conjugada con peroxidasa de rábano. El resultado fue leído por un equipo específico (ATR-01 ArrayTube Reader) y analizado por el software correspondiente (Iconoclust Software, versión 2.3).
HistologíaSe realizó nuevamente una evaluación histológica de las lesiones presentes mediante la tinción con hematoxilina-eosina de secciones de todos los órganos, para cada uno de los casos con detección de Chlamydia spp.
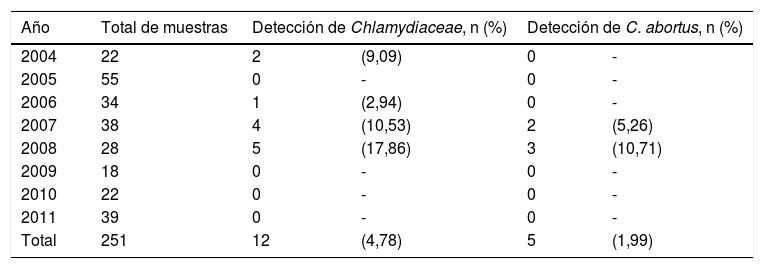
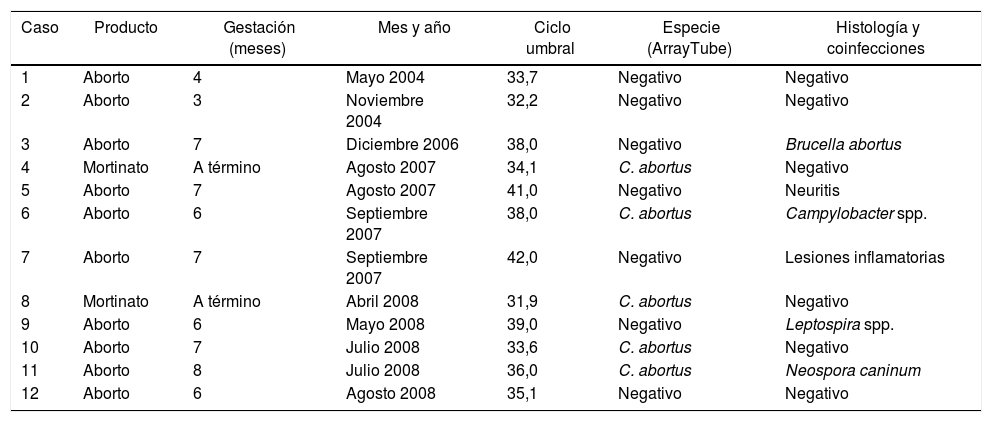
ResultadosNinguna muestra presentó inhibidores. Se detectó ADN de la familia Chlamydiaceae en 12 muestras del total analizadas a lo largo de todo el período estudiado (4,78%); en la tabla 1 se muestra la frecuencia de detección por año. Los valores del ciclo umbral fueron de 31,9 a 42. El 83,33% (10/12) correspondió a abortos y el 16,66% (2/12) a mortinatos. En la tabla 2 se presentan los 12 casos detectados, con los meses de gestación, el año del suceso, los resultados del ArrayTube y las enfermedades asociadas. El caso 7 reveló lesiones inflamatorias localizadas en pulmón e hígado y en el caso 5 se detectó neuritis. En el caso 11 se encontraron lesiones compatibles con N. caninum; este patógeno fue confirmado por serología. En el caso 3 se detectaron anticuerpos específicos contra B. abortus y en el caso 9 contra Leptospira spp. En el caso 6 se detectó Campylobacter spp. mediante inmunofluorescencia directa (datos previos no mostrados). En las otras 6 muestras (50%) no se encontró ninguna lesión o diagnóstico de enfermedad asociada (casos 1, 2, 4, 9, 10 y 12).
Detección de Chlamydiaceae y de C. abortus en muestras de pérdidas reproductivas bovinas de La Pampa, período 2004-2011
| Año | Total de muestras | Detección de Chlamydiaceae, n (%) | Detección de C. abortus, n (%) | ||
|---|---|---|---|---|---|
| 2004 | 22 | 2 | (9,09) | 0 | - |
| 2005 | 55 | 0 | - | 0 | - |
| 2006 | 34 | 1 | (2,94) | 0 | - |
| 2007 | 38 | 4 | (10,53) | 2 | (5,26) |
| 2008 | 28 | 5 | (17,86) | 3 | (10,71) |
| 2009 | 18 | 0 | - | 0 | - |
| 2010 | 22 | 0 | - | 0 | - |
| 2011 | 39 | 0 | - | 0 | - |
| Total | 251 | 12 | (4,78) | 5 | (1,99) |
Todas las muestras analizadas provinieron de la Estación Experimental Agropecuaria (EEA) Anguil, del Instituto Nacional de Tecnología Agropecuaria (INTA).
Descripción de los casos correspondientes a las muestras con detección de Chlamydiaceae
| Caso | Producto | Gestación (meses) | Mes y año | Ciclo umbral | Especie (ArrayTube) | Histología y coinfecciones |
|---|---|---|---|---|---|---|
| 1 | Aborto | 4 | Mayo 2004 | 33,7 | Negativo | Negativo |
| 2 | Aborto | 3 | Noviembre 2004 | 32,2 | Negativo | Negativo |
| 3 | Aborto | 7 | Diciembre 2006 | 38,0 | Negativo | Brucella abortus |
| 4 | Mortinato | A término | Agosto 2007 | 34,1 | C. abortus | Negativo |
| 5 | Aborto | 7 | Agosto 2007 | 41,0 | Negativo | Neuritis |
| 6 | Aborto | 6 | Septiembre 2007 | 38,0 | C. abortus | Campylobacter spp. |
| 7 | Aborto | 7 | Septiembre 2007 | 42,0 | Negativo | Lesiones inflamatorias |
| 8 | Mortinato | A término | Abril 2008 | 31,9 | C. abortus | Negativo |
| 9 | Aborto | 6 | Mayo 2008 | 39,0 | Negativo | Leptospira spp. |
| 10 | Aborto | 7 | Julio 2008 | 33,6 | C. abortus | Negativo |
| 11 | Aborto | 8 | Julio 2008 | 36,0 | C. abortus | Neospora caninum |
| 12 | Aborto | 6 | Agosto 2008 | 35,1 | Negativo | Negativo |
El análisis por ArrayTube detectó C. abortus en 5 muestras (1,99% del total analizado y 41,67% de las muestras con detección de Chlamydiaceae). Los valores del ciclo umbral se ubicaron entre 32 y 38. Las muestras 4 y 6 pertenecieron a casos del año 2007 y las muestras 8, 9 y 11 a casos del año 2008. La infección por C. abortus se detectó en 3 abortos y 2 mortinatos (casos 4 y 8).
DiscusiónLa pérdida reproductiva de etiología desconocida en el ganado bovino representa un problema económico importante y de difícil estimación.
A pesar de que el rol patogénico de las especies del género Chlamydia y su capacidad para producir infecciones en distintas especies animales es conocido, la búsqueda de estos microorganismos no es realizada habitualmente.
Aunque las clamidias no son consideradas como una amenaza para la industria ganadera, ya que los abortos son esporádicos y las tormentas de abortos son eventos pocos frecuentes, podrían afectar al 20% de las vacas preñadas25. Por otra parte, se debe considerar el efecto subclínico de las infecciones clamidiales, las cuales tienen repercusiones notables en términos económicos26.
En rodeos con un alto porcentaje de portadores de clamidias se ha observado una disminución de la ganancia diaria de peso26, la obstrucción de las vías respiratorias16, la presencia de clamidias en semen17 y la reducción del rendimiento de leche3.
Este trabajo presenta la primera confirmación de la presencia de ADN de Chlamydiaceae y de C. abortus en muestras de pérdidas reproductivas de ganado bovino en la provincia de La Pampa, Argentina.
El valor de prevalencia de infección por Chlamydia spp. (4,78%) debe ser tomado como un valor basal, ya que este trabajo se realizó de manera retrospectiva sobre muestras previamente fijadas en formol, parafinadas y luego desparafinadas, procesos agresivos que pudieron haber dañado el ADN y, por lo tanto, disminuido la sensibilidad de la detección.
Por otra parte, no pudieron ser analizadas las placentas, material considerado de elección para el diagnóstico de aborto por clamidias, debido a que, al tratarse de un sistema de cría extensiva, estas fueron consumidas por animales carroñeros5,15,23.
Los datos publicados en las últimas décadas proponen una elevada prevalencia de infección por clamidias en bovinos, que oscilaría entre el 45 y el 100%26. Si bien C. abortus es endémica en rumiantes menores de todo el mundo, también se ha documentado su presencia en bovinos y ha sido asociada con abortos5.
El valor de prevalencia de infección por C. abortus (1,99%) también debe ser tomado como mínimo por los motivos antes citados. De acuerdo con la bibliografía25,26, el aborto causado por C. abortus en el ganado bovino ocurre entre el sexto y octavo mes de gestación, dato que coincide con los resultados obtenidos en este trabajo. Sin embargo, la detección de ADN de C. abortus en 2 mortinatos a término, sin otra enfermedad asociada, podría manifestar alguna diferencia en cepas autóctonas.
La detección de C. abortus en una muestra con diagnóstico de infección por N. caninum y en otra muestra infectada por Campylobacter spp., así como la presencia de B. abortus y de Leptospira spp. en muestras con hallazgo de miembros de la familia Chlamydiaceae, refleja una situación que es frecuente4. Por este motivo, la etiología de la pérdida reproductiva debería analizarse dentro del estudio de otras coinfecciones presentes.
La presencia en ciertos casos de material genético de Chlamydiaceae que no coincidió con ninguna de las especies conocidas podría deberse a variantes intraespecie o a especies autóctonas, aún no descriptas.
Por otra parte, los valores de ciclo umbral altos (mayores de 38) podrían no ser conclusivos y la cantidad de ADN podría ser demasiado escasa como para poder determinar la especie involucrada mediante la metodología de microarray.
La infección zoonótica por C. abortus es considerada de riesgo ocupacional2,14,37. Por lo tanto, la demostración de la circulación de C. abortus refuerza la necesidad de extremar las condiciones de bioseguridad y protección de los veterinarios y del personal de campo involucrados en las actividades de parto y en la manipulación de materiales productos del aborto.
Este trabajo demuestra por primera vez la presencia de miembros de la familia Chlamydiaceae y la posible participación de estos, y de C. abortus en particular, en pérdidas reproductivas en la provincia de La Pampa.
Es necesario avanzar en estudios prospectivos, clínicos y microbiológicos para poder determinar la dimensión de la infección por Chlamydiaceae y las especies involucradas, a fin de analizar la relevancia económica y zoonótica de este problema y planear medidas de prevención y control.
FinanciaciónFuentes de financiación: INTA, Universidad Nacional del Sur (SECyT) y Universidad de Buenos Aires (UBACyT).
Conflicto de interesesLos autores declaran no tener conflictos de intereses.
A la técnica Christine Grajetzki, por procesamiento de muestras. A la Dra. María Lucía Gallo Vaulet, por la lectura crítica del manuscrito.