La identificación rápida de microorganismos causantes de bacteriemias y de sus posibles marcadores de resistencia antimicrobiana es de suma importancia para el inicio oportuno de una terapia antibiótica eficaz. El sistema FilmArray® panel BCID2 (ensayo rápido automatizado por PCR multiplex) detecta microorganismos y genes de resistencia a partir de hemocultivos positivos en una hora. El objetivo de este estudio fue comparar los tiempos de informe y los resultados obtenidos mediante FilmArray® BCID2 y el cultivo convencional en hemocultivos de pacientes pediátricos. Se incluyeron 60 resultados de FilmArray® BCID2. El ensayo automatizado demostró tener alta concordancia con el cultivo en la identificación de microorganismos en bacteriemias monomicrobianas. En hemocultivos polimicrobianos, BCID2 detectó mayor cantidad de microorganismos frente al cultivo (1 Staphylococcus aureus, 3 Staphylococcus epidermidis, 1 Enterococcus faecium, 2 Klebsiella oxytoca, 1 complejo Acinetobacter calcoaceticus-baumannii, 1 Bacteroides fragilis y 1 Haemophilus influenzae). El 88,3% (IC 95%: 78,7-94,8) de los FilmArray® coincidieron con el cultivo y en las bacteriemias polimicrobianas, BCID2 detectó mayor cantidad de microorganismos con respecto al cultivo en un 70% (IC 95%: 39,3-91,5) de los casos. La concordancia de los genes de resistencia fue buena, con algunas excepciones: una BLEE no detectada por FilmArray® y una cepa de S. aureus caracterizada como meticilino-resistente mediante BCID2 y como meticilino-sensible por cultivo. Al comparar el tiempo desde que el hemocultivo fue informado como positivo hasta los resultados de identificación, BCID2 tuvo una mediana de 2h-50min (RIC: 1h-58min/9h-27min) frente a 45h-20min (RIC: 24h-57min/63h-50min) para el cultivo.
The rapid identification of microorganisms that cause bacteremia and their possible resistance markers are extremely important for the timely initiation of effective antibiotic therapy.
The FilmArray® panel BCID2 (an automated rapid multiplex PCR assay) detects microorganisms and resistance genes from positive blood cultures within one hour. The aim of this study was to compare the results obtained from the FilmArray® Panel BCID2 and conventional culture in pediatric patients, as well as the reporting times of both methods. Sixty (60) FilmArray® results were included in the analysis. BCID2 showed high agreement with culture in the identification of microorganisms in monomicrobial bacteremias. However, in polymicrobial blood cultures, BCID2 detected a greater number of microorganisms compared to conventional culture, specifically,1 Staphylococcus aureus, 3 Staphylococcus epidermidis, 1 Enterococcus faecium, 2 Klebsiella oxytoca, 1 Acinetobacter calcoaceticus-baumannii complex, 1 Bacteroides fragilis and 1 Haemophilus influenzae. Furthermore, 88.3% (95%CI: 78.7-94.8) of the FilmArray® results coincided with conventional culture, while in polymicrobial bacteremias, BCID2 detected a greater number of microorganisms with respect to conventional culture [70% (95%CI: 39.3-91.5)]. The agreement of resistance genes was good with a few exceptions (one ESBL was not detected by FilmArray® and one S. aureus strain was characterized as methicillin-resistant by BCID2 and methicillin-sensitive by culture). When comparing the time elapsing since the blood culture was reported as positive up to the results were obtained, BCID2 had a median of 2h 50min (IQR of 1 h 58min to 9h 27min) while the conventional culture had a median of 45h 20min (IQR of 24h 57min to 63h 50min).
La bacteriemia es una de las principales causas de muerte en todo el mundo1,6. En la Argentina, la sepsis grave pediátrica es una entidad frecuente (13% de los ingresos a las unidades de cuidados intensivos pediátricos), con una mortalidad del 31%10.
Diferentes publicaciones han evidenciado la importancia de iniciar precozmente el tratamiento antibiótico apropiado para reducir la mortalidad en pacientes con sepsis o shock séptico, cuya mortalidad se incrementa un 7,6% por hora de tratamiento inadecuado12,13. Sumado a esto, la eficacia de las terapias empíricas se ve significativamente afectada debido a la propagación mundial de bacterias gram negativas y gram positivas portadoras de determinantes de resistencia adquiridos6.
La obtención de resultados rápidos a partir de hemocultivos positivos facilita la identificación temprana de microorganismos causantes de infecciones del torrente sanguíneo, así como de genes de resistencia, para lograr una optimización del tratamiento antibiótico16.
El panel de identificación de cultivos de sangre 2 (BCID2) FilmArray® BioFire® es un ensayo rápido automatizado, basado en la reacción en cadena de la polimerasa (PCR) multiplex para la detección cualitativa de microorganismos y genes de resistencia, a partir de hemocultivos identificados como positivos mediante un sistema de monitorización continua. Los resultados se obtienen en aproximadamente una hora9. Fue aprobado en 2020 por la Administración de Alimentos y Medicamentos de los EE. UU. (FDA) y por la Administración Nacional de Medicamentos, Alimentos y Tecnología Médica de Argentina (ANMAT).
El panel BCID2 representa una mejora de la versión anterior BCID. Contiene 43 blancos, que incluyen 11 bacterias gram positivas, 15 bacterias gram negativas, 7 levaduras y 10 genes de resistencia a los antimicrobianos. Este panel incluye la detección de microorganismos a nivel de género y especie. Entre los cocos gram positivos que puede detectar se incluyen Staphylococcus spp., Staphylococcus aureus, Streptococcus spp., Streptococcus pneumoniae, Streptococcus pyogenes, Streptococcus agalactiae, Listeria monocytogenes, Enterococcus faecalis, Enterococcus faecium (la detección a nivel de género de enterococos, que estaba incluida en BCID, se eliminó de la actual versión), Staphylococcus epidermidis y Staphylococcus lugdunensis (que no estaban incluidos en la versión anterior). En cuanto a los blancos de levaduras incluidos, el BCID2 detecta las 5 especies de Candida más comunes asociadas con enfermedades del torrente sanguíneo (Candida albicans, Candida glabrata, Candida krusei, Candida parapsilosis y Candida tropicalis), el patógeno emergente Candida auris y 2 especies de Cryptococcus (Cryptococcus neoformans/gattii), que fueron agregadas en la actual versión del panel.
En cuanto a las bacterias gram negativas, las especies que se detectan de manera individual son Escherichia coli, Haemophilus influenzae, Klebsiella oxytoca, Neisseria meningitidis, Pseudomonas aeruginosa y Serratia marcescens, sumando en la nueva versión Klebsiella aerogenes, Bacteroides fragilis y Stenotrophomonas maltophilia. También se detectan microorganismos como resultados de complejo, grupo o género: complejo Acinetobacter calcoaceticus-baumannii, complejo Enterobacter cloacae, grupo Klebsiella pneumoniae, Proteus spp. y Salmonella spp. (agregado en la actual versión del panel). Además, detecta un gran número de especies gram negativas identificadas como Enterobacterales9.
El panel BCID2 incluye pruebas para la detección de determinantes genéticos relacionados con varias clases de antibióticos. Solo se informará el determinante genético cuando se detecte una bacteria que pueda ser portadora de este. Con respecto a las bacterias gram positivas, detecta mecA/C y vanA/B. Esta nueva versión agrega la detección del locusmec right-extremity junction (MREJ), lo que asegura que, cuando se detecta el gen mecA/C en un cultivo mixto de S. aureus y Staphylococcus spp., este proviene de S. aureus; es decir que para asignar la resistencia a meticilina a este microorganismo, se deben detectar simultáneamente ambos loci (mecA/C y MREJ). El panel BCID2 solo informará mecA/C para S. epidermidis y S. lugdunensis, que son los únicos blancos específicos de especie de estafilococos coagulasa negativos. Con respecto a las bacterias gram negativas, el panel detecta β-lactamasas de espectro extendido (CTX-M) y genes de resistencia a carbapenemasas (KPC y los agregados en la actual versión VIM, IMP, NDM y OXA-48-like) y de resistencia plasmídica a colistina (mcr-1)9.
El Hospital General de Niños Pedro de Elizalde (HGNPE) cuenta con el panel BCID desde diciembre de 2019, y en marzo de 2021 se comenzó a utilizar la versión BCID2. Son escasos los estudios que demuestran la experiencia de uso de la actual versión del panel sepsis (BCID2) de FilmArray® BioFire® en la población pediátrica, motivo por el cual se investigó este uso en el HGNPE.
ObjetivosComparar los resultados en cuanto a los microorganismos y genes de resistencia identificados mediante FilmArray® panel BCID2 y los métodos de cultivo convencional.
Comparar el tiempo hasta el informe de resultados según el método empleado.
Materiales y métodosSe llevó a cabo un estudio observacional, transversal y retrospectivo en el HGNPE de la Ciudad Autónoma de Buenos Aires, Argentina.
Se revisaron en el Sistema Informático de Gestión del Laboratorio de Microbiología R•E•A•L (BioMérieux) todos los resultados de hemocultivos positivos procesados mediante FilmArray® panel BCID2 y cultivo convencional durante el período comprendido entre marzo de 2021 y marzo de 2022. Se excluyeron los resultados del panel BCID2 que no correspondían a muestras de hemocultivos (como líquidos de punción) y aquellos resultados de BCID2 o cultivo convencional que no fueron jerarquizados clínicamente, es decir, aquellos resultados informados como probables contaminantes.
Procesamiento de los hemocultivos mediante el cultivo convencionalLas botellas de hemocultivos (BacT/ALERT® PF PLUS) fueron incubadas y monitorizadas de manera continua en el equipo BacT/ALERT® (BioMérieux). Cuando un hemocultivo fue identificado como positivo por el software, se retiró y se le realizó la coloración de Gram, un subcultivo en agar chocolate y otro en EMB Levine. La incubación de las placas de agar chocolate se llevó a cabo a 37°C en atmósfera de CO2 al 5% y las placas de EMB Levine fueron incubadas a 37°C en aerobiosis. Cuando se visualizaron levaduras mediante la tinción de Gram, se añadió una placa de CHROMagar Candida®. Los medios de cultivo se incubaron durante al menos 48h. La identificación de las colonias que desarrollaron en las placas de cultivo se realizó mediante VITEK® MS (BioMérieux), que utiliza la tecnología matrix assisted laser desorption ionization time-of-flight (MALDI-TOF) a partir de la pátina obtenida mediante un tiempo de incubación corto (3 o 4h) o tras 24h. Las pruebas de sensibilidad se realizaron mediante el sistema VITEK® 2 Compact (BioMérieux) o mediante difusión en agar (método Kirby-Bauer).
Se utilizó el panel BCID2 en hemocultivos positivos que cumplían con los criterios previamente establecidos por el servicio de microbiología e infectología: pacientes inmunosuprimidos con sepsis, pacientes internados en la unidad de cuidados críticos con sepsis, shock séptico en pacientes inmunocompetentes y sospecha de bacteriemia polimicrobiana.
Procesamiento de los hemocultivos mediante FilmArray® panel BCID2Acorde con las indicaciones del fabricante, el panel BCID2 fue primero rehidratado con 300μl de solución de rehidratación y luego fue inoculado con 200μl de la muestra de sangre positiva diluida con 500μl de tampón de dilución, lo que requiere alrededor de 2min. Por último, el panel fue cargado en el instrumento BioFire® FilmArray® Torch para la posterior extracción, amplificación y análisis de ácidos nucleicos, que se efectúa en 1 hora aproximadamente9.
Los resultados obtenidos, tanto por FilmArray® BCID2 como por cultivo convencional, fueron comunicados mediante el Sistema Informático de Gestión del Laboratorio de Microbiología R•E•A•L (BioMérieux), al cual acceden las salas de internación y los diferentes servicios hospitalarios de manera on line.
Obtención de los registrosEl tiempo transcurrido desde que los hemocultivos fueron informados como positivos hasta que se obtuvieron los resultados, tanto mediante FilmArray® como mediante el cultivo convencional, fue calculado con los datos extraídos del sistema informático del laboratorio (R•E•A•L).
Se utilizaron la historia clínica única electrónica (SIGEHOS) y la historia clínica en papel para recolectar datos clínicos de los pacientes: edad y diagnóstico o condición del paciente.
Análisis estadísticoLas proporciones de microorganismos detectados y de los resultados coincidentes fueron expresadas en porcentaje, con sus respectivos intervalos de confianza (IC), calculados según la prueba de máxima verosimilitud con el programa RStudio Cloud. Se calculó el porcentaje de concordancia como la cantidad de coincidencias en el resultado final de los métodos estudiados respecto del total de frascos de hemocultivos analizados, teniendo en cuenta los blancos incluidos en el BCID2.
En cuanto al tiempo transcurrido en horas, se expresó mediante la mediana y el intervalo intercuartílico (RIC). La diferencia de tiempo de comunicación de resultados entre BCID2 y el cultivo convencional se analizó utilizando la prueba de Wilcoxon para muestras pareadas. Se consideró un nivel de significación de 0,05.
ResultadosSe realizó FilmArray® panel BCID2 a 86 frascos de hemocultivos detectados como positivos. Se excluyeron 26, 13 líquidos de punción y 13 resultados informados mediante cultivo como probables contaminantes. Es por ello que se consideraron 60 frascos de hemocultivos positivos clínicamente relevantes, los que fueron procesados por FilmArray® BCID2 y por cultivo convencional. Estos correspondieron a 52 pacientes (25 de género femenino y 27 de género masculino). La mediana de edad fue de 4,5 meses (rango etario: 11 días de vida a 17 años). Treinta y cinco bacteriemias fueron de pacientes internados en las unidades cerradas; 25 de pacientes en las demás salas de internación. Los diagnósticos o condiciones que presentaron los pacientes fueron las siguientes: posquirúrgicos (n=30), enfermedad oncológica (n=5), cuadro respiratorio (n=3), síndrome febril (n=3), artritis séptica (n=2), meningitis (n=2), dermatitis sobreinfectada (n=1), diarrea de la comunidad (n=1), endocarditis infecciosa (n=1), politraumatismo (n=1), pioventriculitis asociada a ventilación mecánica (n=1), reacción adversa medicamentosa (n=1) y neurológico (n=1).
De los 60 frascos de hemocultivos jerarquizados clínicamente, 40 se remitieron de pares de botellas ambas con resultado positivas, 11 fueron bacteriemias asociadas a catéteres, 7 se aislaron de uno solo de los frascos positivos y hubo solo 2 casos en los que se remitió un solo frasco.
Al comparar los resultados obtenidos de los 60 frascos de hemocultivos procesados por ambos métodos, se identificaron 85 microorganismos en total: 78 mediante FilmArray® panel BCID2 y 71 mediante cultivo (tabla 1). Hubo 3 microorganismos aislados en el cultivo que no fueron detectados por BCID2 (Enterococcus raffinosus, Pseudomonas fluorescens y Burkholderia contaminans) debido a que no están incluidos entre los blancos del panel. Si se hubiera utilizado la versión anterior (BCID), la cepa de E. raffinosus podría haberse detectado a nivel de género. Por otro lado, Pantoea agglomerans, si bien no está incluido como blanco en el panel BCID2, fue detectado dentro de Enterobacterales, ya que se incluye la detección de un gran número de especies gram negativas dentro de este orden9. En el caso de Proteus mirabilis y Salmonella enterica subp. enterica, identificados como tales a partir del cultivo, el panel BCID2 los detectó a nivel de género como Proteus spp. y Salmonella spp., respectivamente. Lo mismo sucedió con los aislamientos pertenecientes al grupo estreptococos viridans, que BCID2 los detectó a nivel de género.
Microorganismos en hemocultivos positivos
| Microorganismos | Detectados por FilmArray® panel BCID2(n) | Aislados por cultivo(n) |
|---|---|---|
| Gram positivos | ||
| Staphylococcus aureus | 8 | 7 |
| Staphylococcus epidermidisa | 5 | 2 |
| Grupo estreptococos viridans | 2 (Streptococcus spp.) | 2 |
| Streptococcus agalactiae | 1 | 1 |
| Enterococcus faecalis | 2 | 2 |
| Enterococcus faecium | 2 | 1 |
| Enterococcus raffinosusb | 0 | 1 |
| Gram negativos | ||
| Grupo Klebsiella pneumoniae | 21 | 21 |
| Klebsiella oxytoca | 2 | 0 |
| Escherichia coli | 9 | 9 |
| Complejo Enterobacter cloacae | 3 | 3 |
| Serratia marcescens | 2 | 2 |
| Proteus mirabilis | 1 (Proteus spp.) | 1 |
| Pantoea agglomerans | 1 (Enterobacterales) | 1 |
| Salmonella enterica subesp. entericaa | 1 (Salmonella spp.) | 1 |
| Pseudomonas aeruginosa | 3 | 3 |
| Pseudomonas fluorescensb | 0 | 1 |
| Complejo Acinetobacter calcoaceticus-baumanii | 2 | 1 |
| Stenotrophomonas maltophiliaa | 2 | 2 |
| Burkholderia contaminansb | 0 | 1 |
| Bacteroides fragilisa | 1 | 0 |
| Haemophilus influenzae | 5 | 4 |
| Levaduras | ||
| Candida albicans | 4 | 4 |
| Candida tropicalis | 1 | 1 |
El 86,4% (IC 95%: 77,9-92,7) de los microorganismos detectados por BCID2 fueron aislados posteriormente en el cultivo convencional y el 94,6% (IC 95%: 87,9-98,3) de los microorganismos aislados del cultivo fueron detectados también por FilmArray®.
Considerando solo los blancos incluidos en el panel BCID2, la concordancia entre el panel BCID2 y el cultivo fue del 88,3% (IC 95%: 78,7-94,8).
En cuanto a las bacteriemias monomicrobianas, considerando solo los blancos incluidos en el panel, los microorganismos detectados por FilmArray® panel BCID2 (n=50) coincidieron en su totalidad con los resultados del cultivo (tabla 2).
Microorganismos en bacteriemias monomicrobianas
| Microorganismos | Detectados por FilmArray® Panel BCID2(n) | Aislados por cultivo(n) |
|---|---|---|
| Staphylococcus aureus | 3 | 3 |
| Staphylococcus epidermidis | 2 | 2 |
| Grupo estreptococos viridans | 2 (Streptococcus spp.) | 2 |
| Enterococcus faecalis | 2 | 2 |
| Grupo Klebsiella pneumoniae | 14 | 14 |
| Escherichia coli | 7 | 7 |
| Serratia marcescens | 1 | 1 |
| Proteus mirabilis | 1 (Proteus spp.) | 1 |
| Pantoea agglomerans | 1 (Enterobacterales) | 1 |
| Salmonella enterica subesp. enterica | 1 | 1 |
| Pseudomonas aeruginosa | 3 | 3 |
| Pseudomonas fluorescensa | 0 | 1 |
| Complejo Acinetobacter calcoaceticus-baumannii | 1 | 1 |
| Stenotrophomonas maltophilia | 2 | 2 |
| Burkholderia contaminansa | 0 | 1 |
| Haemophilus influenzae | 4 | 4 |
| Candida albicans | 3 | 3 |
| Candida tropicalis | 1 | 1 |
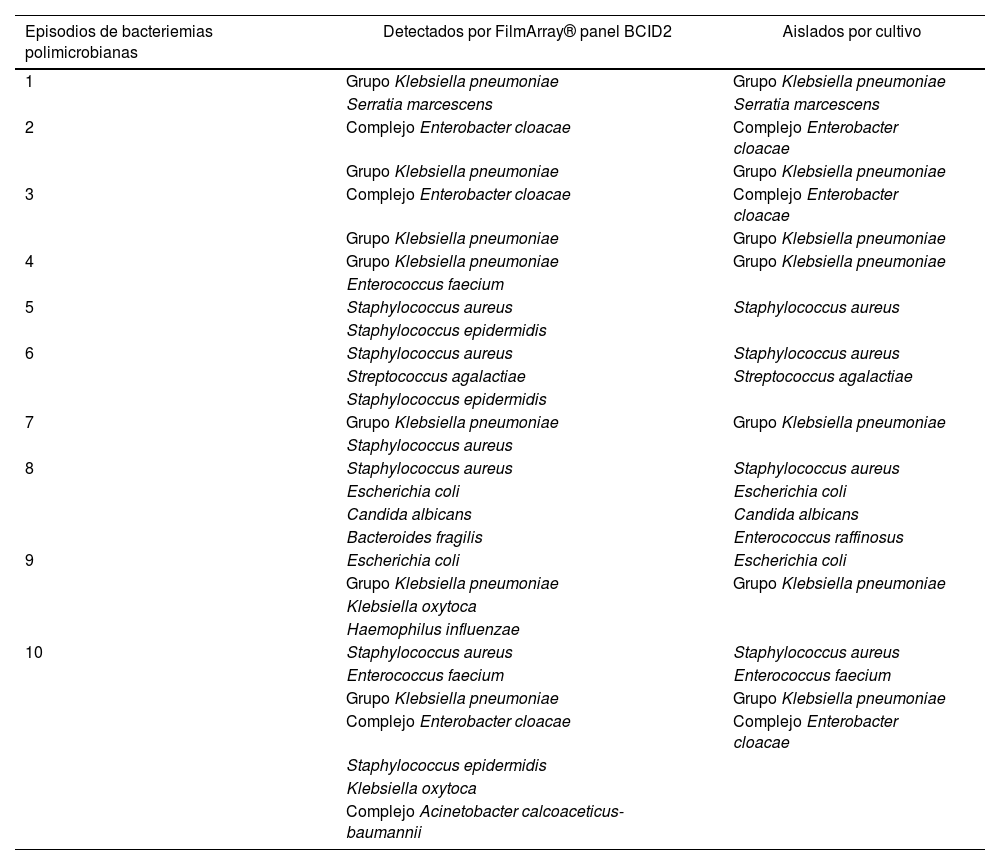
Con respecto a las bacteriemias polimicrobianas, el panel BCID2 detectó 10 episodios, mientras que el cultivo detectó 7 (tabla 3). FilmArray® detectó 10 microorganismos que no desarrollaron en el cultivo convencional (1 S. aureus, 3 S. epidermidis, 1 E. faecium, 2K. oxytoca, 1 complejo Acinetobacter calcoaceticus-baumannii, 1 B. fragilis y 1 H. influenzae). En el 70% de los hemocultivos (IC 95%: 39,3-91,5), el panel BCID2 detectó mayor cantidad de microorganismos con respecto al cultivo (p<0,001). Si se hubiera utilizado la versión anterior del panel (BCID), 9 microorganismos no hubieran sido detectados por el FilmArray® (5 S. epidermidis, 2 S. maltophilia, 1 Salmonella spp. y 1 B. fragilis).
Microorganismos en bacteriemias polimicrobianas
| Episodios de bacteriemias polimicrobianas | Detectados por FilmArray® panel BCID2 | Aislados por cultivo |
|---|---|---|
| 1 | Grupo Klebsiella pneumoniae | Grupo Klebsiella pneumoniae |
| Serratia marcescens | Serratia marcescens | |
| 2 | Complejo Enterobacter cloacae | Complejo Enterobacter cloacae |
| Grupo Klebsiella pneumoniae | Grupo Klebsiella pneumoniae | |
| 3 | Complejo Enterobacter cloacae | Complejo Enterobacter cloacae |
| Grupo Klebsiella pneumoniae | Grupo Klebsiella pneumoniae | |
| 4 | Grupo Klebsiella pneumoniae | Grupo Klebsiella pneumoniae |
| Enterococcus faecium | ||
| 5 | Staphylococcus aureus | Staphylococcus aureus |
| Staphylococcus epidermidis | ||
| 6 | Staphylococcus aureus | Staphylococcus aureus |
| Streptococcus agalactiae | Streptococcus agalactiae | |
| Staphylococcus epidermidis | ||
| 7 | Grupo Klebsiella pneumoniae | Grupo Klebsiella pneumoniae |
| Staphylococcus aureus | ||
| 8 | Staphylococcus aureus | Staphylococcus aureus |
| Escherichia coli | Escherichia coli | |
| Candida albicans | Candida albicans | |
| Bacteroides fragilis | Enterococcus raffinosus | |
| 9 | Escherichia coli | Escherichia coli |
| Grupo Klebsiella pneumoniae | Grupo Klebsiella pneumoniae | |
| Klebsiella oxytoca | ||
| Haemophilus influenzae | ||
| 10 | Staphylococcus aureus | Staphylococcus aureus |
| Enterococcus faecium | Enterococcus faecium | |
| Grupo Klebsiella pneumoniae | Grupo Klebsiella pneumoniae | |
| Complejo Enterobacter cloacae | Complejo Enterobacter cloacae | |
| Staphylococcus epidermidis | ||
| Klebsiella oxytoca | ||
| Complejo Acinetobacter calcoaceticus-baumannii |
En cuanto a los mecanismos de resistencia, el panel BCID2 detectó 16, mientras que el cultivo caracterizó 13 (tabla 4). FilmArray® no detectó una BLEE que sí fue caracterizada como tal en el cultivo, correspondiente a una cepa de K. pneumoniae.
Mecanismos de resistencia a antimicrobianos
| Mecanismos de resistencia | Detectados por FilmArray® BCID2 | Caracterizados por cultivo |
|---|---|---|
| BLEEa | 6 | 7 |
| Resistencia a meticilina en ECNb: mecA/C | 5 | 2b |
| Resistencia a meticilina en S. aureus: mecA/C y MREJ | 5 | 4 |
| Carbapenemasas: KPC, NDM, IMP, VIM y OXA-48-like | 0 | 0 |
| Resistencia a vancomicina: vanA/B | 0 | 0 |
| Resistencia a colistina: mcr-1 | 0 | 0 |
ECN: estafilococos coagulasa negativos.
Por otro lado, se observó otra discrepancia con respecto a una cepa de S. aureus, que se caracterizó como meticilino-resistente mediante FilmArray® BCID2 y como meticilino-sensible mediante cultivo convencional. Dicha cepa se encontraba formando parte de una bacteriemia polimicrobiana (más de 4 microorganismos).
En el período evaluado, no se detectó por BCID2 ni se caracterizó por cultivo resistencia a carbapenems (KPC, NDM, IMP, VIM y OXA-48-like), a vancomicina en enterococos (vanA/vanB), ni resistencia a colistina mediada por mcr-1.
La mediana de tiempo para la comunicación de resultados desde que el hemocultivo se informó como positivo fue de 2h-50min (RIC: 1h-58min/9h-27min) con BCID2 y de 45h-20min (RIC: 24h-57min/63h-50min) mediante el cultivo convencional, lo que significa una diferencia estimada de 45h-15min (IC 95%: 35h-4min/54h-22min), que fue estadísticamente significativa según la prueba de Wilcoxon para muestras pareadas (p<0,0001).
DiscusiónLa identificación rápida de microorganismos causantes de bacteriemias y de sus posibles marcadores de resistencia es de suma importancia para el inicio oportuno de una terapia antibiótica eficaz. Esto podría mejorar la evolución y el pronóstico del paciente, reducir la mortalidad y evitar el uso de antibióticos de amplio espectro.
El porcentaje de concordancia estimado en este trabajo es comparable con lo informado en otros estudios similares3,5,7,19.
La discordancia observada respecto de 3 microorganismos que fueron aislados en el cultivo y no fueron detectados por FilmArray® BCID2 se explica como una limitación de la técnica, dado que no estaban incluidos entre los blancos de detección del panel, situación ya observada en varios estudios3,5,8,11,18–20.
En cuanto a la detección de E. coli por el panel BCID2, se debe tener en cuenta que presenta reactividad cruzada con especies de Shigella (Shigella boydii, Shigella dysenteriae, Shigella flexneri y Shigella sonnei)10. Esta situación ya fue reportada en un estudio de Holma et al.8, donde S. flexneri aislada de una bacteriemia monomicrobiana se detectó de manera incorrecta en el panel BCID2 como E. coli. Si bien la incidencia de bacteriemia en pediatría es de 0,4-7,3%2, este es un dato para tener en cuenta. El fabricante del panel BCID2 menciona que una situación similar sucede con las levaduras C. parapsilosis y C. tropicalis, que pueden dar reacción cruzada cuando el inóculo es elevado, o que BCID2 puede informar ambas cuando solamente una de las 2 especies está presente. Esto no ha sucedido en este estudio: en la única bacteriemia detectada por BCID2 como C. tropicalis, esta fue posteriormente aislada del cultivo.
Debido a que los blancos que incluye BCID2 están limitados a los microorganismos más habituales causantes de bacteriemias, es importante resaltar que un resultado negativo no excluye la posibilidad de infección del torrente sanguíneo. Asimismo, resultados positivos no descartan la infección simultánea con otros microorganismos no incluidos en el panel. Además, podrían producirse resultados negativos de BCID2 debido a la presencia de variantes de secuencia en la región a la que se dirige el test, a la presencia de inhibidores o a errores técnicos9.
En cuanto a los microorganismos detectados por BCID2 y que no se aislaron del cultivo, todos estaban formando parte de bacteriemias polimicrobianas, que representan entre el 10 y el 20% de todas las bacteriemias, pero que suelen asociarse a tasas de mortalidad incluso más altas que las monomicrobianas4,14,15,17. Esta situación en bacteriemias polimicrobianas ya fue comunicada en otras publicaciones3,17–19.
En el estudio multicéntrico de Soloaga et al.18 que abarcó 5 hospitales de nuestro país (incluido un hospital pediátrico), sobre un total de 200 microorganismos, 9 fueron detectados por el panel BCID2, pero no desarrollaron en el cultivo convencional (1 S. aureus, 2 S. epidermidis, 1K. pneumoniae, 1 P. aeruginosa, 2 E. faecium, 1 B. fragilis y 1 Streptococcus spp.). Sparks et al.19 informaron resultados discrepantes en los hemocultivos polimicrobianos debido a que BCID2 detectó una cepa de K. oxytoca que no desarrolló en el cultivo y también al aislamiento por parte del cultivo de microorganismos que no forman parte de los blancos. En el estudio de Benrison et al.3, BCID2 detectó de manera adicional S. epidermidis en bacteriemias polimicrobianas (n=3), que no se aisló en el cultivo, e incluso observaron discordancias en la identificación de estafilococos coagulasa negativos entre el cultivo y FilmArray®, tanto en cultivos mixtos como en monomicrobianos.
Tal como señala el fabricante de BCID2, la ausencia de desarrollo mediante cultivo de microorganismos que son detectados por FilmArray®, cuando estos forman parte de hemocultivos polimicrobianos, podría deberse al crecimiento más rápido de bacterias gram negativas que, posiblemente, inhiban o enmascaren el crecimiento de gram positivas, o a la mayor sensibilidad de la metodología. Otras causas podrían ser la toma del hemocultivo una vez implementado el tratamiento antibacteriano o antifúngico, la presencia de material genético de bacterias no viables o su liberación desde un foco infeccioso, sin estar presente el microorganismo en el torrente sanguíneo9.
En estos casos, donde BCID2 detecta más de un microorganismo y estos no se aíslan del cultivo (bacteriemias polimicrobianas), sería de utilidad repetir la siembra del hemocultivo sobre medios de cultivo selectivos o con discos de antibióticos para inhibir a la bacteria que desarrolla primero, permitiendo así el crecimiento de aquellas no aisladas por cultivo. Los medios de cultivo y los discos de antibióticos por emplear dependerán de las especies bacterianas detectadas.
Debido a la baja prevalencia de carbapenemasas en hemocultivos en el hospital, no se detectaron KPC, NDM, IMP, VIM y OXA-48-like. Lo mismo sucede con los genes de resistencia a vancomicina en los enterococos (vanA/B) y con la resistencia a colistina mediada por mcr-1, que tampoco fueron detectados en el período evaluado. En un estudio llevado a cabo en un hospital infantil de EE. UU., Graff et al.7 tampoco detectaron dichos genes de resistencia (KPC, NDM, IMP, VIM, OXA-48-like, vanA/B y mcr-1) sobre un total de 191 FilmArray® BCID2. En cambio, Soloaga et al.18 detectaron por BCID2 16 carbapenemasas (12 KPC, 3 NDM y 1 VIM), además 18 CTX-M y 3 vanA/B. La detección rápida de los genes de resistencia a carbapenems tendría un beneficio adicional potencial en aquellas instituciones con tasas más altas.
En el presente estudio, se aisló una cepa de K. pneumoniae productora de BLEE en el cultivo y que no fue detectada por el panel BCID2; lo más probable es que no se tratase de una enzima de tipo CTX-M, que es el único blanco de este tipo de enzimas que posee el panel, aunque no se realizó el análisis molecular para identificar a la enzima involucrada. Berinson et al.3 también informaron que 4 aislamientos (2K. pneumoniae, 1K. oxytoca y 1 E. coli) sobre un total de 16 fueron reportados fenotípicamente como resistentes a las cefalosporinas y el panel BCID2 no detectó el gen CTX-M.
Se observó una discrepancia con un aislamiento de S. aureus, que fue detectado por FilmArray® BCID2 como meticilino-resistente (mecA/C y MREJ detectado), pero que por cultivo fue caracterizado como meticilino-sensible. Posteriormente a la obtención de este resultado, se repitió la siembra del hemocultivo en agar manitol salado con discos de cefoxitina para evaluar la posibilidad de que existieran 2 poblaciones, pero no se pudo visualizarlas. Es posible que se tratara de una población heterogénea de S. aureus (resistente y sensible) en la muestra clínica, que los métodos de cultivo no permitan aislar o diferenciar. Por ello, el aislado fue informado e interpretado como meticilino-resistente. Cabe mencionar que este aislamiento de S. aureus se encontraba formando parte de una bacteriemia polimicrobiana con más de 4 microorganismos detectados por BCID2, incluyendo una cepa de S. epidermidis meticilino-resistente (mecA/C detectado). El fabricante describe como una situación infrecuente que una cepa de S. aureus que lleva el casete SCCmec pueda perder el gen mecA/C («casete vacío»), por lo que resultaría meticilino-sensible, pero cuando hay una co-detección adicional de Staphylococcus spp. que lleva el gen mecA/C, S. aureus podría ser caracterizado de manera incorrecta como meticilino-resistente.
El FilmArray® BCID2 detectó 3 genes de resistencia a meticilina correspondientes a estafilococos coagulasa negativos, que no se observaron en el cultivo. Esto se debió a que el panel detectó 3 S. epidermidis que no se aislaron en el cultivo.
El panel BCID2, al ser una técnica rápida, disminuyó significativamente el tiempo para la comunicación de resultados de hemocultivos positivos con respecto al cultivo convencional, lo cual contribuiría a la optimización del tratamiento antibiótico. En otros estudios como en el de Soloaga et al.18, la mediana en horas para BCID2 fue de 3,2h y para los métodos fenotípicos de 33,1h.
La limitación de este estudio radica en el hecho de que al ser monocéntrico, el número de especies y genes de resistencia es limitado y característico de la epidemiología del HGNPE. A su vez, el tipo de diseño utilizado no permitió asegurar que en todos los casos se realizó de manera adicional la siembra en medios selectivos.
Por otra parte, sería interesante evaluar a futuro el impacto clínico de la implementación de esta técnica rápida. Es de esperar que, contando con resultados más rápidos, el personal de salud que atiende al paciente pueda adecuar tempranamente el tratamiento antibiótico empírico. Destacamos la importancia de implementar programas de optimización de uso de antimicrobianos (PROA) en aquellas instituciones que cuentan con esta posibilidad de lograr resultados rápidos y fiables.
ConclusionesEl método FilmArray® panel BCID2 demostró tener excelente concordancia con el cultivo convencional en la identificación de microorganismos en hemocultivos provenientes de bacteriemias monomicrobianas. En hemocultivos polimicrobianos, BCID2 detectó mayor cantidad de microorganismos con respecto al cultivo. En cuanto a los genes de resistencia, la concordancia fue aceptable, con excepciones particulares. La introducción de esta herramienta rápida ha disminuido significativamente el tiempo transcurrido en la emisión de los resultados ante un hemocultivo positivo.
Consideraciones éticasEl estudio fue desarrollado de acuerdo con lo establecido en las normativas que rigen la investigación en salud en el ámbito de los establecimientos dependientes del Gobierno de la Ciudad de Buenos Aires y fue aprobado por del Comité de Ética en Investigación del HGNPE (Código de PRIISA BA: 7180).
FinanciaciónLa presente investigación no ha recibido ayudas específicas provenientes de agencias del sector público, sector comercial o entidades sin ánimo de lucro.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.