Las infecciones respiratorias agudas producen una importante morbimortalidad y comúnmente son causadas por virus. En Argentina, los programas de vigilancia epidemiológica se basan en la detección de antígenos virales por inmunofluorescencia (IF), aunque es bien conocido que los métodos moleculares son más sensibles. El panel respiratorio (PR) FilmArray (PR-FilmArray) es un equipo comercial automatizado de PCR múltiples que detecta 17 virus respiratorios y 3 bacterias, en un sistema cerrado que requiere 5min de procesamiento y una 1h de instrumentación. Se evaluó un total de 315 muestras respiratorias de niños menores de 6 años con infecciones respiratorias agudas por IF para 8 virus respiratorios y por RT-PCR para rinovirus. Posteriormente, estas muestras se estudiaron con el PR-FilmArray. La frecuencia de positividad al considerar los 9 virus estudiados por IF y RT-PCR fue del 75%; por PR-FilmArray fue del 92%. El porcentaje de acuerdo positivo entre ambas metodologías fue del 70,5% y el de acuerdo negativo fue del 99,6% (intervalo de confianza 95%: 65,5-75,1 y 99,2-99,8, respectivamente). El PR-FilmArray permitió obtener un mayor diagnóstico positivo (97%) y detectó otros virus, como los coronavirus NL63, 229E, OC43 y HKU1 (10%) y los bocavirus (18%). Además, permitió identificar coinfecciones múltiples (39%) con 2, 3, 4 y hasta 5 virus. Actualmente, la IF continúa siendo el método más utilizado en los países latinoamericanos para el diagnóstico de virus respiratorios por su bajo costo, por su capacidad para procesar un alto número de muestras simultáneamente y porque los resultados de los virus más frecuentes están disponibles en 5h. Sin embargo, la futura incorporación de métodos moleculares aumentaría notablemente la capacidad diagnóstica.
Acute respiratory infections, which are commonly caused by viruses, are an important cause of morbidity and mortality in children. In Argentina, national surveillance programs for the detection of respiratory viruses are usually performed by using immunofluorescence (IF) assays, although it is well known that molecular methods are more sensitive. An automated multiplex PCR device, the FilmArray-Respiratory Panel (FilmArray-RP), can detect 17 viral and 3 bacterial pathogens in a closed system that requires only 5min of hands-on time and 1h of instrumentation time. A total of 315 respiratory samples from children under 6 years of age suffering from acute respiratory infections, were studied by IF for 8 respiratory viruses and by RT-PCR for rhinoviruses. Later, these samples were tested by the FilmArray-RP. The positivity frequency obtained for the 9 viruses tested was 75% by IF/RT-PCR and 92% by the FilmArray-RP. The positive and negative percent agreement between both methods was 70.5% whereas the negative percent agreement was 99.6% (95% confidence interval:65.5-75.1 and 99.2-99.8 respectively). The FilmArray-RP allowed a higher positive diagnosis (97%) and detected other viruses such as coronavirus NL63, 229E, OC43, HKU1 (10%) and bocavirus (18%). In addition, this method identified multiple coinfections (39%) with 2, 3, 4 and up to 5 different viruses. At present, IF is still the most frequently used method in most Latin American countries for respiratory viruses diagnosis due to its low cost, its capability to process a high number of samples simultaneously and the fast determination of results for the most frequent viruses, which are available within 5h. However, the coming incorporation of molecular methods in routine procedures will significantly increase the diagnostic yield of these infections.
Las infecciones respiratorias agudas (IRA) son una de las principales causas de consultas médicas y de hospitalización en niños, ancianos y en pacientes inmunocomprometidos, con mayor frecuencia durante el otoño e invierno1,10. La mayoría de estas infecciones son causadas por virus respiratorios, que pueden ser detectados por métodos clásicos, como el cultivo o la identificación de antígenos virales por inmunofluorescencia (IF); por pruebas rápidas de inmunocromatografía o por métodos moleculares que amplifican el genoma viral, como la PCR.
Tanto en Argentina como en muchos países latinoamericanos, los programas nacionales de vigilancia epidemiológica se basan en técnicas de IF. Este método ha sido el de elección por su bajo costo, su capacidad para procesar un gran número de muestras simultáneamente y su simplicidad. Sin embargo, requiere un operador entrenado, un microscopio de fluorescencia de calidad y el uso de controles adecuados. Aunque es bien sabido que los métodos moleculares son más sensibles que la IF, no han sido aún ampliamente incorporados debido al mayor costo de los reactivos y del equipamiento, y al tiempo requerido para la extracción, amplificación y detección del genoma viral. Sin embargo, a partir de la pandemia de influenza en 2009, la implementación de métodos moleculares ha aumentado significativamente, incluyendo las RT-PCR para influenza y los rinovirus en el diagnóstico de rutina.
En la actualidad, existe una variedad de ensayos comerciales de PCR múltiples que permiten la detección de los virus respiratorios, con una alta sensibilidad y especificidad12. Uno de ellos es el panel respiratorio (PR) FilmArray (FilmArray Respiratory Panel [BioFire Diagnostics, UT, EE. UU.]) (PR-FilmArray), que ha sido aprobado recientemente (2012) por la FDA. Este es un equipo automatizado que utiliza un cartucho plástico con diferentes compartimentos que incluyen todos los reactivos para realizar la extracción de los ácidos nucleicos y PCR múltiples. El PR-FilmArray permite la detección de 17 virus respiratorios y 3 patógenos bacterianos, y requiere 5min de procesamiento manual y 65min de tiempo de instrumentación13.
El objetivo de este estudio fue comparar el desempeño de los métodos habitualmente utilizados en nuestro laboratorio (IF y RT-PCR) con el PR-FilmArray para el diagnóstico viral de las IRA en niños. Asimismo, se analizó la capacidad del ensayo comercial para detectar otros patógenos respiratorios que generalmente no son incluidos en el diagnóstico de rutina de las IRA en Argentina.
Materiales y métodosSe realizó un estudio retrospectivo en 315 muestras respiratorias congeladas a −70°C para evaluar el PR-FilmArray y compararlo con los métodos utilizados en nuestro laboratorio (IF y RT-PCR).
Muestras respiratoriasLas muestras a analizar consistieron en 133 aspirados nasofaríngeos de pacientes pediátricos internados y 182 hisopados nasofaríngeos de pacientes pediátricos ambulatorios. En todos los casos, las muestras fueron obtenidas de niños menores de 6 años con IRA. Estos pacientes habían sido enrolados en un estudio previo para determinar la etiología de las IRA, realizado de junio de 2008 a mayo de 2010 en el Centro de Investigaciones Médicas e Investigaciones Clínicas (CEMIC)7. El proyecto fue aprobado por el Comité de Ética del CEMIC; todas las muestras fueron codificadas para asegurar el anonimato, de acuerdo a la Declaración de Helsinki y la Ley de Habeas Data Argentina (n.o 25326).
Todas las muestras se obtuvieron al ingreso y se enviaron al Laboratorio de Virología Clínica para la detección por IF indirecta (Chemicon/Millipore, CA, EE. UU.) de los antígenos virales del virus sincicial respiratorio (Human respiratory syncytial virus [RSV]), influenza A y B (FluA/FluB), parainfluenza (PIV) 1-3, adenovirus (AdV); y por IF directa (Argene, Verniolle, Francia) para los metapneumovirus humanos (Human metapneumovirus [HMPV]). Los rinovirus humanos (Human rhinoviruses [HRV]) se estudiaron utilizando una RT-PCR en tiempo real9.
Las 133 muestras correspondientes a pacientes internados se seleccionaron según los siguientes criterios: a) muestras con un resultado positivo para FluA, FluB, AdV o PIV por IF (n=37); b) muestras respiratorias que fueron positivas para RSV (n=31) y HMPV (n=10) por IF, y muestras positivas para HRV (n=64) por RT-PCR; c) muestras negativas para todos los virus estudiados (n=14) por IF y RT-PCR.
De las 133 muestras respiratorias seleccionadas de los niños internados, 23 tenían coinfección viral. Para las muestras de los puntos b y c, se utilizó un método de selección al azar sistemático, representativo de todos los meses de estudio. De los pacientes ambulatorios se incluyeron todas las muestras (n=182).
Panel respiratorio FilmArrayEl PR-FilmArray permite realizar la detección simultánea de 20 patógenos respiratorios incluyendo RSV, FluA, FluA/H1, FluA/H1 2009, FluA/H3, FluB, AdV, PIV 1, 2, 3 y 4, HRV/enterovirus humanos (Human enterovirus [HEV]), HMPV, bocavirus (Human bocavirus [HBoV]), coronavirus humano (Human coronavirus [HCoV]) OC43, 229E, NL63 y HKU1, Bordetella pertussis, Mycoplasma pneumoniae y Chlamydophila pneumoniae13.
El ensayo se inicia con la introducción de 300μl de la muestra en un cartucho, el cual se inserta en el instrumento donde comienza la lisis celular, seguida por la extracción de ácidos nucleicos. Posteriormente se realiza una transcripción inversa y una primera PCR múltiple, y por último, una etapa de PCR individuales. El instrumento se conecta a un software que analiza las curvas de fluorescencia en tiempo real en conjunto con las curvas de fusión de punto final o melting. De este modo, informa finalmente un resultado positivo o negativo para cada patógeno.
Análisis estadísticoPara los 9 virus respiratorios detectados por nuestros métodos de rutina y por el PR-FilmArray, se calcularon los porcentajes de acuerdo positivo y negativo con sus respectivos intervalos de confianza 95% (IC 95%). Además, se utilizó la prueba de McNemar para evaluar la concordancia de los resultados obtenidos por ambos métodos. Los patógenos respiratorios no estudiados por nuestros métodos de laboratorio, pero sí incluidos en el PR-FilmArray, específicamente HCoV, HBoV y bacterias, no fueron considerados discordantes. El porcentaje de acuerdo positivo (sensibilidad) es el número de veces que nuestros métodos detectaron un virus cuando el PR-FilmArray también lo detectó, mientras que el porcentaje de acuerdo negativo (especificidad) es el número de veces que nuestros métodos no detectaron un virus cuando el PR-FilmArray tampoco lo detectó. Se asumió significación estadística para valores de p<0,05. Los análisis estadísticos se realizaron utilizando el programa STATA 11.1 (Stata Corp., TX, EE. UU.).
ResultadosDe las 315 muestras respiratorias de niños con IRA estudiadas, 235 (74,6%) fueron positivas con los ensayos utilizados en nuestro laboratorio (IF para 8 virus y RT-PCR para HRV). Cuando las mismas muestras se estudiaron con el PR-FilmArray (para 17 virus y 3 bacterias), la frecuencia de positividad aumentó al 96,8% (305/315). Sin embargo, si consideramos solo los 9 virus respiratorios que detectan ambos métodos (RSV, AdV, FluA, FluB, PIV 1-3, HMPV, HRV), el PR-FilmArray permitió un diagnóstico positivo en el 92,1% (290/315) de las muestras.
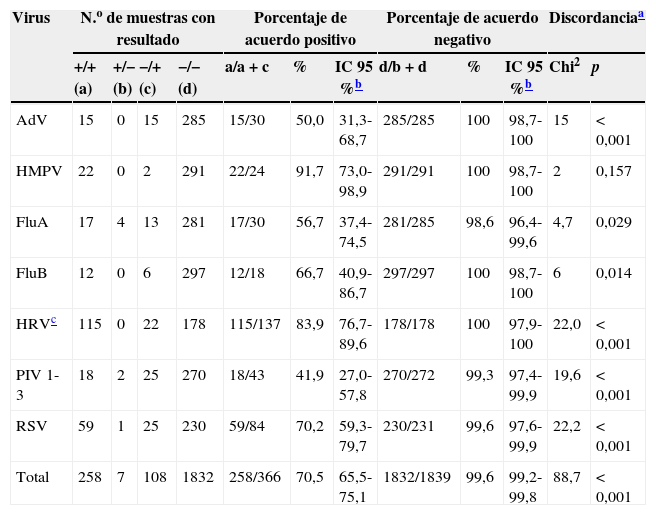
El porcentaje de acuerdo positivo (sensibilidad) de nuestros métodos con el PR-FilmArray varió desde el 41,9% hasta el 91,7% dependiendo del virus (tabla 1). Sin embargo, solo para 2 virus, HMPV y HRV, el porcentaje superó el 80%. Los virus con el acuerdo positivo más bajo fueron PIV 1-3 (41,9%) y AdV (50,0%).
Comparación del desempeño de la inmunofluorescencia (IF) o la RT-PCR frente al panel respiratorio FilmArray (PR-FilmArray) en la detección de virus respiratorios en niños con infección respiratoria aguda (n=315)
| Virus | N.o de muestras con resultado | Porcentaje de acuerdo positivo | Porcentaje de acuerdo negativo | Discordanciaa | ||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| +/+ (a) | +/− (b) | −/+ (c) | −/− (d) | a/a+c | % | IC 95%b | d/b+d | % | IC 95%b | Chi2 | p | |
| AdV | 15 | 0 | 15 | 285 | 15/30 | 50,0 | 31,3-68,7 | 285/285 | 100 | 98,7-100 | 15 | <0,001 |
| HMPV | 22 | 0 | 2 | 291 | 22/24 | 91,7 | 73,0-98,9 | 291/291 | 100 | 98,7-100 | 2 | 0,157 |
| FluA | 17 | 4 | 13 | 281 | 17/30 | 56,7 | 37,4-74,5 | 281/285 | 98,6 | 96,4-99,6 | 4,7 | 0,029 |
| FluB | 12 | 0 | 6 | 297 | 12/18 | 66,7 | 40,9-86,7 | 297/297 | 100 | 98,7-100 | 6 | 0,014 |
| HRVc | 115 | 0 | 22 | 178 | 115/137 | 83,9 | 76,7-89,6 | 178/178 | 100 | 97,9-100 | 22,0 | <0,001 |
| PIV 1-3 | 18 | 2 | 25 | 270 | 18/43 | 41,9 | 27,0-57,8 | 270/272 | 99,3 | 97,4-99,9 | 19,6 | <0,001 |
| RSV | 59 | 1 | 25 | 230 | 59/84 | 70,2 | 59,3-79,7 | 230/231 | 99,6 | 97,6-99,9 | 22,2 | <0,001 |
| Total | 258 | 7 | 108 | 1832 | 258/366 | 70,5 | 65,5-75,1 | 1832/1839 | 99,6 | 99,2-99,8 | 88,7 | <0,001 |
AdV: adenovirus; HMPV: metapneumovirus; Flu: influenza; HRV: rinovirus; PIV: parainfluenza; RSV: virus sincicial respiratorio.
(a) +/+: positivo por ambas metodologías; (b) +/−: positivo por nuestros métodos de rutina y negativo por PR-FilmArray; (c) −/+: negativo por nuestros métodos de rutina y positivo por el PR-FilmArray; (d) −/−: negativo por ambas metodologías.
El porcentaje de acuerdo negativo (especificidad) fue alto para todos los virus respiratorios, y varió entre el 98,6% y el 100%.
Al comparar los diagnósticos obtenidos por nuestros ensayos con los del PR-FilmArray, se observaron resultados significativamente discordantes entre ambas metodologías para todos los virus, excepto para HMPV. La diferencia entre ambas metodologías fue más evidente en los niños ambulatorios que en los internados (tabla 2). En estos últimos, las frecuencias de positividad obtenidas con nuestros ensayos y con el PR-FilmArray fueron del 89,5% y del 99,2%, respectivamente, mientras que en ambulatorios estas fueron del 63,7% y del 95,1%, respectivamente.
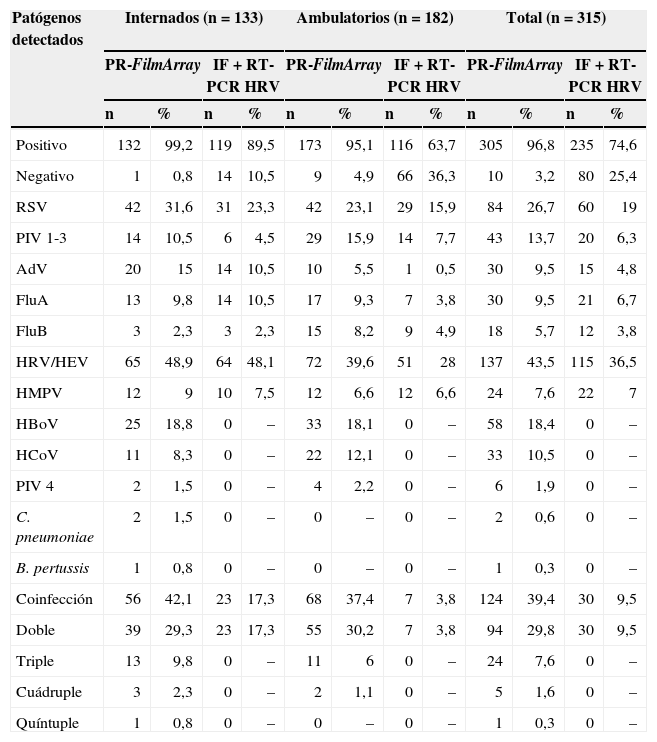
Detección de patógenos respiratorios en niños con infección respiratoria aguda por inmunofluorescencia (IF)/RT-PCR y por el panel respiratorio FilmArray (PR-FilmArray)
| Patógenos detectados | Internados (n=133) | Ambulatorios (n=182) | Total (n=315) | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| PR-FilmArray | IF+RT-PCR HRV | PR-FilmArray | IF+RT-PCR HRV | PR-FilmArray | IF+RT-PCR HRV | |||||||
| n | % | n | % | n | % | n | % | n | % | n | % | |
| Positivo | 132 | 99,2 | 119 | 89,5 | 173 | 95,1 | 116 | 63,7 | 305 | 96,8 | 235 | 74,6 |
| Negativo | 1 | 0,8 | 14 | 10,5 | 9 | 4,9 | 66 | 36,3 | 10 | 3,2 | 80 | 25,4 |
| RSV | 42 | 31,6 | 31 | 23,3 | 42 | 23,1 | 29 | 15,9 | 84 | 26,7 | 60 | 19 |
| PIV 1-3 | 14 | 10,5 | 6 | 4,5 | 29 | 15,9 | 14 | 7,7 | 43 | 13,7 | 20 | 6,3 |
| AdV | 20 | 15 | 14 | 10,5 | 10 | 5,5 | 1 | 0,5 | 30 | 9,5 | 15 | 4,8 |
| FluA | 13 | 9,8 | 14 | 10,5 | 17 | 9,3 | 7 | 3,8 | 30 | 9,5 | 21 | 6,7 |
| FluB | 3 | 2,3 | 3 | 2,3 | 15 | 8,2 | 9 | 4,9 | 18 | 5,7 | 12 | 3,8 |
| HRV/HEV | 65 | 48,9 | 64 | 48,1 | 72 | 39,6 | 51 | 28 | 137 | 43,5 | 115 | 36,5 |
| HMPV | 12 | 9 | 10 | 7,5 | 12 | 6,6 | 12 | 6,6 | 24 | 7,6 | 22 | 7 |
| HBoV | 25 | 18,8 | 0 | – | 33 | 18,1 | 0 | – | 58 | 18,4 | 0 | – |
| HCoV | 11 | 8,3 | 0 | – | 22 | 12,1 | 0 | – | 33 | 10,5 | 0 | – |
| PIV 4 | 2 | 1,5 | 0 | – | 4 | 2,2 | 0 | – | 6 | 1,9 | 0 | – |
| C. pneumoniae | 2 | 1,5 | 0 | – | 0 | – | 0 | – | 2 | 0,6 | 0 | – |
| B. pertussis | 1 | 0,8 | 0 | – | 0 | – | 0 | – | 1 | 0,3 | 0 | – |
| Coinfección | 56 | 42,1 | 23 | 17,3 | 68 | 37,4 | 7 | 3,8 | 124 | 39,4 | 30 | 9,5 |
| Doble | 39 | 29,3 | 23 | 17,3 | 55 | 30,2 | 7 | 3,8 | 94 | 29,8 | 30 | 9,5 |
| Triple | 13 | 9,8 | 0 | – | 11 | 6 | 0 | – | 24 | 7,6 | 0 | – |
| Cuádruple | 3 | 2,3 | 0 | – | 2 | 1,1 | 0 | – | 5 | 1,6 | 0 | – |
| Quíntuple | 1 | 0,8 | 0 | – | 0 | – | 0 | – | 1 | 0,3 | 0 | – |
RSV: virus sincicial respiratorio; PIV: parainfluenza; AdV: adenovirus; Flu: influenza; HRV: rinovirus; HMPV: metapneumovirus; HBoV: bocavirus; HCoV: coronavirus.
El PR-FilmArray detectó patógenos respiratorios que no son estudiados por nuestros ensayos de rutina (tabla 2). Se observó una alta frecuencia de HBoV, presente en el 18,4% de los niños estudiados. Se detectaron las 4 especies de HCoV (10,5%): 12 HCoV-OC43, 12 HCoV-HKU1, 7 HCoV-NL63 y 2 HCoV-229E. La mayoría se encontraron en infección múltiple, excepto 3 de ellos detectados como agentes únicos. El PIV 4 se detectó en 6 (1,9%) pacientes, en 3 como agente único y en los otros 3 en coinfecciones con HRV y HBoV. Respecto a las bacterias, se detectaron 2 C. pneumoniae y una B. pertussis, ambas en coinfección con algún virus respiratorio, en muestras de niños internados. No se detectó ningún M. pneumoniae.
Con los métodos empleados en nuestro laboratorio se detectaron coinfecciones virales en 30 de 235 (13%) pacientes con diagnóstico positivo; estas fueron significativamente más frecuentes en niños internados que en ambulatorios. La coinfección HRV-RSV fue la más frecuente (n=8), seguida por HRV-AdV (n=6) y HRV-PIV (n=5) (tabla 2).
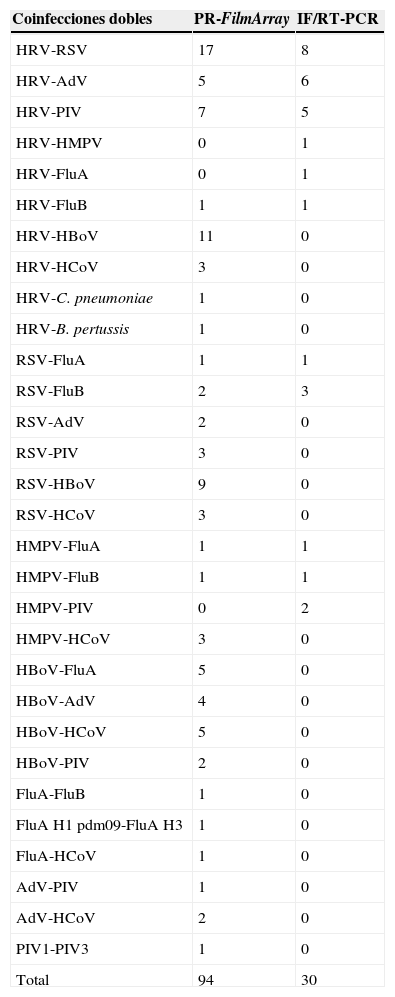
Cuando las muestras se estudiaron con el PR-FilmArray, el número de coinfecciones múltiples fue mayor: 124 de 305 muestras positivas (40,7%). En detalle, 94 (75,8%) muestras fueron positivas para 2 patógenos, 24 (19,4%) fueron positivas para 3 virus, 5 (4%) fueron coinfecciones cuádruples y solo una muestra (0,8%) fue positiva para 5 patógenos. Las coinfecciones dobles más frecuentes fueron HRV-RSV (n=17), HRV-HBoV (n=11), RSV-HBoV (n=9) y HRV-PIV (n=7) (tabla 3). Las coinfecciones múltiples (triples, cuádruples y quíntuples) detectadas pueden observarse en detalle en la tabla 4.
Detección de coinfecciones dobles mediante el panel espiratorio FilmArray (PR-FilmArray) y por inmunofluorescencia (IF)/RT-PCR en niños con infección respiratoria aguda
| Coinfecciones dobles | PR-FilmArray | IF/RT-PCR |
|---|---|---|
| HRV-RSV | 17 | 8 |
| HRV-AdV | 5 | 6 |
| HRV-PIV | 7 | 5 |
| HRV-HMPV | 0 | 1 |
| HRV-FluA | 0 | 1 |
| HRV-FluB | 1 | 1 |
| HRV-HBoV | 11 | 0 |
| HRV-HCoV | 3 | 0 |
| HRV-C. pneumoniae | 1 | 0 |
| HRV-B. pertussis | 1 | 0 |
| RSV-FluA | 1 | 1 |
| RSV-FluB | 2 | 3 |
| RSV-AdV | 2 | 0 |
| RSV-PIV | 3 | 0 |
| RSV-HBoV | 9 | 0 |
| RSV-HCoV | 3 | 0 |
| HMPV-FluA | 1 | 1 |
| HMPV-FluB | 1 | 1 |
| HMPV-PIV | 0 | 2 |
| HMPV-HCoV | 3 | 0 |
| HBoV-FluA | 5 | 0 |
| HBoV-AdV | 4 | 0 |
| HBoV-HCoV | 5 | 0 |
| HBoV-PIV | 2 | 0 |
| FluA-FluB | 1 | 0 |
| FluA H1 pdm09-FluA H3 | 1 | 0 |
| FluA-HCoV | 1 | 0 |
| AdV-PIV | 1 | 0 |
| AdV-HCoV | 2 | 0 |
| PIV1-PIV3 | 1 | 0 |
| Total | 94 | 30 |
HRV: rinovirus; RSV: virus sincicial respiratorio; AdV: adenovirus; PIV: parainfluenza; Flu: influenza; HMPV: metapneumovirus; HBoV: bocavirus; HCoV: coronavirus; pdm09: pandémica 2009.
Detección de coinfecciones múltiples (triples, cuádruples y quíntuples) mediante el panel respiratorio FilmArray (PR-FilmArray) en niños con infección respiratoria aguda
| Coinfecciones | PR-FilmArray |
|---|---|
| Triples | 21 |
| HRV-RSV-FluA | 1 |
| HRV-RSV-PIV | 1 |
| HRV-RSV-HBoV | 3 |
| HRV-RSV-HCoV | 1 |
| HRV-HBoV-FluB | 1 |
| HRV-HBoV-AdV | 1 |
| HRV-HBoV-PIV | 2 |
| HRV-HBoV-HCoV | 1 |
| HRV-AdV-PIV | 1 |
| HRV-HCoV HKU1-HCoV OC43 | 1 |
| RSV-HBoV-FluA | 2 |
| RSV-HBoV-AdV | 1 |
| RSV-HBoV-HCoV | 1 |
| RSV-FluB-C. pneumoniae | 1 |
| HMPV-HBoV-PIV | 1 |
| HMPV-PIV-HCoV | 1 |
| HMPV-FluA-AdV | 1 |
| Cuádruples | 8 |
| HMPV-HCoV HKU1-HCoV OC43 | 1 |
| HBoV-HCoV HKU1-HCoV OC44 | 2 |
| HRV-AdV-HBoV-RSV | 1 |
| HRV-AdV-HBoV-FluA | 1 |
| HRV-AdV-HBoV-FluB | 1 |
| HRV-AdV-HBoV-HCoV | 1 |
| HRV-HMPV-PIV-HCoV | 1 |
| Quíntuples | 1 |
| HRV-AdV-HBoV-HMPV-HCoV | 1 |
| Total | 30 |
HRV: rinovirus; RSV: virus sincicial respiratorio; AdV: adenovirus; PIV: parainfluenza; Flu: influenza; HMPV: metapneumovirus; HBoV: bocavirus; HCoV: coronavirus.
En niños con IRA, el PR-FilmArray permitió un mayor diagnóstico de virus respiratorios (97%) que los métodos de rutina utilizados en nuestro laboratorio (75%). Esto no solamente ocurrió por la detección de virus y bacterias que no se investigan habitualmente, sino también por la mayor sensibilidad del PR-FilmArray en la detección de los virus que se pueden detectar por IF. Si consideramos solo los 8 virus respiratorios estudiados por IF y los HRV por RT-PCR, la frecuencia de positividad alcanzada por el PR-FilmArray fue del 92%.
Las frecuencias de positividad obtenidas en pacientes internados fueron significativamente mayores que en ambulatorios, tanto por nuestros métodos como con el PR-FilmArray. Esto podría deberse a una posible diferencia en la calidad de las muestras analizadas, las cuales variaron de acuerdo a la condición del paciente: aspirados nasofaríngeos en internados e hisopados nasofaríngeos con hisopos flexibles en ambulatorios. Sin embargo, numerosas publicaciones describen que los hisopos flexibles permiten obtener muestras de calidad similar a los aspirados nasofaríngeos2,4.
Debido a que no existe aún un método patrón para la detección de todos los virus respiratorios, cuando comparamos los resultados obtenidos por nuestros procedimientos habituales con el PR-FilmArray empleamos el término «acuerdo positivo» en lugar de sensibilidad, y «acuerdo negativo» por especificidad. El acuerdo positivo obtenido fue muy variable dependiendo del virus estudiado: fue alto para HMPV y HRV, aceptable para RSV y Flu, pero muy bajo para AdV y PIV. Si consideramos el acuerdo negativo observado, este fue muy alto para todos los virus estudiados, del 98-100%. Como era esperable, sobre todo para virus de baja frecuencia, existe una mayor posibilidad de que ambas metodologías coincidan en el diagnóstico de las muestras negativas. Analizando todos los datos en conjunto, pudimos determinar que solamente la IF para HMPV mostró resultados significativamente concordantes con el PR-FilmArray, mientras que para el resto, los resultados fueron significativamente discordantes, lo que implica que usando IF, como es bien sabido, se pierde un elevado número de diagnósticos positivos.
Poritz et al.13 compararon el PR-FilmArray con la IF directa y observaron un amplio rango de acuerdo positivo: para la mayoría de los virus este fue superior al 90%.
En relación con la detección de los HRV, nuestra RT-PCR detectó 115 casos, mientras que el PR-FilmArray detectó 137. Es importante destacar que el PR-FilmArray utiliza una combinación de PCR que detectan HRV y HEV, sin capacidad de distinguir entre ambos virus. Nuestra RT-PCR, que utiliza primers y una sonda adaptados del protocolo del CDC, tiene una mayor especificidad para HRV, aunque puede detectar algún enterovirus si estuviera presente en títulos muy altos9. Por ello, es posible que el PR-FilmArray sea más sensible que nuestra RT-PCR para algunos genotipos de HRV, pero también es probable que algunas muestras negativas por nuestra RT-PCR y positivas por el PR-FilmArray hayan sido HEV y no HRV11.
El PR-FilmArray permitió detectar por primera vez en nuestro medio los 4 HCoV causantes de infección respiratoria (NL63, 229E, OC43 y HKU1), con una frecuencia total del 10%. El trabajo realizado por Xu et al.17 detalla que en su población de estudio los diagnósticos positivos adicionales obtenidos con el PR-FilmArray fueron a expensas de la detección de los HRV y los HCoV, que no son incluidos en la detección por IF directa utilizada de rutina.
Los HBoV se detectaron con una frecuencia del 18% en ambas poblaciones de niños, internados y ambulatorios. Sin embargo, su papel como agente causal de IRA es aún controvertido, ya que se ignora su papel real como patógeno, por sí mismo o en coinfección con otros virus14.
Si bien se detectaron coinfecciones virales con nuestros métodos de rutina, su frecuencia aumentó 4 veces al utilizar el PR-FilmArray. Con este equipo se identificaron coinfecciones con 2, 3, 4 y hasta 5 virus respiratorios, y solo 3 bacterias se detectaron en coinfección. Para determinar si las coinfecciones se asocian con una mayor gravedad de los cuadros clínicos son necesarios nuevos estudios.
Una de las principales ventajas del PR-FilmArray es el corto tiempo de procesamiento manual (5min) y tan solo 1h de instrumentación hasta la obtención del resultado. Esto tiene un alto impacto clínico en el tratamiento de los pacientes, por ejemplo, en casos de B. pertussis en neonatos o en pacientes con Flu. Además, permite realizar el aislamiento de pacientes en cohortes y extremar medidas de higiene y prevención de la diseminación viral. Otra ventaja es su capacidad para detectar 20 patógenos respiratorios simultáneamente con una adecuada sensibilidad y especificidad. Esto fue probado por muchos investigadores que compararon el PR-FilmArray con otros métodos moleculares, ya sean convencionales o múltiples5,6,12. Además, es un sistema totalmente automatizado y cerrado, que disminuye notablemente la posibilidad de errores y contaminaciones comparado con otros ensayos de PCR. Las desventajas que presenta son su capacidad de procesar solo una muestra por corrida y el alto costo del equipo y de los reactivos.
La IF continúa siendo el método más utilizado en países de Latinoamérica, ya que es una técnica sencilla, de bajo costo, que permite procesar un número adecuado de muestras simultáneamente y obtener los resultados dentro de las 5h. Habitualmente se utiliza la IF para el diagnóstico de RSV, Flu, AdV y PIV 1-3, sin discriminar entre los 3 tipos de PIV y sin detectar el PIV 4. Es bien sabido, y en nuestro estudio lo confirmamos, que la sensibilidad de la IF para AdV es baja; por ello se sugiere el uso de un método molecular para obtener un diagnóstico de certeza3,15. Por el contrario, pudimos demostrar que la IF directa para HMPV presenta una elevada sensibilidad y especificidad, comparable a la de un método molecular como el PR-FilmArray.
Los HRV son los virus más frecuentemente detectados en niños con IRA, tanto en internados como en ambulatorios7,8,16. Sin embargo, dado que no existe una IF para detección directa de sus antígenos en muestras respiratorias, es imprescindible utilizar una técnica de RT-PCR para su diagnóstico.
En conclusión, con el presente estudio hemos confirmado que un método molecular como el que emplea el panel respiratorio FilmArray es más efectivo para realizar el diagnóstico de múltiples patógenos en pacientes con IRA. Sin embargo, por el momento, su costo hace difícil su incorporación en la rutina diaria. Por esta razón, la IF continúa siendo un método aceptable para el diagnóstico de los virus respiratorios más frecuentes.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que los procedimientos seguidos se conformaron a las normas éticas del comité de experimentación humana responsable y de acuerdo con la Asociación Médica Mundial y la Declaración de Helsinki.
Confidencialidad de los datosLos autores declaran que han seguido los protocolos de su centro de trabajo sobre la publicación de datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores han obtenido el consentimiento informado de los pacientes y/o sujetos referidos en el artículo. Este documento obra en poder del autor de correspondencia.
FinanciaciónEste trabajo fue financiado con el subsidio PICT 2006-650 otorgado a la Dra. Echavarria por la Agencia Nacional de Promoción Científica y Tecnológica (ANPCYT).
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
La Dra. D.N. Marcone es becaria posdoctoral y la Sra. C. Ricarte es personal técnico, ambas de CONICET; la Dra. G. Carballal es investigadora de CEMIC y la Dra. M. Echavarria es investigadora de CONICET y CEMIC.
A la Dra. C. Videla por la asistencia técnica. A los Dres. S. Vidaurreta, J. Ekstrom y A. Ellis por el reclutamiento de pacientes y toma de muestra. Al Dr. Fernando Poletta por el asesoramiento estadístico. Se agradece a K. Bourzac, M. Poritz y K. Kanack haber contribuido con los reactivos y el equipamiento FilmArray, BioFire Diagnostics.