Los miembros del complejo Trichophyton mentagrophytes representan el segundo grupo en frecuencia de aislamiento de dermatofitos, luego de Trichophyton rubrum. El citado complejo comprende 3grupos principales: 1)Trichophyton benhamiae y especies relacionadas; 2)Trichophyton simii y 2especies relacionadas, Trichophyton quinckeanum y Trichophyton schoenleinii, y 3)T. mentagrophytes, Trichophyton interdigitale y especies relacionadas. Todos estos organismos son difíciles de identificar a través de la morfofisiología. Se presenta en este informe un estudio descriptivo de 17 cepas clínicas aisladas e identificadas en el Laboratorio de Micología de la Universidad de Valparaíso como pertenecientes al complejo T. mentagrophytes, junto con la caracterización de 3cepas de referencia (T. mentagrophytes CBS 318.56, T. interdigitale CBS 428.63, Trichophyton erinacei CBS 511.73), con el objetivo de clasificarlas a nivel de especie. Se realizaron pruebas morfofisiológicas y moleculares por análisis de curvas de melting de alta resolución (high resolution melting analysis) y secuenciación de regiones ITS. Fenotípicamente, se identificaron 3especies incluidas en el complejo. Los análisis moleculares reclasificaron todas las cepas como pertenecientes a T. interdigitale. En conclusión, no se lograron establecer patrones morfofisiológicos confiables para poder diferenciar entre las especies del complejo.
Trichophyton mentagrophytes complex is the most frequent agent found in dermatophyte isolates after Trichophyton rubrum. It is divided into 3main groups: (1) Trichophyton benhamiae and related species; (2) Trichophyton simii and 2related species, Trichophyton quinckeanum and Trichophyton schoenleinii; and (3) T. mentagrophytes, T. interdigitale, and related species. They are all difficult to identify by morphophysiology. With the aim of classifying them at the species level, a descriptive study was performed on 17 isolated clinical strains identified in the Mycology Laboratory of the Universidad de Valparaíso as belonging to the T. mentagrophytes complex. They were compared with 3 reference strains (T. mentagrophytes CBS 318.56, T. interdigitale CBS 428.63, Trichophyton erinacei CBS 511.73). Morphophysiological and molecular tests were performed by high resolution melting analysis curves and ITS regions sequencing. Phenotypically, 3 species of the complex were identified. Molecular analyses reclassified all the species as belonging to T. interdigitale. In conclusion, no reliable morphophysiological patterns were established to differentiate between the species of the complex. Molecularly, all the strains studied were classified as T. interdigitale.
Las especies del complejo Trichophyton mentagrophytes son agentes cosmopolitas y ocupan el segundo lugar en frecuencia entre los aislamientos de dermatofitos, luego de Trichophyton rubrum. Este complejo incluye especies zoofílicas y antropofílicas10,14.
Rutinariamente, la diferenciación morfológica de los dermatofitos se logra por el estudio y la clasificación de los elementos que intervienen en la reproducción asexual (artroconidios, microconidios, macroconidios) y estructuras vegetativas (clamidosporas; hifas en espiral, raqueta o candelabro; órganos nodulares y pectinados). Estas características microscópicas junto con otras macroscópicas, como el tipo de crecimiento de la colonia y la producción de pigmentos, complementadas con pruebas fisiológicas, como perforación del pelo y presencia de ureasa, son útiles para una tipificación morfofisiológica1. Sin embargo, esta metodología puede no ser suficiente en algunos casos debido al pleomorfismo inherente a algunas cepas o al gatillado, principalmente, por el subcultivo sucesivo para lograr la identificación morfológica final8.
El empleo de técnicas de biología molecular para la clasificación filogenética de dermatofitos ha generado un constante reordenamiento taxonómico. Makimura et al.9 y Graser et al.7 reagruparon las especies y dividieron al complejo T. mentagrophytes en 2: grupo Arthroderma benhamiae y grupo Arthroderma vanbreuseghemii/Arthroderma simii. Actualmente, este complejo se divide en 3grupos principales: 1)Trichophyton benhamiae y especies relacionadas; 2)Trichophyton simii y 2especies relacionadas, Trichophyton quinckeanum y Trichophyton schoenleinii, y 3)T. mentagrophytes, Trichophyton interdigitale y especies relacionadas4,13.
La identificación basada en la secuenciación es considerada el gold standard para diferenciar a los miembros del complejo T. mentagrophytes; sin embargo, esta herramienta no está disponible para todos los laboratorios clínicos. La técnica de high resolution melting analysis (HRMA) es una potencial alternativa a la secuenciación. El análisis de curvas de melting de alta resolución (HRMA) se basa en el comportamiento de desnaturalización del ADN de doble cadena debido a pequeños incrementos de la temperatura, dependiendo de su contenido de G+C y la distribución general de las bases. El HRMA es lo suficientemente sensible como para permitir la detección de un cambio de base única entre secuencias de nucleótidos idénticas. Con el software correcto, el análisis de datos es sencillo y permite analizar múltiples muestras simultáneamente12.
Los objetivos del presente trabajo fueron 2: a) clasificar a nivel de especie las cepas clínicas del complejo T. mentagrophytes aisladas en el Laboratorio de Micología de la Universidad de Valparaíso mediante morfofisiología y secuenciación de espaciadores transcriptos internos (ITS), y b) implementar un método de identificación por HRMA para este complejo.
Materiales y métodosSe realizó un estudio descriptivo de 17 cepas clínicas identificadas en el Laboratorio de Micología de la Universidad de Valparaíso como pertenecientes al complejo T. mentagrophytes. Se emplearon 3cepas de referencia, T. mentagrophytes CBS 318.56, T. interdigitale CBS 428.63 y T. erinacei CBS 511.73, adquiridas a la colección del Centraalbureau voor Schimmelcultures, Utrecht, Países Bajos. Las cepas clínicas se aislaron de uñas de pies (n = 12) y de región plantar (n = 3), interdigital (n = 1) y facial (n = 1).
Identificación morfofisiológicaA partir de los cultivos primarios, todas las cepas se resembraron en 3medios de cultivo: agar Sabouraud (2g de glucosa, 1g de peptona y 16g de agar/l agua destilada); agar lactrimel (10g de miel, 7g de leche, 3g de fécula de maíz y 16g de agar/l agua destilada) y agar avena (30g de avena, 1g de extracto de levadura y 16g de agar/l agua destilada). A los 14 días se registraron los datos macroscópicos de color y tipo de colonia, así como de presencia de pigmentos. Al mismo tiempo, se prepararon muestras microscópicas tomadas de la mitad del radio de cada colonia. Se montaron con azul de lactofenol y se observaron en un microscopio óptico Zeiss Axioskop 2 Plus (LLC, EE. UU.), registrando la cantidad y forma de micro y macroconidios, también la presencia de hifas en espiral. Como test fisiológico se empleó la prueba de ureasa. Se utilizó el medio comercial urea agar base (Difco™, Becton Dickinson & Company, EE. UU.) y se consideró reacción positiva cuando el medio pasó de color amarillo a rosado. Como controles se emplearon las cepas de referencia ya mencionadas. La prueba de ureasa se repitió frente a muestras con resultado negativo, a fin de confirmar dicho resultado.
Identificación molecularEl ADN total se preparó a partir de cada aislamiento usando el kit de extracción GeneJET Plant Genomic DNA Purification Mini Kit (Thermo Scientific, MA, EE. UU.), siguiendo las instrucciones del fabricante. Los primers (Forward 5’-CAACAACGGATCTCTTGGTT-3’; Reverse 5’-GAATGGGCAACAAACCAGC-3’, ambos con temperatura de melting [Tm] de 58°C) fueron diseñados para flanquear los principales sitios polimórficos entre el gen 5,8S y la región ITS2 de las secuencias de referencia, generando un producto de 280 pb. Se emplearon las herramientas disponibles en Primer-BLAST (http://www.ncbi.nlm.nih.gov/tools/primer-blast/), Multialin (http://multalin.toulouse.inra.fr/multalin/multalin.html) y Oligo Analyzer Versión 3.1 (http://biotools.idtdna.com/analyzer/Applications/OligoAnalyzer/Default.aspx).
Para el HRMA se aplicó la tecnología de Step One Plus™ real-time PCR, Applied Biosystem, usando el kit MeltDoctor™ HRM Master Mix (Thermo Scientific, MA, EE. UU.). Cada reacción se realizó en tiras de 8 tubos, por triplicado, con un volumen final de 20μl que contenían 10μl de MeltDoctor™ HRM Master Mix, 0,5μl de cada primer (10μM), 1μl de ADN y 8μl de agua. Las condiciones de amplificación y programación del software fueron las siguientes: activación de la enzima a 95°C por 10 min, seguido de 40 ciclos de desnaturalización a 95°C por 15 s, annealing a 59°C por 20 s y extensión a 60°C por 1 min. Para la curva de melting, las temperaturas y los tiempos fueron los siguientes: desnaturalización a 95°C por 10 s, annealing a 60°C por 1 min, HRMA a 95°C por 15 s y annealing a 60°C por 15 s.
SecuenciaciónDe cada extracto se efectuaron 2PCR convencionales, la primera con los primers diseñados para la prueba de HRMA y la segunda con los primers universales ITS1 (5’-TCCGTAGGTGAACCTTGCGG-3’) e ITS4 (5’-TCCTCCGCTTATTGATATGC-3’). Cada reacción empleó un volumen final de 50μl, que contenían 10μl de buffer 5×, 0,2μl de Taq polimerasa (GoTaq), 3μl de MgCl2 (25mM), 1μl de dNTPS (1mM), 2μl de cada primer (10μM), 1μl de ADN y 30,8μl agua. En la PCR con los primers HRMA, las condiciones fueron 95°C por 5 min, seguido de 35 ciclos de desnaturalización a 95°C por 30 s, annealing a 59°C por 45 s y extensión a 72°C por 45 s. En la otra PCR (primers ITS), se emplearon las mismas condiciones, pero disminuyendo la temperatura de annealing a 55,5°C y aumentando el tiempo de extensión a 1 min. La electroforesis se realizó en gel de agarosa al 2% por 50 min a 75 voltios. Una vez visualizadas las bandas, se purificaron con el kit Wizard® SV Gel and PCR Clean-Up System (Promega Corporation, WI, EE. UU.), empleando el protocolo en gel. Los productos se secuenciaron en un equipo 3730 xl/DNA Analyzer (Applied Biosystems, Thermo Fisher Scientific, EE. UU.).
Análisis filogenéticoLas secuencias de las regiones estudiadas por HRMA e ITS fueron alineadas utilizando el software Clustal X (versión 1.8, Toby Gibson EMBL, Heidelberg, Alemania. Des Higgins Conway Institute, UCD, Dublín, Irlanda. Julie Thompson/Francois Jeanmougin IGBMC, Estrasburgo, Francia). Posteriormente, los alineamientos múltiples fueron corregidos utilizando el programa Gblocks (versión 0.91b, Copyright© Jose Castresana). Para la inferencia filogenética de las secuencias, se utilizó el modelo de máxima verosimilitud implementándose en el programa RaxML, haciendo uso de búsquedas heurísticas, comenzando con adiciones de 100 réplicas aleatorias mediante el método de nearest-neighbor interchange. Para la determinación del bootstrap se usaron 1.000 réplicas. El mejor modelo de sustitución nucleotídica se determinó mediante el programa Modeltest, el modelo seleccionado fue el HKY.
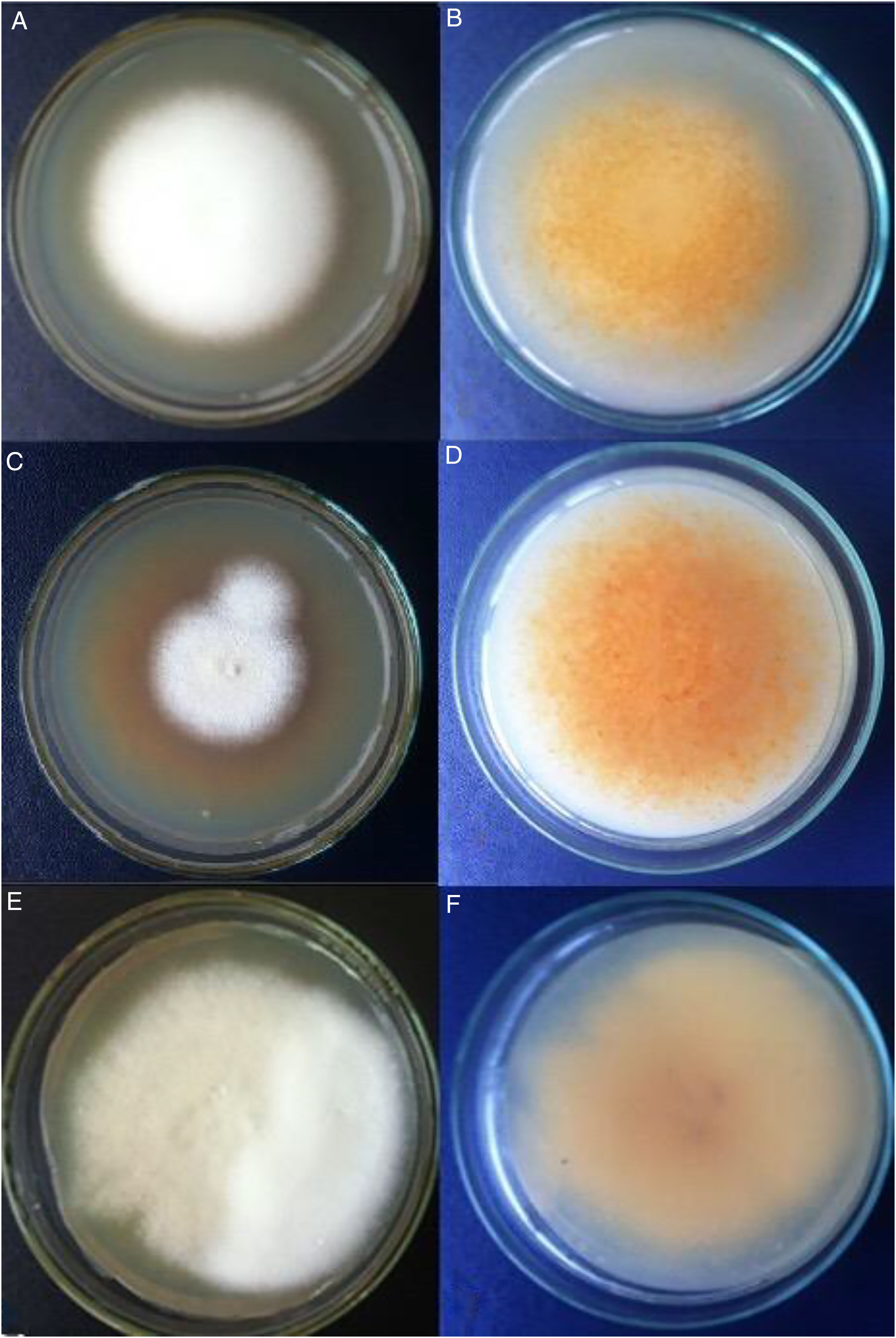
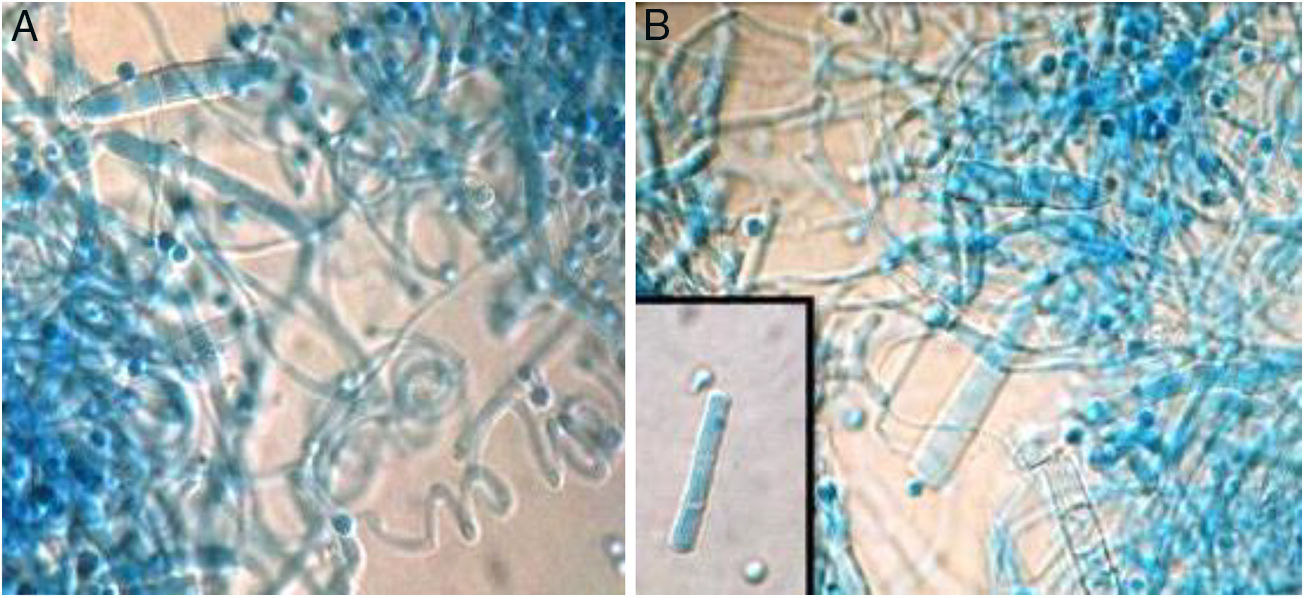
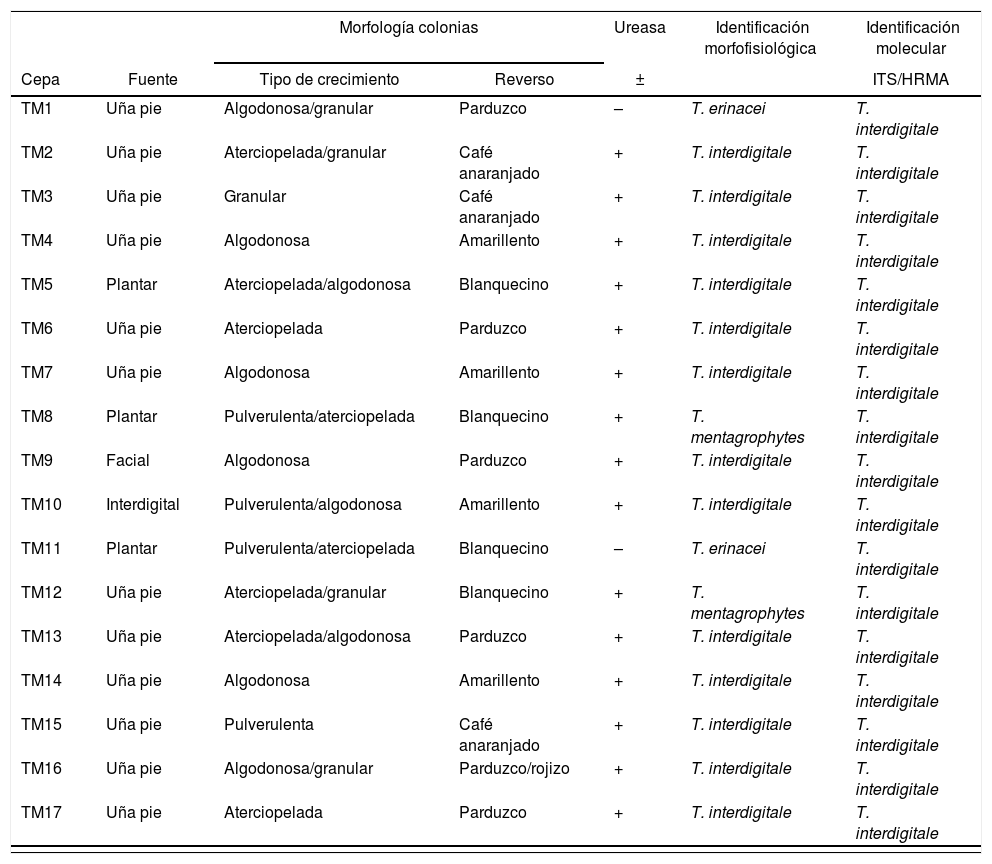
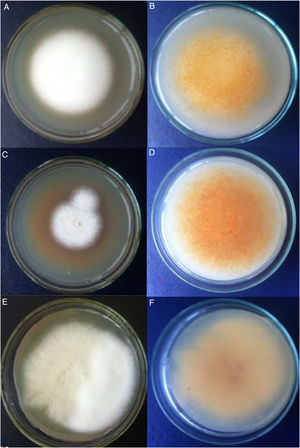
ResultadosAnálisis morfofisiológicoTodas las colonias mostraron heterogeneidad, muchas de ellas con formas algodonosas a granulares o de aterciopeladas a algodonosas (fig. 1). En general, e independientemente de la morfología de la colonia, la producción de conidios fue siempre abundante, estos presentaron formas ovoides a esféricas. La mayor variación se pudo apreciar en la cantidad y la forma de los macroconidios, más bien escasos en general, con forma de cigarro y con bordes rectos, aunque otros fueron de bordes redondeados. Hubo presencia de hifas en espiral (laxas o compactas) en el 60% del total de muestras (fig. 2). Solo en 2muestras la prueba de ureasa fue negativa. Con estos datos y teniendo además las cepas de referencia analizadas bajo las mismas condiciones, se estableció que 13 cepas correspondían a T. interdigitale, 2 a T. mentagrophytes y 2 a T. erinacei (tabla 1).
Registro de las características morfofisiológicas e identificación molecular
| Morfología colonias | Ureasa | Identificación morfofisiológica | Identificación molecular | |||
|---|---|---|---|---|---|---|
| Cepa | Fuente | Tipo de crecimiento | Reverso | ± | ITS/HRMA | |
| TM1 | Uña pie | Algodonosa/granular | Parduzco | – | T. erinacei | T. interdigitale |
| TM2 | Uña pie | Aterciopelada/granular | Café anaranjado | + | T. interdigitale | T. interdigitale |
| TM3 | Uña pie | Granular | Café anaranjado | + | T. interdigitale | T. interdigitale |
| TM4 | Uña pie | Algodonosa | Amarillento | + | T. interdigitale | T. interdigitale |
| TM5 | Plantar | Aterciopelada/algodonosa | Blanquecino | + | T. interdigitale | T. interdigitale |
| TM6 | Uña pie | Aterciopelada | Parduzco | + | T. interdigitale | T. interdigitale |
| TM7 | Uña pie | Algodonosa | Amarillento | + | T. interdigitale | T. interdigitale |
| TM8 | Plantar | Pulverulenta/aterciopelada | Blanquecino | + | T. mentagrophytes | T. interdigitale |
| TM9 | Facial | Algodonosa | Parduzco | + | T. interdigitale | T. interdigitale |
| TM10 | Interdigital | Pulverulenta/algodonosa | Amarillento | + | T. interdigitale | T. interdigitale |
| TM11 | Plantar | Pulverulenta/aterciopelada | Blanquecino | – | T. erinacei | T. interdigitale |
| TM12 | Uña pie | Aterciopelada/granular | Blanquecino | + | T. mentagrophytes | T. interdigitale |
| TM13 | Uña pie | Aterciopelada/algodonosa | Parduzco | + | T. interdigitale | T. interdigitale |
| TM14 | Uña pie | Algodonosa | Amarillento | + | T. interdigitale | T. interdigitale |
| TM15 | Uña pie | Pulverulenta | Café anaranjado | + | T. interdigitale | T. interdigitale |
| TM16 | Uña pie | Algodonosa/granular | Parduzco/rojizo | + | T. interdigitale | T. interdigitale |
| TM17 | Uña pie | Aterciopelada | Parduzco | + | T. interdigitale | T. interdigitale |
| Cepas control | Morfología colonia | Ureasa | |||
| N.° CBS | Especie | Fuente | Tipo de crecimiento | Reverso | ± |
| 318.56 | T. mentagrophytes | Humano | Algodonosa | Parduzco | – |
| 428.63 | T. interdigitale | Humano | Algodonosa/granular | Parduzco | + |
| 511.73 | T. erinacei | Erizo de tierra | Algodonosa | Amarillenta | + |
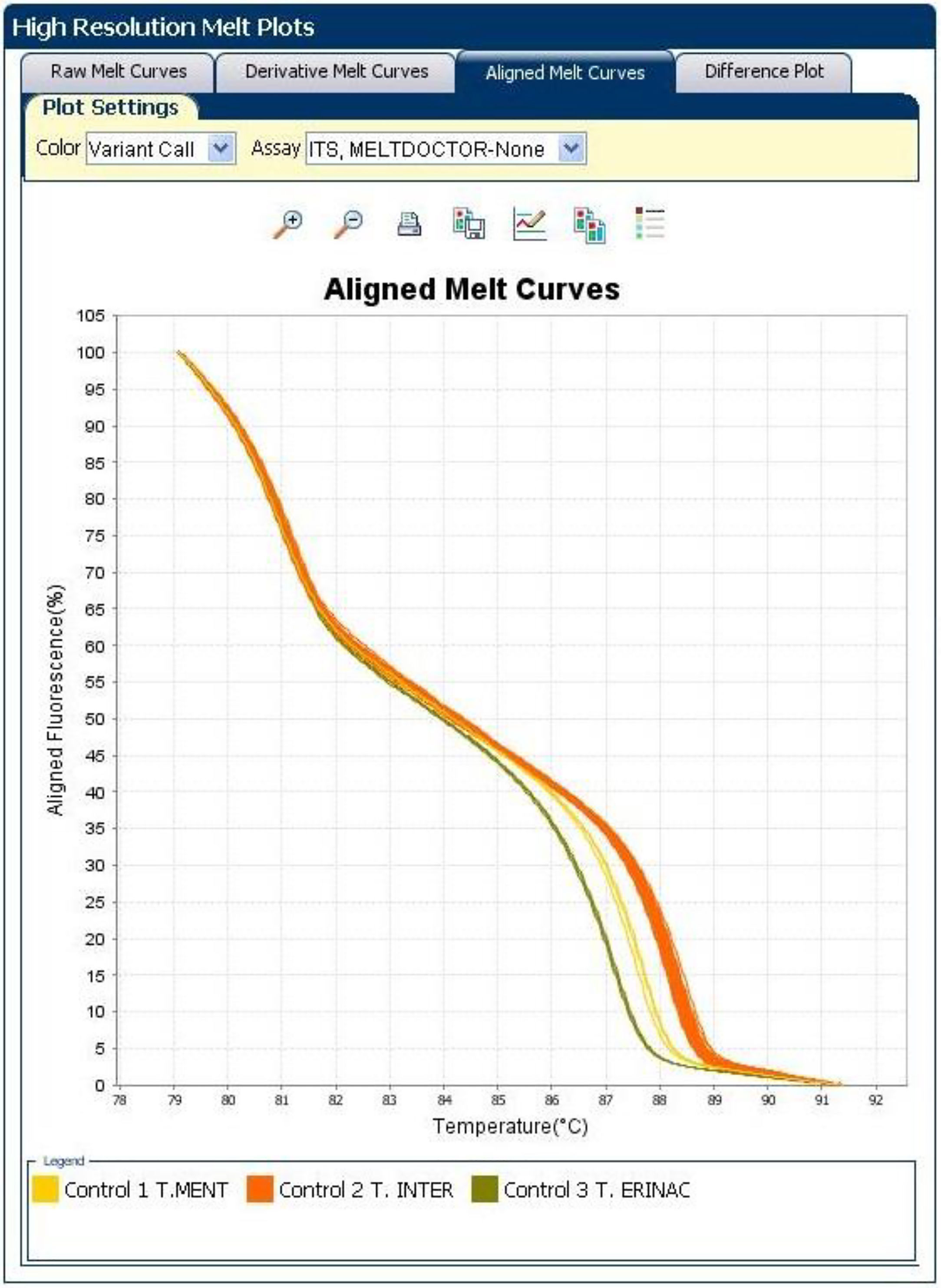
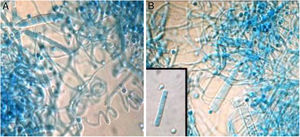
El análisis de HRMA clasificó las 20 muestras (cepas clínicas y cepas de referencia) en 3variantes, con Tm de 87,6°C en el caso de T. mentagrophytes, de 88,2°C en el de T. interdigitale y de 87,1°C en el de T. erinacei. Se distinguieron claramente 3curvas, con altos silhouette scores, entre el 91,2 y el 99,5%, lo que sugiere que las cepas estudiadas corresponden al grupo al que fueron asignadas. Las variantes fueron nombradas de este modo: control 1 TM (T. mentagrophytes); control 2 TI (T. interdigitale), y control 3 TE (T. erinacei) (fig. 3). Todas las cepas clínicas se agruparon en la variante TI.
El alineamiento de las secuencias obtenidas por HRMA e ITS demostró escasa variabilidad nucleotídica entre las muestras. Asimismo, mostró coincidencia con la cepa de referencia de T. interdigitale, exceptuando la muestra TM14, que presentó una transición en la posición 114 del gen 5.8S de una citosina por una timina.
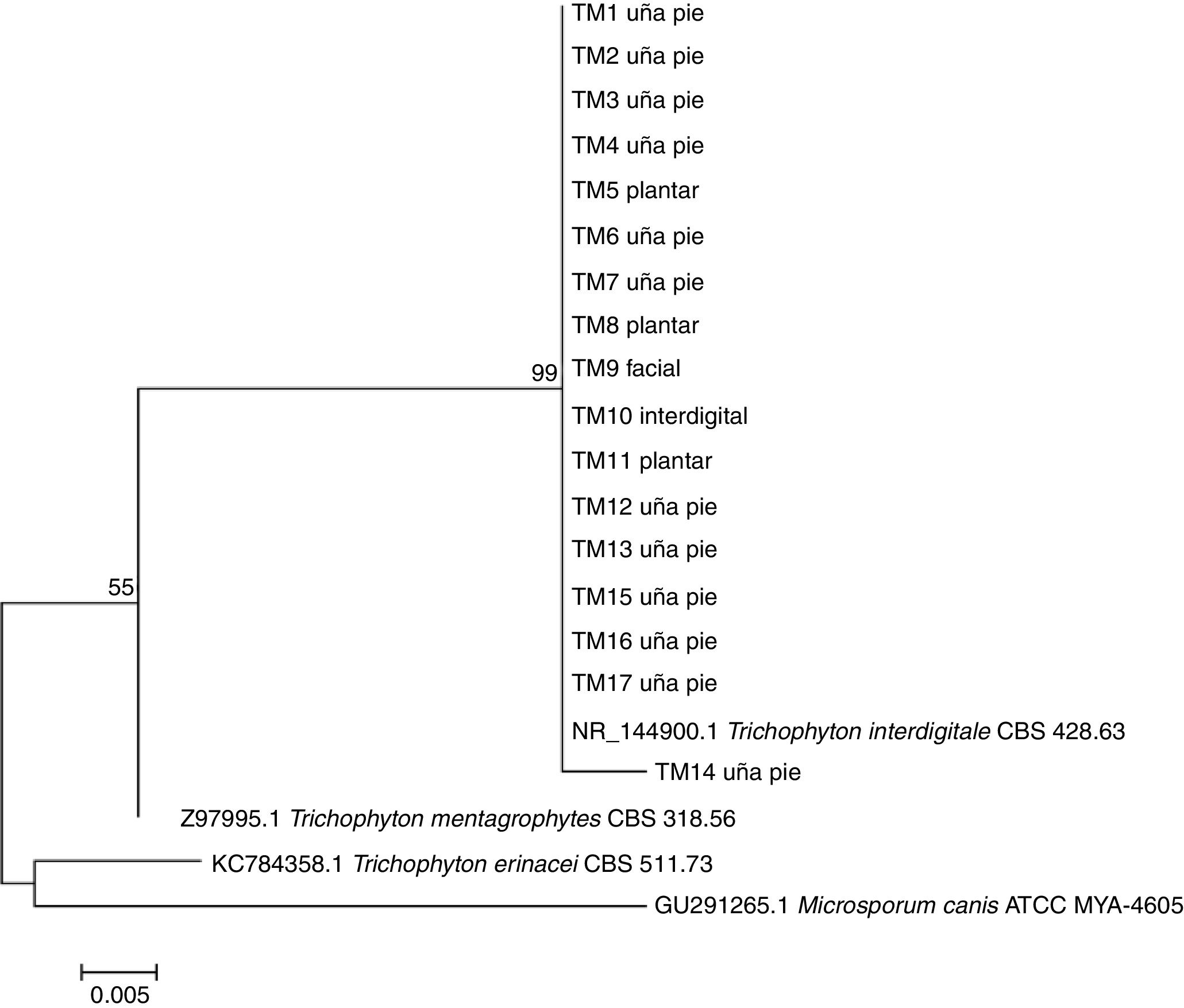
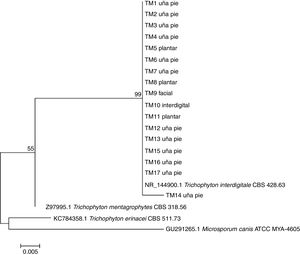
Todas las secuencias fueron analizadas en BLAST y mostraron valores de cobertura entre el 98 y el 100%, E valores cercanos a 0 y entre un 99 y un 100% de identidad con T. interdigitale. Por el método de máxima verosimilitud, T. erinacei fue el organismo más distante de las cepas clínicas, tanto para las secuencias obtenidas por HRMA (fig. 4), como para las obtenidas por ITS (fig. 5).
La identificación morfológica de las especies del complejo T. mentagrophytes no es una tarea fácil debido al pleomorfismo de algunas cepas, lo que complica su diferenciación8. En nuestro estudio, por morfofisiología se detectó principalmente T. interdigitale, seguido por T.mentagrophytes y T. erinacei. La macroscopia reveló una variada morfología de colonias (algodonosas, pulverulentas, aterciopeladas, granulares o mezclas de ellas). Microscópicamente, la cantidad y la forma de los microconidios fueron estables.
A nivel nacional, no hay trabajos que clasifiquen los miembros de este complejo, y en varios de los estudios de alcance internacional la clasificación morfológica es «T. mentagrophytes complex»6,7,8. Sin embargo, existen trabajos como el de Heidemann et al.8, quienes clasificaron inicialmente por morfología 130 especies como T. mentagrophytes para posteriormente reclasificarlas por RFLP, confirmando 102 de ellas como pertenecientes al complejo. Las cepas restantes se identificaron como Fusarium spp.11, Auxarthron spp.2, Penicillium chrysogenum1 y Debaryomyces spp. Asimismo, Drira et al.6 trabajaron la clasificación molecular con 60 cepas inicialmente identificadas por morfofisiología como pertenecientes al complejo, de las cuales 56 fueron confirmadas por el mismo método molecular descripto por Heidemann et al.8, las otras 4 fueron clasificadas como T. rubrum.
Para las especies identificadas como T. interdigitale, se puede encontrar en la literatura subclasificaciones morfológicas que distinguen entre cepas zoofílicas y antropofílicas, con las primeras asociadas a las formas pulverulentas y las segundas a las algodonosas. Sin embargo, como también se ha descrito y se demostró en este trabajo, existen ciertas colonias que muestran dualidad de formas5, por lo que resultan imposibles de clasificar sobre esa base. Dado que la cepa de referencia correspondiente a la especie T. interdigitale también mostró la doble característica en su colonia, se reafirma lo descrito por Nenoff et al.11, que la diferenciación por esta vía es muchas veces imposible. Además, en el momento de comprobar la identidad de la especie por los estudios moleculares, todas mostraron una identidad entre ellas del 99 y el 100%, lo cual demuestra que ese patrón morfológico no es de utilidad. Las 2cepas clasificadas por morfología como T. mentagrophytes se reconocieron dentro de este grupo únicamente dadas sus semejanzas morfofisiológicas con la cepa de referencia. De igual modo, las 2cepas clasificadas como T. erinacei mostraron la prueba de ureasa negativa, dato que, sumado a las características morfológicas, las clasificó como tales.
Existía la posibilidad de encontrar T. erinacei dentro de las cepas clínicas, ya que dicho microorganismo estaría colonizando del 40 al 80% de los erizos de tierra africanos o de vientre blanco (Atelerix albiventris)4, animales que desde hace algún tiempo han pasado a ser domesticados y a convivir con los humanos15.
El alineamiento de secuencias mostró para la cepa TM14 una transición en una base, C por T, la que debería haber sido detectada por HRMA dada la alta sensibilidad descrita para esta técnica. A pesar de esto, el análisis por HRMA logró diferenciar claramente las 3muestras de referencia y clasificar todas las muestras clínicas como T. interdigitale.
La secuenciación es el gold standard para la correcta clasificación de los dermatofitos. Dicha técnica fue la base de múltiples estudios citados en la revisión de Cafarchia et al.2, dentro de los que destacan los publicados por Graser et al.7, Makimura et al.9 y de Hoog et al.4, quienes los agruparon y reclasificaron por secuenciación de regiones ITS. Sin embargo, algunos dermatofitos todavía son difíciles de identificar con precisión a nivel de especie, incluso por la comparación de secuencias ITS. Esto se debe a la existencia de taxones hermanos estrechamente relacionados, a la variación significativa de las secuencias entre cepas dentro de una especie o a la tipificación imprecisa de especies13.
Para este estudio, ambas secuenciaciones corroboraron la clasificación, respaldando y validando la agrupación de las curvas obtenidas por HRMA. Estos resultados se fundamentan en el hecho de que el taxón T. interdigitale posee reservorio antropofílico y en que se analizaron únicamente muestras provenientes de dermatomicosis humanas, contrariamente a la mayoría de los estudios revisados, que incluyeron cultivos provenientes de animales. Sin embargo, como existen reportes de infección en humanos relacionados con la presencia de especies exóticas como mascotas, es probable que las otras especies comiencen a reconocerse con mayor frecuencia como causales de dermatomicosis3. Este primer estudio y sus resultados invitan a ampliar la investigación incluyendo otras especies del mismo complejo u otros dermatofitos.
Tal como se demuestra en este trabajo, es probable que las especies históricamente identificadas como T. mentagrophytes hasta el día de hoy no sean T. mentagrophytes, sino T. interdigitale, situación que debería comenzar a corregirse de aquí en adelante, informando correctamente a nivel de complejo (T. mentagrophytes complex) o, en caso de contar con métodos polifásicos de identificación, informando a nivel de especie.
La taxonomía fúngica ha avanzado considerablemente en los últimos años a partir de los análisis moleculares, pero es necesario desarrollar técnicas complementarias que permitan optimizar la identificación. La falta de estabilidad de los patrones morfológicos entre especies del complejo T. mentagrophytes demuestra la importancia de contar con herramientas moleculares que puedan discriminar sus diferencias, separándolas por genotipo.
FinanciaciónEste estudio fue financiado con fondos propios del laboratorio de micología de la Universidad de Valparaíso.
Conflicto de interesesLos autores declaran no tener conflictos de interés.
A todo el equipo de la unidad de Virología de la Universidad de Valparaíso por permitir el uso de su equipamiento e instalaciones para el análisis molecular.