Se estudiaron 64 aislamientos de K. pneumoniae resistentes a colistina recuperados de materiales clínicos de 57 pacientes atendidos entre los años 2010 y 2012 en el Hospital de Clínicas José de San Martín, Universidad de Buenos Aires, con el objetivo de describir las características microbiológicas y epidemiológicas y los factores relacionados con la emergencia de estos aislamientos. Se incluyeron en el estudio 54 aislamientos de K. pneumoniae sensibles a colistina contemporáneos a los aislamientos resistentes. La relación de similitud genética entre los aislamientos se investigó mediante la técnica de PCR. El 50 % de los aislamientos resistentes presentaron KPC-2, el 45,3 % BLEE y el 4,7 % restante solo presentó resistencia a las aminopenicilinas. Todos los aislamientos portadores de KPC (resistentes o sensibles a colistina), a excepción de uno, fueron indistinguibles genéticamente, mientras que los portadores de BLEE se agruparon en 7 clones distintos, y se distinguieron de los clones recuperados en los aislamientos sensibles a colistina. El uso previo de colistina se asoció como el principal factor vinculado con la adquisición de esta resistencia y con la internación en UCI en los aislamientos sin KPC. A partir del año 2010 la resistencia a colistina comenzó a emerger, alcanzó el 3 % en los aislados nosocomiales y se mantuvo estable en los años subsiguientes, debido a la selección de las subpoblaciones resistentes en el clon epidémico en los aislamientos productores de KPC, y en los no productores de KPC por dispersión de clones resistentes a colistina.
Sixty-four colistin-resistant Klebsiella pneumoniae isolates recovered from clinical specimens from 57 patients admitted to Hospital de Clinicas Jose de San Martin during the period 2010–2012 were studied to describe the microbiological and epidemiological characteristics and factors associated with the emergence of colistin-resistance. Fiftyfour colistin-susceptible K. pneumoniae isolates from the same period were also included in the study. The genetic relatedness among the isolates was studied by a PCR assay. Fifty percent of the resistant isolates were KPC-2 producers, 45.3% were ESBL producers and 4.7% only showed resistance to aminopenicilins. All KPC-producers (resistant and susceptible to colistin) were genotipically indistinguishable except for one, whereas the presence of 7 clonal types, which were different from the ones identified in the colistinsusceptible isolates, were detected among ESBL producers. The previous use of colistin was the main factor associated with the acquisition of resistance, and in the case of non-KPC producers the stay in ICU was another significant factor observed. Colistin resistance emerged in our hospital in the year 2010, reaching 3% in nosocomial isolates and maintaining this rate in successive years, due to the selection of resistant subpopulations in the epidemic clonal type in KPC-producers and due to the dispersion of colistinresistant clonal types in non-KPC producing-isolates.
La emergencia a nivel mundial de bacilos gram negativos multirresistentes ha renovado el interés por los antibióticos polipeptídicos, debido a que frecuentemente son los únicos que mantienen actividad frente a estos aislamientos19. Se han documentado tres tipos de resistencia adquirida a colistina (COL): a) la COL-dependencia, b) la heterorresistencia (HR-COL) y c) la resistencia propiamente dicha, con un valor de CIM > 2μg/ml (COL-R). La COL-dependencia está limitada a pocos aislamientos clínicos de Acinetobacter baumannii9, mientras que la HR-COL, definida por la presencia de subpoblaciones resistentes, ha sido descrita en A. baumannii y Klebsiella pneumoniae12,23. Sin embargo, su impacto clínico todavía no ha sido totalmente establecido21.
La resistencia a COL ha sido comunicada en diferentes regiones del mundo, en A. baumannii, K. pneumoniae y Pseudomonas aeruginosa1,7,11,17. Sin embargo, según nuestro conocimiento no hay al respecto estudios publicados en Argentina. En nuestro hospital, los primeros aislamientos de K. pneumoniae COL-R emergieron en 2010; hacia fines de 2011 se alcanzó el 3 % de resistencia. Este porcentaje se mantuvo estable durante 2012, en un valor de 3,6%. Los objetivos del presente estudio fueron describir las principales características microbiológicas de los aislamientos de K. pneumoniae COL-R y los factores relacionados con su emergencia y diseminación en un hospital universitario de la Ciudad de Buenos Aires.
Materiales y métodosSe estudiaron 64 aislamientos consecutivos de K. pneumoniae COL-R recuperados de materiales clínicos pertenecientes a 57 pacientes atendidos en el Hospital de Clínicas José de San Martín, Universidad de Buenos Aires, entre los años 2010 y 2012. Los aislamientos fueron identificados mediante pruebas bioquímicas; la relación de similitud genética entre ellos se investigó con una técnica de PCR que utiliza primers de oligonucleótidos degenerados (OD-PCR)13.
A la totalidad de los aislamientos se les realizó la prueba de sensibilidad a los antimicrobianos por el método semicuantitativo de dos concentraciones útiles de Marcenac et al.15; se evaluaron ampicilina, piperacilina, piperacilina-tazobactama, cefalotina, cefoxitina, ceftriaxona, ceftacidima, cefepima, amoxicilina-clavulánico, imipenem, meropenem, amicacina, gentamicina, ciprofloxacina y trimetoprima-sulfametoxazol. La concentración inhibitoria mínima (CIM) de COL (colistin sulfate salt, Sigma-Aldrich, EE.UU.) se determinó mediante la técnica de dilución seriada en agar, de acuerdo a las normas establecidas por el Clinical and Laboratory Standards Institute (CLSI) 20116. Para su interpretación se utilizaron los puntos de corte del European Committee on Antimicrobial Susceptibility Testing (EUCAST) 2011 (S≤2μg/ml, R>2μg/ml)8.
La detección de carbapenemasa de tipo KPC se realizó con la sinergia meropenem (10μg)-ácido 3-aminofenilborónico (300μg) (Laboratorio Britania), y se confirmó por PCR con primers específicos20. Debido a la actual importancia epidemiológica y clínica de las carbapenemasas de tipo KPC en nuestro país, para el análisis de los resultados se dividió a los pacientes en dos grupos, según sus aislamientos presentaran o no esta carbapenemasa (grupo KPC+ y KPC−, respectivamente). Se incluyeron 54 aislamientos de K. pneumoniae COL-S (24 KPC(+) y 30 KPC(−)) contemporáneos a los aislamientos resistentes, para evaluar los factores relacionados con la emergencia y diseminación de la resistencia a COL. Para cada paciente se elaboró una ficha clínica en donde se incluyeron los siguientes datos: sexo, edad, espécimen clínico, tratamiento previo con COL, fecha y servicio de internación. En los pacientes del grupo KPC+ se incluyó, además, la enfermedad de base y/o diagnóstico. Para el análisis estadístico de los resultados se utilizó el software GraphPad Prism (versión 6.01 GraphPad Software Inc.). Se realizó el test de Fisher y los resultados se presentaron mediante la odds ratio (OR) y su intervalo de confianza (IC) del 95 %. Se consideró un grado de significación estadística de 5 % (p<0,05).
ResultadosLos aislamientos resistentes a COL presentaron valores de CIM entre 4 y > 64μg/ml. El 81,2 % de los aislamientos presentaron alto nivel de resistencia a COL (CIM≥16μg/ml), mientras que en el 18,2 % restante los valores de CIM se encontraron en el rango de 4 a 8μg/ml.
El 50 % de los aislamientos presentaron KPC-2, el 45,3 % β-lactamasa de espectro extendido (BLEE), y el 4,7 % restante solo presentó resistencia a las aminopenicilinas. Todos los aislamientos portadores de KPC (COL-R o COL-S), a excepción de uno, fueron indistinguibles genotípicamente por OD-PCR.
Los aislamientos COL-R portadores de BLEE se agruparon en 7 clones distintos y los 3 aislamientos con resistencia natural en otros 2 clones distintos; estos 9 clones fueron distintos a los 4 clones recuperados en los 24 aislamientos KPC(−) COL-S (18 portadores de BLEE, 3 portadores de β-lactamasa de amplio espectro [BLEA] y 3 que solo presentaron resistencia a aminopenicilinas).
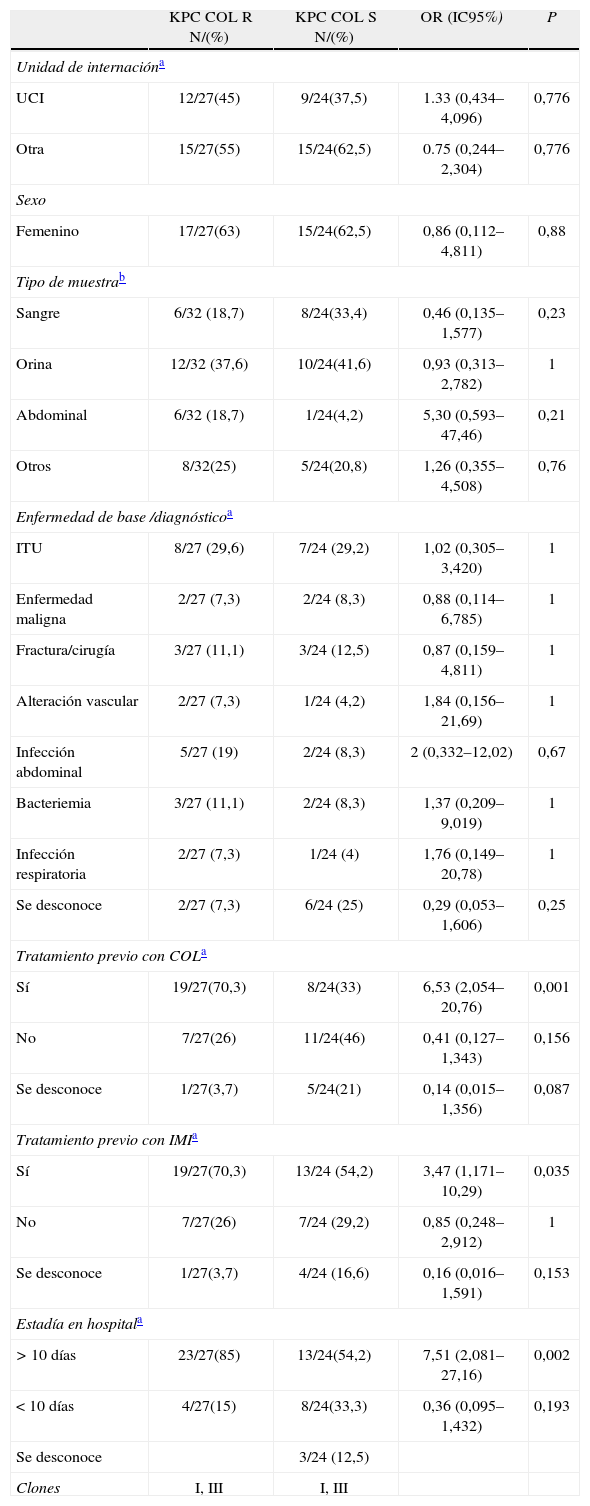
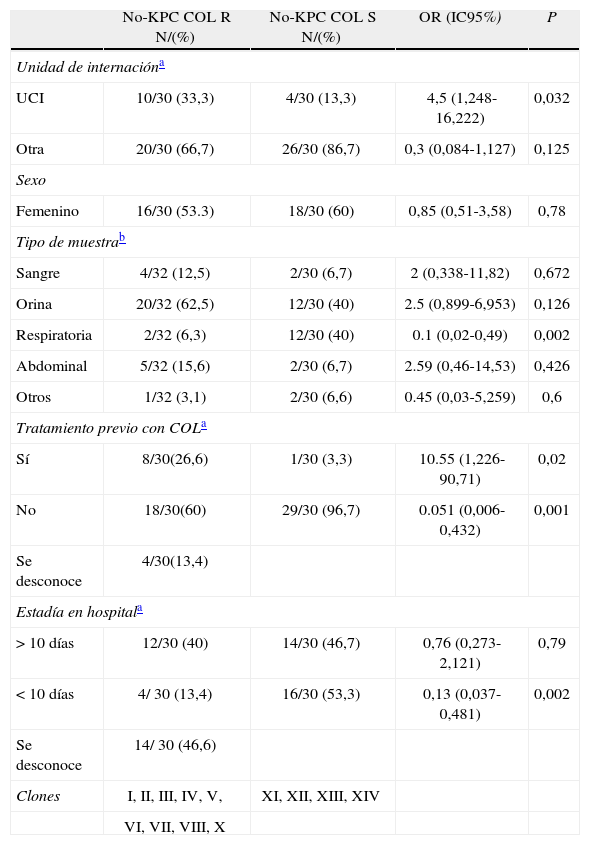
Los factores epidemiológicos asociados a la resistencia a colistina y las características de los pacientes con aislamientos de K. pneumoniae COL-R se detallan en las tablas 1 y 2. En 27/57 pacientes infectados por K. pneumoniae COLR se recuperaron aislamientos KPC(+), mientras que en 30/57 pacientes se recuperaron aislamientos KPC(−). El promedio de edad de los 57 pacientes fue de 54 años (rango: 16-83 años), en su mayoría de sexo femenino. Los aislamientos KPC(+) estuvieron ampliamente distribuidos entre los diferentes servicios del hospital, y se dividieron casi en mitades los recuperados en la Unidad de Cuidados Intensivos (UCI) y los recuperados en otras salas. La presencia en UCI de aislamientos KPC(−) COL R fue estadísticamente significativa respecto de la población COL-S, si bien los aislamientos KPC(−) estuvieron distribuidos de manera similar en los diferentes servicios. Con respecto al tipo de muestra, se destaca una mayor presencia relativa en especímenes clínicos del tracto urinario (62,5 % en los aislamientos KPC(–) y 37,6 % en los KPC(+)). La mayoría de los pacientes de los que se recuperaron aislamientos de K. pneumoniae KPC(–) no habían recibido previamente tratamiento con COL; mientras que ocurrió lo contrario en los pacientes de los cuales se recuperaron aislamientos KPC(+), el 70,3 % había recibido tratamiento previo. La COL-R fue detectada con períodos de internación superiores a los 10 días en la mayoría de los pacientes del grupo KPC+.
Factores epidemiológicos asociados a la resistencia a colistina de pacientes con aislamientos de K. pneumoniae KPC positivos
| KPC COL R N/(%) | KPC COL S N/(%) | OR (IC95%) | P | |
| Unidad de internacióna | ||||
| UCI | 12/27(45) | 9/24(37,5) | 1.33 (0,434–4,096) | 0,776 |
| Otra | 15/27(55) | 15/24(62,5) | 0.75 (0,244–2,304) | 0,776 |
| Sexo | ||||
| Femenino | 17/27(63) | 15/24(62,5) | 0,86 (0,112–4,811) | 0,88 |
| Tipo de muestrab | ||||
| Sangre | 6/32 (18,7) | 8/24(33,4) | 0,46 (0,135–1,577) | 0,23 |
| Orina | 12/32 (37,6) | 10/24(41,6) | 0,93 (0,313–2,782) | 1 |
| Abdominal | 6/32 (18,7) | 1/24(4,2) | 5,30 (0,593–47,46) | 0,21 |
| Otros | 8/32(25) | 5/24(20,8) | 1,26 (0,355–4,508) | 0,76 |
| Enfermedad de base /diagnósticoa | ||||
| ITU | 8/27 (29,6) | 7/24 (29,2) | 1,02 (0,305–3,420) | 1 |
| Enfermedad maligna | 2/27 (7,3) | 2/24 (8,3) | 0,88 (0,114–6,785) | 1 |
| Fractura/cirugía | 3/27 (11,1) | 3/24 (12,5) | 0,87 (0,159–4,811) | 1 |
| Alteración vascular | 2/27 (7,3) | 1/24 (4,2) | 1,84 (0,156–21,69) | 1 |
| Infección abdominal | 5/27 (19) | 2/24 (8,3) | 2 (0,332–12,02) | 0,67 |
| Bacteriemia | 3/27 (11,1) | 2/24 (8,3) | 1,37 (0,209–9,019) | 1 |
| Infección respiratoria | 2/27 (7,3) | 1/24 (4) | 1,76 (0,149–20,78) | 1 |
| Se desconoce | 2/27 (7,3) | 6/24 (25) | 0,29 (0,053–1,606) | 0,25 |
| Tratamiento previo con COLa | ||||
| Sí | 19/27(70,3) | 8/24(33) | 6,53 (2,054–20,76) | 0,001 |
| No | 7/27(26) | 11/24(46) | 0,41 (0,127–1,343) | 0,156 |
| Se desconoce | 1/27(3,7) | 5/24(21) | 0,14 (0,015–1,356) | 0,087 |
| Tratamiento previo con IMIa | ||||
| Sí | 19/27(70,3) | 13/24 (54,2) | 3,47 (1,171–10,29) | 0,035 |
| No | 7/27(26) | 7/24 (29,2) | 0,85 (0,248–2,912) | 1 |
| Se desconoce | 1/27(3,7) | 4/24 (16,6) | 0,16 (0,016–1,591) | 0,153 |
| Estadía en hospitala | ||||
| > 10 días | 23/27(85) | 13/24(54,2) | 7,51 (2,081–27,16) | 0,002 |
| < 10 días | 4/27(15) | 8/24(33,3) | 0,36 (0,095–1,432) | 0,193 |
| Se desconoce | 3/24 (12,5) | |||
| Clones | I, III | I, III | ||
COL: colistina; IC95%: intervalo de confianza del 95%; IMI: imipenem; ITU: infección del tracto urinario; OR: odds ratio; UCI: unidad de cuidados intensivos.
Factores epidemiológicos asociados a la resistencia a colistina de pacientes con aislamientos de K. pneumoniae KPC negativos.
| No-KPC COL R N/(%) | No-KPC COL S N/(%) | OR (IC95%) | P | |
| Unidad de internacióna | ||||
| UCI | 10/30 (33,3) | 4/30 (13,3) | 4,5 (1,248-16,222) | 0,032 |
| Otra | 20/30 (66,7) | 26/30 (86,7) | 0,3 (0,084-1,127) | 0,125 |
| Sexo | ||||
| Femenino | 16/30 (53.3) | 18/30 (60) | 0,85 (0,51-3,58) | 0,78 |
| Tipo de muestrab | ||||
| Sangre | 4/32 (12,5) | 2/30 (6,7) | 2 (0,338-11,82) | 0,672 |
| Orina | 20/32 (62,5) | 12/30 (40) | 2.5 (0,899-6,953) | 0,126 |
| Respiratoria | 2/32 (6,3) | 12/30 (40) | 0.1 (0,02-0,49) | 0,002 |
| Abdominal | 5/32 (15,6) | 2/30 (6,7) | 2.59 (0,46-14,53) | 0,426 |
| Otros | 1/32 (3,1) | 2/30 (6,6) | 0.45 (0,03-5,259) | 0,6 |
| Tratamiento previo con COLa | ||||
| Sí | 8/30(26,6) | 1/30 (3,3) | 10.55 (1,226-90,71) | 0,02 |
| No | 18/30(60) | 29/30 (96,7) | 0.051 (0,006-0,432) | 0,001 |
| Se desconoce | 4/30(13,4) | |||
| Estadía en hospitala | ||||
| > 10 días | 12/30 (40) | 14/30 (46,7) | 0,76 (0,273-2,121) | 0,79 |
| < 10 días | 4/ 30 (13,4) | 16/30 (53,3) | 0,13 (0,037-0,481) | 0,002 |
| Se desconoce | 14/ 30 (46,6) | |||
| Clones | I, II, III, IV, V, | XI, XII, XIII, XIV | ||
| VI, VII, VIII, X | ||||
COL: colistina; IC95%: intervalo de confianza del 95%; UCI: unidad de cuidados intensivos.
Aunque la resistencia adquirida a COL en enterobacterias fue descrita hace varios años17,19, los aislamientos de K. pneumoniae COL-R han sido comunicados con mayor frecuencia en el último lustro, asociados al mayor consumo de polimixinas2,10. En el Hospital de Clínicas José de San Martín, en los últimos años se registra un aumento en el consumo de COL sin que se verifique un incremento en el número de pacientes atendidos (dato no mostrado). Una de las consecuencias de ese fenómeno fue el aumento de la incidencia de infecciones intrahospitalarias por enterobacterias con resistencia intrínseca a polimixinas y la aparición en 2010 de aislamientos de K. pneumoniae COL-R, de modo que en 2011 se alcanzó el 3 % de resistencia en los aislados nosocomiales de esta enterobacteria22.
En nuestro estudio, la mayoría de los aislamientos presentaron elevadas CIM de COL, y resultaron fácilmente detectables por los diferentes métodos de sensibilidad cuantitativa. Sin embargo, el 75 % de los aislamientos con valores cercanos al punto de corte pertenecieron a pacientes que no recibieron COL. Por lo tanto, de manera coincidente con lo expresado por otros autores, se corrobora que la exposición previa a COL tuvo influencia en el grado de expresión de la resistencia10,16.
Tal como ha sido documentado, se observa que el uso previo de COL es uno de los principales factores relacionados con la adquisición de esta resistencia en ambos grupos, y que la internación en UCI lo fue en los pacientes donde se recuperaron los aislamientos KPC−14,25. En este hospital, los primeros aislamientos de K. pneumoniae KPC(+) fueron descritos en enero de 2010, con una posterior y rápida diseminación en los diferentes servicios. La resistencia a COL emergió 4 meses después. Esta evolución de la resistencia fue descrita en otros países y está frecuentemente asociada al secuenciotipo ST2583,14,24. Los primeros aislamientos de K. pneumoniae KPC(+) COL-S estudiados en nuestra institución pertenecieron a este tipo clonal, y resultaron indistinguibles mediante OD-PCR de los aislamientos COL-R, por lo tanto, podríamos afirmar su pertenencia a este tipo clonal de diseminación mundial4. El proceso de selección intratratamiento se vería facilitado por la presencia de subpoblaciones resistentes a COL, las cuales fueron detectadas por nosotros en estudios previos en el 95 % de los aislamientos KPC(+)18. En un 27 % de los pacientes pudo documentarse la presencia previa de aislamientos COL-S, los cuales luego de que los enfermos recibieran el tratamiento con polimixinas se transformaron en COL-R, y ambos pertenecían al mismo clon. Estos resultados explicarían el motivo por el cual la COL-R se detectaría en periodos de internación superiores a 10 días, y también el motivo por el cual todavía se utiliza a la colistina como una de las últimas herramientas en la terapéutica antimicrobiana.
En coincidencia con otros autores, nosotros observamos una mayor dispersión clonal en los aislamientos de K. pneumoniae KPC(−) COL-R5,14. La presencia de diferentes clones entre las poblaciones COL-S y COL-R en los aislamientos KPC(−) descarta el proceso de selección intratratamiento, sino que en este caso el aumento del uso de COL favorecería la aparición de clones previamente menos recuperados en clínica.
En conclusión, la COL-R en K. pneumoniae aparece en los últimos 3 años en nuestro hospital, y el uso previo de COL es el principal factor relacionado con su adquisición. Dicha resistencia emerge mediante dos caminos diferentes: si los aislamientos son KPC(+), debido a la selección de las subpoblaciones resistentes en el clon epidémico, y si los aislamientos son KPC(−), por dispersión de clones COL-R, los cuales son diferentes a los encontrados en la población sensible.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
AgradecimientosEste trabajo fue subvencionado por la Secretaría de Ciencia y Técnica de la Universidad de Buenos Aires (UBACyT B108) a Angela Famiglietti.