En el contexto de la medicina personalizada, la epigenética cobra importancia como base de prevención, diagnóstico, prognosis y tratamiento de enfermedades como el cáncer. De los distintos procesos de control epigenético, el silenciamiento génico por metilación del ADN es el más frecuente en esta patología y aporta aplicaciones clínicas muy variadas como predicción de respuesta terapéutica, prognosis asociada a las características moleculares del tumor y el seguimiento de pacientes tras la intervención quirúrgica o el tratamiento con quimioterapia. Una de las principales ventajas clínicas de estas alteraciones epigenéticas es que pueden revertirse mediante tratamientos farmacológicos, aunque se asocien con múltiples efectos secundarios. Es por ello de gran importancia continuar con el estudio de la regulación epigenética en cáncer, complementado con la biología de sistemas, lo que aportaría conocimiento sobre la implicación biológica real de estos biomarcadores, y la identificación de fármacos específicos que disminuyan estos efectos adversos y nos acerquen a la realidad de una medicina personalizada.
In the context of personalised medicine, epigenetics has an important role as regards the prevention, diagnosis, prognosis, and treatment of diseases such as cancer. Of the different epigenetic control processes, gene silencing by DNA methylation is most frequent in this disease, and contributes to a wide variety of clinical applications such as, prediction of the therapeutic response, the prognosis associated with the molecular characteristics of the tumour, and the follow-up pf patients after treatment by surgery or chemotherapy. One of the main advantages of these epigenetic alterations is that they can be reversed with pharmacological treatments, although they are associated with multiple side effects. It for this reason, it is of great importance to continue studying epigenetic regulation in cancer, complementing with biological systems, as well as the identification of specific drugs that may decrease these adverse effects, and which should help to determine the real biological implications of these biomarkers and may lead to achieving personalised medicine.
El Instituto Nacional de Cáncer en Estados Unidos define la medicina personalizada como una forma de medicina que utiliza la información acerca de los genes y proteínas de un individuo y el ambiente que la rodea para prevenir, diagnosticar y tratar una enfermedad. La medicina personalizada utiliza conceptos tradicionales y conceptos emergentes sobre las bases genéticas de una enfermedad para individualizar la prevención, diagnóstico y tratamiento. Los resultados de multitud de proyectos de investigación sobre la relación entre los cambios genéticos y epigenéticos, así como en las rutas de señalización inducidas por los fármacos y aquellos presentes en tumores de los pacientes, han sido esenciales en la determinación de algunos marcadores de interés que han permitido administrar el mejor tratamiento oncológico para cada paciente.
Las aplicaciones de la genética y la epigenética en la medicina personalizada incluyen áreas básicas de la medicina como son la prevención, diagnóstico, prognosis y tratamiento (tabla 1). El trayecto que va desde un concepto hasta la aplicación clínica en cada una de estas áreas involucra investigación básica, traslacional, clínica y también aspectos regulatorios1. En el estudio de la relevancia de las terapias personalizadas en el tratamiento del cáncer y el impacto de estas nuevas alternativas terapéuticas hay que tener en cuenta, por una parte el valor que puede representar para el paciente en términos de sufrimiento (efectos secundarios, faltas de respuesta), para el sistema nacional de salud en términos de coste farmacéutico (terapias tradicionales vs. personalizadas que incluyen análisis genéticos, etc.) y por último el coste social y personal que implican las horas de trabajo no ejercido, horas de ocio no disfrutado, tiempo y dedicación de los familiares en el cuidado del paciente, etc.
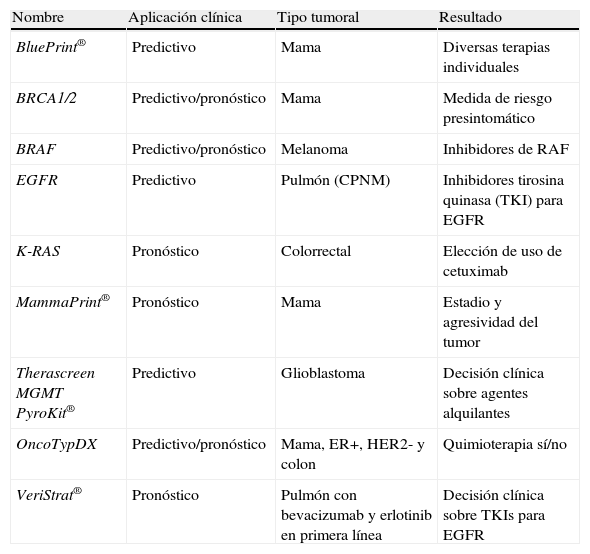
Biomarcadores genéticos/epigenéticos de uso clínico
| Nombre | Aplicación clínica | Tipo tumoral | Resultado |
| BluePrint® | Predictivo | Mama | Diversas terapias individuales |
| BRCA1/2 | Predictivo/pronóstico | Mama | Medida de riesgo presintomático |
| BRAF | Predictivo/pronóstico | Melanoma | Inhibidores de RAF |
| EGFR | Predictivo | Pulmón (CPNM) | Inhibidores tirosina quinasa (TKI) para EGFR |
| K-RAS | Pronóstico | Colorrectal | Elección de uso de cetuximab |
| MammaPrint® | Pronóstico | Mama | Estadio y agresividad del tumor |
| Therascreen MGMT PyroKit® | Predictivo | Glioblastoma | Decisión clínica sobre agentes alquilantes |
| OncoTypDX | Predictivo/pronóstico | Mama, ER+, HER2- y colon | Quimioterapia sí/no |
| VeriStrat® | Pronóstico | Pulmón con bevacizumab y erlotinib en primera línea | Decisión clínica sobre TKIs para EGFR |
Los procesos epigenéticos tienen un papel fundamental en fenómenos fisiológicos como la embriogénesis, la impronta y la inactivación del cromosoma X, pero también en el desarrollo de enfermedades, entre ellas el cáncer. En esta última década, se han establecido los diferentes niveles mecanísticos que participan en la regulación epigenética, entre los que se incluyen la metilación del ADN, la modificación postraduccional de histonas y los micro ARN regulatorios de la expresión génica2. De todos estos mecanismos, el mejor conocido es la metilación del ADN.
Metilación y cáncerLa primera vez que se demostró una relación entre la metilación del ADN y el cáncer fue en 1983, cuando se observó que las células tumorales tenían un grado de metilación menor que las células originales no tumorales. Este fenómeno está relacionado con una pérdida de metilación en las regiones repetitivas del genoma provocando inestabilidad cromosómica en estas células tumorales. Sin embargo el principal evento que está involucrado en el origen de un gran número de cánceres es la hipermetilación de islas CpG en los promotores de genes supresores de tumores (GST)3. Estos genes pueden mediar diferentes funciones celulares implicadas en el desarrollo del cáncer, como son la regulación del ciclo celular, la reparación del ADN, la interacción célula-célula, la apoptosis y la angiogénesis4.
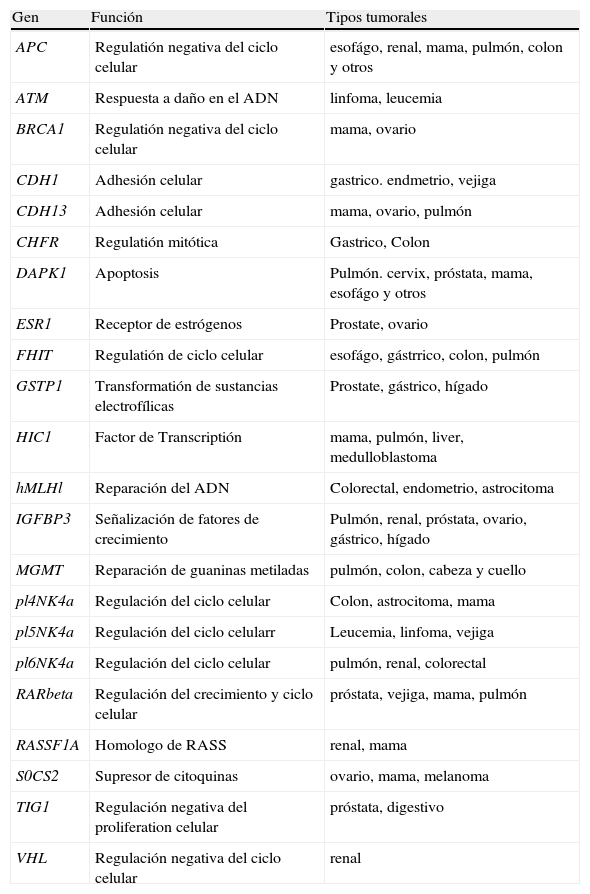
Hoy en día, no hay duda de que alteraciones en la metilación del ADN tienen un papel importante en la tumorogénesis. Una metilación aberrante de las islas CpG de genes específicos, es un mecanismo epigenético tan común como las mutaciones puntuales o pérdidas de heterozigosidad (LOH) que causan el silenciamiento de los GST en cánceres humanos. Entre los diferentes genes alterados por la metilación en diversos tipos tumorales encontramos, RASSF1, BRCA1, GSTP1, CDKN2A, CDH13, MLH1, VHL, APC, MGMT, DAPK, IGFBP-3 o TIMP3 entre otros (tabla 2).
Genes supresores tumorales bajo regulación epigenética en diferentes tipos tumorales
| Gen | Función | Tipos tumorales |
| APC | Regulatión negativa del ciclo celular | esofágo, renal, mama, pulmón, colon y otros |
| ATM | Respuesta a daño en el ADN | linfoma, leucemia |
| BRCA1 | Regulatión negativa del ciclo celular | mama, ovario |
| CDH1 | Adhesión celular | gastrico. endmetrio, vejiga |
| CDH13 | Adhesión celular | mama, ovario, pulmón |
| CHFR | Regulatión mitótica | Gastrico, Colon |
| DAPK1 | Apoptosis | Pulmón. cervix, próstata, mama, esofágo y otros |
| ESR1 | Receptor de estrógenos | Prostate, ovario |
| FHIT | Regulatión de ciclo celular | esofágo, gástrrico, colon, pulmón |
| GSTP1 | Transformatión de sustancias electrofílicas | Prostate, gástrico, hígado |
| HIC1 | Factor de Transcriptión | mama, pulmón, liver, medulloblastoma |
| hMLHl | Reparación del ADN | Colorectal, endometrio, astrocitoma |
| IGFBP3 | Señalización de fatores de crecimiento | Pulmón, renal, próstata, ovario, gástrico, hígado |
| MGMT | Reparación de guaninas metiladas | pulmón, colon, cabeza y cuello |
| pl4NK4a | Regulación del ciclo celular | Colon, astrocitoma, mama |
| pl5NK4a | Regulación del ciclo celularr | Leucemia, linfoma, vejiga |
| pl6NK4a | Regulación del ciclo celular | pulmón, renal, colorectal |
| RARbeta | Regulación del crecimiento y ciclo celular | próstata, vejiga, mama, pulmón |
| RASSF1A | Homologo de RASS | renal, mama |
| S0CS2 | Supresor de citoquinas | ovario, mama, melanoma |
| TIG1 | Regulación negativa del proliferation celular | próstata, digestivo |
| VHL | Regulación negativa del ciclo celular | renal |
La contribución de la epigenética al estudio del cáncer es evidente, ya que la metilación aberrante de determinados genes ocurre muy temprano en la carcinogénesis, incluso cuando las muestras quirúrgicas son histológicamente negativas5. Los marcadores para ADN metilado son por tanto indicadores perfectos para la enfermedad ya establecida.
Además, debido a lo ubicuo que es el ADN hipermetilado y gracias a las nuevas tecnologías, capaces de detectar cambios de metilación en el ADN en diversos fluidos biológicos, hacen que este área de estudio se considere una de las grandes promesas no solo para la detección del cáncer, sino como fuente de identificación de biomarcadores para prognosis e incluso predicción de respuesta a fármacos. La epigenética proporciona gran flexibilidad biológica a la célula tumoral, sin embargo estas características pueden ser modificadas farmacológicamente gracias al uso de agentes desmetilantes e inhibidores de acetilasas de histonas. Esta posibilidad nos permite identificar dianas terapéuticas con uso potencial en clínica, ya que existen en la actualidad cuatro fármacos distintos aprobados con estas características, de los que hablaremos más adelante.
Las características que han de tener las secuencias de ADN metilado para poder ser consideradas biomarcadores clínicos se han definido conjuntamente por el National Cancer Institute's y el Early Detection Research Network6.
La mayoría de los marcadores epigenéticos se han identificado mediante aproximaciones puntuales a genes candidatos; sin embargo en los últimos años, han surgido numerosos estudios combinando arrays de expresión con tratamientos epigenéticos de reactivación de la expresión génica. Estos estudios permiten identificar nuevos genes bajo regulación epigenética con significado en progresión tumoral, que pudieran eventualmente comportarse como biomarcadores en diferentes tipos tumorales como el de vejiga, colorrectal esofágico, renal o pulmón7. Recientemente se han sumado a estas aproximaciones otros estudios apoyados en técnicas de secuenciación masiva del genoma completo, combinada con perfiles de expresión diferencial, que se han enfocado en tumores de origen epitelial, identificando marcadores que pudieran iluminar las rutas moleculares involucradas en tumorogénesis. De los diferentes estudios realizados podemos resaltar los siguientes candidatos: BCN1, MSX1, CCNA1, ALDH1A3, genes cuya expresión está bajo regulación epigenética, y que han sido validados en más de cien muestras quirúrgicas procedentes de diferentes tipos tumorales como mama, colon, próstata y pulmón. Estos resultados indicarían que diversos tipos tumorales de origen epitelial pueden compartir alteraciones en las mismas rutas celulares. Actualmente se argumenta que los patrones de ADN metilado podrían estratificar el cáncer de pulmón no microcítico en dos subtipos fenotípicamente diferentes capaces de predecir respuesta a inhibidores de la ruta del EGFR8.
Por otra parte, la eficacia de un ensayo con biomarcadores viene determinada por su sensibilidad y especificidad. Para un ensayo con biomarcadores de hipermetilación del ADN, la sensibilidad se describe como la cantidad mínima de ADN metilado (diana) capaz de ser detectada por el ensayo. En términos clínicos, se describe como la proporción de pacientes con enfermedad confirmada, para los cuales el test con el biomarcador resulta positivo; mientras que la especificidad del ensayo viene determinada por la proporción de sujetos controles para los que el test resulta negativo. La sensibilidad óptima se alcanza cuando el número de falsos negativos se aproxima a cero. Por otra parte, la especificidad es alta cuando el número de falsos positivos es bajo.
Actualmente, la sensibilidad en cuanto a la detección de los biomarcadores de metilación de ADN recae principalmente en las técnicas de PCR de metilación específica (MSP) o en la versión cuantitativa de esta técnica (qMSP), junto con el origen y la calidad del ADN de la muestra de partida. La orina o la saliva, parecen ser los fluidos biológicos que proporcionan mayor sensibilidad clínica para detectar cánceres de vejiga y de pulmón respectivamente; sin embargo, son el suero y el plasma las fuentes que mejores datos proporcionan en cuanto a la especificidad tumoral. Desafortunadamente, surge un problema inherente asociado a la alta sensibilidad que proporcionan estos biomarcadores de ADN metilado, y es que esta es mayor que la que actualmente proporcionan los métodos diagnósticos por lo que se podrían tener casos de falsos positivos. Por lo que si un caso clínico es detectado por un marcador de metilación de alta sensibilidad, antes que por cualquier otra tecnología existente, lo más frecuente es que este caso sea clasificado como un falso positivo, restándole futura aplicabilidad al biomarcador identificado.
Modificación de histonas y cáncerLas modificaciones de histonas consisten en reacciones covalentes que afectan a sus regiones amino-terminales. Estas modificaciones incluyen la acetilación, la metilación, la fosforilación, la ubiquitinación, la sumoilación, y la ADP-ribosilación y pueden tener efectos directos sobre diferentes procesos nucleares, incluyendo la transcripción génica, la reparación y replicación del ADN y la organización de cromosomas. Las enzimas que catalizan estas reacciones son las histonas acetiltransferasas (HAT), las desacetilasas de histonas (HDAC), las histonas metiltransferasas (HMT) y las histonas desmetilasas (HDMT). Estas pueden funcionar como activadores o represores transcripcionales dependiendo del residuo sobre el que actúen. Generalmente la acetilación de histonas está asociada con la activación transcripcional, en cambio la metilación de histonas depende del tipo de aminoácido que se metile y de su posición. En cáncer, la modificación más común de histonas es la reducción en la acetilación de la lisina 16 de la histona H49, reacción mediada por las HDAC, que se encuentran sobreexpresadas o mutadas en diferentes tipos tumorales10,11. Además en algunos tipos de cáncer (colon, útero, pulmón y leucemia) se han observado translocaciones que dan lugar a la formación de proteínas aberrantes, mutaciones o deleciones de HAT y de genes relacionados, contribuyendo a una acetilación aberrante de histonas12. Las modificaciones postranscripcionales de histonas están íntimamente relacionadas con metilación y participan en la regulación de la expresión génica, mostrando un valor pronóstico y predictivo en muchos tipos de cáncer, entre los que se encuentra el cáncer de pulmón y ovario. En los últimos años se ha visto como estas modificaciones pueden contribuir a la tumorogénesis y se ha comprobado la existencia de un patrón de expresión alterado de enzimas modificadoras de histonas en los tumores humanos.
miARN y cáncerEl control epigenético más recientemente descubierto es la regulación génica postranscripcional mediada por miARN13. Esta regulación se lleva a cabo gracias a la unión por complementariedad de secuencias del miARN a la región 3’UTR de sus ARNm diana, provocando la degradación de dicho ARNm o inhibiendo la traducción proteica. Existen más de 1.000 miARN maduros en el genoma humano (mirbase.org), cada uno de los cuales presenta múltiples tránscritos diana. La especificidad entre miARN y ARNm diana viene determinada por la complementariedad de secuencia entre ambas moléculas. Un solo miARN puede tener como diana cientos de ARN mensajeros que pueden estar implicados en numerosos procesos biológicos incluidos diferenciación, ciclo celular, y apoptosis14. A su vez, un ARNm puede ser diana de distintos miARN. Por lo tanto, la capacidad de alterar la expresión en una célula que posee un solo miARN depende tanto de su propia concentración como de la concentración de sus ARNm diana, y pequeños cambios en la expresión de un miARN puede tener efectos importantes sobre el fenotipo, puesto que puede influir simultáneamente en la expresión de cientos de genes.
La primera evidencia de la correlación entre cáncer y miARN apareció en el año 2002 cuando se observó que el miR-15a y el miR16-1 estaban codificados por una región frecuentemente delecionada del cromosoma 13, en leucemia linfocítica crónica de células B. Actualmente se conocen miARN que se expresan de forma aberrante o que están mutados en una gran variedad de tumores, lo cual sugiere su implicación en iniciación y progresión del cáncer, además estos tienen una función dual, ya que pueden actuar como oncogenes o genes supresores de tumores en función de sus ARNm diana.
Las alteraciones en la expresión de los miARN se han estudiado en diversos tipos tumorales como en cáncer de pulmón15 y ovario14, permitiendo distinguir entre subtipos celulares. Los miARN no solo sirven para perfilar distintos tipos tumorales, sino que también pueden servir para predecir respuesta a fármacos. Se ha comprobado que en tumores de ovario aparecen regulados negativamente el miR-100 y miR-214, habiéndose demostrado que este último tiene como diana al gen supresor tumoral PTEN asociado con la resistencia a platino16. También se ha descrito el papel de determinados miARN, como el miR-34c, como posibles biomarcadores de riesgo en estudios de quimio-prevención en cáncer de pulmón17. Es necesario remarcar que la expresión de los miARN puede estar regulada epigenéticamente a través de la metilación de islas CpG localizadas en regiones reguladoras de la expresión del miARN, pero a su vez, los miARN pueden actuar regulando la maquinaria epigenética. Por ejemplo, en CNMP se encuentra disminuida la expresión de la familia miR-29, que tiene como diana el ARNm de las metiltransferasas DNMT3A y -3B. Cuando se restablecen los valores normales de estos miR-29s se obtienen unos patrones normales de metilación de ADN, inhibiendo en parte la tumorigenicidad celular.
Aplicaciones clínicasDetección y estratificación del cáncerEntre las aplicaciones clínicas de los marcadores de metilación se incluyen la predicción de respuesta terapéutica (marcadores predictivos), la capacidad pronóstica, asociada con las características intrínsecas de cada tumor (marcadores pronósticos) y la monitorización de pacientes para identificar la presencia de recidivas de la enfermedad tras intervención quirúrgica, o tras tratamiento con quimioterapia para evaluar su efectividad, especialmente si se ha definido el biomarcador de antemano por biopsia.
Se han descrito unos 30 GST regulados epigenéticamente, aunque pocos de ellos son específicos para un tipo tumoral en concreto, por ejemplo, DAPK y p16 son posibles marcadores pronósticos de fenotipos más agresivos en cáncer de pulmón, colorrectal y tumores cerebrales; mientras que VHL por ejemplo parece específico de carcinoma de células renales y BRCA1 de cáncer de mama y de ovario. Sin embargo, algunos genes muestran diferentes grados de metilación según el tipo tumoral: GSTP1 está metilado en un 90% de cáncer de próstata, 30% de cáncer de mama y 25% de cáncer hepático. Esta última característica de algunos marcadores de metilación ha permitido a los científicos desarrollar paneles de metilación con GST diferentes, cubriendo en la mayoría de los casos la detección del 100% de las muestras tumorales; por ejemplo, para la detección precoz del cáncer de mama se han testado diferentes paneles de metilación en muestras con diferente origen como aspirado de mama, lavado ductal o suero. Evron et al18, compararon la metilación de los promotores de ciclina D2, Twist y RAR-β utilizando células procedentes de lavado ductal. Estos investigadores encontraron metilación anormal en las células de algunas mujeres sanas, que luego desarrollaron cáncer de mama, proporcionando una evidencia directa del uso de los marcadores de metilación para la detección del cáncer en individuos asintomáticos. El cáncer de mama también se ha podido identificar a través del análisis de los genes RASSF1A, APC, DAPK, GSTP1, beta RAR, p16 y p14 en muestras de suero y aspirado de mama. También se han descrito diferentes aproximaciones para la detección molecular de cáncer de ovario. El estado de hipermetilación de los genes BRCA1 y RASFF1A se ha valorado en 50 pacientes con cáncer de ovario o con tumores primarios peritoneales. La hipermetilación de uno o ambos genes se encontró en un 68% del ADN tumoral. Un examen adicional de uno o más de los siguientes GST; APC, p14, ARF, p16 y DAPK completó el 100% de cobertura, encontrándose un patrón idéntico de hipermetilación del gen en el ADN de origen sérico (82% de sensibilidad), incluyendo estadios de enfermedad I y II19.
La alteración epigenética más común en el cáncer de próstata es la pérdida de la expresión del gen GSTP1 asociada a la presencia de hipermetilación en su promotor. Esta puede valorarse en orina, pudiendo discriminar entre diferentes grados de malignidad tumoral20. También se ha descrito la correlación entre metilación del gen RARb y discriminación entre tejido neoplásico o no neoplásico.
Uno de los problemas asociados al cáncer de pulmón es el fracaso en su diagnóstico temprano. Sin embargo, el material biológico presente en esputo o en aspirado bronquial permite nuevas aproximaciones diagnósticas. Los genes APC, p16 y RARβ están frecuentemente metilados en este tipo tumoral, lo que ha hecho que se diseñen diferentes ensayos utilizando la técnica de MSP para diagnóstico temprano en pacientes con este tipo tumoral. Por ejemplo, la metilación aberrante en los genes p16 y MGMT en el ADN de esputo se identificó en un 100% de los pacientes con carcinoma de pulmón de células escamosas hasta 3 años antes de su diagnóstico clínico. Por otra parte, el descenso en la expresión del gen IGFBP3, mediada por la metilación de su promotor es un evento temprano en la carcinogénesis de pulmón. Más recientemente se ha descrito que la metilación de este gen, no solo sirve para detección temprana de cáncer de pulmón, sino que también es un marcador de respuesta terapéutica al agente CDDP7.
La alteración epigenética más común en gliomas afecta a la enzima O6-metilguanina-ADN metiltransferasa MGMT,esta actúa eliminando el grupo metilo añadido a la posición O6 de las guaninas del ADN. Se ha descrito que la metilación de las islas CpG localizadas en la zona del promotor de este gen reprime la expresión génica; en el caso de los gliomas malignos, el promotor de MGMT está metilado en un 40-68% de los casos por lo que gana eficacia en tratamiento quimioterápico basado en agentes alquilantes y metilantes21.
La ventaja clínica que tiene la pérdida de la expresión génica por metilación del ADN es, en contraste con las mutaciones genéticas, la posibilidad de revertir el estado de metilación mediante tratamiento farmacológico con inhibidores de la desacetilación de histonas y agentes desmetilantes. En la actualidad hay cuatro medicamentos aprobados por la FDA con acción epigenética: los inhibidores de la ADN metiltransferasa 5-azacitidina (Vidaza) y decitabina (2-desoxi-5-azacitidina, Dacogen) y los inhibidores de acetilasas de histonas (HDAC), ácido hidroxámicosuberoilanilida (SAHA, Zolinza) y romidepsina (Istodax). Varios fármacos adicionales desmetilantes e inhibidores de HDAC se están evaluando en estudios preclínicos y en ensayos clínicos. Sin embargo, no hay en la actualidad ningún ensayo clínico con fármacos candidatos dirigidos contra otros objetivos epigenéticos.
Tanto 5-azacitidina como decitabina han demostrado un beneficio clínico significativo en el tratamiento del síndrome mielodisplásico y en leucemia mieloide. Aunque se haya confirmado por varios estudios que se produce una desmetilación en los pacientes tratados, sin embargo, aún no se ha podido demostrar una relación directa entre la desmetilación del ADN y la respuesta clínica, ya que no hay establecidos biomarcadores de metilación del ADN que predigan con precisión las respuestas del paciente22. Este hecho tan relevante tiene abiertos muchos frentes de estudio en busca de biomarcadores para establecer la prueba de concepto que falta con la terapia epigenética de estos fármacos.
Los fármacos epigenéticos más utilizados en la clínica son 5-azacitidina y decitabina. La incorporación de las bases de azacitosina en el ADN y ARN induce la formación de aductos covalentes con las metiltransferasas, resultando en la depleción de las DNMT, y en la consecuente reducción global de la metilación del ADN. Este descenso en los niveles globales de metilación tiene efectos antitumorales según se ha descrito en diferentes modelos murinos. En general se supone que el beneficio terapéutico de la desmetilación global del ADN está relacionado con la desmetilación y la reactivación de los genes aberrantemente silenciados. Sin embargo, como veremos más adelante, la desmetilación global no es selectiva de los marcadores tumorales que se encuentran metilados en cáncer, efecto que podría restringir severamente la especificidad de la terapia con estos fármacos.
Los inhibidores de HDAC, SAHA y romidepsina se han aprobado para el tratamiento de linfoma cutáneo de células T23. En la actualidad hay numerosos inhibidores de HDAC que se están testando en diferentes ensayos clínicos, debido a la gran farmacobilidad de la familia de enzimas HDAC, que permite el desarrollo de potentes inhibidores específicos. Es importante resaltar que la desacetilación de histonas interactúa sinérgicamente con la metilación del ADN en el silenciamiento epigenético en cáncer, por lo que se han justificado diferentes ensayos clínicos combinando inhibidores de HDAC y DNMT. Sin embargo, los estudios no han revelado efectos clínicos sinérgicos, que podrían estar relacionados con la complejidad de los modos de acción de los fármacos que aún no se han dilucidado. Por una parte, cada vez está más claro que la actividad enzimática de HDAC no está restringida a las proteínas histonas, ya que el tratamiento de líneas celulares de cáncer humano con inhibidores de HDAC altamente específicos, inducen la hiperacetilación de 1.750 proteínas24. Este hallazgo sugiere fuertemente que la gran mayoría de los sustratos de los fármacos de HDAC son proteínas no-histonas.
Efectos secundarios sobre la desmetilación global del ADNEl tratamiento de reactivación epigenética basado en agentes desmetilantes, puede inducir como efectos secundarios inestabilidad genómica y/o aumento de la expresión de protooncogenes como resultado de la hipometilación inducida por estos fármacos.
Además, la hipometilación global del DNA se ha asociado tradicionalmente con inestabilidad cromosómica y propensión al desarrollo de neoplasias, y también se ha relacionado más recientemente, una desregulación en los elementos repetitivos del genoma25. La desmetilación de una región LINE-1 situada en un intrón del protooncogen cMet, resulta en un transcrito aberrante de cMET, que se asocia con la reducción de la expresión de la proteína y de la señalización del receptor de cMET. Estos resultados proporcionan una importante fuente de información, por ejemplo sobre la alteración de la regulación transcripcional que se induce por la desmetilación de elementos repetitivos, lo que sugiere que muchos loci genómicos podrían estar sujetos a los efectos secundarios de la desmetilación epigenética global del ADN26. Por otra parte, es bien sabido que la desmetilación de ADN está estrechamente controlada durante la diferenciación celular, por lo que la desmetilación global que inducen estos fármacos epigenéticos y/o la pérdida de las modificaciones de histonas podrían afectar negativamente a las funciones en las poblaciones de células progenitoras en los pacientes27. Por lo que es crítico la identificación de drogas epigenéticas con dianas más específicas que no actuaran sobre las DNMT, sino sobre otros factores o rutas epigenéticas, como podrían ser enzimas que actúan sobre metilación de novo específica en cáncer. Por ejemplo, la metiltransferasa EZH2 es una enzima crítica en la metilación de la lisina 27 de la histona 3, involucrada en el desarrollo de distintos tipos tumorales. Una terapia dirigida contra esta diana podría redirigir el establecimiento de programas epigenéticos específicos en cáncer28.
La medicina personalizada está tomando cada vez más fuerza dentro de los sistemas hospitalarios público y privados, ya que su uso permite optimizar e implementar tratamientos que disminuyen tanto el coste de los gastos médicos asociados a la quimioterapia como al coste personal y social que deriva de estos tratamientos y del desarrollo de la enfermedad. Gracias a la introducción de las nuevas técnicas genómicas y epigenómicas de nueva generación como la secuenciación masiva, se está acelerando exponencialmente la identificación de nuevos parámetros moleculares; sin embargo sigue siendo de gran importancia dedicarle tiempo y esfuerzo al estudio de la biología de sistemas, ya que puede explicar la implicación biológica real de cada uno de estos potenciales biomarcadores, así como las rutas celulares que se alteran en cada caso, que en definitiva es lo que puede permitir la identificación y selección de fármacos específicos.
FinanciaciónFinanciado por FIS PS09/00472 y PI12/00386, Ibanez de Cáceres I, disfruta de un contrato «Miguel Servet» del ISCIII (CP 08/000689; PI-717). O. Pernía disfruta de un contrato con el Ministerio de Economía y Competitividad PTA2012/7141-I.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.