La reacción en cadena de la polimerasa cuantitativa y fluorescente (QF-PCR) identifica, en menos de 24h, las anomalías cromosómicas más frecuentes, buscadas en el cariotipo prenatal convencional: las alteraciones numéricas de los cromosomas 13, 18, 21, X e Y.
En la QF-PCR se utilizan varios marcadores polimórficos por cromosoma, permitiendo una alta fiabilidad, rapidez, bajo costo y automatización. En cambio, no permite detectar anomalías estructurales, mosaicismos de bajo nivel ni cromosomas marcadores.
ObjetivoDesarrollar y valorar un método rápido, practicable y fiable para la detección de aneuploidías de los cromosomas 13,18, 21, X e Y en las muestras recibidas para estudio de cariotipo prenatal en un laboratorio de citogenética.
Material y métodosAdemás de en los líquidos amnióticos recibidos en el último año (465), la QF-PCR se ha ensayado también, de forma retrospectiva, en las muestras de ADN almacenadas de las enfermedades detectadas en los 2 años anteriores (que corresponden al estudio de unas 1.000 muestras prenatales).
La extracción del ADN se realiza de forma automatizada. Los marcadores utilizados son de alta heterocigosidad y están consensuados internacionalmente (Association for Clinical Cytogenetics. QF-PCR for the diagnosis of aneuploidy best practice guidelines (2007). Disponible en: http://www.cytogenetics.org.uk/prof_standards/professional_standards.htm). El test tiene las mismas condiciones de amplificación para las 4 mezclas multiplex utilizadas. En cada una de ellas se han incluido todos los constituyentes necesarios, excepto la muestra, que se añade en el momento de usarse. Cada amplificado se analiza en el ABI 3130 Genetic Analyzer, de Applied Biosystems. El análisis de los fragmentos obtenidos en la amplificación se realiza siguiendo las recomendaciones publicadas.
ResultadosTodos los resultados de las muestras analizadas, normales o patológicas, han coincidido con el resultado del cariotipo.
En total, se detectaron 12 trisomías 21; 5 trisomías 18; 3 monosomías 45,X; 2 trisomías 13; 2 trisomías 47,XYY; 2 trisomías 47,XXX; 2 trisomías 47,XXY, y 2 triploidías. En todos ellos la QF-PCR propuesta fue diagnóstica. No se detectó una monosomía parcial del cromosoma 18, pero sí pudo ponerse de manifiesto una trisomía parcial del cromosoma 18.
ConclusionesLa QF-PCR propuesta es una herramienta útil para el diagnóstico rápido de aneuploidías. Es extremadamente valiosa en el caso de que no crezcan los cultivos de amniocitos, o de contaminación microbiológica de éstos. Sirve para rebajar la ansiedad materna hasta que el cariotipo tradicional esté informado y para tomar decisiones rápidas cuando se encuentran anomalías ecográficas avanzado el segundo trimestre del embarazo.
Quantitative fluorescence polymerase chain reaction (QF-PCR) identifies in less than 24h the most common chromosomal anomalies looked for in conventional prenatal karyotyping: numerical alterations of chromosomes 13, 18, 21, X and Y.
Several polymorphic markers are used in QF-PCR for each chromosome, allowing a high reliability of results, speed, low cost and automation. Nevertheless, QF-PCR can not detect structural abnormalities, low-level mosaicism or marker chromosomes.
ObjectiveTo develop and evaluate a fast, reliable and feasible method for detecting aneuploidies of chromosomes 13, 18, 21, X and Y, in the samples received for prenatal karyotyping in a cytogenetics department.
Materials and methodsIn addition to the 465 amniotic fluids received during last year, stored DNA samples in which a disease was detected in the previous two years were assayed (corresponding to the study of around 1,000 prenatal samples).
DNA extraction is automated. The STR polymorphic markers used, with high heterozygosity, are accepted internationally (Association for Clinical Cytogenetics. QF-PCR for the diagnosis of aneuploidy best practice guidelines (2007). Available at http://www.cytogenetics.org.uk/prof_standards/professional_standards.htm). The test has the same PCR amplification conditions for all four multiplex mixtures used. In each of them All the necessary components were included in each of them, except the sample, which was added at the time of the assay. Each amplified mixture is analyzed into the ABI3130 Genetic Analyzer, Applied Biosystems. Fragments analysis is achieved following the published recommendations.
ResultsThe results of all the tested samples, normal or pathological, matched the outcome of the conventional karyotype.
In total the QF-PCR assay detected: 12 trisomy 21; 5 trisomy 18; 3 women 45,X ; 2 trisomy 47,XXY ; 2 trisomy 13 ; 2 Triple XXX ; 2 men XYY; and 2 triploid pregnancies. In each of these cases the proposed QF-PCR was diagnostic. A partial monosomy of chromosome 18 could not be detected, but a partial trisomy of chromosome 18 was identified.
ConclusionsThe evaluated QF-PCR is a useful tool for the rapid diagnosis of aneuploidies (24h). It is extremely valuable when the amniocyte cultures fail to grow, or suffer microbiological contamination. It serves to reduce maternal anxiety until the conventional karyotype is reported, and to take quick decisions when ultrasound anomalies are found in the second trimester of pregnancy.
Durante la pasada década se han realizado grandes progresos en la detección de las enfermedades fetales de forma no invasiva, gracias a los estudios ecográficos y a los test bioquimícos integrados1. Pero ello, junto con la mayor edad de las gestantes, ha conducido a un aumento importante del número de procedimientos invasivos para obtener muestras fetales, que se analizan en los laboratorios de genética.
El intervalo de tiempo entre la obtención de la muestra y la emisión del informe del cariotipo fetal (2 a 3 semanas) es un período de gran ansiedad para los padres2.
En estos años y de modo paralelo se han desarrollado ensayos como la reacción en cadena de la polimerasa cuantitativa y fluorescente (QF-PCR), que permiten la detección rápida de las aneuploidías más frecuentes, las de los cromosomas 13, 18, 21, X e Y2–10. Este estudio prenatal permite, en tan sólo 24h, detectar la mayoría de las anomalías cromosómicas que se observan en el cariotipo y que causan enfermedad en el recién nacido.
Se utilizan 4 o más marcadores por cromosoma1, que habitualmente se amplifican en reacción en cadena de la polimerasa (PCR) multiplex, con cebadores marcados con fluoróforos, y que después se analizan en un secuenciador. La mayor parte de estos marcadores son STR (short tandem repeats) muy polimórficos y que nos proporcionan señales 1:1 en la mayor parte de los casos, mientras que las muestras trisómicas nos dan señales 1:1:1 (trisómicas trialélicas) o 2 picos con una relación 2:1 (trisómicos dialélicos)3.
Hay otras muestras, como los restos fetales, que también se remiten al laboratorio para cariotipado y que tienen un crecimiento celular dificultoso o nulo y en los que este test molecular nos da a veces la única respuesta posible11.
Las principales ventajas del ensayo son su rapidez, bajo costo, automatización y alta fiabilidad. Sin embargo, no puede detectar anomalías estructurales, mosaicismos de bajo nivel y cromosomas marcadores.
Tradicionalmente se ha reservado esta prueba y la hibridación in situ fluorescente (FISH)12 para pacientes en circunstancias especiales2, pero dado el bajo costo de ésta y su automatización podría hacerse a todas muestras de amniocentesis13.
Ha sido nuestra intención el desarrollar y valorar un método rápido y practicable para la detección de aneuploidías de los cromosomas estudiados en todas las muestras recibidas para estudio de cariotipo prenatal en nuestro laboratorio de citogenética.
Material y métodosMuestrasSe han ensayado las 465 muestras de líquido amniótico recibidas en el último año, y hemos analizado también todas las muestras de ADN almacenadas de las enfermedades que se habían detectado en los 2 años anteriores mediante el cariotipo, que corresponden al estudio de unos 1.000 líquidos amnióticos más.
Para las muestras contaminadas con sangre, visible a simple vista, el tocólogo que hace la extracción nos remite un enjuagado bucal de la madre. Se utiliza este tipo de muestra porque contiene un número suficiente de células de descamación de la boca y es muy cómoda para la paciente, para el transporte y para el procesamiento en el laboratorio.
En algunos casos es necesario solicitar también una muestra paterna para completar el estudio.
A todas las madres se les informa por escrito que se va a realizar una QF-PCR de forma simultánea al inicio del cultivo celular y que el resultado de ésta estará disponible en 24–48h.
Las muestras se reciben en el laboratorio en 3 tubos estériles: 2 se dedican al cultivo y el otro, que contiene sólo 3–4ml, se destina a la QF-PCR.
Se anota el aspecto de la muestra, sobre todo en cuanto a la presencia de contaminación materna, que evaluamos en una escala de 0 a 4 según se observen más o menos hematíes antes y después de la centrifugación.
Los tejidos de restos fetales se homogeneizan en salino. Las células procedentes de cultivos se desprenden con tripsina y se lavan con salino.
Métodos y reactivosLa extracción del ADN de las diferentes muestras está automatizada en el MagNA Pure Compact de Roche Diagnostics. Se siguen las instrucciones del fabricante.
Para el líquido amniótico y el enjuagado bucal partimos de 1,5ml de muestra. Tras el proceso de extracción obtenemos 100μl de eluido que contienen 1–4ng/μl de ADN. No es necesaria la cuantificación del ADN extraído, en la rutina diaria, porque se ha comprobado que la extracción es muy reproducible.
Las secuencias de los cebadores se muestran en la tabla 1 y están ampliamente descritas en la literatura médica2,4–6,9,10,12–15. En la tabla 2 se describen las características de cada uno de ellos y su distribución en las 4 mezclas multiplex, así como las referencias bibliográficas.
Secuencia de los cebadores y concentración de éstos en cada mezcla de reacción multiplex
| Nombre | Mezcla | Secuencia cebadores | Concentración |
| FOR XKRY | A | VIC-CACTCATGGAGAAGGGTAGG | 40nM |
| REV XKRY | A | GTCACACTCAGCCTCTTTAC | 40nM |
| FOR D18S535 | A | FAM-TCATGTGACAAAAGCCACAC | 75nM |
| REV D18S535 | A | AGACAGAAATATAGATGAGAATGCA | 75nM |
| FOR D21S11 | A | VIC-TATGTGAGTCAATTCCCCAAGTGA | 180nM |
| REV D21S11 | A | GTTGTATTAGTCAATGTTCTCCAG | 180nM |
| FOR SRY | A | NED-AGTAAAGGCAACGTCCAGGAT | 180nM |
| REV SRY | A | GCTGGTGCTAAATTCTTGAG | 180nM |
| FOR D13S634 | A | FAM-TCCAGATAGGCAGATTCAAT | 200nM |
| REV D13S634 | A | CCTTCTTCTTCCCATTGATA | 200nM |
| FOR D21S1435 | B | PET-CCCTCTCAATTGTTTGTCTACC | 100nM |
| REV D21S1435 | B | GCAAGAGATTTCAGTGCCAT | 100nM |
| FOR D13S258 | B | VIC-ACCTGCCAAATTTTACCAGG | 50nM |
| REV D13S258 | B | GACAGAGAGAGGGAATAAACC | 50nM |
| FOR XHPRT | B | FAM-ATGCCACAGATAATACACATCCCC | 110nM |
| REV XHPRT | B | CTCTCCAGAATAGTTAGATGTAGGTAT | 110nM |
| FOR D18S386 | B | VIC-TCAGGAGAATCACTTGGAAC | 100nM |
| REV D18S386 | B | TCCATGAAGTAGCTAAGCAG | 100nM |
| FOR D13S256 | C | NED-CCTGGGCAACAAGAGCAAA | 90nM |
| REV D13S256 | C | AGCAGAGAGACATAATTGTG | 90nM |
| FOR D18S858 | C | FAM-AGCTGGAGAGGGATAGCATT | 90nM |
| REV D18S858 | C | TGCATTGCATGAAAGTAGGA | 90nM |
| FOR X22 | C | PET-TAATGAGAGTTGGAAAGAAA | 230nM |
| REV X22 | C | CCCATTGTTGCTACTTGAGA | 230nM |
| FOR D21S1412 | C | FAM-CGGAGGTTGCAGTGAGTTG | 135nM |
| REV D21S1412 | C | GGGAAGGCTATGGAGGAGA | 135nM |
| FOR AMEL | D | FAM-CCCTGGGCTCTGTAAAGAATAGTG | 50nM |
| REV AMEL | D | ATCAGAGCTTAAACTGGGAAGCT | 50nM |
| FOR D18S391 | D | VIC-GGACTTACCACAGGCAATGTGACT | 90nM |
| REV D18S391 | D | TAGACTTCACTATTCCCATCTGAG | 90nM |
| FOR D13S631 | D | NED-GGCAACAAGAGCAAAACTCT | 90nM |
| REV D13S631 | D | TAGCCCTCACCATGATTGG | 90nM |
| FOR D21S1411 | D | FAM-ATGATGAATGCATAGATGGATG | 100nM |
| REV D21S1411 | D | AATGTGTGTCCTTCCAGGC | 100nM |
| FOR DX8377 | D | VIC-CACTTCATGGCTTACCACAG | 100nM |
| REV DX8377 | D | GACCTTTGGAAAGCTAGTGT | 100nM |
Características de los marcadores utilizados en cada mezcla multiplex y referencia bibliográfica de las secuencias de los cebadores
| Nombre | Mezcla | Marcaje | Tamaño | Heterocigosidad | Localización | Tándem | Referencia cebadores |
| XKRY | A | VIC | 91 | ***** | Yq11.221 | ***** | NM_004677 GenBank |
| D18S535 | A | 6-FAM | 115-160 | 0,78 | 18q12.2 | 4 | Pertl et al, 1999 |
| D21S11 | A | VIC | 202-280 | 0,90 | 21q21 | 4 | Pertl et al, 1996 |
| SRY | A | NED | 380 | ***** | Yp11.2 | ***** | Ogilvie et al, 2005 |
| D13S634 | A | 6-FAM | 453-512 | 0,83 | 13q14.3 | 4 (y 2) | Pertl et al, 1997 |
| D21S1435 | B | PET | 156-185 | 0,77 | 21q21 | 4 | Cirigliano et al, 2001 |
| D13S258 | B | VIC | 230-267 | 0,86 | 13q21 | 4 (y 2) | Pertl et al, 1999 |
| XHPRT | B | 6-FAM | 270-300 | 0,74 | Xq26.1 | 4 | Pertl et al, 1997 |
| D18S386 | B | VIC | 314-402 | 0,89 | 18q22.1 | 4 | Pertl et al, 1999 |
| D13S256 | C | NED | 115-178 | 0,90 | 13q21 | 4 | Schmidt et al, 2000 |
| D18S858 | C | 6-FAM | 184-220 | 0,74 | 18q21.1 | 4 | Hulten et al, 2003 |
| X22 | C | PET | 192-258 | 0,88 | Xq28/Yq12 | 5 | Cirigliano et al, 1999 |
| D21S1412 | C | 6-FAM | 378-432 | 0,79 | 21q22.2 | 4 | Pertl et al, 1997 |
| AMEL | D | 6-FAM | 103 | ***** | Xp22.1-23.31 | ***** | Sullivan et al, 1993 |
| AMEL | D | 6-FAM | 109 | ***** | Yp11.2 | ***** | Sullivan et al, 1993 |
| D18S391 | D | 6-FAM | 140-180 | 0,75 | 18pter-18p11.22 | 4 | Mann et al, 2001 |
| D13S631 | D | NED | 178-230 | 0,80 | 13q31-32 | 4 | Pertl et al, 1997 |
| DXS8377 | D | VIC | 213-252 | 0,9 | Xq28 | 3 | Schmidt et al, 2000 |
| D21S1411 | D | 6-FAM | 253-326 | 0,93 | 21q22.3 | 4 | Pertl et al, 1996 |
La reacción de PCR de cada marcador se ha estudiado individualmente en el termociclador con gradiente de temperatura Thermo Hybaid Px2®.
El análisis tiene las mismas condiciones de amplificación para las 4 mezclas. En la tabla 3 se indican esas condiciones de reacción.
Las concentraciones finales en el tubo de reacción son: 250μM de desoxinucleótido trifosfato (dNTP), 2,25mM de Mg++, 1X de PCR Gold Buffer, 40-230 nM de cebadores (tabla 1), y contiene 1 U de AmpliTaq Gold por reacción. Todos los reactivos fueron suministrados por Applied Biosystems.
Cada mezcla (A, B, C y D) (16μl) contiene todos los constituyentes necesarios, excepto la muestra (4μl) que se añade en el momento de usarse. Dado que la polimerasa utilizada es hot-start, no son necesarias precauciones en cuanto a temperatura de trabajo.
Hemos incluido un control negativo (H2O) en todas las series de trabajo.
Cada amplificado se diluye 1/10 con un mezcla de Hi-Di® Formamida, que contiene el marcador de tamaño GS 500 LIZ® de Applied Biosystems, y se analiza en el ABI3130 Genetic Analyzer, programado convenientemente y usando el programa informático GeneMapper® V4.0 de Applied Biosystems.
El análisis de los fragmentos obtenidos se hace siguiendo las recomendaciones de la guía antes indicada1 y considerando los posibles problemas de interpretación: amplificación preferencial de alelos, picos stutter, duplicación o mutación de microsatélites8,16.
ResultadosTodos los resultados (en cuanto se refiere a aneuploidías) de las muestras analizadas, normales o patológicos, han coincidido con el resultado del cariotipo.
En la tabla 4 se muestran los resultados obtenidos en el último año y en la tabla 5 se indican los casos analizados por QF-PCR a posteriori en muestras congeladas de nuestro archivo, correspondientes a las anomalías numéricas detectadas por el cariotipo en los 2 años anteriores. Nuestra QF-PCR es diagnóstica en todos los casos donde debe serlo.
Descripción de las enfermedades detectables por reacción en cadena de la polimerasa cuantitativa y fluorescente (QF-PCR), encontradas en el último año en líquido amniótico y restos fetales
| Descripción caso | Fórmula cromosómica | Número | QF-PCR diagnóstica |
| Síndrome de Down | 47,XX,+21 | 3 | Sí |
| Síndrome de Down | 47,XY,+21 | 2 | Sí |
| Síndrome de Down+Síndrome de Klinefelter | 48,XXY,+21 | 1 | Sí |
| Síndrome de Edwars | 47,XY,+18 | 1 | Sí |
| Síndrome de Edwars | 47,XX,+18 | 2 | Sí |
| Síndrome de Patau | 47,XX,+13 | 1 | Sí |
| Síndrome triple X | 47,XXX | 1 | Sí |
| Síndrome XYY | 47,XXY | 1 | Sí |
| Síndrome de Turner mosaico (isocromosoma) | 45,X[19]/46,X,i(Xq)[81] | 1 | No |
| Síndrome de Turner mosaico (isodisómico) | 45,X[15]/46,X,upd(X)mat[85] | 1 | Sí |
| Triploidía (en restos fetales) | 69,XXY | 1 | Sí |
| Triploidía (en restos fetales) | 69,XXX | 1 | Sí |
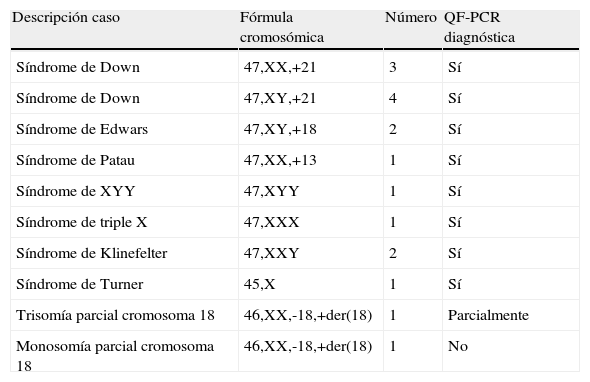
Descripción de las enfermedades detectables por reacción en cadena de la polimerasa cuantitativa y fluorescente (QF-PCR), encontradas en los 2 años anteriores por cariotipado, y a las que se les ha realizado QF-PCR a posteriori
| Descripción caso | Fórmula cromosómica | Número | QF-PCR diagnóstica |
| Síndrome de Down | 47,XX,+21 | 3 | Sí |
| Síndrome de Down | 47,XY,+21 | 4 | Sí |
| Síndrome de Edwars | 47,XY,+18 | 2 | Sí |
| Síndrome de Patau | 47,XX,+13 | 1 | Sí |
| Síndrome de XYY | 47,XYY | 1 | Sí |
| Síndrome de triple X | 47,XXX | 1 | Sí |
| Síndrome de Klinefelter | 47,XXY | 2 | Sí |
| Síndrome de Turner | 45,X | 1 | Sí |
| Trisomía parcial cromosoma 18 | 46,XX,-18,+der(18) | 1 | Parcialmente |
| Monosomía parcial cromosoma 18 | 46,XX,-18,+der(18) | 1 | No |
Un caso de monosomía parcial del cromosoma 18 no pudo detectarse en la QF-PCR, pero sí pudo detectarse una trisomía parcial del cromosoma 18. En el caso del síndrome de Turner, en mosaico y con un isocromosoma X el análisis no fue concluyente.
En las figuras 1 y 2 se muestran los trazados electroforéticos típicos correspondientes a un feto varón normal y a un feto XX con trisomía 21, respectivamente.
Trazados electroforéticos de la mezclas A, B, C y D correspondientes a un feto XX con trisomía 21. Todos los marcadores del cromosoma 21 son informativos. En la mezcla A se ve un patrón 2:1, en la mezcla B se ve un patrón 2:1, en la mezcla C se ve un patrón 1:1:1 y en la mezcla D se ve un patrón 2:1.
En nuestras condiciones de trabajo se ha observado una amplificación preferencial del alelo de menor tamaño más acusada en el caso de los marcadores X22, D13S258, D18S386, lo que ha de tenerse en cuenta a la hora de la interpretación de resultados ambiguos.
Los marcadores que presentan más picos –A son el HPRT, D13S258, D13S256, D18S858 y D13S631, aunque este fenómeno se ve muy minorado si se dejan las muestras para inyectar hasta el día siguiente.
El marcador DX8377 es el único que presenta picos stutter de consideración, ya que el tándem de repetición es trinucleótido.
También hemos observado, en este tiempo, 2 duplicaciones de microsatélites. Una de ellas fue del X22, que tenía origen materno, y otra fue en el D21S1411, también de origen materno.
DiscusiónLos resultados de sensibilidad y especificidad de la prueba en cuanto a la detección de las trisomías estudiadas es del 100% en esta serie y están de acuerdo con los señalados en la literatura médica7,8,17, muy próximos al 100%.
La detección del síndrome de Turner es mayor del 99%, dada la alta heterocigosidad de los marcadores usados: X22, DX8377 y HPRT12,15.
Varios autores4,5,9,14,15 han estudiado en profundidad la detección de aneuploidías que implican a los cromosomas sexuales y éstas se han resuelto con el uso de marcadores específicos del cromosoma Y, del cromosoma X y con marcadores seudoautosómicos de ambos15. Hemos escogido los marcadores señalados en esa literatura médica.
En el caso frecuente de mosaicismo de los cromosomas sexuales, la interpretación puede ser muy dificultosa o no concluyente. Baste como ejemplo el caso de síndrome de Turner, en mosaico, que presenta un isocromosoma X de este trabajo. Todos los marcadores del cromosoma X usados en nuestras mezclas están en el brazo largo. Con las señales obtenidas en el electroferograma: señales únicas o muchas señales aproximadamente 2:1, no era concluyente de que se tratara de un Turner en mosaico o de un feto triple X.
Las monosomías o trisomías parciales de los cromosomas estudiados pueden no detectarse, como ha ocurrido en el caso de monosomía parcial del cromosoma 18 presentado en este trabajo. Lógicamente, los casos de cromosomas marcadores, translocaciones recíprocas o robertsonianas, deleciones e inversiones observadas en este período de tiempo no los ha detectado esta técnica.
Las trisomías en mosaico, si no presentan ningún marcador trialélico, no pueden detectarse, salvo que el mosaico lo sea en un porcentaje muy alto (>40%). Si tienen algún marcador trialélico, se pueden detectar con un porcentaje mucho más bajo. No tenemos ningún caso en el presente estudio.
Las recomendaciones para la obtención de resultados óptimos están recogidas en la guía1 de buenas prácticas y se han seguido al pie de la letra en el presente estudio, ya que recogen el conocimiento de autores que han realizado gran cantidad de este tipo de análisis7. Señalaremos algunas de ellas:
Debe obtenerse ADN de calidad y con la mínima manipulación; siempre debe ponerse un control negativo, debe usarse un mínimo de 4 marcadores por cromosoma, deben ser de alta heterocigosidad y consensuarse para este fin1.
Un resultado normal puede informarse con sólo un marcador dialélico informativo y los demás presentando una única señal, pero es deseable hacerlo con 2 marcadores dialélicos1. Un resultado patológico siempre ha de informarse con al menos 2 marcadores informativos de trisomía y con todos los demás no informativos1.
El ratio alélico se calcula dividiendo el área del alelo de menor tamaño por el área del alelo de mayor tamaño. El rango normal es de 0,8 a 1,4 y el de los trisómicos dialélicos es de 0,45 a 0,65 o de 1,8 a 2,4. Los rangos intermedios de 1,4 a 1,8 y de 0,65 y 0,8 son inconclusivos1.
Se pueden obtener ocasionalmente patrones normales y anormales para un determinado cromosoma. Esto puede deberse a una alteración cromosómica desbalanceada clínicamente relevante, a variantes del número de copias del STR, a polimorfismos en el lugar de unión de los cebadores o a mutaciones somáticas del microsatélite1,8,16.
A pesar del escaso número de muestras, hemos detectado 2 mutaciones somáticas de los microsatélites X22 y D21S1411.
Algunas estrategias de diseño incluyen todos los marcadores de un cromosoma en la misma mezcla13, pero nos parece más adecuada la utilizada aquí, ya que cada mezcla contiene un marcador de cada cromosoma estudiado, de modo que la información proporcionada por cada una de ellas ha de ser obligatoriamente compatible con la que den las otras. Eso proporciona un elemento de control de calidad adicional respecto a la manipulación de tubos.
Como ya se ha mencionado, este análisis se ha reservado tradicionalmente a pacientes en las que concurrían determinadas circunstancias2, como estar cerca del límite temporal de las 22 semanas o presentar anomalías ecográficas que indican una alta probabilidad de aneuploidía.
Las principales causas de petición de cariotipo prenatal en nuestro Hospital son la edad materna, el cribado bioquímico positivo y las cifras elevadas de translucencia nucal. La suma de todas ellas es de más del 90%. Con esa casuística, la presencia de aneuploidías es el hallazgo patológico mayoritariamente esperado y como la QF-PCR es capaz de confirmar o descartar la inmensa mayoría de ellas, parece razonable realizarla en todos los casos13, teniendo en cuenta que es rápida y barata. Es una decisión local y de acuerdo con los clínicos implicados1.
Además, si falla el cultivo celular por falta de crecimiento o contaminación microbiológica, se puede, al menos, ofrecer una respuesta parcial.
En los casos en que la muestra está parcialmente contaminada con sangre materna y disponemos de material materno podemos dar el resultado de la QF-PCR10,11 y además garantizar que el cariotipo realizado, si es XX, corresponde al feto y no al de la madre o a una mezcla de ambas, realizando una QF-PCR de las células del tubo de cultivo una vez finalizado el estudio citogenético1.
Si disponemos de material genético parental, podemos asegurar (confrontando el tamaño de los alelos de cada STR del feto y de los padres) que un feto con una aneuploidía pertenece a esos padres con una probabilidad cercana al 100%.
Se descarta así cualquier cambio de muestras en cualquier punto desde la extracción hasta el resultado final1. Esta seguridad es muy importante ante la posible decisión de interrupción legal del embarazo.
Lógicamente también nos permite saber el origen del cromosoma extra en el caso de una trisomía, lo que tiene un interés limitado.
Hemos calculado el porcentaje de heterocigosidad de cada uno de los STR estudiados en nuestra población y es sustancialmente el mismo que el señalado en la literatura médica12,17.
Aunque el número de muestras es pequeño (corresponde a unos 2.800 nacimientos/año), nos ha sorprendido el alto número de aneuploidías detectadas, que corresponde a una frecuencia mayor que la reportada en la literatura médica18. Esto podría deberse a la mayor edad media de las gestantes actuales, comparado con la que tenían las gestantes en las que se hicieron los estudios de frecuencia.
Actualmente, también se usan otras técnicas moleculares, como FISH o MLPA (multiplex ligation-dependent probe amplification) para la detección de aneuploidías19,20.
Históricamente, el FISH ha sido la primera técnica molecular utilizada para el estudio de aneuploidías y aunque presenta una sensibilidad y especificidad cercana al 100% tiene algunas desventajas frente a la QF-PCR: necesita núcleos de células en interfase conservados, consume mucho tiempo de personal técnico cualificado, no puede automatizarse, no permite detectar la contaminación materna y los reactivos son mucho más costosos. La técnica FISH también se está usando eficazmente para el diagnóstico de varios síndromes de microdeleción cromosómica20.
Con respecto a la detección de aneuploidías, la MLPA no aporta ventajas respecto a la QF-PCR: no descubre todos los casos de triploidías, la sensibilidad para la detección de mosaicismos es baja, requiere más tiempo de trabajo y no permite revelar la contaminación materna20.
Otra técnica de futuro es la array CGH (array comparative genomic hybridization `hibridación genómica comparada en micromatrices'), que en un trabajo preliminar se ha comparado con la QF-PCR en la detección de aneuploidías21, y que tiene un potencial enorme en el diagnóstico prenatal, pues permite en un solo abordaje detectar incluso pequeñas pérdidas o ganancias de material cromosómico.
En conclusión, pensamos que se han conseguido los objetivos planteados en este trabajo: rapidez (24h), practicabilidad, bajo costo de reactivos (menos de 12euros, incluida la extracción de ADN) y fiabilidad (detección del 100% de las aneuploidías estudiadas, que corresponden al 79% de todas las enfermedades que hemos detectado en el cariotipo).
Este trabajo corresponde a una comunicación científica presentada y premiada en el II Congreso Nacional del Laboratorio Clínico celebrado en La Coruña del 4 al 7 de junio de 2008.