El pronóstico de las infecciones fúngicas invasivas (IFI) depende de la rapidez de su diagnóstico y tratamiento. Los métodos diagnósticos convencionales son poco sensibles, laboriosos y demasiado lentos; por lo tanto, existe la necesidad de nuevas estrategias diagnósticas más rápidas y eficaces.
Para diagnosticar una candidemia, existen diferentes técnicas más rápidas que el hemocultivo (HC) convencional. Una vez detectado el crecimiento de levaduras en HC, la identificación de la especie puede acelerarse mediante espectrometría de masas (30minutos), técnicas moleculares comercializadas (60-80minutos) o hibridación fluorescente in situ (90minutos). La detección combinada de biomarcadores (antimicelio, manano y antimanano o β-glucano) ha demostrado mayor utilidad que su uso individualizado. Las técnicas comercializadas de amplificación de ácidos nucleicos (Septifast®, T2Candida®) son alternativas muy fiables al HC. La detección del antígeno capsular de Cryptococcus, mediante aglutinación de látex o inmunocromatografía, es una valiosa técnica para el diagnóstico de la criptococosis.
La microscopía directa y cultivo de muestras representativas constituyen el diagnóstico convencional de la IFI por mohos. La detección de galactomanano y de β-glucano se considera criterio diagnóstico probable de aspergilosis invasora y de la IFI, respectivamente. La detección en aliento de compuestos orgánicos volátiles fúngicos es una interesante estrategia diagnóstica en infecciones pulmonares. Las técnicas de detección de ácidos nucleicos no se consideran un criterio diagnóstico de la IFI por mohos en los documentos de consenso debido a su falta de estandarización. No obstante, son la única alternativa al cultivo en las infecciones por Scedosporium/Lomentospora, Fusarium, zigomicetos u hongos dematiáceos.
The prognosis of invasive fungal infections (IFI) depends on the speed of diagnosis and treatment. Conventional diagnostic methods are of low sensitivity, laborious and too slow, leading to the need for new, faster, and more efficient diagnostic strategies.
There are several techniques for diagnosing a candidemia that are faster than the conventional blood culture (BC). Once yeast growth in BC is detected, species identification can be speeded up by mass spectrometry (30minutes), commercialised molecular techniques (60-80minutes) or fluorescent in situ hybridization (90minutes). The combined detection of biomarkers (antimicellium, mannan and anti-mannan or β-glucan) has shown to be of greater use than their individual use. Commercialised nucleic acid amplification techniques (Septifast®, T2Candida®) are very reliable alternatives to BC. The detection of the capsular antigen of Cryptococcus, by means of latex agglutination or immuno-chromatography, is a valuable technique for cryptococcosis diagnosis.
Direct microscopic examination and culture of representative specimens is used for the conventional diagnosis of IFI by filamentous fungi. Detection of galactomannan and β-glucan are considered diagnostic criteria for probable invasive aspergillosis and probable IFI, respectively, despite the lack of specificity of the latter. The detection of fungal volatile organic compounds in breath is an interesting diagnostic strategy in pulmonary infections. Although widely used, nucleic acid detection techniques are not considered diagnostic criteria for IFIs caused by moulds in consensus documents, due to their lack of standardisation. However, they are the only alternative to culture methods in invasive infections by Scedosporium/Lomentospora, Fusarium, zygomycetes, or dematiaceous fungi.
Candida es el género fúngico que con más frecuencia produce micosis invasora en el ser humano. Las principales formas de candidiasis invasora se manifiestan como candidemias o infecciones intraabdominales9. Por otra parte, hace ya varios años que se demostró la relación directa entre el tiempo de inicio de una terapia antifúngica adecuada y el pronóstico del paciente22. Teniendo en cuenta que los métodos tradicionales de diagnóstico microbiológico basados en el cultivo son poco sensibles, laboriosos y demasiado lentos16, existe una necesidad urgente de pruebas diagnósticas que mejoren lo considerado como referencia hasta ahora. Por eso han ido apareciendo diferentes estrategias diagnósticas que disminuyen los tiempos y mejoran la sensibilidad.
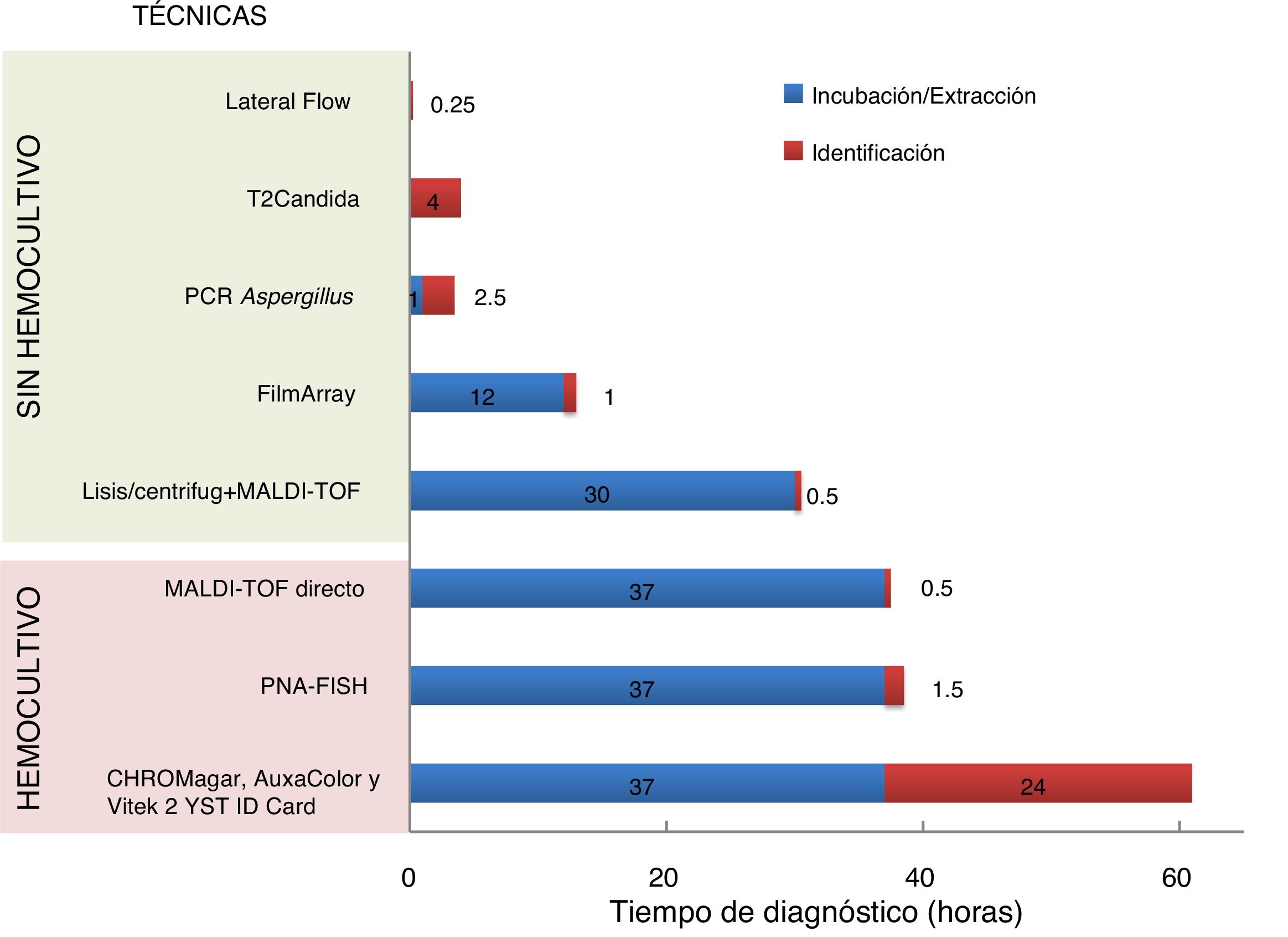
Optimización de los métodos diagnósticos convencionalesCultivoEn la década de 1990 surgieron las primeras propuestas para mejorar el rendimiento de los hemocultivos en la recuperación de hongos en sangre. Estos sistemas basados en técnicas de lisis-centrifugación44 son laboriosos y en algunos grupos de población parece que muestran peores resultados12, por lo que su uso ha quedado restringido. Recientemente, se han utilizado en combinación con la espectrometría de masas MALDI-TOF (Matrix Assisted Laser Desorption/Ionization-Time of Flight), obteniendo resultados positivos en hemocultivos simulados hasta 7h antes del crecimiento en el frasco de hemocultivo28 (fig. 1). Asimismo, se han desarrollado medios enriquecidos específicos (BACTECTM Mycosis-IC/F), para ser utilizados en los sistemas de incubación automatizados de hemocultivos, que adelantan hasta 48 y 64h la detección de Candida glabrata y Cryptococcus neoformans, respectivamente48.
Representación gráfica del tiempo necesario para realizar el diagnóstico en el laboratorio de las infecciones fúngicas invasoras, diferenciando entre métodos dependientes e independientes del hemocultivoFuente: adaptado de Ibáñez-Martínez et al.27.
De igual forma, a partir de los hemocultivos crecidos, han aparecido importantes novedades que eliminan las 18-48h necesarias de crecimiento del subcultivo en medio sólido para su posterior identificación. La espectrometría de masas ha demostrado su utilidad en este contexto, obteniéndose en 30 minutos, a partir de los sedimentos del centrifugado de hemocultivos crecidos, tasas de identificaciones correctas de hasta el 100%53 (fig. 1). Otro sistema basado en la hibridación fluorescente in situ con sondas peptídicas (Yeast Traffic Light PNA FISH®, AdvanDx, Inc., Woburn, EE.UU.) consigue diferenciar mediante diferentes colores en 90 minutos las cinco especies de Candida más frecuentes: Candida albicans, Candida parapsilosis, Candida tropicalis, C. glabrata y Candida krusei. Una evaluación reciente de esta técnica permitió identificar correctamente la especie en un 95,8% cuando se aisló una sola especie, reduciendo el tiempo de inicio de la terapia adecuada en pacientes críticos33 (fig. 1). Otros sistemas basados en la amplificación de una región conservada del genoma de la levadura y la secuenciación posterior del amplicón arrojó hasta un 100% de identificaciones correctas en unas 4h54. También se ha desarrollado recientemente un sistema comercial cerrado mediante una PCR múltiple que permite la identificación en alrededor de 60 minutos (tras 12h de incubación) de 24 patógenos entre los que se incluyen las cinco especies de Candida más frecuentes (FilmArray Blood Culture Panel®, BioFire Diagnostics LLC, Salt Lake City, EE.UU.) (fig. 1). El sistema ha mostrado una sensibilidad del 96,7-100% dependiendo de la especie y una especificidad superior al 99%56. Recientemente se ha descrito una tecnología de hibridación con sondas de captura del ARN ribosómico fúngico y una amplificación de la señal mediante un polímero polisacárido de alto peso molecular conjugado con múltiples moléculas de biotina. Los autores fueron capaces de identificar hasta siete especies de Candida de los hemocultivos en menos de 80 minutos3.
Detección directa de la sensibilidad antifúngica en el hemocultivoAunque en el género Candida la sensibilidad antifúngica es bastante predecible en función de la especie, en ocasiones se describen aislamientos que han desarrollado mecanismos de resistencia secundaria por mutaciones que reducen la utilidad de los antifúngicos de elección57. Por todo ello, diferentes grupos de investigadores están poniendo a punto métodos que permiten o bien realizar antifungigramas directamente del hemocultivo crecido6 o detectar rápidamente mediante biología molecular las mutaciones responsables de las resistencias a los antifúngicos63.
Detección de biomarcadoresLa distribución de pruebas comerciales que detectan en suero componentes estructurales de algunos hongos o anticuerpos frente a los mismos se ha consolidado con éxito. Destacan, entre otros, para el diagnóstico de la candidemia la detección de anticuerpos antimicelio (CAGTA), la detección de manano y anticuerpos antimanano (MN/acMN) y la detección de β(1,3)-D-glucano (BDG). Cada una de estas técnicas por sí sola no supera el 90% de sensibilidad con especificidades variables en torno al 80%7. Por eso se han realizado estudios combinando los diferentes tests para mejorar el rendimiento diagnóstico. En un estudio reciente combinando CAGTA con BDG o MN/acMN, se describió que las combinaciones de CAGTA con BDG o CAGTA con MN/acMN fueron las que mejores resultados ofrecieron para descartar la candidemia, aumentando significativamente el valor predictivo negativo (VPN) que, en función de la prevalencia y la combinación utilizada, osciló entre el 95,6% y el 99,8%. Sin embargo, su papel en el diagnóstico de la candidemia fue más discreto, con valores predictivos positivos (VPP) que variaron entre el 82,9% y el 19,4%38.
La detección de antígeno de Cryptococcus en la meningitis constituye un ejemplo de cómo un test diagnóstico que detecta en suero o líquido cefalorraquídeo un componente polisacárido de la cápsula del hongo (glucuronoxilomanano) presenta tan buen resultado que ha superado al cultivo como técnica de elección en el diagnóstico de la criptococosis. Las técnicas basadas en aglutinación con látex permiten la detección y cuantificación del antígeno criptocócico en muy pocos minutos con una sensibilidad por encima del 97%47. Recientemente, el diagnóstico se ha visto mejorado con la comercialización de un test de inmunocromatografía basado en lateral flow assay (LFA), más barato y más sensible (>99%) que los clásicos métodos de aglutinación con látex47. Este sistema permite una semicuantificación, aunque ya se han descrito falsos positivos en pacientes con títulos bajos20 y falsos negativos por fenómenos de prozona35.
Detección de ácidos nucleicosLas técnicas de amplificación de ácidos nucleicos (TAAN) aportan rapidez, sensibilidad y especificidad en el diagnóstico de las micosis y particularmente en las candidiasis invasoras. Existen dos aproximaciones diagnósticas con diferentes resultados: técnicas de amplificación isotérmica y técnicas no isotérmicas (fig. 1).
La amplificación isotérmica no requiere ciclos de temperatura, ni equipamientos caros y por tanto es coste-efectiva y robusta. Se han ensayado diferentes tecnologías: NASBA (nucleic acid sequence-based amplification), RCA (rolling cycle amplification), y LAMP (loop-mediated isothermal amplification), con resultados muy interesantes tanto en la identificación rápida de las levaduras como en la detección directa en sangre51.
La amplificación no isotérmica se basa en la reacción de PCR en diferentes formatos. Lo más utilizado actualmente son plataformas de PCR múltiple en tiempo real que detectan un panel de diferentes microorganismos, entre ellos algunas especies de Candida y Aspergillus. Destaca fundamentalmente el sistema LightCycler® SeptiFast-MG (Roche Molecular, Pleasatont, EE.UU.) porque lleva más tiempo en el mercado. Un metaanálisis mostró resultados discretos de sensibilidad para la detección de fungemia con cifras de sensibilidad del 61% aunque con una especificidad del 99%14.
Más prometedor, sin embargo, parecía el sistema que combinaba la amplificación genómica con la espectrometría de masas mediante electro-espray (PCR/ESI-MS) comercializado con el nombre de Iridica® (Abbot Molecular, Des Plaines, EE.UU.). El sistema se podía utilizar con diferentes tipos de muestras y era capaz de detectar más de mil patógenos dependiendo de su base de datos. Lamentablemente, los resultados no han sido los esperados, sobre todo en la detección de especies de Candida en sangre29, y recientemente la compañía canceló su desarrollo49.
La introducción reciente de la tecnología de PCR asociada a resonancia magnética T2 (T2Candida panel®, T2 Biosystems, Lexington, EE.UU.) podría cambiar el panorama y revolucionar el diagnóstico de la candidiasis invasiva. Se trata de un sistema totalmente automatizado de PCR múltiple combinado con resonancia magnética para identificar directamente de sangre total las cinco especies de Candida más frecuentes (C. albicans, C. parapsilosis, C. tropicalis, C. glabrata y C. krusei). Su utilidad ha quedado demostrada en recientes estudios en los que mostró una sensibilidad del 91,1% y un VPN entre el 99-99,5% para una prevalencia del 10 y 5%, respectivamente45. Algunos autores han subrayado la superioridad del sistema T2 respecto al hemocultivo en el seguimiento de los pacientes con candidemia probada, llegando a encontrar diferencias significativas en la mejoría postratamiento en aquellos pacientes seguidos con T2 frente a los monitorizados con hemocultivo46.
Finalmente, otras tecnologías de detección genómica basadas en microarrays han sido aplicadas con aparentemente buenos resultados, no solo para la detección e identificación de especies de Candida sino de otros hongos, incluidos los filamentosos51.
Diagnóstico de las micosis invasoras por hongos filamentososCultivo y examen microscópico directoEl aislamiento de hongos filamentosos (mohos) en muestras no estériles (exudados, esputo, etc.) no permite diferenciar entre colonización e infección fúngica; por lo tanto, el diagnóstico convencional de las micosis invasoras por mohos precisa la obtención de muestras clínicas lo más representativas posibles del área anatómica afectada para su cultivo y estudio anatomopatológico: punción-aspiración con aguja fina (PAAF), biopsias, lavado broncoalveolar (LBA) o líquidos orgánicos estériles. Desafortunadamente, la obtención de este tipo de muestras no siempre es posible debido a las comorbilidades del paciente (neutropenia, gravedad, etc.). La sensibilidad global y el VPP del cultivo micológico es <70% (o incluso menor en pacientes no hematológicos o en terapia antifúngica) y la identificación del agente causal a partir del mismo precisa un mínimo de 48-72h4. A pesar de sus limitaciones, el cultivo micológico es la única técnica diagnóstica que permite el aislamiento del agente causal y el posterior estudio de la sensibilidad antifúngica para seleccionar la terapia más apropiada.
Para valorar el significado clínico del crecimiento de Aspergillus en muestras respiratorias se han propuesto sistemas de puntuación (scores) y algoritmos que ayudan a definir mejor las subpoblaciones que necesitan un diagnóstico más inmediato para la confirmación de la enfermedad8,10.
El crecimiento de hongos filamentosos en hemocultivos debe ser considerado, habitualmente, como una contaminación a excepción de Fusarium, Scedosporium/Lomentospora o Acremonium, cuya recuperación en sangre no es infrecuente en el contexto de una infección diseminada.
La identificación de los hongos filamentosos aislados en cultivo se realiza en función de la morfología macro y microscópica de sus colonias, requiriéndose para ello una adecuada experiencia micológica. Actualmente la secuenciación de ácidos nucleicos o la espectrometría de masas ayudan a la identificación definitiva y se han convertido en una alternativa muy atractiva a la identificación morfológica, si bien la utilidad de la espectrometría de masas viene determinada por el mayor o menor número de espectros en su librería de referencia. En cualquier caso, los resultados de estas técnicas deben validarse a la luz del conocimiento micológico.
Además del cultivo, y aunque su sensibilidad diagnóstica es baja, se recomienda realizar siempre un examen microscópico directo (preferiblemente con blanco de calcoflúor) de las muestras clínicas más valiosas y representativas, ya que una muestra positiva puede guiar la instauración del tratamiento antifúngico en menos de 15 minutos. En las infecciones por hifomicetales (Aspergillus, Fusarium, Scedosporium, Acremonium, etc.) se observan hifas delgadas, tabicadas y ramificadas en ángulo agudo; las hifas de los zigomicetos (Mucor, Rhizopus, Lichtheimia, entre otros) son más anchas, poco tabicadas y ramificadas en ángulo recto, y las de los hongos dematiáceos (Lomentospora, Alternaria, Exserohilum, etc.) están pigmentadas en marrón, tabicadas y ramificadas.
Métodos alternativos al cultivo para el diagnóstico de la aspergilosis invasivaDebido a las limitaciones del cultivo micológico (lentitud y moderada sensibilidad), se han desarrollado diferentes técnicas que intentan ofrecer un diagnóstico más rápido y eficaz de las micosis invasoras por hongos filamentosos. Algunas ya se han implantado en la rutina asistencial, como la detección del antígeno galactomanano (GM), pero otras, como Aspergillus LFA, la detección de BDG o la de ácidos nucleicos, no han terminado de incorporarse en muchos centros hospitalarios por diferentes motivos.
Antígeno galactomanano (GM)La detección de GM en suero y LBA, mediante técnicas de enzimoinmunoanálisis (ELISA), se ha convertido en una práctica habitual en pacientes con sospecha de aspergilosis invasora (AI) y desde el año 2008 es un criterio micológico de AI probable aceptado por la EORTC18. Sin embargo, esta herramienta diagnóstica está lejos de ser perfecta: su sensibilidad y especificidad son <85%, y la profilaxis con los fármacos antimohos (habitual en pacientes neutropénicos) reduce considerablemente su sensibilidad, desaconsejando su uso19; además, la administración concomitante de betalactámicos o la infección por otros hongos pueden ser causa de falsos positivos. No obstante, la utilidad de la detección de GM en suero es incuestionable en pacientes onco-hematológicos de alto riesgo con sospecha de AI18, al igual que la detección de GM en LBA en pacientes críticos con sospecha de infección pulmonar por Aspergillus1, donde ha demostrado una mayor sensibilidad diagnóstica que en suero (88% vs. 42%)42.
Aspergillus lateral flow device (LFD)Aspergillus lateral flow device (LFD) es una técnica inmunocromatográfica en papel que detecta la unión del Ac monoclonal JF5 con una glicoproteína extracelular secretada durante el crecimiento de Aspergillus. Es una prueba rápida (15 minutos) diseñada en formato individual, lo que permite una mayor facilidad de uso en los laboratorios de microbiología ya que, a diferencia del GM, no precisa instrumentos de lectura ni procesar las muestras en lotes para optimizar los reactivos26. En un metaanálisis que incluía siete estudios publicados entre 2008 y 2015 se describió una sensibilidad y una especificidad diagnósticas superiores en LBA que en suero (86% vs. 68%, y 93% vs. 87%, respectivamente)50. Además, mientras que las muestras de suero precisan ser calentadas, centrifugadas y tamponadas previamente, las muestras de LBA pueden ser analizadas directamente sin ningún tipo de pretratamiento. Aunque, problemas de fabricación han impedido su distribución comercial en los últimos años la técnica acaba de ser relanzada al mercado (AspLFD®, OLM Diagnostics, Newcastle, UK).
β-glucano (BDG)Esta molécula es un componente de la pared celular de la mayoría de los hongos (excepto zigomicetos y Cryptococcus) y puede detectarse en el suero y los líquidos orgánicos mediante ELISA en pacientes con infecciones invasivas causadas por diferentes especies de Aspergillus, Fusarium, Acremonium, Candida, o Pneumocystis jirovecii, entre otros. En un metaanálisis que incluyó 16 estudios y 2.979 pacientes con infección fúngica invasiva, la sensibilidad y especificidad global de BDG fue del 75±6% y 85±3%, respectivamente, y no fue significativamente diferente en el subgrupo de estudios que evaluaron el rendimiento de la prueba en AI31. En otro metaanálisis posterior realizado por los mismos autores con relación al diagnóstico de la neumonía por P. jirovecii, la sensibilidad y especificidad del BDG fue superior: 95±3% y 86±4%, respectivamente30. La detección en suero de BDG ha sido incorporada como criterio microbiológico de infección fúngica por la EORTC18. Entre las limitaciones en la práctica asistencial de esta técnica que han impedido su uso generalizado en los centros hospitalarios hay que destacar los numerosos falsos positivos detectados (hemodiálisis, inmunoglobulinas, albúmina, bacteriemia, gasas quirúrgicas, contaminación ambiental, etc.), ser una técnica panfúngica que no diferencia el agente causal (precisando de otras técnicas más específicas) y las complejidades técnicas para su realización en un laboratorio asistencial.
Compuestos orgánicos volátiles (COV)En las infecciones y otras enfermedades respiratorias, y posiblemente también en las sistémicas, se producen compuestos orgánicos volátiles (COV) que posteriormente son exhalados por el paciente y pueden ser detectados en su aliento sin necesidad de obtener muestras invasivas (LBA, biopsias)60. Varios autores han detectado 2-pentilfurano en la respiración de pacientes con AI13,59, y en una reciente prueba de concepto utilizando la tecnología de nariz electrónica para detectar la «huella» COV de Aspergillus, la sensibilidad de la técnica fue del 100% y su especificidad del 83% en pacientes con alto riesgo de AI17. Con anterioridad, y utilizando una tecnología más sofisticada (cromatografía de gases y espectrometría de masas) para detectar cuatro COV (α- y β-trans-bergamoteno, β-vatireneno, y trans-geranilacetona), Koo et al. obtuvieron una elevada sensibilidad y especificidad diagnóstica (94 y 93%, respectivamente) en 64 pacientes con riesgo elevado de AI reclutados prospectivamente34. Estos alentadores resultados necesitan ser confirmados en series más amplias, incluyendo diferentes poblaciones de pacientes, más patógenos y la posibilidad de interacciones con agentes antifúngicos u otros fármacos que puedan alterar la composición de los COV25.
Ácidos nucleicosEn los últimos 20 años se han desarrollado múltiples técnicas in-house de amplificación de ácidos nucleicos (TAAN) para detectar ADN de Aspergillus y otros mohos con gran sensibilidad diagnóstica, ya que son capaces de detectar 1-10fg de ADN fúngico en diferentes muestras clínicas (sangre total, suero, líquidos orgánicos LBA, etc.). Para disminuir la heterogeneidad metodológica y mejorar el rendimiento diagnóstico de las TAAN in-house, la Iniciativa Europea de Aspergillus PCR (EAPCRI) ha evaluado, en dos estudios multicéntricos, diferentes métodos de extracción y amplificación de ADN de Aspergillus en sangre total y suero, dando un paso importante para la estandarización definitiva de estas técnicas61,62. A pesar de ello, las TAAN todavía no se han incluido como criterio diagnóstico en los documentos de consenso de la enfermedad fúngica invasiva40. En un metaanálisis de 25 estudios con un total de 2.595 pacientes hematológicos con alto riesgo de AI, la sensibilidad y la especificidad de las TAAN en sangre/suero para detectar AI con un resultado positivo fueron 84 y 76%, respectivamente, y con dos resultados positivos 64 y 95%, respectivamente5.
Desde hace años se han comercializado diferentes TAAN para el diagnóstico de AI. Quizás la más completa es AsperGenius® (Pathognostics, Maastricht, NL), capaz de detectar ADN de A. fumigatus complex, A terreus, A flavus, A niger y A. nidulans en LBA, además de cuatro genes que codifican resistencia a los azoles (TR34, L98H, Y121F y T289A). Chong et al. han validado esta técnica en LBA de pacientes con elevado riesgo de AI: 37 hematológicos (sensibilidad: 88,9%, especificidad: 89,3%, VPP: 72,7% y VPN: 96,2%) y 40 pacientes críticos (sensibilidad: 80%, especificidad: 93,3%, VPP: 80% y VPN: 93,3%)15.
Métodos alternativos al cultivo para el diagnóstico de mucormicosisA diferencia de la AI, los biomarcadores séricos como el GM y el BDG no son útiles en el diagnóstico de las mucormicosis, y las TAAN in-house basadas en PCR en tiempo real son la única alternativa válida al cultivo convencional. Estos métodos emplean cebadores dirigidos a genes multicopias como la unidad 28S del ARNr, espaciadores de transcripción internos y sondas específicas de especie32. Millon et al. evaluaron una combinación de tres PCR cuantitativas con sondas de Mucor/Rhizopus, Lichtheimia y Rhizomucor para detectar mucorales en suero de 10 pacientes con mucormicosis confirmada por histopatología y/o cultivo, y encontraron ADN de mucorales en 9 de ellos43. También se ha demostrado la utilidad de combinar PCR con análisis de fusión de alta resolución (HRMA) en 99 muestras de LBA para diagnosticar mucormicosis en 86 pacientes inmunodeprimidos con infiltrados pulmonares, con una sensibilidad y especificidad de la técnica del 100 y 93%, respectivamente36.
Métodos alternativos al cultivo para el diagnóstico de la hialohifomicosisEn nuestro entorno, Scedosporium apiospermum complex y Fusarium son los agentes causales de hialohifomicosis más habituales, y para su diagnóstico, al igual que en las mucormicosis, las PCR a tiempo real in-house son la única alternativa válida al cultivo convencional.
EscedosporiosisLas especies del complejo S. apiospermum son la segunda causa de colonización de la vía aérea por hongos filamentosos en pacientes con fibrosis quística y son responsables de muchas infecciones respiratorias en receptores de trasplante pulmonar. Para identificar de forma rápida y sencilla las seis especies más habituales se han desarrollado TAAN dirigidas al gen de la β-tubulina mediante PCR cuantitativa a tiempo real37. Sin embargo, para la completa identificación y genotipificación de los aislamientos del cultivo, la amplificación por PCR de secuencias repetitivas (rep-PCR) diferencia mejor entre las especies del complejo, e incluso entre cepas de la misma especie, y parece una técnica más eficaz que las usualmente utilizadas basadas en amplificación aleatoria de ADN polimórfico (RAPD) o tipificación de secuencia multilocus (MLST)39.
FusariosisAunque las especies del género Fusarium liberan GM y BDG, estos biomarcadores no permiten diagnosticar definitivamente una fusariosis debido a su falta de especificidad. Para la identificación de Fusarium en cortes histológicos se han desarrollado técnicas de hibridación in situ basadas en el gen del factor de elongación 1α (tef1α); sin embargo, su uso no se ha extendido en la práctica clínica24,41. También se ha descrito la utilidad de la PCR múltiple cuantitativa, con cebadores de los genes 5.8S y 28S del ADNr, para distinguir entre géneros de varios patógenos fúngicos pulmonares, incluyendo Fusarium, Aspergillus y mucorales23.
Métodos alternativos al cultivo para el diagnóstico de micosis por hongos dematiáceosEstos hongos son una causa poco frecuente de micosis y, en ausencia de un brote, como el de de meningitis iatrogénica por Exserohilum rostratum21, es improbable que se desarrollen TAAN para detectar estos patógenos, aunque estas técnicas son quizás las únicas que pueden ser rápidamente implementadas para dar una adecuada respuesta diagnóstica en emergencias epidemiológicas.
Detección múltiple de hongos filamentosos mediante métodos comercializadosHasta la fecha se han intentado comercializar, con mayor o menor éxito, varias técnicas de diagnóstico panfúngico que incluyen hongos filamentosos en su base de datos. La tecnología de arrays en fase líquida soluciona las limitaciones de las TAAN convencionales, incluida la PCR en tiempo real, para diferenciar gran número de dianas diferentes. Aplicando una variante de esta metodología (sondas ligadas a microesferas fluorescentes y detectadas mediante citometría de flujo) a una PCR múltiple (Luminex Co., Austin, EE.UU.) Buelow et al. han podido detectar e identificar 11 patógenos fúngicos causantes de infecciones respiratorias, incluido Fusarium solani11. Con el nombre de Iridica® (Abbot Molecular, Des Plaines, EE.UU.) se desarrolló una plataforma diagnóstica basada en la combinación de PCR múltiple con ionización mediante electro-espray y detección del amplificado por espectrometría de masas (PCR/ESI-MS). Esta tecnología era capaz de identificar, en muestras clínicas y en menos de 6h, más de 750 especies bacterianas, 150 virus y 200 patógenos fúngicos, pero su comercialización se paralizó definitivamente en 2017, como ya se ha comentado antes49. Los pocos estudios realizados con esta metodología demostraron su ventaja frente al cultivo para detectar hongos filamentosos en muestras clínicas con elevada presencia bacteriana (esputo, LBA)58 así como mucorales en biopsias tisulares2.
Perspectivas futurasEn los años próximos nos esperan interesantes propuestas derivadas de las nuevas tecnologías. Sistemas basados en nanotecnología, microfluídica, o microchips ya se han ensayado con éxito para la identificación de diferentes especies de hongos en muestras clínicas, incluidas levaduras. El desarrollo tecnológico se encamina a la miniaturización de sistemas compactos con pocos requerimientos en infraestructura que permitan la descentralización de las áreas diagnósticas a través de los llamados point of care52,55. Sin embargo, todavía estamos lejos de encontrar el método diagnóstico ideal que se convierta en la nueva referencia y que incluya todas las características exigibles:
- 1.
Fiables en términos de sensibilidad y especificidad.
- 2.
Precoces: con resultados positivos antes incluso de la aparición de signos y síntomas.
- 3.
Monitorización de la respuesta al tratamiento.
- 4.
Valor pronóstico.
- 5.
Comerciales: estandarizados que permitan la comparación entre diferentes laboratorios.
- 6.
Económicos: coste-efectivos considerando el impacto en hospitalización y tratamiento.
- 7.
Rápidos: el tiempo del análisis en una jornada de trabajo y en pocas horas.
- 8.
Automatizables: sistemas simples con mínimo requerimiento de infraestructuras.
- 9.
No invasivos: sobre muestras de fácil obtención.
- 10.
Información rápida de especie y sensibilidad.
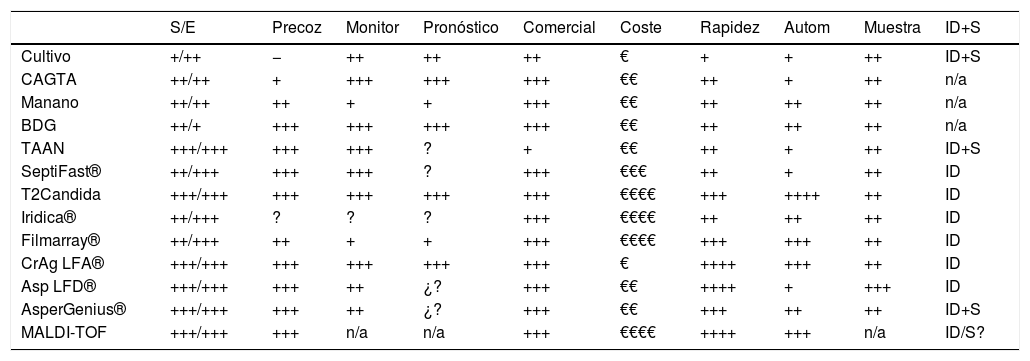
En la tabla 1 se detallan las principales características de los métodos descritos.
Comparación de las características de los diferentes métodos diagnósticos
| S/E | Precoz | Monitor | Pronóstico | Comercial | Coste | Rapidez | Autom | Muestra | ID+S | |
|---|---|---|---|---|---|---|---|---|---|---|
| Cultivo | +/++ | − | ++ | ++ | ++ | € | + | + | ++ | ID+S |
| CAGTA | ++/++ | + | +++ | +++ | +++ | €€ | ++ | + | ++ | n/a |
| Manano | ++/++ | ++ | + | + | +++ | €€ | ++ | ++ | ++ | n/a |
| BDG | ++/+ | +++ | +++ | +++ | +++ | €€ | ++ | ++ | ++ | n/a |
| TAAN | +++/+++ | +++ | +++ | ? | + | €€ | ++ | + | ++ | ID+S |
| SeptiFast® | ++/+++ | +++ | +++ | ? | +++ | €€€ | ++ | + | ++ | ID |
| T2Candida | +++/+++ | +++ | +++ | +++ | +++ | €€€€ | +++ | ++++ | ++ | ID |
| Iridica® | ++/+++ | ? | ? | ? | +++ | €€€€ | ++ | ++ | ++ | ID |
| Filmarray® | ++/+++ | ++ | + | + | +++ | €€€€ | +++ | +++ | ++ | ID |
| CrAg LFA® | +++/+++ | +++ | +++ | +++ | +++ | € | ++++ | +++ | ++ | ID |
| Asp LFD® | +++/+++ | +++ | ++ | ¿? | +++ | €€ | ++++ | + | +++ | ID |
| AsperGenius® | +++/+++ | +++ | ++ | ¿? | +++ | €€ | +++ | ++ | ++ | ID+S |
| MALDI-TOF | +++/+++ | +++ | n/a | n/a | +++ | €€€€ | ++++ | +++ | n/a | ID/S? |
Autom: automatizado; Asp LFD®: Aspergillus lateral flow device; BDG: β-D-glucano; CAGTA: anticuerpos anti-micelio; CrAg LFA®: Cryptococcus Ag lateral flow assay; ID+S: identificación y sensibilidad antifúngica; MALDI-TOF: espectrometria de masas; Manano: Ag/Ac/Manano; Monitor: monitorización; Muestra: facilidad de obtención de la muestra; n/a: no aplica; S/E: sensibilidad/especificidad; TAAN: técnicas amplificación ácidos nucleicos.
Los autores declaran no tener ningún conflicto de intereses.