Las infecciones fúngicas invasivas han ido aumentando y las levaduras del género Candida son la principal causa de las mismas. Las especies distintas de Candida albicans son cada vez más frecuentes y algunas pueden presentar patrones variables de sensibilidad a los antimicóticos, por lo que es importante la correcta identificación de la especie. Los métodos de identificación convencionales con los que cuentan la mayoría de los laboratorios pueden presentar inconvenientes. La espectrometría de masas (MALDI-TOF MS) surgió como una alternativa a los mismos.
ObjetivosEvaluar la concordancia de los resultados de identificación de diferentes especies de Candida mediante dostécnicas: una convencional (galerías API) y MALDI-TOF MS.
MétodosSe analizaron las siguientes especies y número de aislamientos: Candida parapsilosis (28), Candida glabrata (34), Candida krusei (24), Candida tropicalis (45), Candida guilliermondii (30), C. albicans (28), Candida dubliniensis (6), Candida kefyr (1) y Candida lipolytica (1) del cepario de RMCABA; también se utilizaron las cepas C. parapsilosis 22019, C. glabrata 90030, C. krusei 6258 y C. albicans 68548, pertenecientes a la American Type Culture Collection (ATCC). Las discrepancias en la identificación se resolvieron mediante genotipificación.
Resultados y conclusionesLa concordancia directa entre la identificación convencional y MALDI-TOF MS fue del 92,5% (186/201).
Invasive fungal infections are increasing, and Candida yeasts are the main cause. Species other than Candida albicans are becoming more frequent, and some of them may have variable patterns of susceptibility to antifungal agents, making it important to identify them correctly. Conventional identification methods used by most laboratories may present with drawbacks. Mass spectrometry (MALDI-TOF MS) has emerged as an alternative method.
AimsThe aim of this study was to evaluate the concordance of the identification, at species level, by conventional methods (API) and MALDI-TOF MS.
MethodsThe following species and number of isolates were studied: Candida parapsilosis (28), Candida glabrata (34), Candida krusei (24), Candida tropicalis (45), Candida guilliermondii (30), C. albicans (28), Candida dubliniensis (6), Candida kefyr (1), and Candida lipolytica (1) from the strain collection of Autonomous City of Buenos Aires Mycology Network (RMCABA). The strains C. parapsilosis 22019, C. glabrata 90030, C. krusei 6258 and C. albicans 68548 from the American Type Culture Collection (ATCC) were also included. Discrepancies were resolved by genotyping.
Results and conclusionsThe direct concordance between the conventional identification method and MALDI-TOF MS was 92.5% (186/201).
En los últimos años se ha observado un incremento en las infecciones fúngicas invasivas. Las especies de Candida distintas de Candida albicans se recuperan cada vez con más frecuencia y pueden presentar patrones variables de sensibilidad a los antifúngicos, por lo que es muy importante identificarlas correctamente1,5,12.
Los métodos de identificación convencionales incluyen estudios micromorfológicos, medios cromogénicos y sistemas comerciales basados en pruebas bioquímicas. Estos presentan inconvenientes, tales como un tiempo de identificación prolongado, bases de datos limitadas e identificaciones incorrectas14. La espectrometría de masas (MALDI-TOF MS) surgió como una alternativa a los mismos1,10.
La mayoría de los laboratorios asistenciales de nuestro país cuenta con métodos convencionales para la identificación de levaduras de importancia clínica. El objetivo del estudio fue evaluar la concordancia en los resultados de la identificación de especies de Candida obtenidos por dosmetodologías: una convencional (galerías API) y otra novedosa (MALDI-TOF MS).
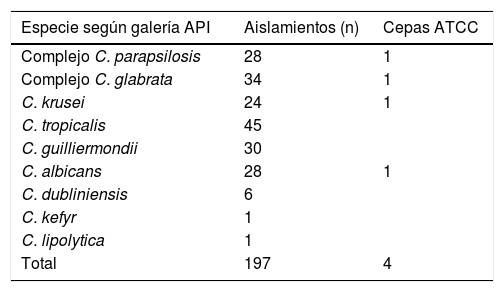
Materiales y métodosSe analizaron 197 aislamientos de Candida: 28 Candida parapsilosis, 34 Candida glabrata, 24 Candida krusei, 45 Candida tropicalis, 30 Candida guilliermondii, 28C. albicans, 6 Candida dubliniensis, 1 Candida kefyr y 1 Candida lipolytica, provenientes de materiales clínicos; se incluyeron las cepas de la colección ATCC C. parapsilosis 22019, C. glabrata 90030, C. krusei 6258 y C. albicans 68548 (tabla 1). Todos los aislamientos fueron identificados por métodos convencionales y MALDI-TOF MS. Las discrepancias se resolvieron mediante genotipificación.
Aislamientos clínicos y cepas ATCC evaluados de especies de Candida
| Especie según galería API | Aislamientos (n) | Cepas ATCC |
|---|---|---|
| Complejo C. parapsilosis | 28 | 1 |
| Complejo C. glabrata | 34 | 1 |
| C. krusei | 24 | 1 |
| C. tropicalis | 45 | |
| C. guilliermondii | 30 | |
| C. albicans | 28 | 1 |
| C. dubliniensis | 6 | |
| C. kefyr | 1 | |
| C. lipolytica | 1 | |
| Total | 197 | 4 |
A todos los aislamientos se les realizó un estudio micromorfológico en agar harina de maíz con 1% de Tween 80 (Corn-Meal agar, Oxoid, Reino Unido) y se sembraron en agar cromogénico para levaduras (CHROMagarTM Candida, París, Francia). Para la identificación según el patrón de asimilación de azúcares se utilizaron las galerías API® ID 32C o API® 20C AUX (bioMérieux, Marcy l’Etoile, Francia). Además, se incluyeron otras pruebas bioquímicas para diferenciar C. albicans de C. dubliniensis13.
Análisis por MALDI-TOF MSLos resultados se leyeron y analizaron por el sistema Bruker Microflex LT versión 3.1 (Bruker Daltonics, Alemania). Se consideró un score mayor o igual a 1,7 como identificación aceptable de la especie, entre 1,5 y 1,6 identificación aceptable del género, y menor o igual a 1,5 como identificación no fiable. La identificación se realizó a partir de un extendido directo de la colonia con 1μl de ácido fórmico al 70% (Sigma-Aldrich, Lyon, Francia) y 1μl de matriz alfa-ciano-4 hidroxi-ácido cinámico (Bruker Daltonics, Alemania). Cuando los scores fueron < 1,70, se realizó el método de extracción en tubo con etanol absoluto (Sigma-Aldrich, Francia) y acetonitrilo al 100% (Sigma-Aldrich), siguiendo las indicaciones dadas por el fabricante Bruker Daltonics.
Identificación genotípicaPara cada aislamiento discordante se realizó la extracción de ADN según Silva et al.17. Se emplearon los primers universales ITS1 (5’-TCCGTAGGTGAACCTGCGG-3’) e ITS4 (5’TCCTCCGCTTATTGATATGC-3’). Los productos de amplificación fueron purificados y secuenciados con ABI 3730xl (Applied Biosystems, EE. UU.). Para la identificación se empleó la herramienta BLAST y se estableció como criterio una concordancia > 97% y una cobertura de al menos el 99%.
Métodos estadísticosSe aplicó la prueba de concordancia kappa8 para la estimación de la concordancia entre ambas técnicas diagnósticas, y el programa estadístico EpiDat 4.1 versión 2014 (Consellería de Sanidade, Xunta de Galicia, España; Organización Panamericana de la Salud; Universidad CES, Colombia; http://dxsp.sergas.es).
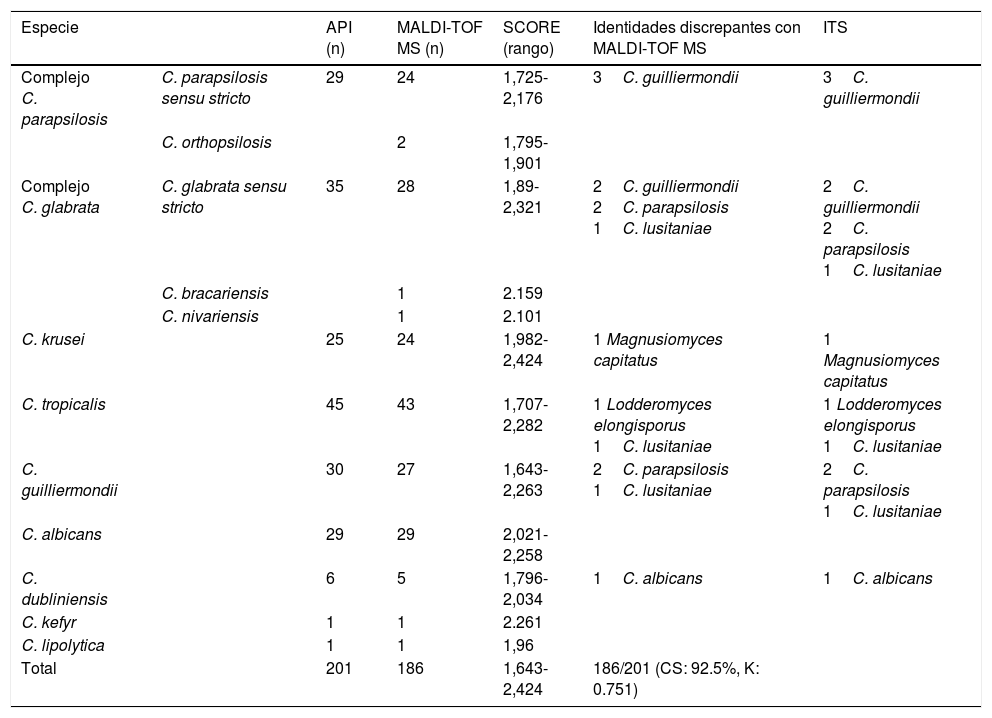
Resultados y discusiónLa tabla 2 muestra los resultados obtenidos con ambas metodologías. El porcentaje de acuerdo o concordancia directa entre la identificación convencional y la espectrometría de masas (MALDI-TOF MS) fue del 92,5% (IC del 95%: 91,64-93,43) (186 aislamientos de 201), y un índice kappa igual a 0,751 (IC del 95%: 0,633-0,869), lo que indica una buena concordancia entre ambas técnicas diagnósticas. Se observaron discrepancias con las siguientes especies y número de aislamientos: C. parapsilosis (3/29); C. glabrata (5/35); C. krusei (1/25); C. tropicalis (2/45); C. guilliermondii (3/30) y C. dubliniensis (1/6). No hubo discordancia entre los resultados obtenidos mediante secuenciación y los de MALDI-TOF MS.
Resultados de la identificación de levaduras según el método convencional (API), por técnica proteómica (MALDI-TOF MS) e identidades discrepantes confirmadas por genómica (ITS)
| Especie | API (n) | MALDI-TOF MS (n) | SCORE (rango) | Identidades discrepantes con MALDI-TOF MS | ITS | |
|---|---|---|---|---|---|---|
| Complejo C. parapsilosis | C. parapsilosis sensu stricto | 29 | 24 | 1,725-2,176 | 3C. guilliermondii | 3C. guilliermondii |
| C. orthopsilosis | 2 | 1,795-1,901 | ||||
| Complejo C. glabrata | C. glabrata sensu stricto | 35 | 28 | 1,89-2,321 | 2C. guilliermondii 2C. parapsilosis 1C. lusitaniae | 2C. guilliermondii 2C. parapsilosis 1C. lusitaniae |
| C. bracariensis | 1 | 2.159 | ||||
| C. nivariensis | 1 | 2.101 | ||||
| C. krusei | 25 | 24 | 1,982-2,424 | 1 Magnusiomyces capitatus | 1 Magnusiomyces capitatus | |
| C. tropicalis | 45 | 43 | 1,707-2,282 | 1 Lodderomyces elongisporus 1C. lusitaniae | 1 Lodderomyces elongisporus 1C. lusitaniae | |
| C. guilliermondii | 30 | 27 | 1,643-2,263 | 2C. parapsilosis 1C. lusitaniae | 2C. parapsilosis 1C. lusitaniae | |
| C. albicans | 29 | 29 | 2,021-2,258 | |||
| C. dubliniensis | 6 | 5 | 1,796-2,034 | 1C. albicans | 1C. albicans | |
| C. kefyr | 1 | 1 | 2.261 | |||
| C. lipolytica | 1 | 1 | 1,96 | |||
| Total | 201 | 186 | 1,643-2,424 | 186/201 (CS: 92.5%, K: 0.751) | ||
CS: porcentaje de concordancia directa; ITS: secuenciación de los espacios intergénicos; K: índice kappa; MALDI-TOF MS: matrix-assisted laser desorption/ionization time-of-flight mass spectrometry.
Como ya ha sido publicado por otros autores, una limitación de los métodos convencionales es la incapacidad de identificar especies crípticas de los complejos C. parapsilosis y C. glabrata4,9,16. Por el contrario, MALDI-TOF MS permitió diferenciar entre especies del complejo C. parapsilosis, como Candida orthopsilosis y C. parapsilosis sensu stricto, y las del complejo C. glabrata, como Candida bracariensis, Candida nivariensis y C. glabrata sensu stricto. La identificación de las especies crípticas de estos complejos es importante no solo desde el punto de vista epidemiológico, sino también por las posibles diferencias de sensibilidad a los antifúngicos y la virulencia de las especies que los integran9. Otro inconveniente que presentan los métodos convencionales es el de no poder diferenciar C. dubliniensis de C. albicans, cuya importancia radica en la diferente sensibilidad al fluconazol entre ambas especies9,15. En este trabajo, MALDI-TOF MS permitió identificar correctamente la levadura Magnusiomyces capitatus, erróneamente identificada por los métodos convencionales como C. krusei. Aunque M. capitatus es un hongo poco frecuente puede causar infecciones graves. Su identificación puede ser difícil y requerir mucho tiempo si no se cuenta con la tecnología MALDI-TOF MS2,7,11.
La dificultad para identificar C. guilliermondii por métodos fenotípicos es bien conocida. En el presente trabajo, 3 de los 30 aislamientos de C. guilliermondii estaban erróneamente identificados; algunos aislamientos identificados como C. parapsilosis (3) y C. glabrata (2) por métodos convencionales resultaron ser C. guilliermondii por MALDI-TOF MS y secuenciación3,9. Otra especie que presenta limitaciones con la metodología convencional es C. lusitaniae, que en este estudio fue identificada por los métodos convencionales como C. guilliermondii, C. tropicalis y complejo C. glabrata9. Un aislamiento identificado como C. tropicalis por el sistema API fue identificado por MALDI-TOF MS como Lodderomyces elongisporus, levadura a la que las últimas revisiones taxonómicas describen como una especie estrechamente relacionada con C. parapsilosis6. Los scores obtenidos por MALDI-TOF MS estuvieron comprendidos entre 1,643 y 2,424. C. guilliermondii fue la especie con scores más bajos: en 7 aislamientos de esta especie se requirió extracción en tubo y solo en un caso de estos no se llegó al score de identificación aceptable. La extracción en tubo se realizó solo en 8 ocasiones (7C. guilliermondii y 1C. tropicalis).
Estos resultados permiten concluir que las metodologías convencionales siguen siendo útiles para identificar en forma confiable las especies más frecuentemente aisladas de muestras clínicas, pero cuando se trata de especies de Candida poco comunes o raras, o especies crípticas, es importante su confirmación utilizando tecnologías como MALDI-TOF MS.